離子液體[BMIm][PF6]增強抗性質(zhì)粒RP4介導(dǎo)的接合轉(zhuǎn)移基因mRNA的表達水平
汪慶,羅義,崔玉曉,齊麗英,周爍磊,楊光,*
1. 河北工程大學(xué) 能源與環(huán)境工程學(xué)院,河北省水污染控制與水生態(tài)修復(fù)工程技術(shù)研究中心,邯鄲 0560382. 環(huán)境污染過程與基準(zhǔn)教育部重點實驗室,南開大學(xué),天津300071
離子液體[BMIm][PF6]增強抗性質(zhì)粒RP4介導(dǎo)的接合轉(zhuǎn)移基因mRNA的表達水平
汪慶1, 2,羅義2,崔玉曉2,齊麗英1,周爍磊1,楊光1,*
1. 河北工程大學(xué) 能源與環(huán)境工程學(xué)院,河北省水污染控制與水生態(tài)修復(fù)工程技術(shù)研究中心,邯鄲 0560382. 環(huán)境污染過程與基準(zhǔn)教育部重點實驗室,南開大學(xué),天津300071
環(huán)境中廣泛存在的抗生素和抗生素抗性基因會導(dǎo)致很嚴(yán)重的人類健康風(fēng)險。在我們前期研究中發(fā)現(xiàn),離子液體1-丁基-3-甲基咪唑六氟磷酸鹽([BMIm][PF6])作為環(huán)境選擇性壓力可以促進抗生素抗性基因的水平轉(zhuǎn)移。本研究以E. coli DH5α (RP4) 和同屬的E. coli HB101,以及E. coli DH5α (RP4)和跨屬的Salmonella enterica之間的純菌接合轉(zhuǎn)移體系為考察對象,從mRNA基因表達調(diào)控水平角度闡明離子液體[BMIm][PF6] (0.001~2.5 g·L-1)影響質(zhì)粒RP4接合轉(zhuǎn)移的機理。結(jié)果表明,離子液體[BMIm][PF6]通過抑制基因korA, korB和trbA的mRNA表達水平來提高接合和跨膜轉(zhuǎn)運基因trbBp和trfAp的表達;增強水平轉(zhuǎn)移基因traF的mRNA表達水平,并通過增強負(fù)調(diào)控基因kilA和kilB的mRNA表達水平抑制質(zhì)粒的垂直傳遞過程;通過抑制基因trbK的mRNA表達水平降低接合轉(zhuǎn)移體系的排斥效果,從而促進質(zhì)粒RP4的接合轉(zhuǎn)移。該結(jié)果有助于為抗生素抗性基因在環(huán)境中水平轉(zhuǎn)移擴散的機理研究提供理論依據(jù)。
離子液體;抗生素抗性基因;質(zhì)粒RP4;接合轉(zhuǎn)移;mRNA表達
Received12 January 2017accepted13 March 2017
Abstract: Widespread antibiotic and antibiotic resistance genes (ARGs) can pose serious health risks. In our previous study, an ionic liquid (IL) 1-butyl-3-methylimidazolium hexafluorophosphate ([BMIm][PF6]) exerted selective pressure promoting the dissemination of ARGs via horizontal gene transfer. To further investigate the molecular mechanisms of an IL [BMIm][PF6] (0.001-2.5 g·L-1) facilitate the conjugative transfer of ARGs with the mRNA expression levels of conjugative transfer genes, plasmid RP4 conjugative transfer were setup from Escherichia coli DH5α (as the donor) to E. coli HB101 (as the recipient, same species) and Salmonella enterica (as the recipient, across genera). Results of the mRNA expression levels of conjugative transfer genes showed that alleviation of the repression of korA, korB, and trbA genes significantly triggered the expression of trbBp and trfAp, promoting the conjugative transfer of plasmid RP4; Enhanced traF mRNA expression levels for facilitating horizontal transfer and increased kilA and kilB mRNA expression levels for inhibiting the genes responsible for vertical transfer; Inhibited trbK mRNA expression levels for decreasing the entry exclusion phenotype, promoting the conjugative transfer of plasmid RP4. It is helpful to provide theoretical basis for the study of the mechanism of ARGs transfer in the environment.
Keywords: ionic liquid; antibiotic resistance genes; conjugative transfer; plasmid RP4; mRNA expression
抗生素和抗生素抗性基因的廣泛存在和傳播會引起很嚴(yán)重的環(huán)境生態(tài)風(fēng)險[1-2]。環(huán)境中殘留的抗生素所產(chǎn)生的選擇性壓力被認(rèn)為是抗生素抗性基因出現(xiàn)、存在和傳播的最主要因素[3]。但是,很多報道發(fā)現(xiàn)在從來沒有使用過抗生素的環(huán)境介質(zhì)中也檢測到了抗性基因的存在[4]。抗生素抗性基因主要通過接合、轉(zhuǎn)化和轉(zhuǎn)導(dǎo)的方式在相同種屬或者不同種屬的細菌之間發(fā)生水平轉(zhuǎn)移,從而導(dǎo)致抗性基因在環(huán)境中的傳播擴散[5]。除了抗生素對抗性基因的篩選作用外,其他環(huán)境選擇性壓力如重金屬、消毒劑、洗滌劑、生物殺滅劑和納米材料等都可以通過增強水平轉(zhuǎn)移來促進抗生素抗性基因在環(huán)境中的傳播[5-6]。
離子液體作為一種“環(huán)境友好型”溶劑,已經(jīng)廣泛應(yīng)用于能源[7]、生物技術(shù)[8]、化學(xué)工程[9]、高分子材料[10]、納米技術(shù)[11]等領(lǐng)域。Pham等[12]綜述了離子液體在環(huán)境中的毒理、降解、歸趨和環(huán)境風(fēng)險,離子液體的環(huán)境毒性與離子液體碳鏈長度和陰、陽離子種類有關(guān)。通常,水環(huán)境中微生物、水生植物考察濃度在0.001~10 g·L-1(碳鏈長度在4~10之間)[12-13]。通過在植物、動物、微生物、細胞和分子層面對離子液體毒理的研究發(fā)現(xiàn),離子液體可以通過攻擊細胞膜的脂質(zhì)結(jié)構(gòu)使細胞膜受損傷,導(dǎo)致細胞壁不均勻或改變細胞膜成分[14-15]。細胞膜是細菌同屬或跨屬之間通過水平轉(zhuǎn)移進行基因交換的重要阻礙[5]。在前期研究工作中,我們初步發(fā)現(xiàn)一種離子液體1-丁基-3-甲基咪唑六氟磷酸鹽([BMIm][PF6])作為環(huán)境選擇性壓力,可以促進抗生素抗性基因的水平轉(zhuǎn)移[16]。但離子液體[BMIm][PF6]暴露怎樣通過影響接合轉(zhuǎn)移基因mRNA表達水平來達到促進抗生素抗性基因的水平轉(zhuǎn)移的目的還有待進一步深入研究。
在抗性質(zhì)粒RP4介導(dǎo)的接合轉(zhuǎn)移體系中,主要需要兩大調(diào)控系統(tǒng)基因[17]:一個是調(diào)控供體菌與受體菌接合配對形成系統(tǒng)(Mpf)的基因trbBp,它主要負(fù)責(zé)供體菌和受體菌細胞的接觸和DNA跨膜轉(zhuǎn)運過程[18]。另一個是DNA轉(zhuǎn)移與復(fù)制系統(tǒng)(Dtr)的基因trfAp,它主要負(fù)責(zé)質(zhì)粒RP4在供體菌細胞內(nèi)單鏈的切割, DNA的跨膜轉(zhuǎn)運和單鏈DNA在受體菌細胞內(nèi)雙鏈的復(fù)制過程[19-20]。這2個基因在調(diào)控過程中又都受到負(fù)調(diào)控因子korA、 korB和trbA基因的控制[21]。另外,質(zhì)粒RP4的接合轉(zhuǎn)移還受細菌垂直傳遞(自我復(fù)制)基因kilA和kilB的負(fù)調(diào)控以及水平轉(zhuǎn)移正調(diào)控基因traF的影響[22]。在質(zhì)粒RP4進入受體菌過程中,又會受體細胞進入排斥系統(tǒng)基因trbK的抑制作用[23-24]。
本文主要以E. coli DH5α (RP4) 和同屬的E. coli HB101,以及E. coli DH5α (RP4)和跨屬的Salmonella enterica之間的純菌接合轉(zhuǎn)移體系為考察對象,研究離子液體[BMIm][PF6] (0.001~2.5 g·L-1) 對質(zhì)粒RP4在同屬和跨屬之間接合轉(zhuǎn)移的影響。并從mRNA基因表達調(diào)控水平來闡明離子液體[BMIm][PF6]促進質(zhì)粒RP4接合轉(zhuǎn)移的機理。本研究有助于為抗生素抗性基因在環(huán)境中水平轉(zhuǎn)移擴散的機理研究提供理論依據(jù)。
1 材料與方法(Materials and methods)
1.1 試劑與儀器
試劑:離子液體1-丁基-3-甲基咪唑六氟磷酸鹽([BMIm][PF6])購自中國科學(xué)院蘭州理化研究所(純度>99.9%)。氨芐青霉素、卡那霉素、鹽酸四環(huán)素、鏈霉素購自天津鼎國生物技術(shù)公司。
儀器:定量PCR儀(CFX Connect,BIO-RAD);PCR梯度基因擴增儀(S1000 Thermal,BIO-RAD);紫外凝膠成像儀(Tanon1600,天能科技有限公司);電泳儀(DY-A2,上海西巴斯生物技術(shù)公司);恒溫培養(yǎng)箱(GNP-9160, 上海精宏實驗設(shè)備有限公司)。
1.2 實驗質(zhì)粒和菌株
(1)質(zhì)粒RP4:實驗中的質(zhì)粒RP4由中國人民解放軍軍事醫(yī)學(xué)科學(xué)院衛(wèi)生學(xué)環(huán)境醫(yī)學(xué)研究所李君文研究員饋贈。該質(zhì)粒同時攜帶有氨芐青霉素、卡那霉素和四環(huán)素的3種抗生素抗性(ApR, KmR和TcR),能夠在多個細菌種屬間進行水平轉(zhuǎn)移。使用前保存于-80 ℃冰箱。
(2)含質(zhì)粒RP4的細菌E. coli DH5α:通過轉(zhuǎn)化將質(zhì)粒RP4轉(zhuǎn)入感受態(tài)的E.coli DH5α中,從而使E. coli DH5α同時攜帶有氨芐青霉素、卡那霉素和鏈霉素3種抗性。
(3)細菌E. coli HB101為實驗室保藏所有。Salmonella enterica為前期實驗篩選獲得的環(huán)境土著菌。2種菌都攜帶鏈霉素抗性(StrR)。
1.3 實驗方法
1.3.1 建立質(zhì)粒RP4純菌接合轉(zhuǎn)移體系
利用接合轉(zhuǎn)移實驗考察離子液體1-丁基-3-甲基咪唑六氟磷酸鹽([BMIm][PF6]) 對質(zhì)粒RP4從供體E. coli DH5α(ApR, KmR和TcR)分別向受體E. coli HB101 (StrR,同屬接合轉(zhuǎn)移) 和受體Salmonella enterica(StrR, 跨屬接合轉(zhuǎn)移)的影響。供體菌(E. coliDH5α)和受體菌(E. coli HB101和Salmonella enterica)分別過夜培養(yǎng)后,菌液經(jīng)離心后調(diào)整菌液濃度至OD600≈0.400(對應(yīng)的菌液濃度為108cfu·mL-1)。供體菌和受體菌中各加入0.5 mL,混合均勻,并添加離子液體[BMIm][PF6]使其最終濃度為0, 0.001, 0.01, 0.1, 0.5和 2.5 g·L-1,每個濃度設(shè)置3組平行。
水平轉(zhuǎn)移體系置于30 ℃的恒溫培養(yǎng)箱中靜置進行。實驗結(jié)束后,利用10倍梯度稀釋法將接合液涂布到LB固體選擇性培養(yǎng)基(100 mg·L-1Ap, 60 mg·L-1Km, 10 mg·L-1Tc,30 mg·L-1Str)篩選接合子。接合子(NT, ApR, KmR, TcR和StrR) 用4種抗性的固體培養(yǎng)基篩選,并以平板上生長的每毫升菌落數(shù)(cfu·mL-1)進行計數(shù);受體菌(NS, StrR) 用鏈霉素抗性(30 mg·L-1Str)固體培養(yǎng)基篩選,并以平板上生長的每毫升菌落數(shù)(cfu·mL-1)進行計數(shù)。接合轉(zhuǎn)移頻率為接合子與受體菌數(shù)量的比值,計算公式為: f = NT(cfu·mL-1)/NS(cfu·mL-1)。
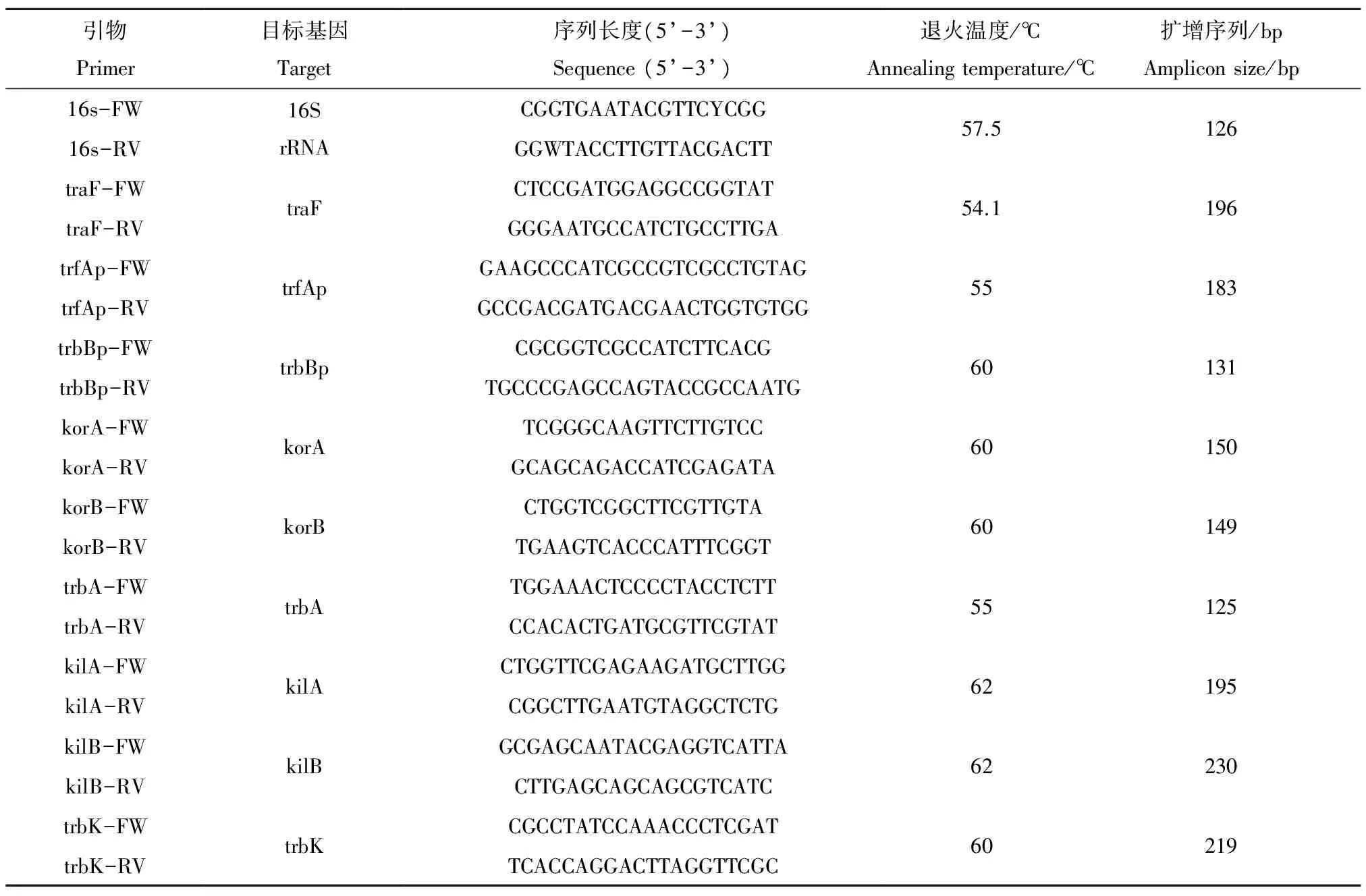
表1 目標(biāo)基因PCR引物和PCR條件Table 1 PCR primers and PCR conditions
注:FW是前引物, RV是后引物。
Note: FW, forward; RV, reverse.
同時,作為陰性對照,供體菌(E. coli DH5α)和受體菌(E. coli HB101和Salmonella enterica)也分別涂布到含4種抗性的LB固體培養(yǎng)基(100 mg·L-1Ap, 60 mg·L-1Km, 10 mg·L-1Tc,30 mg·L-1Str)上,以排除自發(fā)突變產(chǎn)生的具有4種抗性的假接合子。
1.3.2 接合體系RNA的提取
取上述接合菌液,4 ℃下離心收集菌體用于RNA提取。細菌RNA利用Takara的細菌RNA提取試劑盒進行。利用紫外分光光度計測定RNA的純度和濃度,采用TaKaRa第一鏈cDNA合成試劑盒,反轉(zhuǎn)錄合成cDNA第一鏈,逆轉(zhuǎn)錄的反應(yīng)體系和反應(yīng)條件均參考試劑盒的操作說明。
1.3.3 熒光定量PCR檢測mRNA的表達
檢測mRNA表達的目的基因為: (1) 調(diào)控質(zhì)粒RP4配對和轉(zhuǎn)移復(fù)制的調(diào)控基因trfAp和trbBp,以及3個相應(yīng)的全調(diào)控因子基因korA、 korB 和trbA; (2) 影響質(zhì)粒RP4接合轉(zhuǎn)移的基因traF、 kilA和kilB;(3) 進入阻礙體系基因trbK。所有基因的引物是根據(jù)有關(guān)文獻的報道或根據(jù)Genebank中該基因的全序列,利用DNAStar的PrimerSdecte程序進行設(shè)計。所有基因的引物序列及擴增產(chǎn)物片段長度見表1。
熒光定量PCR方法及體系、標(biāo)準(zhǔn)曲線的制備和基因表達量的計算參考文獻[25-26]。
1.4 數(shù)據(jù)統(tǒng)計與分析
采用SPSS 20.0對數(shù)據(jù)進行統(tǒng)計分析。實驗組和對照組數(shù)據(jù)進行 One way ANOVA檢驗差異性分析,并利用Student-Newman-Keuls (S-N-K) 檢驗,P< 0.05 (*)、P< 0.01(**)為具有顯著性差異。
2 結(jié)果與討論 (Results and discussion)
2.1 離子液體[BMIm][PF6]對質(zhì)粒RP4接合轉(zhuǎn)移的影響
同屬和跨屬接合轉(zhuǎn)移體系中,離子液體1-丁基-3-甲基咪唑六氟磷酸鹽([BMIm][PF6])都能促進質(zhì)粒RP4的接合轉(zhuǎn)移(圖1)。接合轉(zhuǎn)移頻率隨離子液體[BMIm][PF6]濃度的增加而增加(0.001~0.5 g·L-1),在同屬的從E. coli DH5α到E. coli HB101接合轉(zhuǎn)移中,0.5 g·L-1[BMIm][PF6] [(1.82±0.38)×10-3]的接合頻率是對照組[(2.07±0.62)×10-5]的80倍。另外,離子液體[BMIm][PF6]同樣促進跨屬的從E. coli DH5α到Salmonella enterica的接合轉(zhuǎn)移,并且接合轉(zhuǎn)移頻率隨離子液體[BMIm][PF6]濃度的增加而增加(0.001~0.5 g·L-1),在濃度0.5 g·L-1[(5.27±0.14)×10-5]時達到最大值,約為對照組[(3.33±0.67)×10-6]的16倍(圖1)。
相反,與0.5 g·L-1離子液體[BMIm][PF6]濃度相比,在離子液體[BMIm][PF6]最高的實驗濃度2.5 g·L-1時,接合轉(zhuǎn)移頻率開始降低(圖1)。這可能是因為在高濃度(2.5 g·L-1)時,高濃度[BMIm][PF6]對細菌的毒理效應(yīng)使體系中供體菌和受體菌的濃度過低。同屬和跨屬兩組接合轉(zhuǎn)移體系中,供體菌、受體菌和接合轉(zhuǎn)化子在濃度0~2.5 g·L-1離子液體[BMIm][PF6]范圍內(nèi)生存率可以看出,濃度在0~0.5 g·L-1時,體系中供體菌和受體菌數(shù)量基本沒變化,而在2.5 g·L-1高濃度時,供體菌存活率只有對照組的19.65%,受體菌存活率也只有對照組的19.90% (數(shù)據(jù)未列出)。邱志剛等[27]也報道過同樣的研究結(jié)果,在納米材料影響的接合轉(zhuǎn)移中,高濃度的納米材料使接合轉(zhuǎn)移頻率降低也是由于在高濃度的納米下,供體菌和受體菌的存活率過低。供體菌和受體菌的菌液濃度是接合轉(zhuǎn)移過程中的極其重要的因素,過低的菌液濃度會直接導(dǎo)致接合轉(zhuǎn)移轉(zhuǎn)移中細菌與細菌接觸的機會,從而導(dǎo)致接合轉(zhuǎn)移的頻率過低。
同時,研究結(jié)果還發(fā)現(xiàn)質(zhì)粒RP4在E. coli DH5α和E. coli HB101同屬之間的接合轉(zhuǎn)移明顯要高于在E. coli DH5α和Salmonella enterica跨屬之間的接合轉(zhuǎn)移(P<0.05, S-N-K 檢驗)。
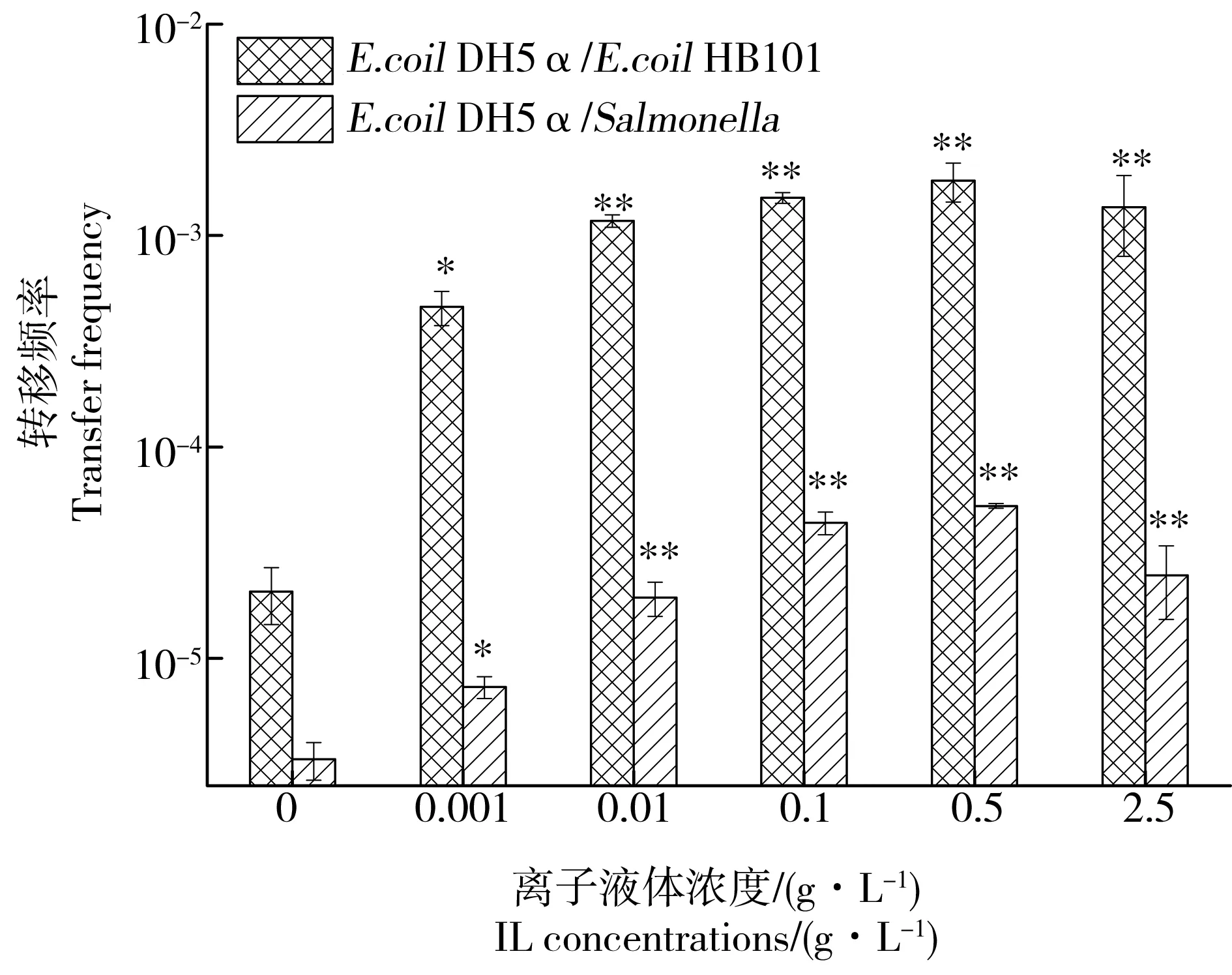
圖1 離子液體[BMIm][PF6]濃度對質(zhì)粒RP4分別 從E. coli DH5α到E. coli HB101和從E. coli DH5α到 Salmonella enterica接合轉(zhuǎn)移的影響注:接合轉(zhuǎn)移條件為pH 7.0, 溫度30 ℃, 接合時間12 h。Fig. 1 The conjugation of plasmid RP4 from E. coli DH5α to E. coli HB101 and from E. coli DH5α to Salmonella enterica was affected by [BMIm][PF6] concentration mated for 12 h at pH 7.0 and 30 ℃.
本文報道的離子液體[BMIm][PF6]促進抗生素抗性基因增強的效果低于0.5 g·L-1納米氧化鋁的效果,0.5 g·L-1納米氧化鋁使質(zhì)粒RP4從 Escherichia coli到Salmonella spp.的水平轉(zhuǎn)移頻率比空白對照組高200倍[27],但是0.5 g·L-1[BMIm][PF6]的促進效果要高于0.16 g·L-1VCl3的促進效果。Ganske等[28]報道了0.16 g·L-1VCl3使Photobacterium到Escherichia coli的水平轉(zhuǎn)移頻率比空白對照組高2倍。本文中離子液體[BMIm][PF6]的暴露濃度比文獻[29]報道的濃度低3~8倍,在這些高于本文濃度的離子液體暴露下,對Escherichia coli只表現(xiàn)出輕微抑制作用,并且對酵母Saccharomyces cerevisiae和細菌Pichia pastoris、Bacillus cereus的細胞生存能力沒有影響[30]。
通過水平轉(zhuǎn)移產(chǎn)生的獲得性抗性在抗生素抗性基因從外來菌向土著菌群的轉(zhuǎn)移和傳播過程中起著非常重要的作用[31-32]。在微生物面臨的眾多新的環(huán)境選擇性壓力時,抗生素、重金屬、異性生物質(zhì)、殺蟲劑、洗滌劑和有機溶劑等都對抗生素抗性基因起著很強的環(huán)境選擇性[6]。在本文中,離子液體作為一種選擇性壓力促進抗生素抗性基因在細菌間中的水平轉(zhuǎn)移,從而導(dǎo)致抗性基因在環(huán)境介質(zhì)中的傳播擴散。尤其值得注意的是,和抗生素不同,離子液體在環(huán)境介質(zhì)中不容易降解,會作為一種持續(xù)存在的環(huán)境選擇性壓力,長期起到促進抗性基因水平轉(zhuǎn)移的作用,從而增強離子液體這一新型環(huán)境污染物的環(huán)境生態(tài)風(fēng)險。
2.2 離子液體[BMIm][PF6]對全調(diào)控基因表達的影響
接合轉(zhuǎn)移受控于供體菌和受體菌之間接合橋的形成,該過程受到雜交對形成系統(tǒng)基因trbBp和質(zhì)粒轉(zhuǎn)移復(fù)制基因系統(tǒng)基因trfAp的控制[18-20]。它們分別調(diào)控質(zhì)粒RP4的接合和跨膜轉(zhuǎn)運過程。有文獻研究了在離子液體(1-ethyl-3-methylimidazolium chloride)暴露下,細菌Enterobacter lignolyticus轉(zhuǎn)運蛋白和藥物外排基因上調(diào),但是離子液體暴露下,接合轉(zhuǎn)移基因的表達調(diào)控還未見報道。在本文中,我們首次研究了離子液體[BMIm][PF6]通過調(diào)節(jié)接合基因的表達調(diào)控來增強質(zhì)粒的接合轉(zhuǎn)移。與空白對照組(0 g·L-1[BMIm][PF6])相比,基因trbBp的mRNA表達水平均明顯升高,0.5 g·L-1[BMIm][PF6]中基因trbBp的表達水平最高,同屬E. coli HB101中[(1.33±0.06)×10-4/16S rRNA]和跨屬Salmonella enterica [(9.18±0.86)×10-5/16S rRNA]中分別是空白對照組的18倍和12倍(圖2)。同樣,基因trfAp的 mRNA表達水平在同屬和跨屬中都隨離子液體濃度的增加而升高(圖2),這將有助于質(zhì)粒RP4的轉(zhuǎn)移和復(fù)制,促進抗生素抗性基因在環(huán)境介質(zhì)中的傳播擴散。
離子液體[BMIm][PF6]抑制全調(diào)控基因korA、 korB和trbA的表達。與空白對照組(0 g·L-1[BMIm][PF6])相比,在同屬和跨屬中,全調(diào)控基因korA、 korB和trbA的表達水平都隨著離子液體[BMIm][PF6]濃度的升高而明顯降低(0.001~0.5 g·L-1)(圖3)。基因korA和korB共同抑制trfAp, 而基因korB和trbA共同抑制trbBp。離子液體[BMIm][PF6]通過抑制korA和korB的mRNA表達使trfAp的表達上調(diào); 同時,通過抑制korB和trbA的mRNA表達使trbBp的表達上調(diào)。離子液體[BMIm][PF6]通過抑制基因korA、 korB和trbA的表達來提高接合和跨膜轉(zhuǎn)運基因的表達,從而促進質(zhì)粒RP4的接合轉(zhuǎn)移。

圖2 離子液體[BMIm][PF6]對細菌同屬和跨屬接合轉(zhuǎn)移中調(diào)控基因trbBp和跨膜轉(zhuǎn)運復(fù)制基因trfAp mRNA表達水平的影響Fig. 2 The mRNA expression levels of regulatory genes trbBp and transfer and replication gene trfAp of the plasmid RP4 in E. coli DH5α to E. coli HB101 and E. coli DH5α to Salmonella enterica transfers was affected by [BMIm][PF6] concentration
2.3 離子液體[BMIm][PF6]對水平轉(zhuǎn)移基因表達的影響
抗生素抗性基因的傳播主要通過細菌菌落的垂直傳遞(自我復(fù)制)和水平轉(zhuǎn)移2種途徑[5]。基因traF對水平轉(zhuǎn)移過程具有正調(diào)控作用,而基因kilA和kilB對垂直傳遞過程具有負(fù)調(diào)控作用[22]。在本文中,我們發(fā)現(xiàn)離子液體[BMIm][PF6]增強水平轉(zhuǎn)移基因的表達并抑制質(zhì)粒的垂直傳遞過程。與空白對照組(0 g·L-1[BMIm][PF6])相比,在同屬和跨屬中,基因traF的表達水平都顯著升高。在0.5 g·L-1[BMIm][PF6]實驗組,同屬E. coli HB101中基因traF的表達水平是對照組的80倍,跨屬Salmonella enterica中基因traF的表達水平是對照組的65倍(圖4)。同時,接合轉(zhuǎn)移頻率與基因traF的mRNA表達水平在同屬(圖5 a)和跨屬(圖5 b)接合轉(zhuǎn)移體系中都具有很好的正相關(guān)性。由此可見,同屬E. coli HB101和跨屬Salmonella enterica接合轉(zhuǎn)移中增強的基因trbK的mRNA表達有助于促進體系中質(zhì)粒RP4的接合轉(zhuǎn)移。
高濃度(2.5 g·L-1)的離子液體[BMIm][PF6]抑制細菌的存活率。在本文中,kilA和kilB的mRNA 表達水平隨離子液體濃度的升高而升高(圖4),說明離子液體[BMIm][PF6]使基因kilA和kilB對垂直傳遞的負(fù)調(diào)控作用隨濃度增加而增強。這也很好地解釋了離子液體通過抑制細菌生長來減少質(zhì)粒RP4的垂直傳遞。
2.4 離子液體[BMIm][PF6]對外組排基因表達的影響
進入排斥體系是受體菌對供體質(zhì)粒進入受體菌過程的阻礙作用。基因trbK主要調(diào)控質(zhì)粒RP4的進入排斥過程[23-24]。基因trbK的mRNA表達水平隨著離子液體[BMIm][PF6]濃度的增加而顯著降低(0.001~0.5 g·L-1)。在同屬的E. coli HB101接合轉(zhuǎn)移中,對照組基因trbK的mRNA表達水平比0.5 g·L-1[BMIm][PF6] [(1.68±0.32)×10-6/16S rRNA]高94倍,但是,在跨屬的Salmonella enterica接合轉(zhuǎn)移中對照組基因trbK的mRNA表達水平僅僅比0.5 g·L-1[BMIm][PF6] [(1.81±0.09)×10-4/16S rRNA]高5倍(圖6 a)。同時,接合轉(zhuǎn)移頻率與基因trbK的mRNA表達水平在同屬(圖6 b)和跨屬(圖6 c)接合轉(zhuǎn)移體系中都具有很好的負(fù)相關(guān)性。由此可見,同屬的E. coli HB101接合轉(zhuǎn)移中基因trbK的mRNA過低表達是同屬的接合轉(zhuǎn)移頻率比跨屬的接合轉(zhuǎn)移頻率高的主要原因。
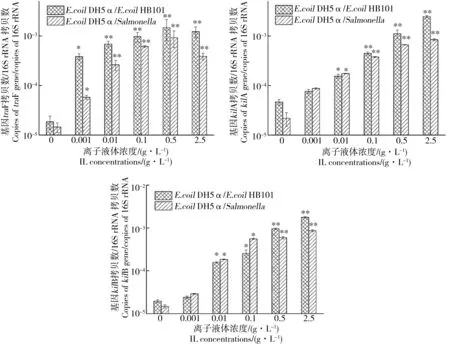
圖4 離子液體[BMIm][PF6]對細菌同屬和跨屬接合轉(zhuǎn)移中接合基因traF , kilA , kilB mRNA表達水平的影響Fig. 4 The mRNA expression levels of conjugative genes traF , kilA , kilB of the plasmid RP4 in E. coli DH5α to E. coli HB101 and E. coli DH5α to Salmonella enterica transfers was affected by [BMIm][PF6] concentration
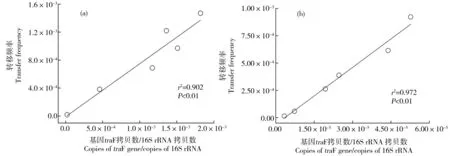
圖5 離子液體[BMIm][PF6]對細菌同屬(a)和跨屬(b)接合轉(zhuǎn)移的轉(zhuǎn)移頻率與基因traF相對含量的相關(guān)性Fig. 5 Correlation between the transfer frequency versus relative abundance of traF gene of the plasmid RP4 in E. coli DH5α to E. coli HB101(a) and E. coli DH5α to Salmonella enterica transfers (b) was affected by [BMIm][PF6] concentration
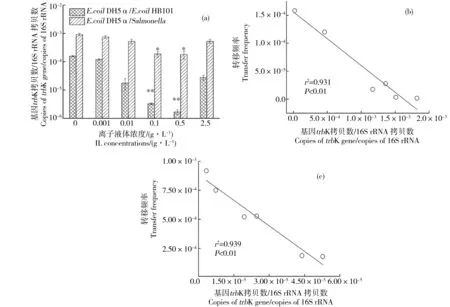
圖6 離子液體[BMIm][PF6]對細菌同屬和跨屬接合轉(zhuǎn)移中進入排斥體系基因trbK mRNA表達水平的影響(a); 離子液體[BMIm][PF6]對細菌同屬(b)和跨屬(c)接合轉(zhuǎn)移的轉(zhuǎn)移頻率與基因trbK相對含量的相關(guān)性Fig. 6 The mRNA expression levels of entry exclusion system gene trbK of the plasmid RP4 in E. coli DH5α to E. coli HB101 and E. coli DH5α to Salmonella enterica transfers was affected by [BMIm][PF6] concentration (a); Correlation between the transfer frequency versus relative abundance of trbK gene of the plasmid RP4 in E. coli DH5α to E. coli HB101 (b) and E. coli DH5α to Salmonella enterica (c) transfers was affected by [BMIm][PF6] concentration
[1] Zhu Y G, Zhao Y, Li B, et al. Continental-scale pollution of estuaries with antibiotic resistance genes [J]. Nature Microbiology, 2017, 2: 1-7
[2] Zhu B, Chen Q, Chen S, et al. Does organically produced lettuce harbor higher abundance of antibiotic resistance genes than conventionally produced? [J]. Environment International, 2017, 98: 152-159
[3] 蘇建強, 黃福義, 朱永官. 環(huán)境抗生素抗性基因研究進展[J]. 生物多樣性, 2013, 21(4): 481-487
Su J Q, Huang F Y, Zhu Y G. Antibiotic resistance genes in the environment [J]. Biodiversity Science, 2013, 21(4): 481-487 (in Chinese)
[4] Pallecchi L, Bartoloni A, Gotuzzo E, et al. Antibiotic Resistance in the Absence of Antimicrobial Use [M]// Microbial Drug Resistance. 2013: 120-132
[5] Thomas C M, Nielsen K M. Mechanisms of, and barriers to, horizontal gene transfer between bacteria[J]. Nature Reviews Microbiology, 2005, 3(9): 711-721
[6] Alonso A, Sánchez P, Martinez J L. Environmental selection of antibiotic resistance genes [J]. Environmental Microbiology Reports, 2001, 3(1): 1-9
[7] Wishart J F. Energy applications of ionic liquids [J]. Energy Environmental Science, 2009, 2(9): 956-961
[8] María D D, Maugeri P Z. Ionic liquids in biotransformations: From proof-of-concept to emerging deep-eutectic-solvents [J]. Current Opinion in Chemical Biology, 2011, 15(2): 220-225
[9] Welton T. Room-temperature ionic liquids. Solvents for synthesis and catalysis [J]. Chemical Reviews, 1999, 99(8): 2071-2084
[10] Lu J, Yan F, Texter J. Advanced applications of ionic liquids in polymer science [J]. Progress in Polymer Science, 2009, 34(5): 431-448
[11] Li Z, Jia Z, Luan Y, et al. Ionic liquids for synthesis of inorganic nanomaterials [J]. Current Opinion in Solid State and Materials Science, 2008, 12(1): 1-8
[12] Pham T P T, Cho C W, Yun Y S. Environmental fate and toxicity of ionic liquids: A review [J]. Water Research, 2010, 44(2): 352-372
[13] Zhao D, Liao Y, Zhang Z. Toxicity of ionic liquids [J]. Clean-Soil, Air, Water, 2007, 35(1): 42-48
[14] Khudyakov J I, Dhaeseleer P, Borglin S E, et al. Global transcriptome response to ionic liquid by a tropical rain forest soil bacterium, Enterobacter lignolyticus [J]. Proceedings of the National Academy of Sciences, 2012, 109(32): 2173-2182
[15] Xue Z, Sendamangalam V R, Gruden C L, et al. Multiple roles of extracellular polymeric substances on resistance of biofilm and detached clusters[J]. Environmental Science and Technology, 2012, 46(24): 13212-13219
[16] Yi L, Wang Q, Lu Q, et al. An ionic liquid facilitates the proliferation of antibiotic resistance genes mediated by class I integrons [J]. Environmental Science Technology Letters, 2014, 1(5): 266-270
[17] Haase J, Lurz R, Grahn A M, et al. Bacterial conjugation mediated by plasmid RP4: RSF1010 mobilization, donor-specific phage propagation, and pilus production require the same Tra2 core components of a proposed DNA transport complex [J]. Journal of Bacteriology, 1995, 177(16): 4779-4791
[18] Jagura-Burdzy G, Thomas C M. kfrA gene of broad host range plasmid RK2 encodes a novel DNA-binding protein [J]. Journal of Molecular Biology, 1992, 225(3): 651-660
[19] Antonenka U, Nlting C, Heesemann J, et al. Horizontal transfer of Yersinia high‐pathogenicity island by the conjugative RP4 attB target‐presenting shuttle plasmid [J]. Molecular Microbiology, 2005, 57(3): 727-734
[20] Llosa M, Gomis-R F X, Coll M, et al. Bacterial conjugation: A two-step mechanism for DNA transport [J]. Molecular Microbiology, 2002, 45(1): 1-8
[21] Jagura-Burdzy G, Khanim F, Smith C A, et al. Crosstalk between plasmid vegetative replication and conjugative transfer: Repression of the trfA operon by trbA of broad host range plasmid RK2 [J]. Nucleic Acids Research, 1992, 20(15): 3939-3944
[22] Sakai H, Komano T. DNA replication of IncQ broad-host-range plasmids in gram-negative bacteria [J]. Bioscience Biotechnology and Biochemistry, 1996, 60(3): 377-382
[23] Lyras D, Chan A W, Mcfarlane J, et al. The surface exclusion system of RP1: Investigation of the roles of trbJ and trbK in the surface exclusion, transfer, and slow-growth phenotypes [J]. Plasmid, 1994, 32(3): 254-261
[24] Haase J, Kalkum M, Lanka E. TrbK. A small cytoplasmic membrane lipoprotein, functions in entry exclusion of the IncP-alpha plasmid RP4 [J]. Journal of Bacteriology, 1996, 178(23): 6720-6729
[25] Chen H, Zhang M M. Occurrence and removal of antibiotic resistance genes in municipal wastewater and rural domestic sewage treatment systems in eastern China [J]. Environment International, 2013, 55: 9-14
[26] Luo Y, Mao D, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China [J]. Environmental Science and Technology, 2010, 44(19): 7220-7225
[27] Qiu Z G, Yu Y M, Chen Z, et al. Nanoalumina promotes the horizontal transfer of multiresistance genes mediated by plasmids across genera [J]. Proceedings of the National Academy of Sciences, 2012, 109(13): 4944-4949
[28] Suzuki S, Kimura M, Agusa T, et al. Vanadium accelerates horizontal transfer of tet(M) gene from marine Photobacterium to Escherichia coli [J]. FEMS Microbiology Letters, 2012, 336(1): 52-56
[29] Ganske F, Bornscheuer U T. Growth of Escherichia coli, Pichia pastoris and Bacillus cereus in the presence of the ionic liquids [BMIm][BF4] and [BMIm][PF6] and organic solvents [J]. Biotechnology Letters, 2006, 28(7): 465-469
[30] Song X L, Ye S Y, Xie R, et al. Effects of BMIm [PF6] treatments with different concentrations on microbial activity of Saccharomyces cerevisiae [J]. Korean Journal of Chemical Engineering, 2011, 28(9): 1902-1907
[31] Van Hoek A H, Mevius D, Guerra B, et al. Acquired antibiotic resistance genes: An overview [J]. Frontiers in Microbiology, 2011, 3(2): 1-17
[32] Li B, Zhang T. Biodegradation and adsorption of antibiotics in the activated sludge process [J]. Environmental Science and Technology, 2010, 44(9): 3468-3473
◆
AnIonicLiquid[BMIm][PF6]EnhancedthemRNAExpressionLevelsofConjugativeTransferGenesMediatedbyPlasmidRP4
Wang Qing1, 2, Luo Yi2, Cui Yuxiao2, Qi Liying1, Zhou Shuolei1, Yang Guang1,*
1. College of Energy and Environmental Engineering, Hebei Engineering Research Center for Water Pollution Control and Water Ecological Remediation, Hebei University of Engineering, Handan 056038, China2. Ministry of Education Key Laboratory of Pollution Processes and Environmental Criteria, Nankai University, Tianjin 300071, China
10.7524/AJE.1673-5897.20170112006汪慶, 羅義, 崔玉曉, 等. 離子液體[BMIm][PF6]增強抗性質(zhì)粒RP4介導(dǎo)的接合轉(zhuǎn)移基因mRNA的表達水平[J]. 生態(tài)毒理學(xué)報,2017, 12(3): 273-281
2017-01-12錄用日期2017-03-13
1673-5897(2017)3-273-09
X171.5
A
楊光(1987-),男,環(huán)境科學(xué)博士,講師,主要研究方向為環(huán)境生態(tài)學(xué)。
國家自然科學(xué)基金 (41473085);環(huán)境污染過程與基準(zhǔn)教育部重點實驗室開放基金(KL-PPEC-2016-1);河北省自然科學(xué)基金(No. D2017402109);河北省教育廳青年基金(No. QN2017035);邯鄲市科技計劃項目(1621212047)
汪慶(1985-),男,講師,研究方向為環(huán)境生態(tài)毒理學(xué),E-mail: wangqing@hebeu.edu.cn
*通訊作者(Corresponding author), E-mail: yang87537@hebeu.edu.cn
Wang Q, Luo Y, Cui Y X, et al. An ionic liquid [BMIm][PF6] enhanced the mRNA expression levels of conjugative transfer genes mediated by plasmid RP4 [J]. Asian Journal of Ecotoxicology, 2017, 12(3): 273-281 (in Chinese)

