基于DNA條形碼技術(shù)對(duì)蒼南海域仔稚魚(yú)的物種鑒定*
李 淵, 張麗艷, 張 然, 宋普慶, 王良明, 張 立, 林龍山**
(1. 國(guó)家海洋局第三海洋研究所,福建 廈門 361005; 2. 福建海洋研究所,福建 廈門 361013;3. 海洋赤潮災(zāi)害立體監(jiān)測(cè)技術(shù)與應(yīng)用國(guó)家海洋局重點(diǎn)實(shí)驗(yàn)室,上海 200137)
基于DNA條形碼技術(shù)對(duì)蒼南海域仔稚魚(yú)的物種鑒定*
李 淵1, 3, 張麗艷2, 張 然1, 宋普慶1, 王良明1, 張 立1, 林龍山1**
(1. 國(guó)家海洋局第三海洋研究所,福建 廈門 361005; 2. 福建海洋研究所,福建 廈門 361013;3. 海洋赤潮災(zāi)害立體監(jiān)測(cè)技術(shù)與應(yīng)用國(guó)家海洋局重點(diǎn)實(shí)驗(yàn)室,上海 200137)
采用DNA條形碼技術(shù)對(duì)蒼南海域冬季采集的仔稚魚(yú)進(jìn)行物種鑒定,以提高物種鑒定的準(zhǔn)確性。所有仔稚魚(yú)通過(guò)形態(tài)特征鑒定出10個(gè)形態(tài)種,但經(jīng)條形碼技術(shù)可鑒定出11個(gè)有效種。序列相似性結(jié)果顯示11個(gè)形態(tài)種中10種可鑒定到種的水平,1種無(wú)法匹配到相應(yīng)的物種。條形碼間隙分析結(jié)果顯示各物種的最小種間遺傳距離均高于該物種的最大種內(nèi)遺傳距離,存在明顯的條形碼間隙。由系統(tǒng)發(fā)育樹(shù)可以看出同一形態(tài)種的不同個(gè)體聚為一支系,各物種間能明顯區(qū)分開(kāi)。同時(shí)結(jié)合本次漁業(yè)資源現(xiàn)場(chǎng)調(diào)查結(jié)果,發(fā)現(xiàn)11種仔稚魚(yú)中僅有5種的成魚(yú)出現(xiàn)在本次調(diào)查中,剩余6種為新出現(xiàn)種類。可見(jiàn),DNA條形碼技術(shù)除了能提高仔稚魚(yú)物種鑒別的準(zhǔn)確性外,還能對(duì)物種多樣性調(diào)查起到一定的補(bǔ)充作用。
DNA條形碼;生物多樣性;仔稚魚(yú);序列相似性;條形碼間隙
作為魚(yú)類種群補(bǔ)充重要來(lái)源的魚(yú)類浮游生物主要是指魚(yú)類生活史早期發(fā)育階段,包括魚(yú)卵和仔稚魚(yú)[1]。魚(yú)類浮游生物的正確鑒定、分布和數(shù)量變化能更好地幫助我們了解魚(yú)類產(chǎn)卵場(chǎng)及早期的洄游路線,更有利于漁業(yè)資源的監(jiān)測(cè)、管理和補(bǔ)充量及海洋保護(hù)區(qū)的建設(shè)[2-3]。魚(yú)類浮游生物的傳統(tǒng)鑒定方法比較粗放,主要依靠其外部形態(tài)特征,并不能全部鑒定到種的水平,特別是隱存種和新記錄種[4-5];同時(shí)魚(yú)類浮游生物的形態(tài)特征復(fù)雜多變,而有的物種在發(fā)育期間變化較大,甚至存在變態(tài)發(fā)育,再加上鑒定者自身具備的知識(shí)參差不齊、主觀性較強(qiáng)及參考資料不全等因素,導(dǎo)致種類鑒定結(jié)果存在一定出入[6-8]。一種準(zhǔn)確、快速、高效、穩(wěn)定的鑒定方法就顯得十分必要。隨著DNA條形碼技術(shù)的提出,其在物種鑒定方面的優(yōu)勢(shì)越來(lái)越明顯[9-11],因?yàn)樵摷夹g(shù)突破了對(duì)經(jīng)驗(yàn)過(guò)度依賴和樣品完整性的局限,實(shí)現(xiàn)了樣品鑒定的標(biāo)準(zhǔn)化。一些學(xué)者嘗試用該技術(shù)來(lái)解決魚(yú)類浮游生物的物種鑒定和物種多樣性的調(diào)查[2-3, 7-8, 12-13]。
蒼南海域位于浙江省南端,是東海中部和南部的交界海域,屬中亞熱帶海洋性氣候區(qū),初級(jí)生產(chǎn)力水平較高,物種豐富,是許多游泳動(dòng)物的重要產(chǎn)卵場(chǎng)[14]。有關(guān)該海域的研究?jī)H見(jiàn)于游泳動(dòng)物生態(tài)位及種間的聯(lián)結(jié)性[14],而有關(guān)魚(yú)類仔稚魚(yú)鑒定的研究尚屬空白。本研究利用DNA條形碼技術(shù)對(duì)采集于蒼南海域的仔稚魚(yú)進(jìn)行種類鑒定,以明確仔稚魚(yú)種類與現(xiàn)場(chǎng)資源調(diào)查捕獲魚(yú)類種類的關(guān)系,為條形碼技術(shù)應(yīng)用于魚(yú)類浮游生物種類鑒定提供參考,并探討該技術(shù)對(duì)物種多樣性調(diào)查的補(bǔ)充作用。
1 材料與方法
1.1樣品采集與鑒定
于2016年2月20日在浙江省蒼南海域進(jìn)行底拖網(wǎng)調(diào)查,所有樣品低溫保存,帶回實(shí)驗(yàn)室進(jìn)行種類鑒定,在樣品分析時(shí)發(fā)現(xiàn)漁獲物中夾雜大量的仔稚魚(yú),依據(jù)外部形態(tài)特征(體型、鰭條發(fā)育、體色等)對(duì)其進(jìn)行初步分類,測(cè)量其體長(zhǎng),并進(jìn)行標(biāo)號(hào),留待后續(xù)分子遺傳學(xué)研究。
為了進(jìn)一步確定仔稚魚(yú)鑒定的準(zhǔn)確性,本研究采用DNA條形碼技術(shù)對(duì)其做進(jìn)一步的分析。對(duì)數(shù)量較多的形態(tài)種隨機(jī)選取10或15尾,數(shù)量較少的則全部用于后續(xù)的分子遺傳學(xué)研究,同時(shí)取在資源調(diào)查中出現(xiàn)過(guò)仔稚魚(yú)種類的成魚(yú)1尾和GenBank數(shù)據(jù)庫(kù)中的參考序列用于后續(xù)的物種鑒定(見(jiàn)表1)。

表1 蒼南調(diào)查海域仔稚魚(yú)種類鑒定信息及遺傳多樣性信息Table 1 Information and molecular indices of fish larvae identified in the investigated waters of Cangnan
1.2實(shí)驗(yàn)方法
取仔稚魚(yú)和成魚(yú)適量肌肉組織,采用海洋動(dòng)物組織DNA提取試劑盒提取基因組DNA,4℃保存?zhèn)溆谩S糜跀U(kuò)增COI片段的引物為[9]:F1:5′-TCAACCAACCACAAAGACATTGGCAC-3′;R1:5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′。PCR反應(yīng)體系為25 μL[11]:0.15 μLTaqDNA聚合酶,2.5 μL dNTP(2 mmol/L),2 μL 10×Taq buffer(含Mg2+),正反引物各1 μL(2 mmol/L),DNA模板1 μL,其余dd H2O補(bǔ)足。反應(yīng)條件為:95 ℃預(yù)變性5 min;95 ℃變性30 s,50 ℃退火30 s,72 ℃延伸30 s,30個(gè)循環(huán);72 ℃延伸10 min。以上反應(yīng)均設(shè)陰性對(duì)照以排除DNA污染的情況。取3 μL PCR擴(kuò)增產(chǎn)物進(jìn)行1.5%瓊脂糖凝膠電泳檢測(cè)(U=5 V/cm),對(duì)符合測(cè)序濃度的PCR產(chǎn)物送生物公司進(jìn)行純化和序列雙向測(cè)定。
1.3數(shù)據(jù)處理
對(duì)獲得的全部序列進(jìn)行人工比對(duì)并輔以校正,截取有效、準(zhǔn)確的目的片段用于后續(xù)分析。將所有序列在NCBI數(shù)據(jù)庫(kù)中進(jìn)行BLAST序列相似性比對(duì),采用兩兩序列間遺傳相似度>98%的為同一物種,92%~98%為同一屬,85%~92%為同一科的標(biāo)準(zhǔn)對(duì)蒼南海域的幼魚(yú)進(jìn)行種類鑒定[2-3],選取最佳匹配值(The best match)和種間最佳匹配值(Interspecific best match)(序列匹配百分?jǐn)?shù)為數(shù)值),選取2%為種間的遺傳分化界限,對(duì)所有比對(duì)結(jié)果劃分為三種模式[3, 15]:(Ⅰ)準(zhǔn)確比對(duì)(Match to species):最佳比對(duì)值(The best match)低于遺傳分化界限,最佳鄰接比對(duì)值(The nearest neighbor best match)高于遺傳分化界限,該模式下能鑒定到種;(Ⅱ)不確定比對(duì)(Uncertain match):最佳比對(duì)值和最佳鄰接比對(duì)值均低于遺傳分化界限,該模式下鑒定到的種是不確定的或者錯(cuò)誤的;(Ⅲ)無(wú)法比對(duì)(Unmatched):最佳比對(duì)值和最佳鄰接比對(duì)值均高于遺傳分化界限,該模式下由于條形碼參考數(shù)據(jù)庫(kù)(Reference library)的限制導(dǎo)致物種無(wú)法鑒定。基于K2P模型利用Mega 4.0軟件計(jì)算各物種間的條形碼間隙(Barcoding gap)和構(gòu)建鄰接關(guān)系樹(shù)[15-16];利用Arlequin軟件計(jì)算各物種的單倍型數(shù)量和單倍型多樣度。
2 結(jié)果
2.1魚(yú)類種類組成
本次調(diào)查漁獲成魚(yú)種類36種,隸屬于9目16科29屬(附錄1),其中鱸形目占絕對(duì)優(yōu)勢(shì),為17種,占總種類數(shù)的47.22%,其次是鯡形目和鰈形目分別為7種和6種,占19.44%和16.67%,其余各目種類數(shù)均為1種。在科級(jí)水平上,鰈形目的舌鰨科種類數(shù)最多,為6種,占總種類數(shù)的16.67%,其次為鳀科和蝦虎魚(yú)科種類,分別為5種,占13.89%。在屬級(jí)水平上,舌鰨屬的種類數(shù)最多,為6種,其余各屬分布較為平均。

附錄1:蒼南調(diào)查海域漁獲魚(yú)類名錄Appendix 1:List of fish in the investigated waters of Cangnan in Winter
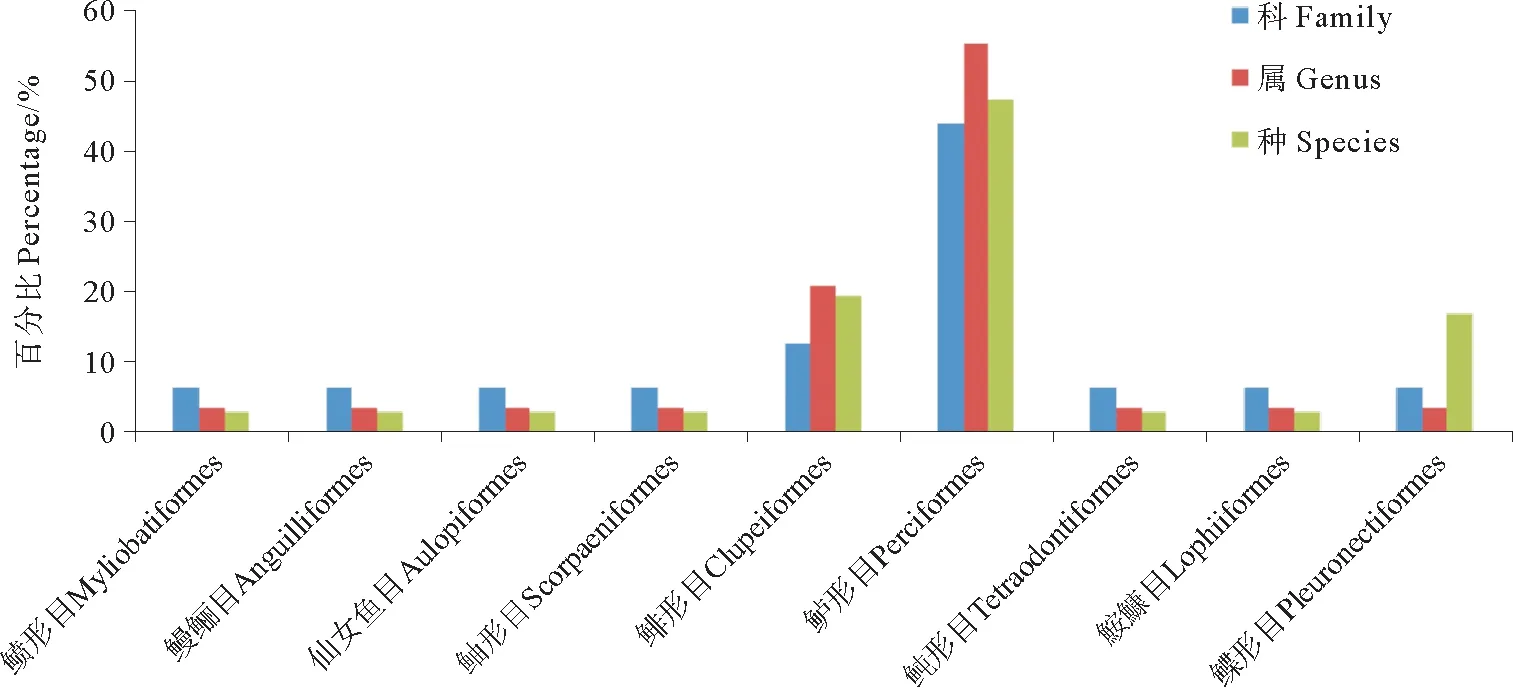
圖1 蒼南調(diào)查海域漁獲魚(yú)類物種組成
除上述魚(yú)類外,尚有一定數(shù)量的仔稚魚(yú)存在,體長(zhǎng)范圍在26.4~133.6 mm,依據(jù)其外部形態(tài)特征對(duì)其進(jìn)行初步鑒定,分為10個(gè)形態(tài)種,其中數(shù)量最多的為鱸形目魚(yú)類(見(jiàn)表1)。
2.2 COI序列相似性比對(duì)
對(duì)所有88條仔稚魚(yú)的目的序列進(jìn)行相似性比對(duì)發(fā)現(xiàn),序列相似度在98%~100%之間的有85條,占總數(shù)的96.59%;相似性在92%~98%的有3條,占總數(shù)的3.41%;無(wú)相似性在85%~92%的序列。
每一個(gè)體的最佳匹配值和種間最佳匹配值之間的比較能更直接地反映比對(duì)的結(jié)果(見(jiàn)圖2)。以相似度98%作為同一物種的界限對(duì)所有個(gè)體進(jìn)行分析,結(jié)果顯示有85個(gè)體屬于模式Ⅰ(96.59%),能準(zhǔn)確鑒定到種;有2個(gè)體屬于模式Ⅱ(2.27%),屬于模糊種;有1個(gè)體屬于模式Ⅲ(1.14%),無(wú)法匹配到種的水平。
由序列相似性比對(duì)結(jié)果可以看出(見(jiàn)表1),只有鐮鯧、龍頭魚(yú)和六絲鈍尾蝦虎魚(yú)(小個(gè)體)的鑒定結(jié)果與形態(tài)鑒定結(jié)果一致;形態(tài)種棘頭梅童魚(yú)實(shí)際為大黃魚(yú),綠鰭魚(yú)實(shí)際上為小眼綠鰭魚(yú);鯛科魚(yú)類為二長(zhǎng)棘犁齒鯛,鲉科魚(yú)類為褐菖鲉,髭鯛屬魚(yú)類為橫帶髭鯛,鮨科魚(yú)類為花鱸。模糊種(形態(tài)種食蟹豆齒鰻)與裸鰭蟲(chóng)鰻(Muraenichthysgymnopterus,KX215178)的相似性為99%~100%,而與西里伯蛇鰻(Ophichthuscelebicus,KX215180)的相似性為99%。在形態(tài)特征上,裸鰭蟲(chóng)鰻與西里伯蛇鰻二者最明顯的區(qū)別在于有無(wú)尾鰭,從本研究的樣品可以明顯觀察到尾鰭的存在,因此可以推斷該種為裸鰭蟲(chóng)鰻。無(wú)法匹配種(形態(tài)種六絲鈍尾蝦虎魚(yú)大個(gè)體)相似性最高為85%,相似性最高的物種為鱷鲬(Cociellacrocodila,KP267636),隸屬于鲉形目鲬科,依據(jù)遺傳相似性鑒定標(biāo)準(zhǔn)可以推斷該物種應(yīng)屬于鲬科(Platycephalidae)。
經(jīng)序列比對(duì)和種類整理發(fā)現(xiàn),所有仔稚魚(yú)存在11個(gè)種(見(jiàn)表1),隸屬于4目11科11屬,其中10個(gè)種能確定到種,1個(gè)未確定。鱸形目所占種類最多為6種,分屬于6科6屬,鲉形目包含3種,分屬于3科3屬,仙女魚(yú)目和鰻鱺目分別包含1種,分屬于1科1屬。11種仔稚魚(yú)中僅有5種的成魚(yú)在本次資源調(diào)查中出現(xiàn),分別為大黃魚(yú)(Larimichthyscrocea)、褐菖鲉(Sebastiscusmarmoratus)、花鱸(Lateolabraxmaculatus)、龍頭魚(yú)(Harpadonnehereus)和六絲鈍尾蝦虎魚(yú)(Amblychaeturichthyshexanema),剩余6種為新出現(xiàn)種,分別為鐮鯧(Pampusechinogaster)、二長(zhǎng)棘犁齒鯛(Evynniscardinalis)、橫帶髭鯛(Hapalogenysanalis)、小眼綠鰭魚(yú)(Chelidonichthysspinosus)、裸鰭蟲(chóng)鰻和鲬科魚(yú)類。
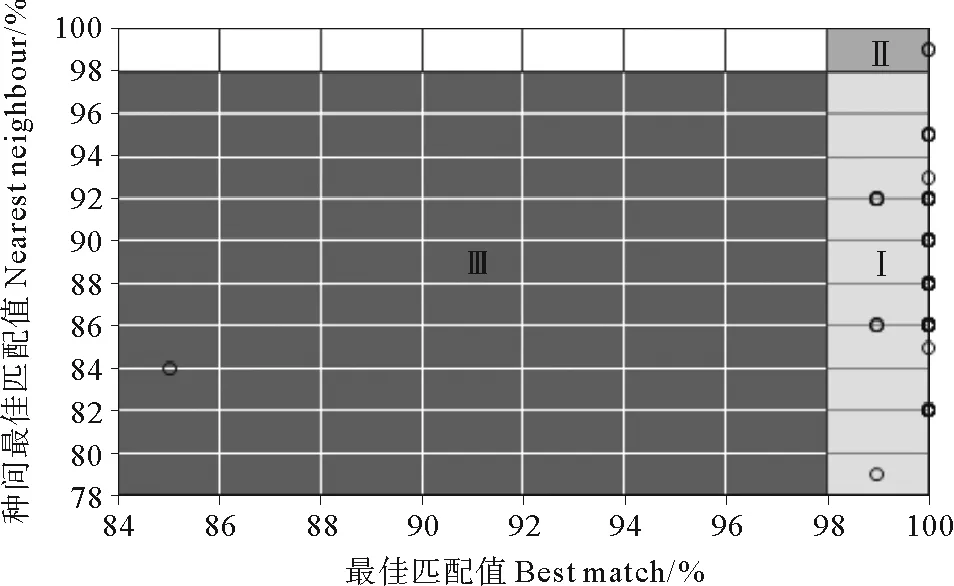
(Ⅰ-鑒定到種;Ⅱ-模糊種;Ⅲ-無(wú)法匹配種。Ⅰ- match to species, Ⅱ- ambiguous species, Ⅲ- unmatched species.黑色線代表物種間分化的界限。Dotted lines correspond to 2% divergence threshold for species boundaries.)
圖2 每一個(gè)體最佳匹配值和種間最佳匹配值之間的比較
Fig.2 Best match compared with nearest neighbor (similarity percentage) for each specimen
2.3條形碼間隙和遺傳多樣性
基于K2P模型計(jì)算各種間的遺傳距離范圍為0.21~0.35,平均遺傳距離為0.26,明顯達(dá)到種的水平;各種內(nèi)的遺傳距離均低于0.017(六絲鈍尾蝦虎魚(yú)),符合種內(nèi)界限。種內(nèi)遺傳距離和種間遺傳距離基本符合“10乘法則”[17]。各物種的最小種間遺傳距離均高于該物種的最大種內(nèi)遺傳距離,由圖3可以明顯看出種內(nèi)和種間遺傳距離間存在條形碼間隙,不存在重疊,證實(shí)各物種的有效性。
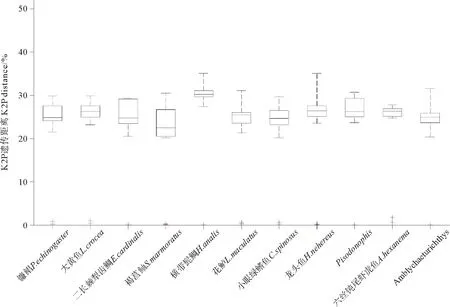
(種間遺傳距離的最大值和最小值分別用上下橫杠進(jìn)行表示,中間橫杠為中位值,藍(lán)線代表最大種內(nèi)遺傳距離;紅線代表平均種內(nèi)遺傳距離。Median interspecific distances with maximum and minimum values are represented by the upper and lower bars, respectively. Blue line: Maximum intraspecific distance; Red line: Mean intraspecific distance.)
圖3 基于K2P模型計(jì)算各物種的條形碼間隙
Fig.3 DNA barcoding gaps for all of the species based on the K2P model
剔除僅一個(gè)單倍型的4種魚(yú)類,計(jì)算剩余7種魚(yú)類的單倍型數(shù)量和單倍型多樣度(見(jiàn)表1)。六絲鈍尾蝦虎魚(yú)的單倍型數(shù)量最高;其次為鐮鯧、大黃魚(yú)和花鱸均為6;褐菖鲉、小眼綠鰭魚(yú)和龍頭魚(yú)的單倍型數(shù)分別為4、3和2。單倍型多樣度范圍為(0.250±0.180)~(0.867±0.067),最高的為六絲鈍尾蝦虎魚(yú),其次為大黃魚(yú)、花鱸和小眼綠鰭魚(yú),鐮鯧和褐菖鲉的單倍型多樣度較接近,龍頭魚(yú)的最低。
2.4系統(tǒng)發(fā)育樹(shù)
基于11種仔稚魚(yú)COI單倍型、部分成魚(yú)序列和GenBank數(shù)據(jù)庫(kù)中的參考序列構(gòu)建鄰接關(guān)系樹(shù)(見(jiàn)圖4),可以看出大部分同一形態(tài)種的不同個(gè)體聚類到同一支系內(nèi),鲬科魚(yú)類SP. 明顯不同于六絲鈍尾蝦虎魚(yú),獨(dú)自成為一支系。各物種間能明顯分開(kāi),形成獨(dú)立的11個(gè)支系,這一結(jié)果與各物種間的條形碼間隙結(jié)果相對(duì)應(yīng),表明各物種的有效性。各種類的照片同樣在圖4中給出,但是由于前期鑒定和樣品保存的原因,未對(duì)鲬科魚(yú)類SP. 的標(biāo)本進(jìn)行拍照,僅保留其肌肉樣品。
3 討論
專業(yè)分類學(xué)家的減少、隱存種和新種的不斷發(fā)現(xiàn)及參考資料的局限性導(dǎo)致了基于外部形態(tài)特征對(duì)物種進(jìn)行鑒定往往存在一定的出入,而對(duì)仔稚魚(yú)鑒定的準(zhǔn)確性更低,DNA條形碼技術(shù)的出現(xiàn)能在一定程度上很好地解決這一問(wèn)題。本研究從序列相似性比較結(jié)果(見(jiàn)圖2)可以看出,模式Ⅰ下能準(zhǔn)確鑒定到種,本次調(diào)查出現(xiàn)的仔稚魚(yú)大部分(96.59%)處于該模式下,如大黃魚(yú)、龍頭魚(yú)等。模式Ⅱ下的物種比對(duì)結(jié)果比較混亂,同一序列可能與多物種的相似性在98%以上,如六絲鈍尾蝦虎魚(yú)與矛尾蝦虎魚(yú)(Chaeturichthysstigmatias)(KC495071)、裸鰭蟲(chóng)鰻與西里伯蛇鰻(KX215180)的相似性均在99%及以上,出現(xiàn)這種情況可能與漸滲雜交和系統(tǒng)分類有關(guān)[3, 18]。在本研究中漸滲雜交顯然不是造成這種情況的主要原因,更多的可能是與序列提交者分類鑒別水平和條形碼參考數(shù)據(jù)庫(kù)的準(zhǔn)確性有關(guān)。模式Ⅲ下的物種難以找到與之高匹配的數(shù)據(jù),如鲬科魚(yú)類SP.。不管上述哪種模式,要實(shí)現(xiàn)物種的準(zhǔn)確鑒定,必須建立準(zhǔn)確且高覆蓋度的參考序列數(shù)據(jù)庫(kù)。
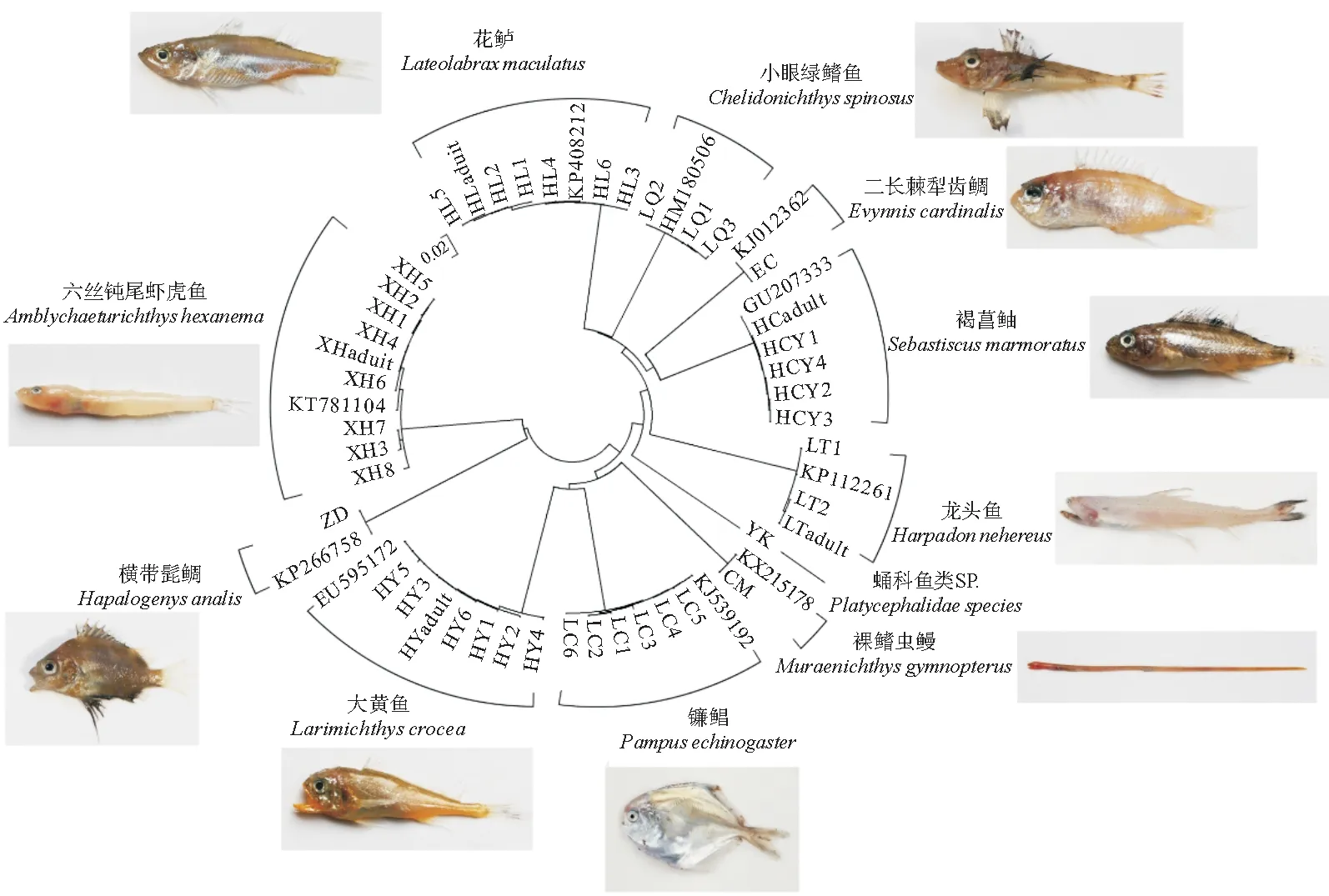
(圖中- adult為仔稚魚(yú)的成魚(yú)序列。-adult represented the adult sequences of fish of larvae.)
利用平均種間遺傳距離判斷種內(nèi)和種間的關(guān)系往往會(huì)造成一定的錯(cuò)誤,但條形碼間隙可以有效的彌補(bǔ)這一缺陷,即比較最低種間遺傳距離和最高種內(nèi)遺傳距離[15-16]。本研究的11種魚(yú)類各種間平均遺傳距離均高于0.2,種內(nèi)平均遺傳距離均低于0.2,且各種間均能發(fā)現(xiàn)清晰的條形碼間隙,在系統(tǒng)發(fā)育樹(shù)上同樣可以看出各物種均各自聚為一支系,能將各種區(qū)分開(kāi),因此能保證各種的有效性和準(zhǔn)確性。
片面的取樣、錯(cuò)誤的種類鑒定及薄弱的分類學(xué)研究均會(huì)阻礙物種的鑒定和新物種的發(fā)現(xiàn),影響調(diào)查區(qū)域內(nèi)生物多樣性的評(píng)估[19]。本研究11種仔稚魚(yú)中有6個(gè)種的成魚(yú)在本次資源調(diào)查中未曾出現(xiàn),進(jìn)一步說(shuō)明了仔稚魚(yú)研究的重要性,同時(shí)也可以看出DNA條形碼技術(shù)對(duì)物種多樣性調(diào)查在一定程度上有補(bǔ)充作用,能更真實(shí)地反映調(diào)查區(qū)域內(nèi)的物種多樣性水平。
同樣,在仔稚魚(yú)的鑒定過(guò)程中不能因?yàn)檎{(diào)查中出現(xiàn)同屬成魚(yú)而將其武斷定種,如橫帶髭鯛和斜帶髭鯛,六絲鈍尾蝦虎魚(yú)和鲬科魚(yú)類SP.。對(duì)外形十分相似物種的鑒定更能考驗(yàn)分類者的知識(shí)水平,在不能準(zhǔn)確定種的情況下應(yīng)將其鑒定到有把握的屬或科級(jí)水平,如大黃魚(yú)和棘頭梅童魚(yú)。應(yīng)加強(qiáng)對(duì)經(jīng)濟(jì)價(jià)值較低且常見(jiàn)物種樣品的采集和保存,以增加條形碼數(shù)據(jù)庫(kù)的有效數(shù)據(jù),如食蟹豆齒鰻(僅有16S和12S片段,無(wú)COI片段)。浙江省多年以來(lái)開(kāi)展大黃魚(yú)幼魚(yú)的增殖放流[20],本次調(diào)查發(fā)現(xiàn)該海域中大黃魚(yú)有一定的產(chǎn)量,同樣在仔稚魚(yú)中也發(fā)現(xiàn)大量的大黃魚(yú),表明增殖放流產(chǎn)生了一定的效果,應(yīng)進(jìn)一步加強(qiáng)大黃魚(yú)的保護(hù)和管理,不能松懈。鲬科魚(yú)類SP. 未發(fā)現(xiàn)與之相匹配的序列,由于仍有很多物種的COI基因序列尚未被報(bào)道,因此無(wú)法確認(rèn)該種是屬于已報(bào)道而未發(fā)表相關(guān)序列信息的種類,還是屬于隱存種。因此對(duì)某些近緣種或外形相似的種類,要同時(shí)結(jié)合形態(tài)學(xué)、分子遺傳學(xué)和地理學(xué)才能對(duì)其分類地位進(jìn)行更準(zhǔn)確地定位[21]。
本研究依據(jù)形態(tài)特征對(duì)蒼南調(diào)查海域仔稚魚(yú)進(jìn)行種類鑒定的準(zhǔn)確率不足三成,表明研究者在仔稚魚(yú)形態(tài)鑒定方面的羸弱,更表明DNA條形碼技術(shù)的有效性。條形碼技術(shù)與傳統(tǒng)分類學(xué)相結(jié)合,不僅可提高物種鑒別的準(zhǔn)確性,而且可以對(duì)生物多樣性調(diào)查起到一定的補(bǔ)充作用,是對(duì)生物多樣性進(jìn)行高效、快速、可靠評(píng)估的新工具,但分類學(xué)家也不能過(guò)分依靠條形碼技術(shù),仍要加強(qiáng)自身分類知識(shí)水平的積累。正確的形態(tài)鑒定提供準(zhǔn)確的條形碼,準(zhǔn)確的條形碼指導(dǎo)后續(xù)的物種鑒定,只有如此才能形成良好的物種鑒定循環(huán),有助于今后的種類鑒定、隱存種的發(fā)現(xiàn)、多樣性調(diào)查及系統(tǒng)發(fā)育等研究。
[1] 張仁齋, 陸穗芬, 趙傳絪, 等. 中國(guó)近海魚(yú)卵與仔魚(yú)[M]. 上海:上海科學(xué)技術(shù)出版社, 1985: 1-12.
Zhang R Z, Lu S F, Zhao C Y, et al. The Fish Eggs and Larvae from Coastal Waters of China[M]. Shanghai: Shanghai Science and Technology Press, 1985: 1-12.
[2] Ko H L, Wang Y T, Chiu T S, et al. Evaluating the accuracy of morphological identification of larval fishes by applying DNA barcoding[J]. PLoS ONE, 2013, 8(1): e53451. doi: 10. 1371/journal. pone. 0053451.
[3] Hubert N, Espiau B, Meyer C, et al. Identifying the ichthyoplankton of a coral reef using DNA barcodes[J]. Molecular Ecology Resources, 2015, 15: 57-67.
[4] Bogorodsky S V, Suzuki T, Mal A O. Description of a new species of Priolepis (Perciformes: Gobiidae) from the Red Sea, a new record ofPriolepiscompita, and a distributional range extension ofTrimmafishelsoni[J]. Zootaxa, 2016, 4150(2): 168-184.
[5] Tornabene L, Van Tassell J L, Gilmore R G, et al. Molecular phylogeny, analysis of character evolution, and submersible collections enable a new classification of a diverse group of gobies (Teleostei: Gobiidae: Nes subgroup), including nine new species and four new genera[J]. Zoological Journal of the Linnean Society, 2016, 177(4): 764-812.
[6] Carvalho D C, Palhares R M, Drummond M G, et al. DNA Barcoding identification of commercialized seafood in South Brazil: a governmental regulatory forensic program[J]. Food Control, 2015, 50: 784-788.
[7] 李海濤, 張保學(xué), 高陽(yáng), 等. DNA條形碼技術(shù)在海洋貝類鑒定中的實(shí)踐: 以大亞灣生態(tài)監(jiān)控區(qū)為例[J]. 生物多樣性, 2105, 23(3): 299-305.
Li H T, Zhang B X, Gao Y, et al. DNA barcoding in species identification of seashells: A case study in the ecological monitoring zone of Daya Bay, Guangdong[J]. Biodiversity Science, 2015, 23(3): 299-305.
[8] 周美玉, 陳驍, 楊圣云. 采用DNA條形碼技術(shù)對(duì)廈門海域魚(yú)卵、仔稚魚(yú)種類的鑒定[J]. 海洋環(huán)境科學(xué), 2015, 34(1): 120-125,135.
Zhou M Y, Chen X, Yang S Y. Identification of several fish eggs and larvae by DNA barcoding in Xiamen water[J]. Marine Environmental Science, 2015,34(1): 120-125,135.
[9] Hebert P D N, Cywinska A, Ball S L. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society of London B: Biological Sciences, 2003, 270(1512): 313-321.
[10] Chen W T, Ma X H, Shen Y J, et al. The fish diversity in the upper reaches of the Salween River, Nujiang River, revealed by DNA barcoding[J]. Scientific Reports, 2015, doi: 10. 1038/srep17437.
[11] 李淵, 宋娜, Khan Fozia Siyal, 等. 銀鯧形態(tài)特征與DNA條形碼研究[J]. 水產(chǎn)學(xué)報(bào), 2013, 37(11): 1-9.
Li Y, Song N, Khan F S, et al. New evidence of morphological and DNA barcoding ofPampusargenteus(Euphrasen, 1788)[J]. Journal of Fisheries of China, 2013, 37(11): 1-9.
[12] Ji Y, Ashton L, Pedley S M, et al. Reliable, verifiable and efficient monitoring of biodiversity via metabarcoding[J]. Ecology Letters, 2013, 16: 1245-1257.
[13] Perl R G B, Nagy Z T, Sonet G, et al. DNA barcoding Madagascar’s amphibian fauna[J]. Amphibia Reptilia, 2014, 35: 197-206.
[14] 胡成業(yè), 水玉躍, 杜肖, 等. 浙江蒼南南部沿岸海域主要游泳動(dòng)物生態(tài)位及種間聯(lián)結(jié)性[J]. 應(yīng)用生態(tài)學(xué)報(bào), 2015, 26(10): 3195-3201.
Hu C Y, Shui Y Y, Du X, et al. Niche and interspecific association of nekton in the south coastal waters of Cangnan, Zhejiang, China[J]. Chinese Journal of Applied Ecology, 2015, 26(10): 3195-3201.
[15] Hebert P D N, Stoeckle M Y, Zemlak T S, et al. Identification of birds through DNA barcodes[J]. PLoS Biology, 2004, 2(10): e312. doi. org/10. 1371/journal. pbio. 0020312.
[16] Shen Y, Guan L, Wang D, et al. DNA barcoding and evaluation of genetic diversity in Cyprinidae fish in the midstream of the Yangtze River[J]. Ecology and Evolution, 2016, 6(9): 2702-2713.
[17] Ward R D, Zemlak T S, Innes B H, et al. DNA barcoding Australia’s fish species[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360: 1847-1857.
[18] Meyer C, Paulay G. DNA barcoding: Error rates based on comprehensive sampling[J]. PLoS Biology, 2005, 3(12): 2229-2238.
[19] 高連明. DNA條形碼在生物多樣性編目與評(píng)價(jià)中的應(yīng)用[J]. 生物多樣性, 2015, 23(3): 286-287.
Gao L M. Applications of DNA barcoding in biodiversity inventory and assessment[J]. Biodiversity Science, 2015, 23(3): 286-287.
[20] 張其永, 洪萬(wàn)樹(shù), 楊圣云, 等. 大黃魚(yú)增殖放流的回顧和展望[J]. 現(xiàn)代漁業(yè)信息, 2010, 25(12): 3-5,12.
Zhang Q Y, Hong W S, Yang S Y, et al. Review and prospects in the restocking of the large yellow croaker (Larimichthyscrocea)[J]. Modern Fisheries Information, 2010, 25(12): 3-5, 12.
[21] 王太, 張艷萍, 管麗紅, 等. 甘肅省魚(yú)類資源現(xiàn)狀及DNA條形碼在魚(yú)類物種鑒定中的應(yīng)用[J]. 生物多樣性, 2015, 23(3): 306-313.
Wang T, Zhang Y P, Guan L H, et al. Current freshwater fish resources and the application of DNA barcoding in species identification in Gansu Province[J]. Biodiversity Science, 2015, 23(3): 306-313.
IdentificationofSeveralFishLarvaeBasedonDNABarcodingintheInvestigatedWatersofCangnan
LI Yuan1, 3, ZHANG Li-Yan2, ZHANG Ran1, SONG Pu-Qing1, WANG Liang-Ming1, ZHANG Li1, LIN Long-Shan1
(1. Third Institute of Oceanographgy, SOA, Xiamen 361005, China; 2. Fujian Institute of Oceanography, Xiamen 361013, China; 3. The Key Laboratory of Integrated Marine Monitoring and Applied Technologies for Harmful Algal Blooms, SOA, Shanghai 200137, China)
The investigated waters of Cangnan located in the south of Zhejiang province, where is an important spawning ground for many marine species. Fishery resources survey in this water was conducted by bottom trawling in the winter of 2016 and a large number of fish larvae were collected. To improve the accuracy of species identification, DNA barcoding based on COI gene was employed to identify fish larvae collected from the investigated waters. Ten morphospecies were identified based on morphological characteristics, while 11 species were identified by DNA barcoding analyses. After running blast searches in NCBI, a match of 98%~100% was found in 96. 59% of all individuals, a match of 92%~98% was 2. 27%, and 85%~92% was 1. 14%. Results of sequence similarity showed that ten of 11 species were identified to the species level, while one species was unmatched. There would be a clear overlap (barcoding gap) between intraspecific and interspecific variabilities in each species, as the smallest interspecific divergences were well below 2%, but the largest intraspecific divergences reached above 2%. Different individuals were clustered together, and different species could be unambiguously identified from NJ tree. Results noted above indicated that there were 11 valid species identified from this survey at last. Compared with the survey results of fishery resources, only five fish larvae species were identified with adult fish species found in this survey, and the six remaining species were new types. The COI barcoding diversity was found to be high, with the exception ofHarpadonnehereus(0. 250±0. 180). It was vital that some protective measures be taken immediately toH.nehereusbecause of low COI barcoding diversity. It is thus clean that DNA barcoding is an effective tool for accurate species identification, and also could play a supplementary role in the species diversity survey. All entire dataset in this study could play an important role in survey of biodiversity and genetic diversity of Cangnan, and all results could provide reference for fisheries management and formulating policy.
DNA barcoding; biodiversity; fish larvae; sequence similarity; barcoding gap
Q174;S917.4
A
1672-5174(2017)12-072-08
責(zé)任編輯 高 蓓
10.16441/j.cnki.hdxb. 20160343
李淵, 張麗艷, 張然, 等. 基于DNA條形碼技術(shù)對(duì)蒼南海域仔稚魚(yú)的物種鑒定[J]. 中國(guó)海洋大學(xué)學(xué)報(bào)(自然科學(xué)版), 2017, 47(12): 72-79.
LI Yuan, ZHANG Li-Yan, ZHANG Ran, et al. Identification of several fish larvae based on DNA barcoding in the investigated waters of Cangnan[J]. Periodical of Ocean University of China, 2017, 47(12): 72-79.
海洋赤潮災(zāi)害立體監(jiān)測(cè)技術(shù)與應(yīng)用國(guó)家海洋局重點(diǎn)實(shí)驗(yàn)室開(kāi)放基金項(xiàng)目(MATHAB201603);雙邊國(guó)際事務(wù)合作—開(kāi)展海峽兩岸合作與交流項(xiàng)目(2200207)資助
Supported by Open Research Fund Program of MATHAB, S.O.A (MATHAB201603);Bilateral Cooperation on International Affairs—Cross-Strait Cooperation and Communication Program (2200207)
2016-10-09 ;
2016-12-14
李 淵(1985-),男,博士生,主要從事海洋生態(tài)學(xué)研究。E-mail:liyuan@tio.org.cn
** 通訊作者:E-mail:linlsh@tio.org.cn

