環境中微生物原位檢測方法研究進展
宋偉鳳 李明聰 高崢
(山東農業大學生命科學學院 作物生物學國家重點實驗室,泰安271000)
環境中微生物原位檢測方法研究進展
宋偉鳳 李明聰 高崢
(山東農業大學生命科學學院 作物生物學國家重點實驗室,泰安271000)
微生物是生態系統物質循環和能量流動的主要參與者,在生態系統中起著重要的作用。但在現有技術條件下可培養微生物所占比例極小,限制了微生物資源的開發利用。目前有許多方法,可以避開微生物不可培養的問題,直接對微生物進行原位檢測。對此,將前人關于微生物生態的原位檢測研究方法進行了綜述,方便以后對這些方法的合理應用。分別從DNA水平、RNA水平和蛋白質水平,介紹了對應的原位微生物檢測方法(如BrdU標記、DNA-SIP、FISH和環境轉錄物組等),比較了它們的優缺點,并介紹了如何將這些方法與目前流行的高通量測序、單細胞測序等技術相結合來捕獲原位活性微生物組等。同時,還對這些方法的特點進行了比較,使得人們可以更清楚地了解在不同場景下對不同方法的選擇。這些經過改進的新興方法及其與其它方法的結合使用將有助于解決微生物生態學研究中出現過或即將出現的很多問題。地球上的各種生態系統復雜而龐大,包含的微生物種群也各有差異。各種原位檢測方法對微生物生理生態做出了更加真實有效的描述,必將成為研究微生物生態的有力手段。
微生物原位檢測;BrdU標記;DNA-SIP;熒光原位雜交;宏基因組;轉錄物組;生物正交反應
微生物是生物地球化學循環的驅動者,在生態系統中扮演著重要角色[1]。微生物多樣性是微生物生態學研究的重要內容,是人們認識、開發和利用微生物資源的前提和基礎,對了解微生物與環境互作具有重要意義。早期的微生物多樣性研究需要通過純培養獲得菌株后才能對其特性進行描述[2],可實際上培養得到的只是自然環境中的少部分微生物(0.001%-15%)[3]。隨著分子生物學技術的不斷發展,直接利用環境樣品的總DNA進行16S rRNA基因的PCR擴增并進行鑒定,開辟了將分子生物學技術直接應用于微生物生態學研究的新紀元[4],解決了環境中未培養微生物研究的難題。然而傳統的16S擴增子測序[5]、宏基因組測序[6]和單細胞基因組測序[6]雖然對微生物群落的分布模式及潛在功能鑒定提供了重要依據,卻不能對其在環境變化的動態響應進行完全檢測[6]。隨著研究的深入,越來越多的研究者選擇使用原位微生物活性檢測與測序組學相結合的方法,對環境中活性微生物的豐度、多樣性、群落結構、功能代謝及環境響應能力進行研究[7]。本文綜述了現有的常用于微生物檢測的一些方法,這些方法可以增加人們對微生物群落成員的不同生態和生理的了解,為人們提供了原位微生物生態群落對環境變化響應的新視角。以下是我們對這一領域中新興技術前景的探討。
1 微生物原位檢測的常用方法
1.1 基于DNA水平的微生物原位檢測方法
DNA 復制是細胞生命周期中的關鍵環節之一,利用胸苷的類似物進入新合成的 DNA 進行檢測一直是研究 DNA 代謝的有效方法[8]。5'-溴脫氧尿苷(BrdU)標記技術是最早開發的可以原位標記活性樣品并能結合組學測序的方法。其原理是利用BrdU在細胞增殖時期代替胸腺嘧啶(T)滲入正在復制的DNA(和RNA)分子中,隨后進行密度梯度離心或者免疫學方法捕獲被BrdU標記的DNA/RNA[9]。BrdU標記法已經成功應用于多種環境中微生物分子生態學的研究。目前,研究者們主要針對細菌、古菌和真菌在不同環境刺激下DNA合成活性的變化進行了研究,研究內容包括森林土壤樣品中不同碳源刺激下細菌活性的變化[10],不同植物接種后叢枝菌根真菌后的反應[11],阿拉斯加北部森林樣品中氮添加后微生物活性群落的變化[12],土壤中活性微生物對不同碳源的反應[9-13]等。雖然BrdU標記實驗可以用來檢測微生物在各種環境刺激條件下的活性反應,但目前發現的BrdU標記技術與高通量測序結合的應用是十分有限的[14-16]。Hamasaki等[16]通過BrdU標記及454高通量測序對太平洋南北橫斷面海水樣品中活性微生物的物種組成進行了研究,研究結果發現由于環境過濾,微生物群落結構發生了動態變化,活躍的細菌群體更加多樣化。然而,由于標記效率較低,需要相對大量的樣品,以獲取測序所需要的DNA量,這使得這種技術相對昂貴且需要大量勞動力。并且由于不同生物細胞的吸收速率不同,重復性不高,也影響了該技術的普遍應用。
穩定性同位素核酸探針技術(DNA-based stable isotope probing,DNA-SIP),是采用穩定性同位素示蹤復雜環境中微生物基因組DNA的分子生態學技術[17]。利用穩定性同位素示蹤復雜環境中微生物基因組DNA,實現了由單一微生物生理過程研究向微生物群落生理生態研究的轉變,能在更高更復雜的整體水平上定向發掘重要微生物資源,推動微生物生理生態的發展。DNA-SIP分析的原理是通過將同位素穩定的結合到特定的底物上來確定環境中微生物的作用。除磷以外,幾乎所有具有生物學意義的元素均有2 種以上的穩定性同位素,而且不同同位素組成的化合物通常具有相同的物理化學及生物學性質,所以微生物可以利用不同的穩定性同位素來進行生長繁殖[17]。Radajewski等[18]將這種方法應用于土壤微生物的甲醇利用的研究,隨后確定了有活性的甲基營養型,發現甲基營養型局限于α-變形菌和乳桿菌屬,這是這種方法的首次應用。最近,Dumont等[19]從湖泊沉積物的甲烷氧化菌中得到了13CH4標記的RNA宏轉錄組序列,發現標記的宏轉錄組序列主要集中于甲基球菌科和甲烷單加氧酶(pmoCAB)基因的轉錄組序列中。但是,這種方法也受到了許多的限制。例如,孵育時間較長,交互共生問題,容易發生污染,依賴于商用標記化合物,潛在的富集偏向性等。近年來,隨著SIP技術的逐漸成熟和不斷優化,其應用范圍也不斷擴展,與其他技術的結合應用也使得DNA-SIP成為重要種群DNA合成序列分析的最好的方法。Huang等[20]將SIP與拉曼光譜和熒光原位雜交技術相結合進行原位雜交,對單個細胞的特性和功能進行了分析。到目前為止,關于DNA-SIP 與宏基因組學結合的研究報道還很少,但從這些研究中可以看出它比傳統宏基因組學具有更大的優勢,通過運用DNA-SIP 可以提高新酶類發現的概率,也可以減少鳥槍測序法很難解決種群復雜的問題。
熒光原位雜交(Fluorescence in situ hybridization,FISH)是在20世紀80年代末在放射性原位雜交技術的基礎上發展起來的一種非放射性分子細胞遺傳技術,該技術是以熒光標記取代同位素標記而形成的一種新的原位雜交方法。1980 年,Roumam等[21]首次報道了用熒光素標記的cDNA 進行的原位雜交,自那以后,FISH逐漸成為在環境生物學中最常用的方法之一。作為一種非放射性的檢測系統,FISH 技術有其特有的屬性和優點:(1)熒光試劑和探針經濟、安全;(2)探針穩定,一次標記后可在兩年內使用;(3)實驗周期短、能迅速得到結果、特異性好、定位準確;(4)FISH可定位長度在1kb的DNA序列,其靈敏度與放射性探針相當;(5)多色FISH通過在同一個核中顯示不同的顏色可同時檢測多條序列;(6)既可以在玻片上顯示中期染色體數量或結構的變化,也可以在懸液中顯示間期染色體DNA的結構。盡管如此,這種方法也有缺點:因為細胞內的靶定分子數量較少,探針在細胞內的滲透性差,雜交效率低,所以可以被檢測的靈敏度不高。這極大程度的限制了這種方法的應用,所以人們想出了很多方法來克服這個問題。其中催化信使沉積法熒光原位雜交(CARD-FISH)的效果最好。CARD的原理是在過氧化氫存在的前提下,辣根過氧化氫酶(HRP)將其轉化成一個酪胺中間體的自由基,這種酪胺可以在細胞或阻斷試劑中與芳香族化合物進行非特異性反應,這種反應只發生在HPR附近,而且時間很短,最終HPR分子周圍會沉積大量的酪胺,從而獲得較強的檢測信號[22]。CARD最早是作為放大免疫分析信號的新方法被提出來的[23-24]。1995年,Kerstens等[25]將其應用于熒光原位雜交的信號增強中,取得了良好的效果。Kawakami等[26]通過雙通路CARD-FISH技術檢測并標記到了mcrA基因;Moraru等[27]使用CARD-FISH和雙鏈DNA探針檢測到了海水樣品中的泉古菌amoA基因;Kenzaka等[28]將CARD-FISH與納米金標記的鏈霉親和素結合,檢測到了質粒上的綠色熒光基因和氨芐青霉素耐藥基因。總體來說,熒光原位雜交技術能夠將微生物環境中的完整細胞景象的信息進行再現還原,精確度較高,因此在現在的微生物多樣性的研究領域中被廣泛的應用。
宏基因組也稱環境微生物基因組或元基因組,是指環境中全部微小生物(目前主要包括細菌和真菌)DNA的總和。宏基因組學誕生于20世紀90年代,是指不經過微生物培養階段,直接提取環境中總DNA,對微生物基因總和進行研究的一門學科。1998 年,Handelsman[29]首次提出了宏基因組(Metagenome)概念,認為應該針對環境樣品中細菌和真菌的基因組總和進行研究。2006年,Leininger等[30]首次將高通量測序技術應用于土壤中微生物群體的研究發現,古菌在土壤原核氨氧化生物中占優勢。2008年,Frias-Lopez等[31]又將該技術應用于對海洋中微生物群落的研究中。隨著宏基因組等技術的發展,其他技術與宏基因組技術的結合使用將有效改善這些問題。Kalyuzhnaya等[32]利用高通量快速測序研究華盛頓湖淤泥的碳循環,他們用13C復合物進行DNA-SIP 實驗,隨后利用純化的標記DNA 建立宏基因組庫,通過回收的13C-DNA 宏基因組庫序列幾乎重建了嗜甲基菌Methylotenera mobilis的完整染色體組。總之,全基因組鳥槍測序的宏基因組學方法可重建低豐度微生物的基因組和新陳代謝通路,但前提是需要結合DNA-SIP 技術。
同時,峰谷比(PTR)也可以用來檢測微生物活性。峰谷比是一種可以在不同的生長條件和復雜的細菌群落中,提供一個定量的體內和體外生長速率測量方法的技術。該技術由Korem等[33]首次發現,隨后Brown等[34]開發了復制指數(iRep)可以改進PRT技術。這個技術需要提供被檢測微生物的復制起點信息,并需要根據實際情況,進行人工校正,目前使用并不廣泛。
1.2 基于RNA水平的微生物原位活性檢測方法
RNA-SIP是與DNA-SIP基本相同的一種分子生態學技術,采用穩定性同位素追蹤環境中微生物的轉錄產物RNA。其原理和優缺點與DNA-SIP完全相同。
轉錄組測序能夠全面快速的獲取某一物種特定組織或器官在某一狀態下的幾乎所有轉錄本序列信息。轉錄組在廣義上是指某一生理條件下,細胞內所有轉錄產物的集合,在狹義上則是指所有mRNA的集合。與基因組不同的是,轉錄組的定義中包含了時間和空間的限定。同一細胞在不同的生長時期及生長環境下,其基因表達情況是不完全相同的。轉錄組學以微生物的全部mRNA為研究對象,所以會存在一些問題,如mRNA的含量較低,結構不穩定容易被降解等。隨著新一代測序技術的產生,RNA-Seq(High-throughput RNA sequencing)隨之而來,轉錄組學也得到了飛速的發展。Voorhies等[35]通過通過對休倫湖島中污水池的藍藻團進行宏基因組分析和環境轉錄組學分析,推斷出了藍藻和病毒的生態與遺傳的互作關系。De Filipps等[36]通過環境轉錄組學對微生物對奶酪成熟速率的影響進行了研究,揭示了在溫度推動下微生物的功能變化。為了了解導致大分子顆粒形成厭氧氨氧化生長增值機制,Bagchi等[37]結合環境轉錄組學、定量PCR和16S rRNA基因測序,對厭氧氨氧化顆粒狀生物反應器中的群落組成、代謝基因組成和表達變化進行了分析,并提出了大顆粒形成的概念模型,這有助于今后采用厭氧氨氧化工藝進行廢水處理。相信在未來的發展中,轉錄組學測序技術將會更好的與其他技術相結合,在生態學等領域獲得更加廣泛的應用。
1.3 基于氨基酸生物合成的微生物原位活性的檢測
生物正交反應(Bioorthogonal reaction)是指一類可以在活體細胞中進行的化學反應。這類反應可以在生物體內的生理條件下發生,不會與體內同時發生的其他生化反應互相干擾,也不會對生物體和目標生物分子產生損傷。要完成生物正交反應,首先需要有可以參與正交反應的一對官能團,將其中一個參與正交反應的官能團引入目標蛋白;另一個參與反應的官能團與標記物進行連接,然后將兩者進行正交反應,最后利用不同標記物的特性對目標蛋白的定位、結構和功能進行檢測[38]。前期研究表明,生物正交非標準氨基酸標記(Bioorthogonal noncanonicalamino acid tagging,BONCAT) 技 術能對環境中功能微生物蛋白進行能有效地標記。Hatzenpichler等[39]用深海沉積物樣品富集好氧嗜甲烷菌群,通過BONCAT標記確定其富集物主導類群為γ變形菌門的甲基球菌屬,這些甲基球菌細胞的轉化活性被認為依賴于富集培養時甲烷的添加。利用這種方法可以有針對性地研究在實驗條件發生改變時特殊的標記蛋白所做出的反應。但這種方法在傳統意義上來說,也只能用來研究某種生物的蛋白質組,一些新興的技術將這種方法與其他方法耦合,希望能夠使得這種方法得到更廣泛的應用。Hatzenpichler等[7]將BONCAT與FACS結合,應用于甲烷厭氧氨氧化的研究中,對甲烷氧化菌的翻譯活性進行了分類,并通過全基因組測序和16S RNA測序發現了一個新的古菌-細菌互作組合。這項技術與高通量技術的結合使用也展現出了非常好的前景,將有助于在微米分辨率下監測細胞翻譯活動對環境信號的響應,并擴大高通量測序的尺度。當然,BONCAT也存在一定的局限性,如環境氨基酸吸收機制的多樣性,添加的氨基酸的潛在影響等。希望在未來的改進或與其他技術結合后,能夠有效地消除或減少這些限制性條件的影響。
2 選擇合適的方法來檢測原位微生物
微生物活性檢測的結果,是由選擇的方法和獲取的參數產生的。在研究微生物在某一過程中所起到的作用時,我們無法對每一個細節都進行完整的評價,但也不可以僅僅在系統水平上進行概括性描述。所以,在我們設計相關的實驗時,要將實際時間和空間條件的限制與各種待選方法的特性綜合考慮,根據各種限制因素選擇合適的方法來檢測原位微生物(各方法比較詳見表1)。在對微生物研究的過程中通常會面臨一個非常重要的挑戰,即確定微生物能產生作用的范圍[44],因為微生物產生影響的范圍會決定檢測方法的選擇。例如,基于單細胞水平的檢測技術不能為系統水平的鑒定與分類提供足夠的數據支撐。因為選擇不同檢測方法對各種微生物細胞的分類可能會產生影響,所以在對實驗設計進行評價之前,應該對每種檢測方法進行評估,以確定特定種類的微生物群落所需要的研究方法。
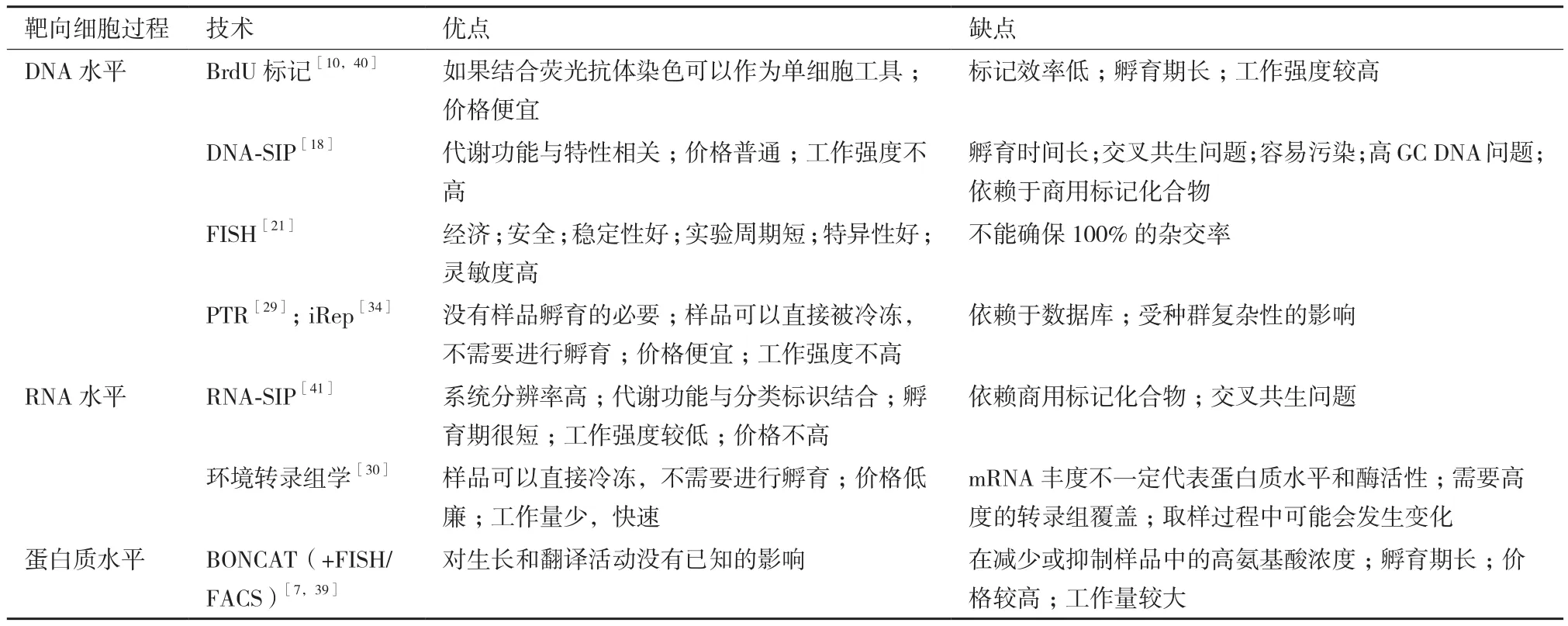
表1 微生物原位檢測方法的特征對比
3 展望
微生物生態學自產生以后,經過許多前輩的探索與發展,已經克服了傳統微生物培養的限制,形成了一些比較系統的研究方法,使得我們對各種生態系統的了解更加的深入。隨著各種檢測方法的發展完善,檢測結果會更加精確,這會讓認識更加明確。同時檢測費用的逐漸降低,也會讓各種檢測方法的使用更加廣泛。地球上的各種生態系統復雜而龐大,包含的微生物種群也各有差異。各種原位檢測方法對微生物生理生態做出了更加真實有效的描述,必將成為研究微生物生態的有力手段。
[1]Canfield DE, Glazer AN, Falkowski PG. The evolution and future of Earth’s nitrogen cycle[J]. Science, 2010, 330(6001):192-196.
[2] Anderson IC, Cairney JWG. Diversity and ecology of soil fungal communities:increased understanding through the application of molecular techniques[J]. Environ Microbiol, 2004, 6(8):769-779.
[3] Schleifer KH. Microbial diversity:facts, problems and prospects[J]. Systematic and Applied Microbiology, 2004, 27(1):3-9.
[4]Pace NR, Stahl DA, Lane DJ, et al. Analyzing natural microbial populations by rRNA sequences[J]. ASM American Society for Microbiology News, 1985, 51(1):4-12.
[5]Tringe SG, Hugenholtz PA renaissance for the pioneering 16S rRNA gene[J]. Current Opinion in Microbiology, 2008, 11(5):442-446.
[6]Handelsman J. Metagenomics:application of genomics to uncultured microorganisms[J]. Microbiology and Molecular Biol Rev, 2005, 69(1):195-195.
[7]Hatzenpichler R, Connon SA, Goudeau D, et al. Visualizing in situ translational activity for identifying and sorting slow-growing archaeal- bacterial consortia[J]. Proc Natl Academy of Sciences,2016, 113(28):E4069-E4078.
[8]Biggs MJP, Richards RG, Dalby MJ. Using immuno-scanning electron microscopy for the observation of focal adhesion-substratum interactions at the nano-and microscale in S-phase cells[J]. 3D Cell Culture:Methods and Protocols, 2011:53-60.
[9]Urbach E, Vergin KL, Giovannoni SJ. Immunochemical detection and isolation of DNA from metabolically active bacteria[J]. Appl Environ Microbiol, 1999, 65(3):1207-1213.
[10]Yin B, Crowley D, Sparovek G, et al. Bacterial functional redundancy along a soil reclamation gradient[J]. Appl Environ Microbiol, 2000, 66(10):4361-4365.
[11]Artursson V, Finlay RD, Jansson JK. Combined bromodeoxyuridine immunocapture and terminal-restriction fragment length polymorphism analysis highlights differences in the active soil bacterial metagenome due to Glomus mosseae inoculation or plant species[J]. Environ Microbiol, 2005, 7(12):1952-1966.
[12]Allison SD, Czimczik CI, Treseder KK. Microbial activity and soil respiration under nitrogen addition in Alaskan boreal forest[J].Global Change Biology, 2008, 14(5):1156-1168.
[13]Goldfarb KC, Karaoz U, Hanson CA, et al. Differential growth responses of soil bacterial taxa to carbon substrates of varying chemical recalcitrance[J]. Frontiers in Microbiology, 2011, 2.
[14]Mou X, Sun S, Edwards RA, et al. Bacterial carbon processing by generalist species in the coastal ocean[J]. Nature, 2008, 451(7179):708-711.
[15]David MM, Cecillon S, Warne BM, et al. Microbial ecology of chlorinated solvent biodegradation[J]. Environ Microbiol, 2015,17(12):4835-4850.
[16]Hamasaki K, Taniguchi A, Tada Y, et al. Active populations of rare microbes in oceanic environments as revealed by bromodeoxyuridine incorporation and 454 tag sequencing[J].Gene, 2016, 576(2):650-656.
[17] 賈仲君. 穩定性同位素核酸探針技術 DNA-SIP 原理與應用[J].微生物學報, 2011, 51(12):1585-1594.
[18] Radajewski S, Ineson P, Parekh NR, et al. Stable-isotope probing as a tool in microbial ecology[J]. Nature, 2000, 403(6770):646-649.
[19] Dumont MG, Pommerenke B, Casper P. Using stable isotope probing to obtain a targeted metatranscriptome of aerobic methanotrophs in lake sediment[J]. Environ Microbiol Reports, 2013, 5(5):757-764.
[20] Huang WE, Stoecker K, Griffiths R, et al. Raman-FISH :combining stable-isotope Raman spectroscopy and fluorescence in situ hybridization for the single cell analysis of identity and function[J]. Environ Microbiol, 2007, 9(8):1878-1889.
[21] Bauman JG J, Wiegant J, Borst P, et al. A new method for fluorescence microscopical localization of specific DNA sequences by in situ hybridization of fluorochrome-labelled RNA[J].Experimental Cell Research, 1980, 128(2):485-490.
[22]Kubota K. CARD-FISH for environmental microorganisms:technical advancement and future applications[J]. Microbes and Environments, 2013, 28(1):3-12.
[23]Bobrow MN, Harris TD, Shaughnessy KJ, et al. Catalyzed reporter deposition, a novel method of signal amplification application to immunoassays[J]. Journal of Immunological Methods, 1989, 125(1):279-285.
[24]Bobrow MN, Shaughnessy KJ, Litt GJ. Catalyzed reporter deposition, a novel method of signal amplification:II. Application to membrane immunoassays[J]. Journal of Immunological Methods, 1991, 137(1):103-112.
[25] Kerstens HM, Poddighe PJ, Hanselaar AG. A novel in situ hybridization signal amplification method based on the deposition of biotinylated tyramine[J]. Journal of Histochemistry &Cytochemistry, 1995, 43(4):347-352.
[26]Kawakami S, Kubota K, Imachi H, et al. Detection of single copy genes by two-pass tyramide signal amplification fluorescence in situ hybridization(Two-Pass TSA-FISH)with single oligonucleotide probes[J]. Microbes and Environments, 2010, 25(1):15-21.
[27]Kawakami S, Kubota K, Imachi H, et al. Detection of single copy genes by two-pass tyramide signal amplification fluorescence in situ hybridization(Two-Pass TSA-FISH)with single oligonucleotide probes[J]. Microbes and Environments, 2010, 25(1):15-21.
[28]Kenzaka T, Ishidoshiro A, Tani K, et al. Scanning electron microscope imaging of bacteria based on DNA sequence[J].Letters in Applied Microbiology, 2009, 49(6):796-799.
[29]Handelsman J, Rondon MR, Brady SF, et al. Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J]. Chemistry & Biology, 1998, 5(10):R245-R249.
[30]Leininger S, Urich T, Schloter M, et al. Archaea predominate among ammonia-oxidizing prokaryotes in soils[J]. Nature, 2006, 442(7104):806-809.
[31]Frias-Lopez J, Shi Y, Tyson GW, et al. Microbial community gene expression in ocean surface waters[J]. Proc Natl Academy of Sciences, 2008, 105(10):3805-3810.
[32]Kalyuzhnaya MG, Lapidus A, Ivanova N, et al. High-resolution metagenomics targets specific functional types in complex microbial communities[J]. Nature Biotechnology, 2008, 26(9):1029-1034.
[33]Korem T, Zeevi D, Suez J, et al. Growth dynamics of gut microbiota in health and disease inferred from single metagenomic samples[J]. Science, 2015, 349(6252):1101-1106.
[34]Brown CT, Olm MR, Thomas BC, et al. In situ replication rates for uncultivated bacteria in microbial communities[J]. BioRxiv,2016:057992.
[35]Voorhies AA, Eisenlord SD, Marcus DN, et al. Ecological and genetic interactions between cyanobacteria and viruses in a lowoxygen mat community inferred through metagenomics and metatranscriptomics[J]. Environ Microbiol, 2016, 18(2):358-371.
[36]De Filippis F, Genovese A, Ferranti P, et al. Metatranscriptomics reveals temperature-driven functional changes in microbiome impacting cheese maturation rate[J]. Scientific Reports, 2016, 6:21871.
[37]Bagchi S, Lamendella R, Strutt S, et al. Metatranscriptomics reveals the molecular mechanism of large granule formation in granular anammox reactor[J]. Scientific Reports, 2016, 6 :28327.
[38]楊麥云, 陳鵬. 生物正交標記反應研究進展[J]. 化學學報,2015, 73(8):783-792.
[39]Hatzenpichler R, Scheller S, Tavormina PL, et al. In situ visualization of newly synthesized proteins in environmental microbes using amino acid tagging and click chemistry[J].Environ Microbiol, 2014, 16(8):2568-2590.
[40]Borneman J. Culture-independent identification of microorganisms that respond to specified stimuli[J]. Appl Environ Microbiol,1999, 65(8):3398-3400.
[41]Radajewski S, McDonald IR, Murrell JC. Stable-isotope probing of nucleic acids:a window to the function of uncultured microorganisms[J]. Current Opinion in Biotechnology, 2003, 14(3):296-302.
[42]Blazewicz SJ, Barnard RL, Daly RA, et al. Evaluating rRNA as an indicator of microbial activity in environmental communities:limitations and uses[J]. The ISME Journal, 2013, 7(11):2061-2068.
[43]Hoehler TM, Jorgensen BB. Microbial life under extreme energy limitation[J]. Nature Reviews Microbiology, 2013, 11(2):83-94.
[44]Paerl HW, Steppe TF. Scaling up:the next challenge in Environ Microbiol[J]. Environ Microbiol, 2003, 5(11):1025-1038.
Research Progress on in situ Detection Methods of Microorganisms
SONG Wei-feng LI Ming-cong GAO Zheng
(School of Life Science,Shandong Agricultural University,State Key Laboratory of Crop Biology,Tai’an 271000)
As major participants in ecosystem material cycle and energy flow,microorganisms play an important role in the ecosystem.However,the proportion of the cultivable microorganism under the existing technology is very small,which limits the exploit of microbial resources. At present,there are a number of approaches that can avoid the problem of uncultured microorganisms,which are designed to study in situ microbial activity. Regarding this,we summarized some research methods of studying in situ microbial ecology,allowing it convenient to reasonably use these methods in the future. This article introduces the corresponding microbial detection methods of BrdU-labeling,DNA-SIP,fluorescence in situ hybridization(FISH),and environmental transcriptome from DNA level,RNA level and protein level,respectively,and compares their advantages and disadvantages. It also introduces how to apply these methods combined with popular high-throughput sequencing and single cell sequencing technology to capture the in situ activity of microbial groups. At the same time,comparing the characteristics of these methods,so that we can more clearly understand the choice of different methods under different scenarios. These modified methods combined with other methods will be conducive to solve many have-been or will-happen problems in the study of microbial ecology. The ecosystems on the earth are complex and huge,in which the microbial populations vary. A variety of in situ detection methods have made a more realistic and effective description for the physiology and ecology of microorganisms,which will become a powerful tool for the study of microorganisms.
microbial detection in sit;BrdU-labeling;DNA-SIP;fluorescence in situ hybridization;metagenome;transcriptome;BONCAT
10.13560/j.cnki.biotech.bull.1985.2017-0550
2017-07-02
國家自然科學基金項目(41306150),山東省優秀中青年科學家科研獎勵基金(BS2012HZ011),河口海岸學國家重點實驗室開放課題(SKLEC-KF201603)
宋偉鳳,女,研究方向:微生物生態;E-mail:songweifeng9719@qq.com,李明聰為共同第一作者
高崢,男,博士,副教授,研究方向:微生物生態與環境微生物學;E-mail:gaozheng@sdau.edu.cn
(責任編輯 狄艷紅)

