黑曲霉天冬氨酰氨肽酶的分子克隆與酶學性質解析
喬雅麗,董自星,2,*,宋鵬,劉曉光,2,路福平
(1.天津科技大學生物工程學院,天津300457;2.天津科技大學化工與材料學院,天津300457)
黑曲霉天冬氨酰氨肽酶的分子克隆與酶學性質解析
喬雅麗1,董自星1,2,*,宋鵬1,劉曉光1,2,路福平1
(1.天津科技大學生物工程學院,天津300457;2.天津科技大學化工與材料學院,天津300457)
以黑曲霉CICIM F0510的互補DNA(complementary DNA,cDNA)為模板,通過PCR擴增出一個新的天冬氨酰氨肽酶基因(vacuolar aspartyl aminopeptidase,vaap),并成功將其在畢赤酵母GS115中進行表達。搖瓶水平上,重組菌GS115(pPIC9K-vaap)的氨肽酶酶活達到39.7 U/mL。該酶的最適反應溫度和pH值分別為55℃和9.5;在90℃或pH 8.0~10.0孵育1 h后,該酶仍能保持20%或80%以上的酶活;Sn2+和Co2+對其酶活有明顯促進作用,Fe3+、Ca2+、EDTA和SDS對其有抑制作用;該酶對L-亮氨酰對硝基苯胺(Leu-pNA)的Km和Vmax分別是12.96 mmol/L和2.14 μg/(mL·min)。在所測的8種底物中,Leu-pNA是Vaap最適底物,且它對丙氨酸對硝基苯胺(Ala-pNA·HCl)和賴氨酸對硝基苯胺(Lys-pNA·2HCl)也有一定的水解能力。
天冬氨酰氨肽酶;黑曲霉;分子克隆;酶學性質
氨肽酶(Aminopetidases,EC 3.4.11)是一類外切蛋白酶,能水解蛋白或多肽鏈的N端特定肽鍵,釋放出相應的游離氨基酸[1]。不同氨肽酶對N端氨基酸殘基的水解能力存在差異性,同一種氨肽酶對不同的N端氨基酸殘基的水解能力也不同。根據底物特異性的不同,可將氨肽酶細分為天冬氨酰氨肽酶、亮氨酸氨肽酶、纈氨酸氨肽酶、苯丙氨酸氨肽酶和脯氨酸氨肽酶等[2]。氨肽酶由于其獨特的水解和脫苦性能,在食品工業中特別是營養保健品和調味品的制造方面潛力巨大。與其它蛋白酶復合使用可以深度水解蛋白質,促進良好風味的形成[3-4];運用氨肽酶和蛋白質酶解程度控制工藝,可制備多種功能性多肽[5];部分特殊種類的氨肽酶還可用作醫學上的解毒劑或環境消毒劑[6]。
氨肽酶廣泛存在于哺乳動物、植物和微生物中。由于動植物體內氨肽酶含量低、成分復雜,氨肽酶的提取成本較高。因此,利用微生物發酵法生產氨肽酶成為當今的研究熱點之一。目前,我國還沒有實現氨肽酶的自主生產,工業上使用的氨肽酶主要由丹麥諾維信、日本田野等公司生產,價格昂貴,影響了我國相關產業的發展。據報道,目前已有多種不同微生物來源的氨肽酶被克隆表達,主要是真核和原核微生物[2]。其中,已被克隆表達的黑曲霉氨肽酶有以下4種:賴氨酸氨肽酶(ApsA)[7]、二肽基氨肽酶(DapB)[8]、脯氨酸氨肽酶(PapA)[9]和苯丙氨酸氨肽酶(ApsC)[10]。
盡管黑曲霉CBS 513.88的基因組序列已經于2007年解析完成并公布[11],但黑曲霉的蛋白數據庫中還有許多蛋白的功能未確定。目前已報道的天冬氨酰氨肽酶主要來源于哺乳動物[12]、酵母[13]和米曲霉[14]等,而黑曲霉、植物和細菌來源的天冬氨酰氨肽酶未見報道。本文通過對黑曲霉基因組信息進行分析,發現了一個新的編碼天冬氨酰氨肽酶的基因序列,通過分子克隆技術成功將該基因在畢赤酵母中進行了克隆與表達,并系統解析了其酶學性質。為進一步闡明其耐熱和耐堿機制以及挖掘其應用價值等奠定了良好的基礎。
1 材料與方法
1.1 菌種與質粒
黑曲霉(Aspergillus niger)CICIM F0510:中國高校工業微生物資源與信息中心;大腸桿菌(Escherichia coli)JM109、畢赤酵母(Pichia pastoris)GS115 和質粒pPIC9k:保藏于天津科技大學生物催化與生物轉化研究室;黑曲霉采用CD培養基進行培養,大腸桿菌的培養用LB培養基;畢赤酵母重組菌的培養基和培養方法按照Invitrogen的畢赤酵母操作手冊進行。
1.2 主要試劑
限制性內切酶 Xba I、Sac I、Stu I 以及 PyrobestTMDNA聚合酶和T4DNA連接酶:寶生物工程(大連)有限公司;蛋白質分子量標準參照物:美國Thermo公司;質粒快速提取試劑盒、DNA純化回收試劑盒:北京莊盟國際生物基因科技有限公司;cDNA合成試劑盒:Roche公司;G418、真菌RNA快速提取試劑盒、反轉錄試劑盒:Invitrogen公司;化學合成底物Leu-pNA、GlupNA、Arg-pNA·2HCl、Lys-pNA·2 HCl、Met-pNA·HCl、Ile-pNA·2HCl、Pro-pNA·HCl和 Ala-pNA·HCl:上海阿拉丁生化科技股份有限公司。
1.3 基因克隆與重組菌的構建
質粒、PCR產物的純化、酶切、連接、電轉化以及轉化子的篩選等均采用實驗室常規方法進行[15]。基因克隆過程中所用引物(vaap1:5’-GTAGCCAAGAAGAACATCATGGGCC-3’;vaap2:5’-TGCTCTAGACTAAAAGTCAGCAAACTCCTTGTCAATCTCC-3’;下劃線部分為人工引入的限制性酶切位點)由生工生物工程(上海)股份有限公司合成。黑曲霉總RNA的提取與cDNA制備按照試劑盒說明書進行。
1.4 重組酶的誘導表達與制備
將畢赤酵母重組菌GS115(pPIC-vaap)在YPD平板上進行純化,挑取單菌落接種于25 mL YPD液體培養基中,于30℃、220 r/min振蕩培養至對數期(OD600=2.6,約18 h~20 h)。按1%接種量轉接于25 mL BMGY培養基,再于30℃、220 r/min振蕩培養至對數期(OD600=2.6,約 16h~18h)。室溫下5 000r/min離心5 min回收酵母細胞,棄上清,將細胞重懸于適當體積的BMMY培養基中,至OD600值為1.0,于30℃繼續培養并開始誘導。每隔24小時補加100%甲醇至終濃度為0.5%以維持誘導。每隔24小時取樣一次,并測定酶活,直至120 h。發酵結束后,離心(4℃,8000r/min)收集上清液,即為粗酶液,于-20℃保存。將粗酶液用30%~70%的硫酸銨分級沉淀后,再用截留分子量為50 kDa的透析袋透析,從而對重組氨肽酶Vaap進行初步純化。
1.5 重組氨肽酶活力測定
重組氨肽酶的酶活測定按照文獻方法進行[17]。其一般步驟是:將酶液用50 mmol/L的Tris-HCl緩沖液(pH 8.0)進行適當稀釋,取稀釋液0.4 mL,加入6 mL 50 mmol/L的Tris-HCl緩沖液(pH 8.0)。40℃下預熱5 min后,加入0.4 mL 26 mmol/L L-亮氨酰對硝基苯胺(LNA)乙醇溶液。空白對照中加入0.4 mL的無水乙醇,水浴反應10 min。反應結束后立即取出,冰浴5 min終止反應。測定反應液在405 nm波長下的吸光值。酶活定義為:在40℃下,單位體積的酶液在單位時間內水解底物生成1 μg對硝基苯胺所消耗的酶量為一個酶活力單位(U)。
1.6 重組酶酶學性質與特征分析
1.6.1 最適溫度的測定
在不同溫度(20、30、40、45、50、55、60、65、70 ℃)pH 8.0的條件下測定酶活,考察溫度對重組酶酶活的影響。
1.6.2 最適pH值的測定
在不同 pH 值(5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0、9.5、10.0、10.5)和50℃測定酶活,考察pH值對酶活的影響。所用緩沖液為:0.1 mol/L磷酸氫二鈉-檸檬酸緩沖液(pH 5.5、6.0、6.5、7.0、7.5、8.0)和 0.05 mol/L 甘氨酸-氫氧化鈉緩沖液(pH 9.0、9.5、10.0、10.5)。
1.6.3 溫度穩定性的測定
分別將經過適當稀釋的重組酶酶液在40℃~90℃下保溫0.5、1、2、3 h,按酶活測定方法在最適pH值條件下測定殘留酶活。以未進行熱處理的酶液酶活力為100%,計算相對酶活,考察重組酶在不同溫度條件下的穩定性。
1.6.4 pH值穩定性的測定
分別將酶液在 pH 7.0、7.5、8.0、8.5、9.0、9.5、10.0的緩沖液中保溫1 h,在最適溫度下測定剩余酶活,考察重組酶在不同pH值條件下的穩定性。
1.6.5 金屬離子和化學試劑對酶活的影響
分別在重組酶與底物進行反應的體系中加入終濃度為0.1 mmol/L和1 mmol/L的金屬離子(Ca2+、Zn2+、Mn2+、Co2+、Sn2+和 Fe3+) 或終濃度為 1 mmol/L 的化學試劑(EDTA和SDS)。以未加金屬離子的反應體系的酶活為100%,計算相對酶活力。
1.6.6 動力學參數的測定
分別以不同濃度(2、4、6、8、10、12 mmol/L)的 L-亮氨酰對硝基苯胺為底物,在重組酶的最適作用溫度和pH值條件下進行反應,然后按照1.5的方法進行酶活測定。并采用雙倒數法(Lineweaver-BurK法)作圖,計算出米氏常數Km及最大反應速度Vmax。
1.6.7 底物特異性分析
分別以 1 mmol/L 的 Leu-pNA、Glu-pNA、ArgpNA·2HCl、Lys-pNA·2HCl、Met-pNA·HCl、Ile-pNA·2HCl、Pro-pNA·HCl和 Ala-pNA·HCl) 為底物進行反應,按1.5所述方法測定酶活。以Leu-pNA為底物時測定的酶活為100%,計算其它底物的相對酶活。
1.7 序列測定與分析
基因的核苷酸序列測定采用Sanger法[16]進行,所測得序列,經DNASTAR拼接,利用DNAMAN初步分析。利用軟件Clustal X2和Bioedit 7.0.9將vaap的氨基酸序列與其它來源天冬氨酰氨肽酶的氨基酸序列進行比對,并通過軟件MEGA 4.0以鄰近法(Neighbourjoining)構建進化樹,分析它們親緣關系的遠近[17]。
2 結果與討論
2.1 黑曲霉天冬氨酰氨肽酶基因vaap的克隆與序列分析
采用BLAST等分析方法對A.niger CBS 513.88的基因組序列(EMBL AM270980-AM270998)進行分析,發現了一個新的氨肽酶基因(vacuolar aspartyl aminopeptidase,vaap)。以此為基礎,設計出相應的核苷酸引物。然后以A.niger CICIM F0510的cDNA為模板對其進行PCR擴增。PCR產物經純化后,用Xba I進行完全酶切,并與經過SnaB I和Avr II酶切的pPIC9K進行連接。再通過Pst I酶切,驗證了重組質粒的正確性,獲得了重組表達質粒pPIC-vaap。進一步通過核苷酸序列測定,確認了所克隆的vaap基因具有完整的開放閱讀框(ORF),且其核苷酸序列和氨基酸序列與A.niger CBS 513.88基因組公布的序列完全一致。該基因的大小為1551 bp,編碼516個氨基酸。
將不同來源的天冬氨酰氨肽酶的氨基酸序列進行比對,結果如圖1所示。
AnVaap 與 AkDap、AfDap、AlDap、OcDap、TmVaap和ScLap的氨基酸序列相似度分別為99.42%、84.77%、84.80%、70.59%、69.17%和40.67%。在它們的氨基酸序列中,保守的組氨酸、天冬氨酸和谷氨酸殘基分別有3個、5個和2個(圖1中方框標出的氨基酸殘基)。組氨酸、半胱氨酸、谷氨酸和天冬氨酸通常是鋅離子的配體[18],所以上述這些保守的氨基酸殘基可能與Vaap的活性中心和鋅離子結合位點有關[13,19]。通過與Homo sapiens天冬氨酰氨肽酶的氨基酸序列進行比對,可知底物結合口袋的關鍵氨基酸殘基為His390、Tyr400、Ser415和Met422[20]。箭頭標出的氨基酸殘基只在天冬氨酰氨肽酶中是保守的(是Lys),而在M18家族的其它酶中不是保守的,而且這個位點的氨基酸殘基可能對酶的底物特異性起決定作用[13]。進化樹構建的結果表明,天冬氨酰氨肽酶Vaap與酵母氨肽酶I(ScLap)的親緣關系比較近,同屬于金屬蛋白酶的M18家族,見圖 2[13]。
2.2 黑曲霉氨肽酶Vaap在畢赤酵母中的高效表達
將重組質粒pPIC-vaap經限制性酶Sal I線性化后,電轉化入畢赤酵母GS115中,并通過G418平板篩選高拷貝轉化子,獲得重組菌GS115(pPIC-vaap)。在250 mL搖瓶培養120 h后,重組菌的胞外氨肽酶酶活達到最高,為39.7 U/mL。通過鹽析和透析,將發酵制備的粗酶液進行了初步純化,SDS-PAGE分析的結果表明(圖3),Vaap的純度已經達到酶學特征分析的要求;其分子量約為55.0 kDa,與理論分子量大小相符。
2.3 重組氨肽酶的酶學特征
2.3.1 最適反應溫度和熱穩定性的測定
溫度對Vaap的酶活和穩定性的影響見圖4。
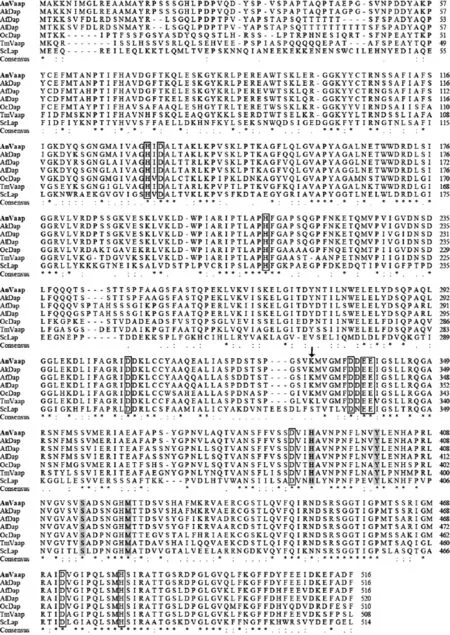
圖1 不同來源天冬氨酰氨肽酶的氨基酸序列比對Fig.1 Multiple sequence alignment of aspartyl aminopeptidases from different microorganisms
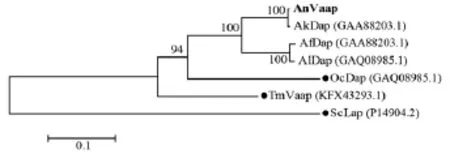
圖2 進化樹描述不同來源的天冬氨酰氨肽酶的遺傳距離Fig.2 Phylogenetic tree describing the genetic distances among various aspartyl aminopeptidases from different microorganisms
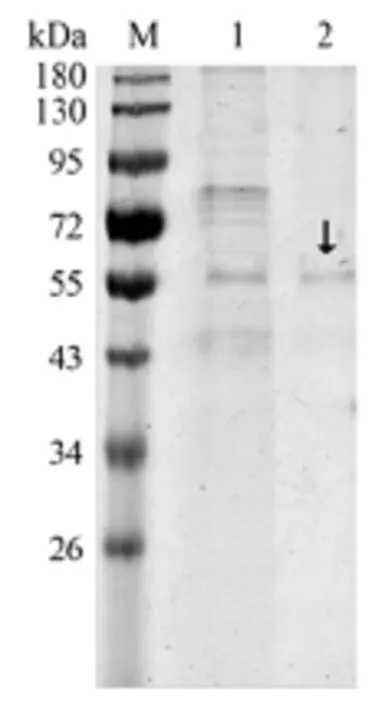
圖3 蛋白電泳分析重組酶Vaap的純化效果Fig.3 SDS-PAGE analysis of the purification of recombinant enzyme Vaap
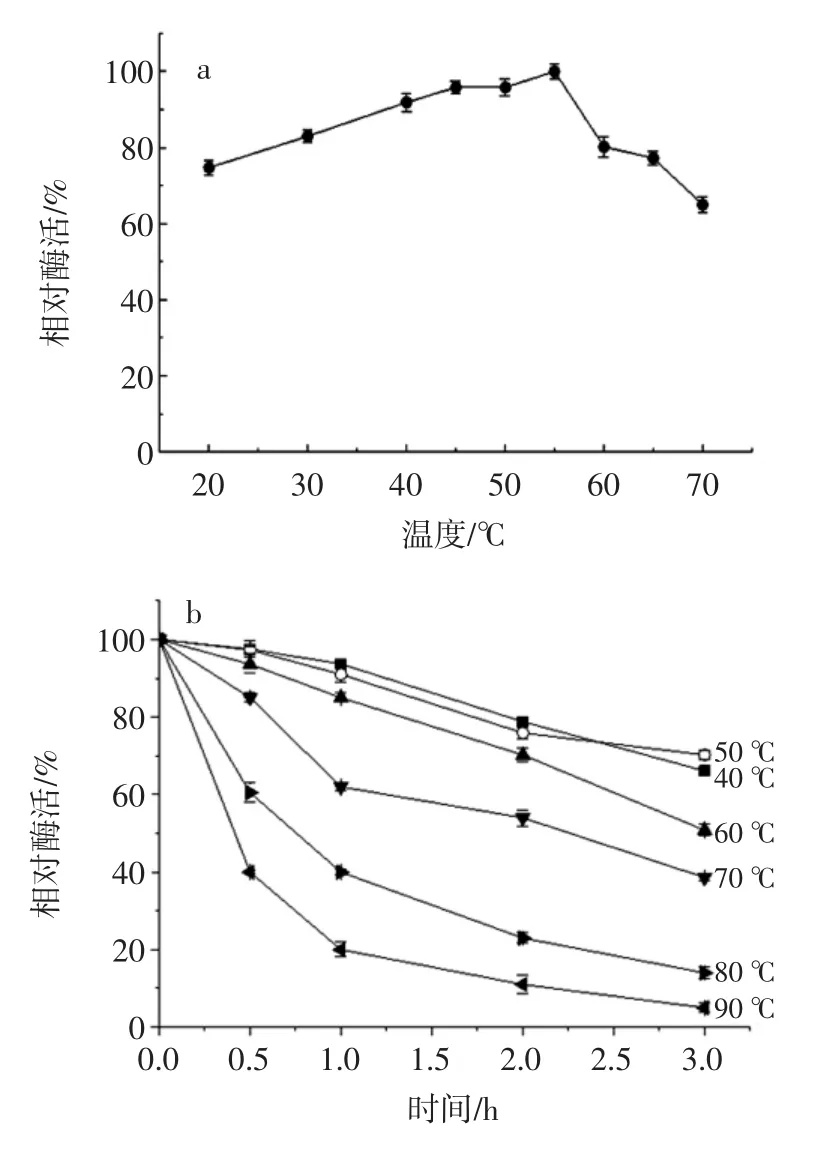
圖4 溫度對Vaap的酶活和穩定性的影響Fig.4 Effect of temperature on the activity and stability of Vaap
在不同溫度(20℃~70℃)下測定氨肽酶的酶活,結果如圖4a所示。Vaap的最適作用溫度是55℃,在30℃~55℃其相對活力達到80%以上,從20℃~70℃相對酶活力均達到60%以上,顯示該酶具有較寬泛的作用溫度范圍。
熱穩定性的研究結果如圖4b所示,Vaap在40℃~80℃保溫30min后,剩余酶活力在60%以上;在40℃~60℃保溫3 h剩余酶活力仍在50%以上;而在90℃孵育30 min后,其酶活仍能保留40%左右,表明其熱穩定性較好。該酶的最適反應溫度和熱穩定性高于A.oryzae天冬氨酰氨肽酶的最適反應溫度(50℃)和熱穩定性(80℃孵育1 h,酶幾乎完全失活)[14]。Vaap是目前已報道的少數耐熱氨肽酶之一,良好的耐熱性使得該酶可以提高化學反應速率、降低成本、簡化工藝、提高產品質量、活性穩定和耐貯藏等優點[21]。因此,Vaap具有更廣闊的應用前景。
2.3.2 最適作用pH值和pH值穩定性的測定
pH值對Vaap的酶活和穩定性的影響見圖5。
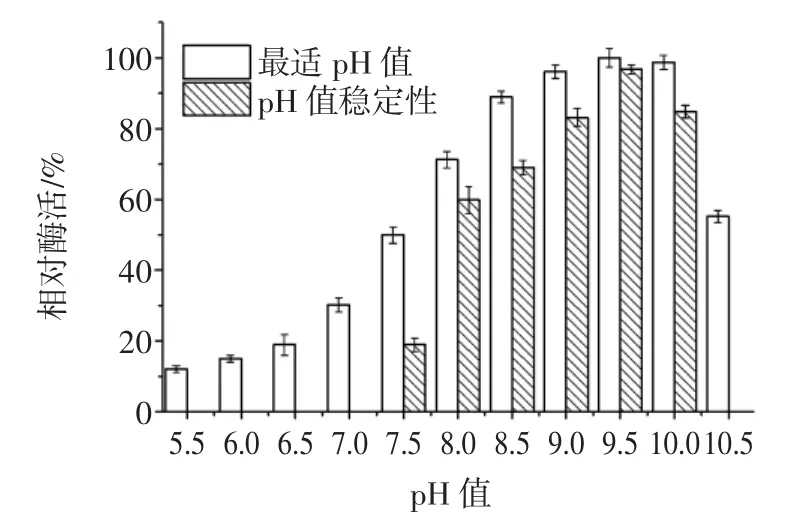
圖5 pH值對Vaap的酶活和穩定性的影響Fig.5 Effect of pH on the activity and stability of Vaap
在不同pH值(pH 5.5~10.5)的反應體系中測定酶活,結果匯總于圖5。Vaap在pH 9.5下表現出最高活力,而在pH 8.5~10.0相對酶活達到80%以上,在酸性條件下水解活性很低,表明該酶是一種堿性氨肽酶。如圖5所示,Vaap在pH 8.0~10.0條件下相對穩定,孵育1 h后殘留酶活仍保持在60%以上;在pH 9.5時最穩定,保留有96.77%的活性,說明該酶的pH值穩定范圍在8.0~10.0,與其發揮活性的pH值范圍相同。與該酶相比,米曲霉、酵母和哺乳動物(如兔子腦細胞)來源的天冬氨酰氨肽酶的最適反應pH值和pH值穩定范圍均偏中性[12-14]。
2.3.3 金屬離子和化學試劑對酶活的影響
不同金屬離子或化合物對酶活的影響如表1所示。
0.1 mmol/L 和 1 mmol/L 的 Zn2+、Co2+以及 1 mmol/L Sn2+對Vaap的酶活有促進作用,而這兩種濃度的Fe3+和Ca2+對酶活有不同程度的抑制作用。SDS對該酶有強烈的抑制作用。與酵母和米曲霉的天冬氨酰氨肽酶[13-14]相同,該酶的活性受到金屬螯合劑EDTA的明顯抑制,而哺乳動物來源的天冬氨酰氨肽酶的酶活不受EDTA的影響[12],表明該酶是一種金屬離子螯合酶。
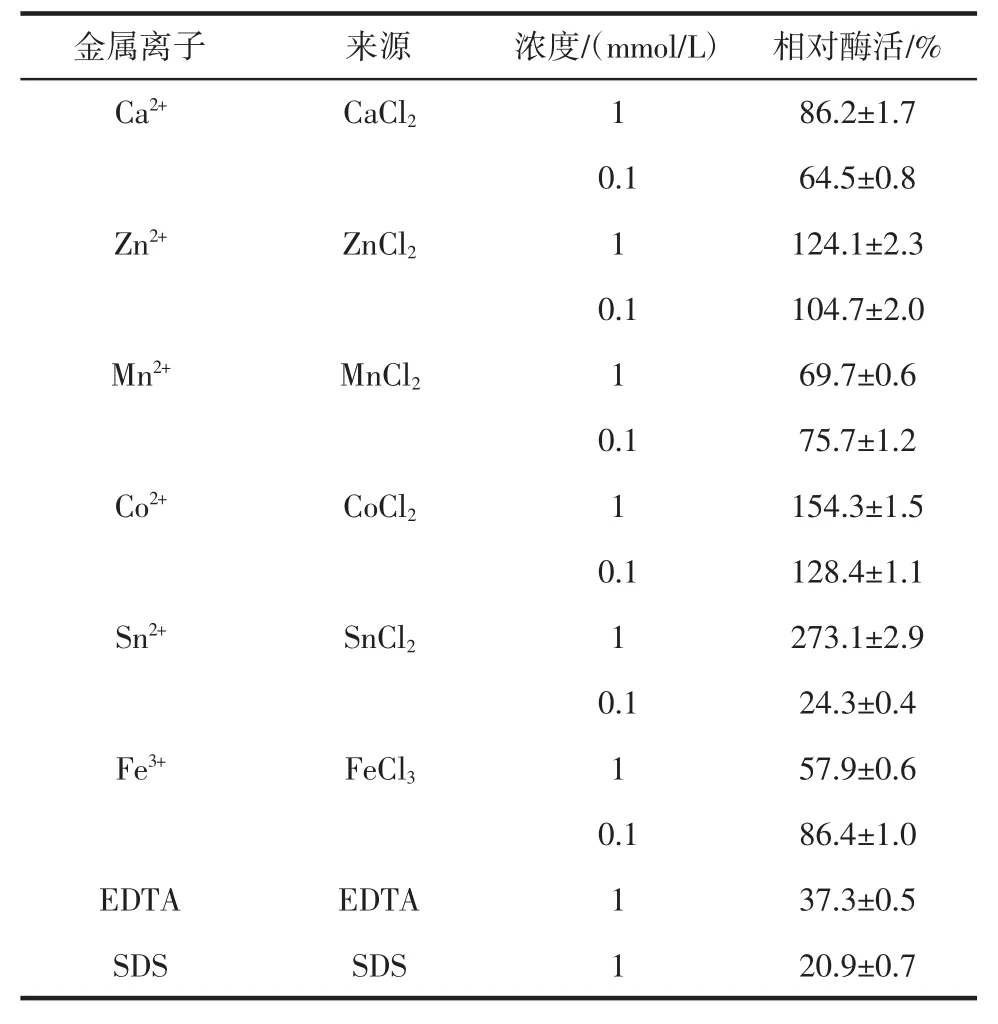
表1 金屬離子或化學試劑對Vaap酶活的影響Table 1 Effect of metal ions or chemicals on the enzymatic activity of Vaap
2.3.4 動力學參數的測定
以不同濃度(2、4、6、8、10、12 mmol/L)的 L-亮氨酰對硝基苯胺為底物,在最適溫度和pH值條件下進行反應,然后按照1.5所述方法測定酶活,以1/V對1/S作雙倒數圖。根據雙倒數圖,計算獲得該酶的米氏常數Km值為12.96 mmol/L,最大反應速度Vmax為2.14 μg/(mL·min)。其中,Km值遠高于兔子腦細胞天冬氨酰氨肽酶對不同底物的 Km值(0.05 mmol/L~8.4 mmol/L)[12],表明其底物親和力較低。這可能是由于所用的底物不同造成的,如Asp-Ala-Ala-Leu和Asp-Ala-Asp-Leu等三肽。
2.3.5 底物特異性研究
分別以8種不同的aminoacyl-pNAs為底物,研究重組氨肽酶的底物特異性,結果匯總于圖6。
由圖6可知,在8種底物中,Leu-pNA為Vaap的最適作用底物,Vaap對 Ala-pNA·HCl和 Lys-pNA·2HCl也有一定的水解能力,對其余底物無水解作用。該酶對 Asp-pNA·HCl、血管緊張素 I(Angiotensin I)和血管緊張素II(Angiotensin II)等底物的水解作用還有待進一步研究。

圖6 重組氨肽酶的底物特異性Fig.6 Substrate specificity of recombinant aminopeptidase
3 結論
本研究首次將黑曲霉CICIM F0510來源的天冬氨酰氨肽酶基因vaap在畢赤酵母中進行了克隆表達,并對其基本酶學性質進行了系統解析,包括最適反應溫度和熱穩定性、最適反應pH值和pH值穩定性、金屬離子對酶活的影響、動力學參數和底物特異性等。搖瓶水平上,重組酶的最高酶活為39.7 U/mL。酶學性質的研究表明,該酶的最適反應溫度和pH值分別為55℃和9.5;在90℃孵育30 min后,其酶活仍能保留40%左右,而且在pH 8.0~10.0孵育1 h后殘留酶活在60%以上。良好的耐熱性和耐堿性使得該酶在工業應用中具有降低成本和簡化工藝等優點,應用前景十分廣闊。這為后續挖掘其應用價值以及解析其耐熱和耐堿機制提供了基礎材料。
[1]梁二賓.氨肽酶產生菌的鑒定、酶的分離純化及性質研究[D].廣州:華南理工大學,2014
[2]高新星.枯草芽孢桿菌氨肽酶分泌表達、分子改造及生理功能研究[D].無錫:江南大學,2014
[3]付靜.食品外肽酶的研究進展[J].食品科學,2013(7):349-354
[4]Zhang L,Cai Q F,Wu G P,et al.Arginine aminopeptidase from white shrimp(Litopenaeus vannamei)muscle:purification and characterization[J].Eur Food Res Technol,2013,236(5):759-769
[5]Wang W Y,De Mejia E G.A new frontier in soy bioactive peptides that may prevent age-related chronic diseases[J].Compr Rev Food Sci F,2005,4(4):63-78
[6]Gonzales T,Robert-Baudouy J.Bacterial aminopeptidases:properties and functions[J].FEMS Microbiol Rev,1996,18(4):319-344
[7]Basten D E J W,Visser J,Schaap P J.Lysine aminopeptidase of Aspergillus niger[J].Microbiology-SGM,2001,147:2045-2050
[8]Jalving R,Godefrooij J,ter Veen W J,et al.Characterisation of the Aspergillus niger dapB gene,which encodes a novel fungal type IV dipeptidyl aminopeptidase[J].Mol Genet Genomics,2005,273(4):319-325
[9]Basten D E,Moers A P,Ooyen A J,et al.Characterisation of Aspergillus niger prolyl aminopeptidase[J].Mol Genet Genomics,2005,272(6):673-679
[10]Basten D E J W,Dekker P J T,Schaap P J.Aminopeptidase C of Aspergillus niger is a novel phenylalanine aminopeptidase[J].Appl Environ Microb,2003,69(2):1246-1250
[11]Pel H J,de Winde J H,Archer D B,et al.Genome sequencing and analysis of the versatile cell factory Aspergillus niger CBS 513.88[J].Nat Biotechnol,2007,25(2):221-231
[12]Wilk S,Wilk E,Magnusson R P.Purification,characterization,and cloning of a cytosolic aspartyl aminopeptidase[J].J Biol Chem,1998,273(26):15961-15970
[13]Yokoyama R,Kawasaki H,Hirano H.Identification of yeast aspartyl aminopeptidase gene by purifying and characterizing its product from yeast cells[J].FEBS J,2006,273(1):192-198
[14]Watanabe J,Tanaka H,Akagawa T,et al.Characterization of Aspergillus oryzae aspartyl aminopeptidase expressed in Escherichia coli[J].Biosci Biotechnol Biochem,2007,71(10):2557-2560
[15]諸葛健,王正祥.工業微生物實驗技術手冊[M].北京:中國輕工業出版社,1994
[16]Zialor.DNA sequencing with chain-terminating inhibitors[J].Proc Natl Acad Sci U S A,2014,5(1233):751-780
[17]Tamura K,Dudley J,Nei M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Mol Biol Evol,2007,24(8):1596-1599
[18]Vallee B L,Auld D S.Zinc coordination,function,and structure of zincenzymesandotherproteins[J].Biochemistry,1990,29(24):5647-5659
[19]Wilk S,Wilk E,Magnusson R P.Identification of histidine residues important in the catalysis and structure of aspartyl aminopeptidase[J].Arch Biochem Biophys,2002,407(2):176-183
[20]Chaikuad A,Pilka E S,De Riso A,et al.Structure of human aspartyl aminopeptidase complexed with substrate analogue:insight into catalytic mechanism,substrate specificity and M18 peptidase family[J].BMC Struct Biol,2012,12:14
[21]吳延濤,丁國偉,席宏星,等.耐熱賴氨酸氨肽酶菌株篩選、鑒定及基因克隆[J].食品與生物技術學報,2014(8):821-826
Molecular Cloning and Biochemical Characterization of An Aspartyl Aminopeptidase from Aspergillus niger
QIAO Ya-li1,DONG Zi-xing1,2,*,SONG Peng1,LIU Xiao-guang1,2,LU Fu-ping1
(1.College of Biotechnology,Tianjin University of Science and Technology,Tianjin 300457,China;2.College of Chemical Engineering and Materials Science,Tianjin University of Science and Technology,Tianjin 300457,China)
In the present study,using complementary DNA (cDNA)of Aspergillus niger CICIM F0510 as the template,a novel gene (vaap)encoding aspartyl aminopeptidase was amplified by PCR and then successfully expressed in Pichia pastoris GS115.At the shake-flask level,the aminopeptidase activity of recombinant strain GS115 (pPIC9K-vaap)was 39.7 U/mL.The optimum temperature and pH of recombinant enzyme Vaap were shown to be 55℃ and 9.5,respectively.After incubation at 90℃or pH 8.0-10.0 for 1 h,this enzyme retained 20%or 80%of its initial activity.The activity of Vaap was significantly enhanced by Sn2+and Co2+,but inhibited by Fe3+,Ca2+,EDTA and SDS.Its Kmand Vmaxvalues towards L-leucine-pnitroanilide (Leu-pNA)were determined to be 12.96 mmol/L and 2.14 μg/(mL·min),respectively.Among the eight substrates tested,LeupNA was the preferred substrate for Vaap,and it also hydrolyzed alanine-pnitroanilide(Ala-pNA·HCl)and Lysine-pnitroanilide(Lys-pNA·2HCl).
aspartyl aminopeptidase;Aspergillus niger;molecular cloning;enzymatic properties
10.3969/j.issn.1005-6521.2017.24.036
喬雅麗(1991—),女(漢),碩士,研究方向:酶工程與技術。
*通信作者:董自星,助理研究員,研究方向:酶工程與技術。
2017-05-12

