大連兩個海區海帶相關可培養細菌群落分析
孫丕海,錢 坤,楊志平,李曉麗,王連順,孫雪瑩,馬悅欣
( 1.大連海洋大學,海珍品苗種培育基地,遼寧 大連116023;2.大連金普新區海洋與漁業局,遼寧 大連116100;3.大連匯新鈦設備開發有限公司,遼寧 大連116039;4.大連海洋大學,農業部北方海水增養殖重點實驗室,遼寧 大連116023 )
大連兩個海區海帶相關可培養細菌群落分析
孫丕海1,錢 坤2,楊志平3,李曉麗4,王連順4,孫雪瑩4,馬悅欣4
( 1.大連海洋大學,海珍品苗種培育基地,遼寧 大連116023;2.大連金普新區海洋與漁業局,遼寧 大連116100;3.大連匯新鈦設備開發有限公司,遼寧 大連116039;4.大連海洋大學,農業部北方海水增養殖重點實驗室,遼寧 大連116023 )
為研究大連兩個海區海帶相關可培養細菌群落,于2014年12月和2015年3月分別自登沙河灣和正明寺海帶養殖區采集健康海帶孢子體和海水,采用2216E涂布平板法,共分離出240株細菌,對所有菌株進行PCR和16S rDNA測序。結果表明,120條海帶相關序列歸屬為4個門、5個綱、8個目、15個科、19個屬、42個種;120條海水序列屬于4個門、6個綱、10個目、18個科、26個屬,46個種。在門水平,海帶相關和海水細菌群落均可分為變形菌門、厚壁菌門、放線菌門和擬桿菌門,其中變形菌門和厚壁菌門為優勢門。在屬水平,海帶相關優勢屬為嗜冷桿菌屬、假單胞菌屬、副球菌屬和Salinibacterium;海水相關優勢屬為芽孢桿菌屬、假單胞菌屬、動球菌屬、假交替單胞菌屬和弧菌屬。只從海帶分離出的細菌屬于微桿菌屬、微小桿菌屬、黃桿菌屬、短小桿菌屬、腸桿菌屬、寡養單胞菌屬和原小單孢菌屬。S?rensen指數表明,兩個海區海帶細菌群落有一定差異,不同海區海水及同一海區海帶與海水細菌群落明顯不同。
海帶孢子體;16S rDNA;細菌群落
海帶(Saccharinajaponica,原名Laminariajaponica)[1]是一種經濟價值較高的大型褐藻, 廣泛應用于食品、醫藥、化工等方面,是大連地區大規模栽培的主要藻類之一。Duan等[2]對膠州灣海帶表面進行了掃描電子顯微鏡觀察和細菌的分離鑒定。從團島灣潮間帶采集的海帶表面分離的細菌種類比較簡單[3]。Balakirev等[4]分析發現太平洋西北地區廣泛分布的海帶典型種及其常見形態變種共生微生物群落存在顯著差異,認為高遺傳相似性形式(海帶典型種和常見形態變種)的表型差異由宿主—共生體相互作用引起,細菌共生體可以作為區分遺傳相似藻類形式的敏感標記。而假交替單胞菌屬(Pseudoalteromonas)、弧菌屬(Vibrio)和鹽單胞菌屬(Halomonas)與海帶病害相關[5-8]。不過目前有關海帶相關細菌群落的研究仍然較少。本試驗采用傳統培養方法對大連登沙河灣和正明寺養殖區健康海帶相關和海水細菌群落進行了比較研究,以期為了解細菌與海帶的相互作用提供參考。
1 材料與方法
1.1 樣品采集
分別于2014年12月和2015年3月自大連登沙河灣和正明寺養殖區采集健康海帶孢子體(1棵)和海水(3個點混合樣)。
1.2 細菌分離純化
剪取海帶葉片生長點附近葉片10 g,經無菌海水沖洗3遍后,按一定比例加入無菌海水磨碎后作為原液,梯度稀釋后涂布2216E平板;水樣直接作為原液,梯度稀釋后涂布2216E平板;室溫(約15 ℃)培養14 d,從各樣品適當稀釋度平板上各隨機挑取30個菌落,在2216E平板上分離純化,獲得純培養于2216E斜面4 ℃保存。
1.3 細菌16S rRNA基因測序
將待測菌株接種2216E液體培養基過夜培養,使用生工生物工程(上海) 股份有限公司的土壤基因組DNA快速提取試劑盒提取細菌基因組DNA,提取方法見試劑盒說明書。用引物為F27/R1492[9]擴增細菌16S rRNA基因片段,用1% (m/V)瓊脂糖凝膠電泳檢測PCR產物,將PCR 產物送生工生物工程(上海) 股份有限公司測序。
1.4 細菌16S rRNA基因序列分析
將各菌株序列于EzBioCloud(http://www.ezbiocloud.net/eztaxon)提供的公共數據庫中進行序列同源性比對,從結果中取相似性最高的序列。將比對后相似性大于97%的序列作為1個種[10],反之則為2個。
為了比較兩個海區海帶和海水細菌群落,計算S?rensen相似性指數[11]。
2 結果與分析
2.1 海帶和海水可培養細菌群落組成分析
自海帶及其周圍海水各分離可培養細菌120株。經過16S rRNA基因序列比對,120條海帶相關序列歸屬為4個門(圖1)、5個綱(圖2)、8個目、15個科(圖3)、19個屬(圖4);120條海水序列屬于4個門(圖1)、6個綱(圖2)、10個目、18個科(圖3)、26個屬(圖4)。
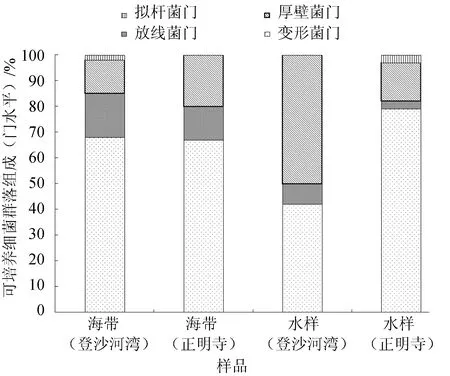
圖1 海帶和海水可培養細菌群落組成(門水平)
由圖1可見,登沙河灣海帶優勢門為變形菌門(68.33%)、放線菌門(16.67%)和厚壁菌門(13.33%);正明寺海帶優勢門為變形菌門(66.67%)、厚壁菌門(20.00%)和放線菌門(13.33%);登沙河灣海區水樣優勢門為厚壁菌門(50.00%)和變形菌門(41.67%);正明寺海區水樣優勢門為變形菌門(78.33%)和厚壁菌門(15.00%)。
由圖2可見,登沙河灣海帶優勢綱為γ-變形菌綱(56.67%)、放線菌綱(16.67%)、芽孢桿菌綱(13.33%)和α-變形菌綱(11.67%);正明寺海帶優勢綱為γ-變形菌綱(45.00%)、α-變形菌綱(21.67%)、芽孢桿菌綱(20.00%)和放線菌綱(13.33%);登沙河灣海區水樣優勢綱依次為芽孢桿菌綱(51.67%)和γ-變形菌綱(31.67%);正明寺海區水樣優勢綱依次為γ-變形菌綱(68.33%)、芽孢桿菌綱(15.00%)和α-變形菌綱(10.00%)。
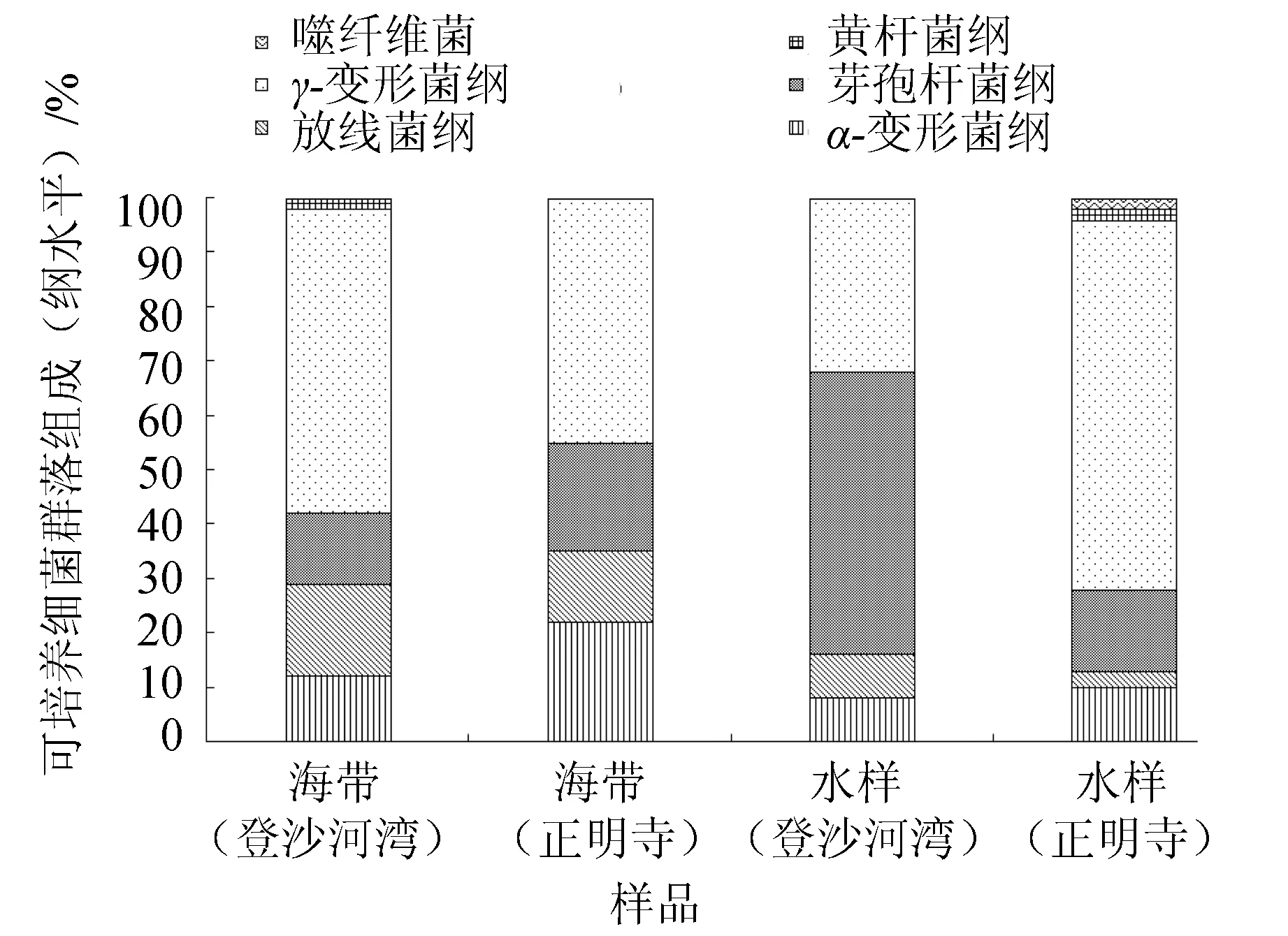
圖2 海帶和海水可培養細菌群落組成(綱水平)
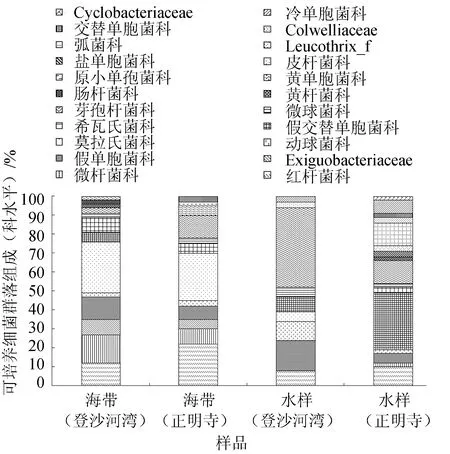
圖3 海帶和海水可培養細菌群落組成(科水平)
由圖3可見,登沙河灣海帶優勢科依次為莫拉氏菌科(26.67%)、微桿菌科(15.00%)、假單胞菌科(13.33%)和紅桿菌科(11.67%);正明寺海帶優勢科為莫拉氏菌科(25.00%)、紅桿菌科(21.67%)和芽孢桿菌科(11.67%);登沙河灣海區水樣優勢科依次為芽孢桿菌科(41.67%)、假單胞菌科(15.00%)和動球菌科(10.00%);正明寺海區水樣優勢科依次為假交替單胞菌科(30.00%)、芽孢桿菌科(13.33%)、弧菌科(11.67%)和紅桿菌科(10.00%)。
由圖4可見,登沙河灣海帶優勢屬依次為嗜冷桿菌屬(Psychrobacter)(26.67%)、假單胞菌屬(Pseudomonas)(13.33%)、副球菌屬(Paracoccus) (11.67%)和Salinibacterium(11.67%); 正明寺海帶優勢屬為嗜冷桿菌屬(25.00%)和副球菌屬(20.00%);登沙河灣海區水樣優勢屬依次為芽孢桿菌屬(Bacillus)(35.00%)、假單胞菌屬(15.00%)和動球菌屬(Planococcus)(10.00%);正明寺海區水樣優勢屬依次為假交替單胞菌屬(30.0%)、芽孢桿菌屬(13.33%)和弧菌屬(10.00%)。
所有菌株與其相關有效發表菌株的序列相似性為99%。按16S rRNA基因序列相似性大于97%的菌株歸于同一種計[10],從海帶和海水分離的細菌分別可以歸為42個種和46個種(表1)。
2.2 海帶和海水細菌群落比較
S?rensen指數可反映不同細菌群落的相似性,S?rensen指數值越小,說明群落間相似性越低,群落間差異越大。兩個海區海帶和海水細菌群落之間S?rensen相似性指數結果見表2,不同海區海帶細菌群落有一定差異,不同海區海水和同一海區海帶與海水細菌群落明顯不同。

圖4 海帶和海水可培養細菌群落組成(屬水平)

樣品細菌群落組成(種水平)登沙河灣海帶地下節桿菌(Arthrobactersubterraneus),Bacillusoryzaecorticis,B.tianshenii,萎蔫短小桿菌(Curtobacteriumflaccumfaciens),Enterobacterxiangfangensis,Exiguobacteriumoxidotolerans,E.sibiricum,Flavobacteriumfrigidarium,Microbacteriumfoliorum,Oceanobacilluspicturae,Paracoccusaestuarii,P.haeundaensis,馬氏副球菌(P.marcusii),P.seriniphilus,Planococcusmaritimus,埃氏假交替單胞菌(Pseudoalteromonasespejiana),Pseudomonasgessardii,P.koreensis,Psychrobacteraquimaris,P.fozii,P.nivimaris,解蛋白嗜冷桿菌(P.proteolyticus),阿穆爾斯克灣鹽地桿菌(Salinibacteriumamurskyense),Shewanellaarctica,S.glacialipiscicola,S.vesiculosa,嗜根寡養單胞菌(Stenotrophomonasrhizophila)正明寺海帶貝熱爾氏節桿菌(Arthrobacterbergerei),海口芽孢桿菌(Bacillushaikouensis),B.oryzaecorticis,Exiguobacteri-umoxidotolerans,E.sibiricu,棲稻黃色單胞菌(Flavimonasoryzihabitans),Microbacteriummaritypicum,氧化微桿菌(M.oxydans),Oceanobacilluspicturae,Paracoccusaestuarii;P.homiensis,P.seriniphilus,Paragla-ciecolamesophila,Planococcusmaritimus,P.plakortidis,Promicromonosporaxylanilytica,Pseudomonasgra-nadensis,Psychrobacteraquimaris,P.arcticus,P.cryohalolentis,P.nivimaris,P.okhotskensis,P.proteolyti-cus,嗜根寡養單胞菌(Salinibacteriumamurskyense),Shewanellaarctica,S.basaltis,S.vesiculosa,嗜根寡養單胞菌(Stenotrophomonasrhizophila),Sulfitobacterlitoralis登沙河灣海區水樣Arthrobacterarilaitensis,海口芽孢桿菌(Bacillushaikouensis),B.oceanisediminis,B.oryzaecorticis,B.vietna-mensis,Brachybacteriumginsengisoli,Nesterenkoniajeotgali,馬氏副球菌(Paracoccusmarcusii),P.seriniphi-lus,Planococcusrifietoensis,Planomicrobiumchinense,P.koreense,P.okeanokoites,北極假交替單胞菌(Pseud-oalteromonasarctica),產黑假交替單胞菌(P.nigrifaciens),Pseudomonascoleopterorum,P.rhodesiae,猴假單胞菌(P.simiae),Psychrobactercibarius,P.nivimaris,肺嗜冷桿菌(P.pulmonis),Ruegeriaatlantica,Sulfito-bacterguttiformis,Virgibacillusnecropolis正明寺海區水樣Algoriphagusaquimarinus,Aliivibriologei,Arthrobacterhumicola,海口芽孢桿菌(Bacillushaikouensis),B.litoralis,B.oryzaecorticis,B.vietnamensis,Cellulophagabaltica,Cobetialitoralis,Colwelliaasteriadis,C.meonggei,Glaciecolapunicea,滸苔緩生菌(Lentibacteralgarum),毛霉亮發菌(Leucothrixmucor),Paracoccuszeaxanthinifaciens,Planococcusdechangensis,北極假交替單胞菌(Pseudoalteromonasarctica),埃氏假交替單胞菌(P.espejiana),產黑假交替單胞菌(P.nigrifaciens),Pseudomonasrhodesiae,猴假單胞菌(P.simiae),Pseu-dophaeobacterarcticus,Psychromonasarctica,阿穆爾斯克灣鹽地桿菌(Salinibacteriumamurskyense),海動物腸希瓦氏菌(Shewanellamarinintestina),Vibriohemicentroti,V.lentus,燦爛弧菌(V.splendidus)
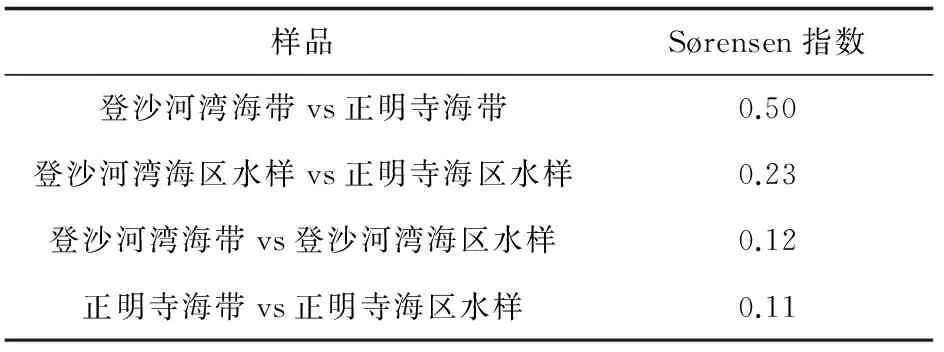
表2 海帶和海水可培養細菌群落的S?rensen指數
3 討 論
3.1 海帶相關可培養細菌群落組成分析
已有研究表明,海帶相關細菌群落包括變形菌門、擬桿菌門(CFB 群)、放線菌門和厚壁菌門[2-4,12]。在門水平,本試驗從海帶分離的細菌和前人研究結果基本一致。海帶屬的其他種如L.saccharina相關細菌群落也屬于上述四個門[13-14],L.digitata附生細菌群落由變形菌門、放線菌門和擬桿菌門組成[15],L.longicruris中發現變形菌門和擬桿菌門[16],細布海帶(L.religiosa)中只有變形菌門[17],而L.hyperborea相關細菌群落也包括變形菌門、擬桿菌門、厚壁菌門、放線菌門[18-21]。在科水平,從健康海帶分離出22個科的細菌(圖3),其中假單胞菌科、假交替單胞菌科、交替單胞菌科、微球菌科、莫拉氏菌科、腸桿菌科、芽孢桿菌科和黃桿菌科為已報道海帶相關細菌群落成員[2,4,12,22-23]。此外,還分離出屬于希瓦氏菌科、微桿菌科、黃單胞菌科、紅桿菌科、動球菌科、原小單胞菌科的細菌。L.saccharina相關細菌群落由希瓦氏菌科、微桿菌科、微球菌科、紅桿菌科和黃單胞菌科的成員組成[14]。從L.digitata上發現屬于微桿菌科、微球菌科和紅桿菌科的細菌[15]。紅桿菌科也可從L.hyperborea上分離到[20]。動球菌科和原小單胞菌科作為海帶屬藻類相關細菌群落組成為國內外首次報道。本試驗從健康海帶分離出共計19個屬的細菌(圖4),其中芽孢桿菌屬、黃桿菌屬(Flavobacterium)、假交替單胞菌屬、假單胞菌屬和嗜冷桿菌屬為已報道海帶相關細菌群落成員[2,4,12]。希瓦氏菌屬(Shewanella)、亞硫酸桿菌屬(Sulfitobacter)、假交替單胞菌屬、假單胞菌屬、寡養單胞菌屬(Stenotrophomonas)、Glaciecola和節桿菌屬(Arthrobacter)在L.saccharina中發現[14]。L.digitata的細菌群落由短小桿菌屬(Curtobacterium)、節桿菌屬、副球菌屬、假交替單胞菌屬和假單胞菌屬組成[15]。從L.longicruris分離出屬于假單胞菌屬和黃桿菌屬的種類[16]。芽孢桿菌屬的細菌也可從L.hyperborea分離獲得[20]。微桿菌屬在澳洲石莼(Ulvaaustralis)和Pachymeniopsislauceolata中發現[24-25]。腸桿菌屬(Enterobacter)、動性球菌屬和Salinibacterium分別屬于藻類Cystoseiramyrica[26]、Gracilariadura[27]、Camphylaephorahyphaeoides[28]和Ahnfeltiasp. (Genbank未發表數據)相關細菌群落成員。原小單胞菌屬(Promicromonospora)、Virgibacillus和Oceanobacillus作為海藻相關細菌群落成員為首次發現,擴展了目前海帶相關微生物群落的知識。
海帶相關細菌可能是潛在的抗菌資源,如一種芽孢桿菌、一種假單胞菌和一種黃桿菌(Flavobacteriumsp.)具有抗微生物活力[29],也可能是潛在病原,如Pseudoalteromonasbacteriolytica[6]和P.elyakovii[7]分別是海帶紅點病和葉斑點傷病害的病原菌。7株假交替單胞菌屬和4株弧菌屬細菌可能為海帶孔爛病的潛在病原[8]。本研究從海帶中發現了芽孢桿菌、假單胞菌、黃桿菌、假交替單胞菌和弧菌屬,其生態作用及其對宿主的影響待于進一步研究。
3.2 海帶和海水細菌群落比較
本試驗中海帶養殖區海水中的細菌群落由變形菌門、厚壁菌門、放線菌門和擬桿菌門組成。這些門廣泛分布在其他海藻生長海域水體中,如L.saccharina生長海域水體中的細菌屬于變形菌門和擬桿菌門[13]。澳洲石莼生長海域水體中優勢門為變形菌門、擬桿菌門和放線菌門,而厚壁菌門豐度較低。盡管在門水平上海帶相關細菌群落與其周圍海水的細菌群落大致相似,但在種水平,海帶和海水細菌群落明顯不同。這一結果與已有的研究結果基本一致[13,19,30]。Staufenberger 等[13]研究結果表明,L.saccharina和海水樣品DGGE 條帶圖譜明顯不同,相似性只有20%。L.hyperborea表面生物膜和其周圍海水細菌群落重疊較少[19]。Burke等[30]通過16S rRNA基因克隆文庫技術發現在門水平澳洲石莼附生細菌群落和和其周圍海水細菌群落類似,但是,Bray-Curtis相似性和聚類分析結果表明,在較低等分類水平(種),兩細菌群落明顯不同。可能是由于兩種環境的物理差異引起群落組成的差異。海藻上細菌的可能來源之一是其周圍的海水,因此海藻表面細菌可能是通過選擇過程或經直接表面接觸輸送細菌,從海水較少的種群中吸收成員[19]。
[1] Lane C E, Mayes C, Druehl L D, et al. A multi-gene molecular investigation of the kelp (Laminariales, Phaeophyceae) supports substantial reorganization [J]. Journal of Phycology, 2006, 42(2):493-512.
[2] Duan D, Xu L, Fei X, et al. Marine organisms attached to seaweed surfaces in Jiaozhou Bay, China[J]. World Journal of Microbiology and Biotechnology, 1995, 11(3):351-352.
[3] Wang Z F, Xiao T, Pang S J, et al. Isolation and identification of bacteria associated with the surfaces of several algal species [J]. Chinese Journal of Oceanology and Limnology, 2009, 27(3):487-492.
[4] Balakirev E S, Krupnova T N, Ayala F J. Symbiotic associations in the phenotypically-diverse brown algaSaccharinajaponica[J]. PloS One, 2012, 7(6):e39587.
[5] Yumoto I, Yamaguchi K, Yamada K, et al. Relationship between bacterial flora and occurrence of theAlteromonassp., the causative agent of red-spots on the culture bed of makonbuLaminariajaponica, in the coastal area of Funka Bay [J]. Nippon Suisan Gakkaishi, 1989, 55(11):1907-1914.
[6] Sawabe T, Makino H, Tatsumi M, et al.Pseudoalteromonasbacteriolyticasp. nov., a marine bacterium that is the causative agent of red spot disease ofLaminariajaponica[J]. International Journal of Systematic Bacteriology, 1998, 48(3):769-774.
[7] Sawabe T, Tanaka R, Christen R. et al. Assignment ofAlteromonaselyakoviiKMM162T and five strains isolated from spot wounded fronds ofLaminariajaponicatoPseudoalteromonaselyakoviicomb. nov. and the extended description of the species [J]. International Journal of Systematic and Evolutionary Microbiology, 2000, 50(1):265-271.
[8] Wang G, Shuai L, Li Y, et al. Phylogenetic analysis of epiphytic marine bacteria on Hole-Rotten diseased sporophytes ofLaminariajaponica[J]. Journal of Applied Phycology, 2008, 20(4):403-409.
[9] Stackebrandt E, Goodfellow M. Nucleic Acid Techniques in Bacterial Systematics [M]. Chichester:John Wiley & Sons Press,1991:115-175.
[10] Stackebrandt E, Goebel B M. Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology [J]. International Journal of Systematic Bacteriology, 1994, 44(4):846-849.
[11] S?rensen T. A method of establishing groups of equal amplitude in plant sociology based on similarity of species and its application to analyses of the vegetation on Danish commons [J]. Kongelige Danske Videnskabernes Selskabs Biologiske Skrifter, 1948, 5(4):1-34.
[12] Dimitrieva G Y, Crawford R L, Yuksel G ü. The nature of plant growth-promoting effects of a pseudoalteromonad associated with the marine algaLaminariajaponicaand linked to catalase excretion [J]. Journal of Applied Microbiology, 2006, 100(5):1159-1169.
[13] Staufenberger T, Thiel V, Wiese J, et al. Phylogenetic analysis of bacteria associated withLaminariasaccharina[J]. FEMS Microbiology Ecology, 2008, 64(1):65-77.
[14] Wiese J,Thiel V, Nagel K, et al. Diversity of antibiotic-active bacteria associated with the brown algaLaminariasaccharinafrom the Baltic Sea [J]. Marine Biotechnology, 2009, 11(2):287-300.
[15] Salaün S, Nelly K, Philippe P, et al. Whole-cell spectroscopy is a convenient tool to assist molecular identification of cultivatable marine bacteria and to investigate their adaptive metabolism [J]. Talanta, 2010, 80(5):1758-1770.
[16] Laycock R A. The detrital food chain based on seaweeds. I. Bacteria associated with the surface ofLaminariafronds [J]. Marine Biology, 1974, 25(3):223-231.
[17] Vairappan C S, Suzuki M, Motomura T, et al. Pathogenic bacteria associated with lesions and thallus bleaching symptoms in the Japanese kelpLaminariareligiosaMiyabe (Laminariales, Phaeophyceae) [J]. Hydrobiologia, 2001, 445(1):183-191.
[18] Bengtsson M M, ?vre?s L. Planctomycetes dominate biofilms on surfaces of the kelpLaminariahyperborea[J]. BMC Microbiology, 2010, 10(3):1-12.
[19] Bengtsson M M, Sjotun K, Ovreas L, et al. Seasonal dynamics of bacterial biofilms on the kelpLaminariahyperborea[J]. Aquatic Microbial Ecology, 2010, 60(1):71-83.
[20] Bengtsson M M, Sj?tun K, Storesund J E, et al. Utilization of kelp-derived carbon sources by kelp surface-associated bacteria [J]. Aquatic Microbial Ecology, 2011, 62(2):191-199.
[21] Bengtsson M M, Sj?tun K, Lanzén A, et al. Bacterial diversity in relation to secondary production and succession on surfaces of the kelpLaminariahyperborea[J]. The ISME Journal, 2012, 6(12):2188-2198.
[22] 徐滌,秦松,龐國興,等. 青島和大連海區海帶(Laminariajaponica) 外生細菌的16SrRNA 基因序列分析[J].高技術通訊, 2004,14(2):81-86.
[23] Hollants J, Leliaert F, De Clerck O, et al. What we can learn from sushi: a review on seaweed bacterial associations [J]. FEMS Microbiology Ecology, 2013, 83(1):1-16.
[24] Kanagasabhapathy M, Sasaki H, Nagata S. Phylogenetic identification of epibiotic bacteria possessing antimicrobial activities isolated from red algal species of Japan [J]. World Journal of Microbiology Biotechnology, 2008, 24(10):2315-2321.
[25] Burm?lle M, Webb J S, Rao D, et al. Enhanced biofilm formation and increased resistance to antimicrobial agents and bacterial invasion are caused by synergistic interactions in multispecies biofilms [J]. Applied and Environmental Microbiology, 2006, 72(6):3916-3923.
[26] Mohammed N A, Hassan H M, Rateb M E, et al. Diketopiperazine derivatives fromEnterobactercloacaeisolated from the Red Sea algaCystoseiramyrica[J]. Egyptian Pharmaceutical Journal, 2013, 12(2):163.
[27] Singh R P, Bijo A J, Baghel R S, et al. Role of bacterial isolates in enhancing the bud induction in the industrially important red algaGracilariadura[J]. FEMS Microbiology Ecology, 2011, 76(2):381-392.
[28] Beleneva I A, Zhukova N V. Bacterial communities of some brown and red algae from Peter the Great Bay, the Sea of Japan [J]. Microbiology, 2006, 75(3):348-357.
[29] Zheng L, Han X, Chen H, et al. Marine bacteria associated with marine macroorganisms: the potential antimicrobial resources[J].Annals Microbiology,2005,55(2):119-124.
[30] Burke C, Thomas T, Lewis M, et al. Composition, uniqueness and variability of the epiphytic bacterial community of the green algaUlvaaustralis[J]. ISME Journal,2011, 5(4):590-600.
CulturableBacteriaCommunityAssociatedwithSaccharinajaponicafromTwoSeaAreasinDalian
SUN Pihai1, QIAN Kun2, YANG Zhiping3, LI Xiaoli4, WANG Lianshun4, SUN Xueying4, MA Yuexin4
( 1.Seafood Seedling Breeding Base, Dalian Ocean University, Dalian 116023, China;2. Dalian Jinzhou District Oceanic and Fishery Administration, Dalian 116100, China; 3. Dalian Huixin Titanium Equipment Development Company Limited, Dalian 116039, China; 4. Key Laboratory of Mariculture and Stock Enhancement in North Chinas Sea, Ministry of Agriculture, Dalian Ocean University, Dalian 116023,China )
The aim of the study was to examine the bacterial community associated with the kelpSaccharinajaponicafrom two sea areas in Dalian. In December 2014 and March 2015, a total of 240 isolates were obtained from the healthy kelp and the surrounding seawater collected from the sea areas of Dengshahe bay and Zhengmingsi using spread plate method relied on Zobell 2216E medium. Polymerase chain reaction and partial 16S rDNA sequencing of DNA from all isolates revealed that 120 kelp-associated sequences were classified as 4 phyla, 5 classes, 8 orders, 15 families, 19 genera and 42 species; that 120 water-derived sequences were classified as 4 phyla, 6 classes, 10 orders, 18 families, 26 genera and 46 species. At phylum level,S.japonicaand seawater communities were composed of Proteobacteria, Firmicutes, Actinobacteria and Bacteroidetes, with dominant phyla of Proteobacteria and Firmicutes. At genus level, the composition of community associated withS.japonicawas represented principally byPsychrobacter,Pseudomonas,SalinibacteriumandParacoccus; the seawater community was dominated byBacillus,Pseudoalteromonas,Pseudomonas,PlanococcusandVibrio. Additionally,Microbacterium,Exiguobacterium,Flavobacterium,Curtobacterium,Enterobacter,StenotrophomonasandPromicromonosporawere exclusively present in community ofS.japonica. As indicated by S?rensen index, there were some differences in bacterial communities between kelp samples from the two different sea areas; while there existed significant differences in communities between seawater samples from the two sea areas and between kelp sample and seawater sample from the same sea area.
Saccharinajaponica;16S rDNA; bacterial community
10.16378/j.cnki.1003-1111.2017.04.016
S917.1
A
1003-1111(2017)04-0498-06
2016-03-10;
2016-12-16.
大連市海洋與漁業局項目(2014115,2014118).
孫丕海(1959-),男,高級工程師;研究方向:海珍品種苗培育及養殖.E-mail:dlsph@aliyun.com.通訊作者:馬悅欣(1963-),女,教授,博士;研究方向:微生物生態學. E-mail:mayuexin@dlou.edu.cn.

