基于非線性降維的合成生物元件可視化
李榮燦, 楊矯云, 王海鵬
(合肥工業大學 計算機與信息學院,安徽 合肥 230009)
基于非線性降維的合成生物元件可視化
李榮燦, 楊矯云, 王海鵬
(合肥工業大學 計算機與信息學院,安徽 合肥 230009)
合成生物學中標準化元件數量多、種類雜,使得構建生物設備時難以選擇標準化元件,將這些元件可視化有助于提高生物設備構建效率。考慮生物元件為長度不一的基因短序列,文章通過結合編輯距離與高斯核函數構建生物元件距離矩陣,使用拉普拉斯特征映射方法將生物元件序列降為二維或三維數據;通過圖形化展示,功能類似的生物元件可有效地聚類,功能差異大的元件可有效地區分,且對降維后數據聚類顯示的二分類精度達到91.6%,三分類精度達到82.4%。實驗結果表明,降維后的數據具有良好的區分度,通過降維可視化將顯著提高標準化元件的選擇效率。
可視化;合成生物學;非線性降維;編輯距離;聚類
當前合成生物學的可視化多集中于設備構造過程的可視化,如Pigeoncad[1]、TinkerCell[2]、VisBOL[3]等軟件。這些軟件通過構建生物元件的可視化符號,將生物設備的構建過程進行形象化展示,從而促進生物設備的設計。當前合成生物學的迅猛發展,標準生物元件庫已積累三萬多個標準生物元件,在構建生物設備時,如何進行元件選擇是一件耗時費力的工作。考慮到合成生物標準元件種類多、數量大,若將生物元件進行可視化展示,具有不同功能的元件可有效區分,則可降低合成生物元件選擇時的復雜程度,提高生物設備的合成效率。
生物元件為基因片段,當前也有若干基因可視化方法,如Cytoscape[4]、ParaView[5]等,這些方法多是對單個基因組可視化,從而形象化展示基因內部結構。而本文期望能夠對生物元件進行可視化聚類,這對當前的方法提出了挑戰。
對生物元件可視化聚類的一個思路是數據降維。當前數據降維主要分為線性降維與非線性降維。線性降維以主成分分析(principal component analysis,PCA)為主要代表[6],通過將原始數據進行線性變換,消除屬性相關項;非線性變換以局部線性嵌入(locally linear embedding,LLE)[7]、拉普拉斯特征映射(Laplacian eigenmaps,LE)[8]為主要代表,通過維持原始數據的流行結構,使得降維后的數據與原始數據維持結構一致。鑒于標準生物元件為長度不一的文本序列,難以直接對其進行線性變換,同時非線性變換中的局部關系構建也不適用于序列文本數據,因此需要構建一種針對長度不一的基因文本序列進行降維可視化的方法。
本文通過改進拉普拉斯特征映射來進行合成生物標準元件可視化。首先采用編輯距離構建生物元件的距離矩陣,并利用高斯核函數進行距離映射,然后借助映射后的距離矩陣構建拉普拉斯矩陣,最后進行特征分解完成數據降維并可視化。通過在合成生物標準元件庫上的應用,實驗結果表明,本文提出的可視化方法可有效區分具有功能差異的生物元件,通過聚類發現,2類元件和3類元件的聚類精度分別達到91.6%和82.4%。這不僅為合成生物學家提供了一種利用可視化快速選擇元件的方法,也提供了一種有效分類生物元件序列的方法。
1 算法流程介紹
標準生物元件為長度不一致的基因片段序列,傳統基于歐氏距離的方法難以有效衡量生物元件的相似性,因此本文算法主要采用編輯距離進行生物元件相似性度量。通過結合編輯距離與拉普拉斯特征映射,對生物元件序列降維,達到序列數據可視化的目的。該主要過程步驟如下:
(1) 相似度計算。使用編輯距離作為衡量數據間距離的標準,并進行歸一化處理,以構建表征數據集的加權無向圖矩陣。
(2) 非線性降維。構建拉普拉斯矩陣,進行矩陣分解,得到降維后的數據。
(3) 可視化。將降維后的數據以圖形化方式進行展示。
1.1 距離矩陣構建
編輯距離,又稱Levenshtein距離,是指2個字串之間由一個轉成另一個所需的最少編輯操作次數[9]。通常,編輯距離越小,2個字符串的相似度越高。例如計算TAGAA→TGACA的編輯距離為2,TAGAA到TGACA編輯操作轉換過程如圖2所示。

圖1 TAGAA到TGACA編輯操作轉換過程
當前編輯距離的計算主要是基于動態規劃算法[10]。給定2條長為m、n的序列x、y,動態規劃算法構建大小為m×n的矩陣E其中的每個值Ei,j表示子序列x1x2…xi和y1y2…yj中xi與yj的最小編輯距離。Ei,j的計算公式為:
其中,δ(xi,-)、δ(-,yj)分別為插入、刪除的得分。若xi=yj,則δ(xi,yj)表示匹配得分;若xi≠yj,則δ(xi,yj)表示錯配得分。
DNA序列TAGAA和TGACA的動態規劃矩陣見表1所列。以(3,3)格計算為例,取如下3個值的最小值填入單元格。
(1) 若最上方的字符等于最左方的字符,則取左上方的數字;否則取左上方的數字加1(對于(3,3)格來說為3)。
(2) 左方數字加1(對于(3,3)格來說為2)。
(3) 上方數字加1(對于(3,3)格來說為2)。
矩陣右下角的值即為2條序列的編輯距離。
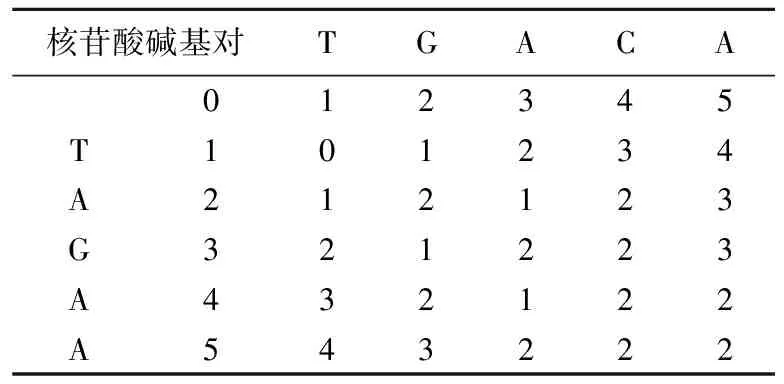
表1 TGACA和TAGAA的動態規劃矩陣
編輯距離與2條序列的長度相關,其長度為:
因為降維可視化是要得到不同序列間的相似程度,所以計算出編輯距離值后應對其進行歸一化處理,即用ED(x,y)值除以2個序列中較長序列的長度(maxLength(|x|,|y|)值)。編輯距離與2條序列的長度相關,長為m、n的序列間的編輯距離最大為max(m,n),即最長序列的長度。
得到歸一化距離后,為使距離矩陣具有更好的局部性,本文對計算得到的編輯距離使用徑向基函數核做高斯化處理,定義為:
1.2 拉普拉斯特征映射
拉普拉斯特征映射是非線性降維的主要方法,其主要思想是保證2個很相似的數據在降維的子空間里盡可能接近。假設數據實例xi、xj降維后數據實例為yi、yj,則拉普拉斯特征映射的目標函數為:
其中,Wi,j為實例xi、xj相似度。傳統拉普拉斯特征映射采用歐氏距離等計算相似度,本文通過(2)式計算得到實例間的距離矩陣,從而更好地刻畫不同基因序列間的相似度。
(4)式中目標函數的求解可轉化為最小化目標函數yTLy,再通過矩陣分解進行計算。因此拉普拉斯特征映射的主要步驟為:
(1) 采用特定的距離衡量方法,得到所有點間的相似度值,并構建一個相似度矩陣W,本文使用編輯距離來確定,即
Wi,j=K(xi,yj)
(5)
(2) 借助W和度矩陣D(D是由di構成的對角矩陣)計算拉普拉斯矩陣L,并計算其特征值與特征向量,即
L=D-W
(7)
Ly=λDy
(8)
(3) 取最小的k個非零特征值對應的特征向量作為LE算法的結果輸出,得到降維后的數據結果。
1.3 算法流程
本文算法的詳細流程為:
(1) 計算任意2條序列x、y間的編輯距離ED(x,y),并依據(2)式進行標準化處理。
(2) 利用步驟(1)的距離計算結果構造距離相似度矩陣W。
(3) 對步驟(2)的矩陣W進行高斯化處理,其中參數σ需要不斷調整以實現好的聚類效果。
(4) 計算度矩陣D。具體公式如下:
(5) 將度矩陣D和鄰接矩陣W相減得到拉普拉斯矩陣L。
L=D-W
(12)
(6) 再通過對相似矩陣進行特征分解得到特征向量。
Ly=λDy
(13)
(7) 取最小的k個非零特征值對應的特征向量作為LE算法的結果輸出,得到降維后的數據結果,并進行可視化展示。
2 實驗結果
本文從合成生物學標準元件數據庫中選取了3類生物元件,即復合部件(composite)、核糖體綁定位點(ribosome binding site,RBS)、引物(primer)來檢驗算法分類效果。其中復合部件數目為200,核糖體綁定位點數目為300,引物數目為300。實驗中的參數σ取值為0.3。
2類組件的可視化結果如圖2所示。圖2a表示復合部件與引物的可視化結果,其中深色代表復合部件,淺色代表引物;圖2b表示復合部件與核糖體綁定位點的可視化結果,其中深色代表核糖體綁定位點,淺色代表復合部件。圖2中不同顏色和符號代表不同類型的元件,可以看出,同一類型的元件會聚集在一起,不同類型間的元件會有較大差距。這說明本文的可視化方法可以很好地區分不同的元件,使得用戶可依據功能差異進行元件選擇。
3類元件的三維可視化結果如圖3所示。由圖3可以看出,淺色代表的復合部件與其他2類元件具有顯著差異性,而深色(左側下方)代表的核糖體綁定位點與深色(左側上方)代表的引物之間差別相對較小,但也可明顯看出兩者之間具有明顯聚類。產生這種現象的原因是復合部件是由不同元件構成的復合體,功能復雜,而核糖體綁定位點與引物相對簡單,差異性較小。這也反映了本文基于編輯距離的可視化很好地區分出了不同元件的功能。
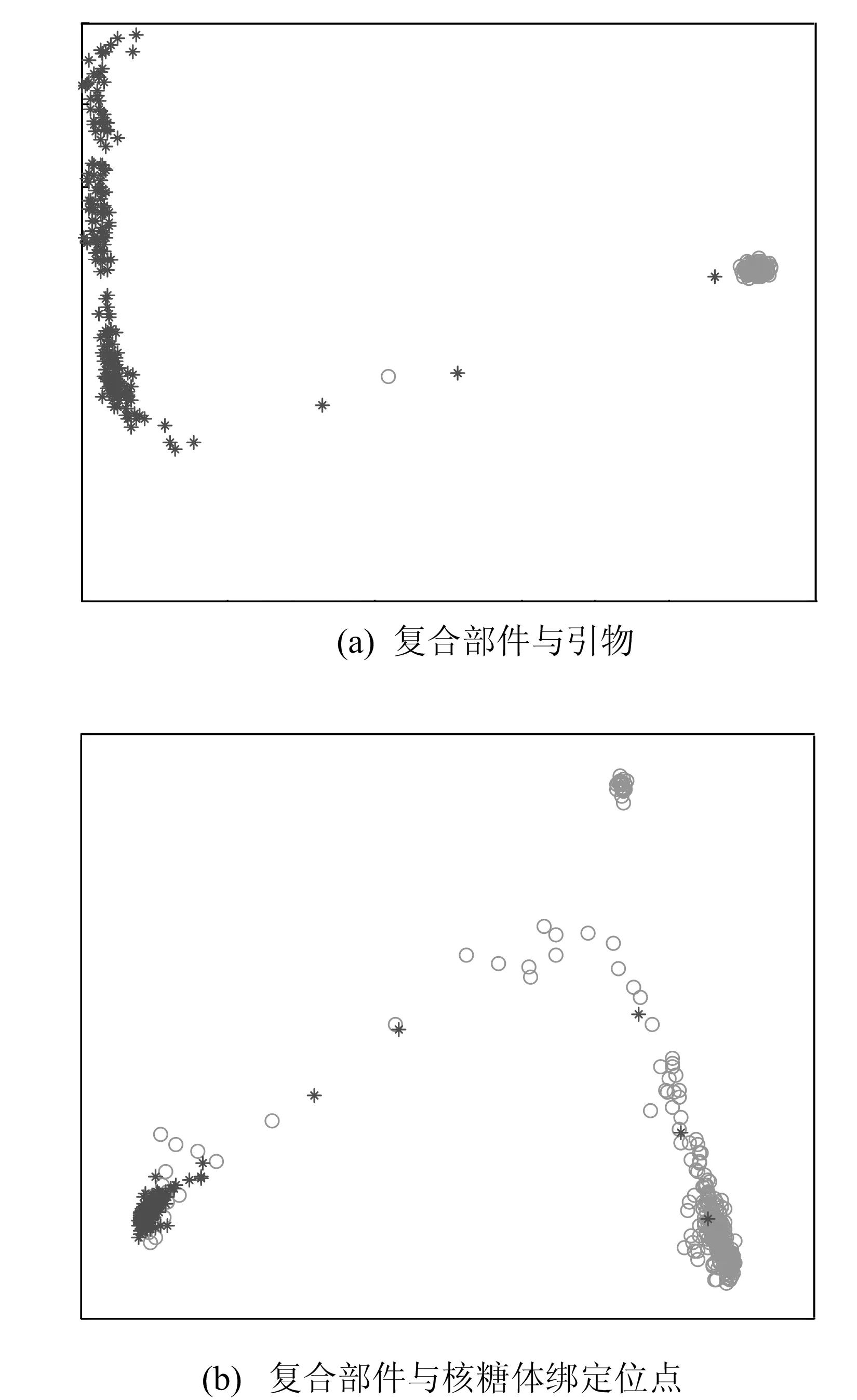
圖2 2類組件的可視化結果
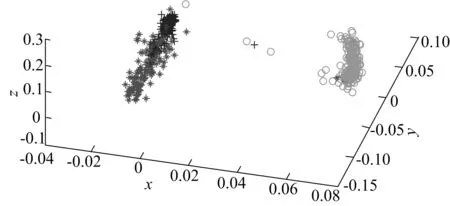
圖3 復合部件、核糖體綁定位點和引物的可視化結果
使用k-means算法對3類降維可視化的數據進行聚類,結果如下:對于2種合成元件的組合,復合部件與引物、復合部件與核糖體綁定位點的分類準確率分別為99.2%、91.6%;對于3種合成元件的組合,復合部件、引物與核糖體綁定位點的分類準確率為82.4%。可見降維后的數據具有較好的區分度。這里的準確率是指聚類后與元件的原類型相比正確分類的比率。
3 結 論
本文針對多類型的大規模合成生物元件數據集進行降維可視化,通過將編輯距離與高斯核函數相結合,建立不同元件間的相似度關系矩陣,然后基于此相似度關系進行拉普拉斯特征映射,達到元件降維目的。通過觀察降維后的數據可視化結果,表明不同類型的元件可構成不同聚類,功能差異性在圖中表現出了距離差異性,從而說明合成生物學者依據可視化的結果幫助進行元件選擇的可能性。
[1] BHATIA S,DENSMORE D.Pigeon:a design visualizer for synthetic biology[J].ACS Synthetic Biology,2013,2(6):348-350.
[2] CHANDRAN D,BERGMANN F T,SAURO H M.TinkerCell:modular CAD tool for synthetic biology[J].Journal of Biological Engineering,2009,3(1):1-17.
[3] MCLAUGHLIN J A,POCOCK M,MISIR G,et al.VisBOL:web-based tools for synthetic biology design visualization[J].ACS Synthetic Biology,2016,5(8):874.
[4] SHANNON P,MARKIEL A,OZIER O,et al.Cytoscape:a software environment for integrated models of biomolecular interaction networks[J].Genome Research,2003,13(11):2498-2504.
[5] HENDERSON A,AHRENS J,LAW C,et al.The paraview guide[M].New York:Kitware,2004.
[6] KARL PEARSON F R S.LIII.On lines and planes of closest fit to systems of points in space[J].Philosophical Magazine Series 6,2010,2(11):559-572.
[7] ROWEIS S T,SAUL L K.Nonlinear dimensionality reduction by locally linear embedding[J].Science,2000,290(5500):2323-2326.
[8] BELKIN M,NIYOGI P.Laplacian eigenmaps and spectral techniques for embedding and clustering[J].Advances in Neural Information Processing Systems,2001(14):585-591.
[9] LI Y J,LIU B.A normalized levenshtein distance metric [J].IEEE Transactions on Pattern Analysis & Machine Intelligence,2007,29(6):1091-1095.
[10] JONES N C,PEVZNER P A.An introduction to bioinformatics algorithms [M]//An introduction to bioinformatics algorithms.Massachusetts:MIT Press,2004:626-626.
Visualizationofstandardsyntheticbiologicalpartsbasedonnonlineardimensionalityreduction
LI Rongcan, YANG Jiaoyun, WANG Haipeng
(School of Computer and Information, Hefei University of Technology, Hefei 230009, China)
In synthetic biology, there are a number of standard parts with a wide variety of categories, making it hard to choose a part when constructing devices. Visualizing these parts could simplify the part selection. Considering that synthetic biological parts are DNA segments with various lengths, the similarity of these parts is evaluated by the integration of edit distance and Gaussian kernel. Based on the similarity, Laplacian Eigenmaps is employed to reduce data dimensions to two or three dimensions. By visualizing the reduced data, the parts with similar functionality could cluster together, and the parts with different functionality could be separated efficiently. Besides, the cluster accuracy for two kinds and three kinds of parts reaches 91.6% and 82.4%, respectively, which proves the discrimination of the reduced data, and this could significantly improve the efficiency of parts selection.
visualization; synthetic biology; nonlinear dimensionality reduction; edit distance; clustering
2016-04-05;
2016-05-16
國家自然科學基金資助項目(61502135);中央高校基本科研業務費專項資金資助項目(JZ2015HGBZ0111)和國家高等學校學科創新引智計劃資助項目(B14025)
李榮燦(1990-),男,福建泉州人,合肥工業大學碩士生;
楊矯云(1987-),男,山東招遠人,博士,合肥工業大學副教授,通訊作者,E-mail:jiaoyun@hfut.edu.edu.cn.
10.3969/j.issn.1003-5060.2017.12.006
TP317.4
A
1003-5060(2017)12-1610-04
(責任編輯胡亞敏)

