基于深度學習的肝臟腫瘤圖像分割
黃佳佳+趙曙光+張笑青+楊峰+許方成

摘要:傳統(tǒng)肝臟CT 圖像腫瘤分割算法常需引進先驗信息,分割效率低,無法滿足臨床實時性要求。針對這些問題,提出基于卷積-反卷積神經網絡的肝臟腫瘤圖像分割算法,自動提取肝臟腫瘤的特征。實驗表明,DSC指標約85.32%,且分割每幅圖像只需用時3至5秒,實時性好。
關鍵詞:肝臟CT圖像;腫瘤分割;反卷積網絡;自動提取特征
中圖分類號:TP391 文獻標識碼:A 文章編號:1007-9416(2017)11-0081-02
近年來,有許多算法實現肝臟及其腫瘤圖像的分割,但這些算法存在的共性是在處理的過程中或多或少的需要人工干預。如Seung-Jin Park等[1]提出基于最優(yōu)閾值的肝臟腫瘤圖像分割,該方法的缺點是必須先將肝臟分割出來,然后才能對腫瘤進行分割。本文提出一種基于卷積-反卷積神經網絡的肝臟腫瘤圖像分割方法,能夠自動分割肝臟腫瘤圖像。
1 卷積神經網絡
卷積神經網絡(Convolutional Neural Networks,CNN)[2]是深度學習算法中的一種,CNN由多個層次組成的可訓練的架構,每一層的輸入和輸出包含多個特征映射層,主要由若干個卷積層、池化層、激活函數層以及全連接層組成。
2 肝臟腫瘤圖像分割方法
2.1 圖像的預處理
如圖1所示,(a)是原始的肝臟CT圖像,在直方圖均衡化之后的(b)中,肝臟腫瘤和其它組織的灰度值有明顯的差異,這將有助于提取有效特征。由于圖像采集時受到設備噪聲的影響,CT圖像中含有噪聲。本文采用中值濾波器對其進行濾波,結果如(c)所示,濾波效果較好。
2.2 卷積-反卷積神經網絡
如前文所述,由于卷積網絡在池化過程中縮小了圖片的尺寸,而對于圖像分割的任務,最終需要得到與原圖尺寸相同的結果,為了解決此問題,文獻[3]引入反卷積網絡,其主要由反卷積層和反池化層組成。
本文在卷積網絡的基礎上加上對稱的反卷積網絡,構成卷積-反卷積網絡。在網絡中,本文增加Batch normalization層[4],使模型盡可能地收斂到全局最優(yōu)解。
3 實驗與結果分析
3.1 實驗數據集
本次實驗采用MICCAI 2017數據集。本實驗中從數據集中篩選出含有腫瘤的樣本,得到訓練圖像4983張,測試圖像2141張,尺寸為像素。
3.2 實驗流程與評價指標
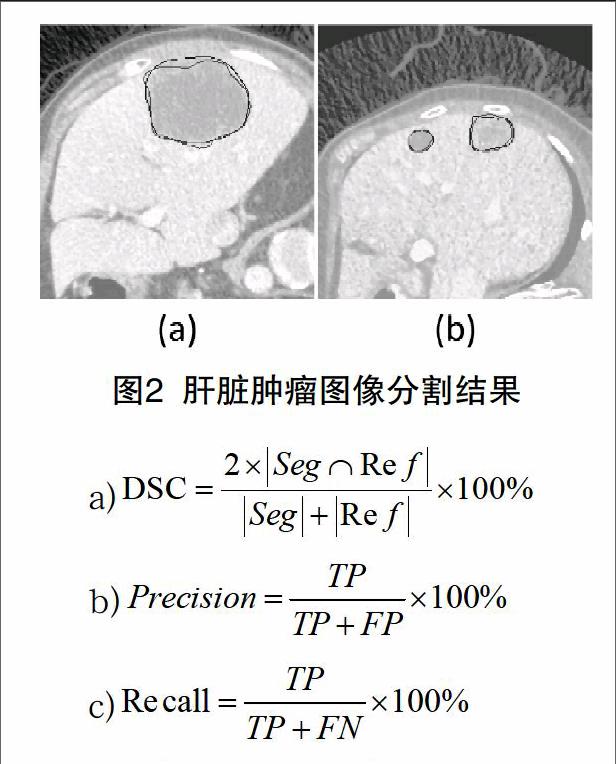
實驗流程是先對圖像進行預處理,再將預處理過后的圖像作為訓練集提供給卷積-反卷積網絡進行訓練。利用訓練得到參數模型對測試圖像進行分割之后,利用MATLAB對分割圖進行后續(xù)處理能得到分割最終結果(如圖2)。本實驗的評價指標分別采用:a)Dice相似系數;b)準確率;c)召回率等醫(yī)學圖像分割的常用指標。Seg表示為算法分割結果,Ref為金標準,TP表示真陽性,FP表示假陽性。FN表示假陰性。
3.3 實驗結果及分析
部分實驗結果如圖2所示。圖中的藍色的實線是標準分割的結果,紅色的線條是本文的分割結果。從圖中可看出,本文的肝臟腫瘤預測接近真實值,分割的準確度較高。
本文的方法與Adaboost[5]、Random Forests[6]等傳統(tǒng)分割方法進行比較,在DSC(%)指標中Adaboost、Random Forests及本文的算法分別是77.22%、78.94%、85.32%。Precision(%)指標中三種算法的指標分別是77.34%、80.99%、86.53%。Recall(%)指標中三種算法的指標分別是82.91%、83.43%、85.37%。本文分割方法的效果好于其它方法。分割一幅圖像只需2至3秒,速度明顯快于其它算法。
4 結語
本文結合肝臟CT圖像的特點,提出了一種基于全卷積-反卷積的肝臟腫瘤圖像分割方法。該方法無需手工設計特征,自動提取的深層特征能很好地描述圖像變化。經過實驗證明,該方法對肝臟腫瘤圖像分割具有較好的準確性和實時性。
參考文獻
[1]Park Seung-Jin,Seo Kyung-Sik,Park Jong-An.Automatic hepatic tumor segmentation using statistical optimal threshold[J].Lecture Notes in Computer Science,2005,3514:275-283.
[2]Y.Le Cun,L.Bottou,Y.Bengio,P.Haffner.Gradient-based Learning Applied to Document Recognition. Proceedings of the IEEE, 86(11):2278-2324,1998.
[3]NohH,HongS,HanB.Learning deconvolution network for semantic segmentation[C]//The IEEE International Conference on Computer Vision .NewYork:IEEE,2015:1520-1528.
[4]S.Ioffe and C.Szegedy.Batch normalization:Accelerating deep network training by reducing internal covariate shift.arXiv preprint arXiv:1502.03167,2015.5.
[5]Y.Freund,R.E.Schapire. A Decision-theoretic Generalization of On-line Learning and An Application to Boosting.Journal of Computer and System Sciences,55,1997.
[6]L.Breiman.Random forests.Mach.Learning,45(1):5-32,2001.
Abstract:The traditional liver CT image segmentation algorithm often needs to introduce prior information, and the segmentation efficiency is low, which can not meet the real-time clinical requirements. To solve these problems, an algorithm of hepatic tumor segmentation based on convolution-deconvolution neural network is proposed to automatically extract features of liver tumors. Experiments show that the DSC index is about 85.32%, and the segmentation of each image takes only 3 to 5 seconds, good real-time.
Key Words:liver CT image; tumor segmentation; deconvolution network; automatic extraction of featuresendprint

