土壤主要還原轉化過程中微生物功能基因多樣性研究進展
馮佳胤,朱敏,何艷*1
土壤是一個復雜的氧化還原體系,存在大量活躍的碳、氮、鐵、硫等生源要素,以及農藥、重金屬等污染物質的生物地球化學循環過程,對自然界中物質代謝和能量流動具有決定性的影響。隨著全社會對于土壤質量的關注度日漸提高,國家在2016年全面啟動了土壤污染防治行動計劃,著重強調土壤環境的改善與良性生態系統的構建。因而,開展自然復雜土壤體系中多介質界面上多要素-多過程的耦合作用與機制的研究,對探索物質循環過程、維持生態環境平衡和實現可持續發展均具有重要的現實意義。
自然環境中的氮循環、Fe/S還原、產甲烷以及有機污染物的還原脫鹵等還原過程的發生本質上是微生物厭氧呼吸作用介導的電子傳遞過程。在該過程中,存在著不同的生源要素、蛋白組分間頻繁而復雜的交互關系,如硝酸鹽、三價鐵、硫酸鹽和鹵代污染物等物質之間存在電子受體間的競爭關系,由功能基因表征的對應的蛋白酶,如亞硫酸鹽還原酶、甲基輔酶M還原酶或還原脫鹵酶等參與并催化著相關物質的呼吸代謝過程。
近年來,隨著研究領域的深入和分子生物學技術的發展,傳統的測序手段已經不能完全滿足對微觀尺度下土壤微生物適應特定環境的變化過程、相關元素物質的代謝機制以及污染物轉化與歸趨等功能性研究。因此,應用先進的分子生物學方法,如功能基因組學技術、微陣列技術、原位活性檢測技術和宏基因組學技術等,利用參與相應代謝過程的功能基因的特異性來指示土壤微生物的生態功能,已成為探究微生物生態信息與特定代謝途徑關系的重要紐帶[1-2]。
現有研究尚未對復雜土壤體系中的物質代謝和元素耦合關系,及其可能影響的有機污染物還原脫鹵過程的相關機制進行過系統的梳理。有鑒于此,本文綜述了幾種在典型土壤還原轉化過程中參與和介導不同代謝過程的關鍵功能基因,以期為進一步研究氮循環、硫酸鹽還原、鐵還原、產甲烷以及還原脫鹵等過程中的微生物生態特性,揭示相關生源要素耦合污染物還原轉化的內在微生物學機制提供參考依據。
1 氮還原轉化過程
土壤中由微生物介導的氮素循環是生物地球化學循環的重要組成部分之一,在穩定農田生態系統、緩解全球溫室效應等方面具有舉足輕重的作用。梳理土壤固氮作用、反硝化作用和異化硝酸鹽還原成銨作用等過程中的微生物功能基因特性,對掌握氮循環過程、提高氮素可利用性以及控制溫室氣體排放等意義重大[3]。
由固氮酶催化進行的生物固氮作用可以將大氣中的氮氣(N2)還原為生物可利用的銨態氮形式,因而是氮素進入土壤、植物等陸地生態系統中的重要途徑之一。固氮酶廣泛存在于固氮菌屬(Azotobacter)、固氮螺菌屬(Azospirillum)和中華根瘤菌屬(Sinorhizobium)等微生物中,主要由2個多亞基的金屬蛋白酶組成。nifD、nifK基因編碼的成分Ⅰ是由2個異二聚體構成的250 kDa蛋白組分,包括N2還原的活性位點。成分Ⅱ(約70 kDa)由nifH基因編碼,能夠將三磷酸腺苷(adenosine triphosphate,ATP)水解與電子轉移過程偶聯。Fe-S中心在2個蛋白組分中共同存在,并且介導著固氮酶的生化代謝過程[4]。由于nifD、nifK基因序列相對較少,限制了對固氮酶多樣性的系統研究,因此nifH基因常被用作研究固氮微生物的系統進化分析要素,為探索固氮微生物的種類和群落分布提供了重要依據[3,5](表1)。
反硝化作用是土壤中N2O最重要的產生途徑,微生物可利用硝酸根作為電子受體,將其呼吸還原為氣態產物,具體途徑為:NO3-→NO2-→NO→N2O→N2,反應過程逐級連續,涉及的關鍵酶包括:硝酸鹽還原酶(nitrate reductase,Nar),亞硝酸還原酶(nitrite reductase,Nir),一氧化氮還原酶(nitric oxide reductase,Nor),氧化亞氮還原酶(nitrous oxide reductase,Nos)等,分別由各自對應的功能基因(narG/napA,nirK/nirS,norC/norB,nosZ/nosR/nosD)所編碼[6-8](表1)。硝酸鹽還原酶可以催化反硝化過程的第一步,根據其附著位置不同,主要分為膜結合型硝酸鹽還原酶(Nar)和周質硝酸鹽還原酶(periplasmic nitrate reductase,Nap)。膜結合型硝酸鹽還原酶(Nar)由α、β、γ 3個亞基構成,分別由narG、narH和narI基因編碼;可溶性的周質硝酸鹽還原酶(Nap)是由napA和napB基因編碼的異質二聚體結構[6]。其中narG和napA基因是研究硝酸鹽還原過程最重要和使用最廣泛的功能基因[3]。nirK和nirS基因也是反硝化微生物群落研究中普遍使用的分子標志物,可分別編碼含銅(Cu)的亞硝酸鹽還原酶(NirK)和細胞色素cd1型亞硝酸鹽還原酶(NirS)[3,9]。由norB和nosZ分別編碼的一氧化氮還原酶(Nor)和氧化亞氮還原酶(Nos)同樣在反硝化過程中發揮著重要作用[10-11]。
中國乳制品進口價格的由乳制品的進口數量和乳制品的進口金額獲得。其選取的樣本區間為2006—2016年,記作ln P。
異化硝酸鹽還原成銨的過程主要為:在硝酸鹽還原酶(Nar)的催化作用下,NO3-還原成NO2-,再由亞硝酸還原酶(Nir)催化還原為NH4+[12-13],其還原酶對應的功能基因分別為(narG/napA,nirB/nirC/cysG)(表1)。盡管該過程與反硝化過程具有相同的硝酸鹽還原酶,但在亞硝酸鹽還原酶的結構和分類上(該過程中的亞硝酸鹽還原酶主要可分為溶解性酶和胞外酶2類)有明顯區別[14-15]。
針對氮還原轉化過程中功能基因多樣性的研究開展較早,同時也最為全面,在不同生態系統和農藝措施管理過程下相關微生物群落變化等研究已較為普遍。基于nirK、nirS、nosZ、nifH等功能基因靶標的分子生物學研究,對探索功能微生物的分布規律、代謝過程以及與土壤氮素循環之間的關系等方面的研究具有重要的指導意義。

表1 土壤中氮還原轉化過程的主要功能基因[2-3,5-13]Table 1 Main functional genes involved in soil nitrogen reduction[2-3,5-13]
2 硫酸鹽還原過程
硫酸鹽還原過程是地球上最古老的呼吸形式之一,也是物質循環的重要組成部分。在自然條件下,硫氧化形成的硫酸鹽一部分可以被植物和微生物吸收利用,另一部分則可在厭氧條件下參與由硫酸鹽還原菌介導的異化硫酸鹽還原過程(形成H2S)或同化還原過程(形成有機硫化物),從而成為構建硫元素循環的關鍵過程[16]。
異化硫酸鹽還原過程是一個復雜的生物化學反應,其本質是由微生物的多種蛋白組分(如Dsr、Qmo、Hmc、Tmc、Qrc、Nuo和Rnf)催化和介導的電子傳遞過程。作為一種穩定的化學成分,SO42-首先要在ATP硫酸化酶和APS還原酶(APS reductase,Apr)的作用下獲得電子轉化為SO32-,再在異化型亞硫酸鹽還原酶(dissimilatory sulfite reductase,Dsr)的催化作用下經過一系列電子轉移過程,最終還原形成S2-[17](圖1)。ATP硫酸化酶是一種含有鈷(Co)和鋅(Zn)的金屬蛋白,由sat基因編碼,同時存在于硫酸鹽同化和異化還原過程中,是活化硫酸鹽的重要物質。aprBA基因編碼的APS還原酶(Apr)是一種重要的可溶性細胞質黃素蛋白,包含2種不同形式的[4Fe-4S]簇和黃素腺嘌呤二核甘酸,廣泛存在于所有的硫酸鹽還原微生物以及無機營養型的硫化物氧化細菌中。異化型亞硫酸鹽還原酶(Dsr)是硫酸鹽還原過程中最為關鍵的功能蛋白之一,由α2β2結構組成,2個亞基分別被鄰近的dsrA和dsrB基因所編碼表征[17](圖1)。dsr基因在同一類群的硫酸鹽還原菌中相當保守,但在不同類群的硫酸鹽還原菌中卻存在著較大的差別[18-20]。NERETIN等[21]通過純培養實驗發現,硫酸鹽還原微生物中dsrAB基因的mRNA水平隨著硫酸鹽還原速率的增加而上升,這表明環境中的硫酸鹽還原速率可以從dsr轉錄水平來進行評價。因此,dsr基因作為應用最為廣泛的、可用于追蹤參與調控硫循環功能微生物的關鍵分子標志物,對于研究硫酸鹽還原菌的微生物群落結構和功能特性具有重要作用[22-24]。
經過對 Desulfococcus multivorans、Desulfococcus oleovorans、Desulfovibriovulgaris、Desulfobacterium autotrophicum、Desulfobacula toluolica 以 及Desulfatibacillum alkenivorans等多種硫酸鹽還原微生物的宏基因組測序[17,25-28],發現其中的dsr操縱子包含多種編碼基因,即dsrABEFHCMKLJOPNRS等,可表征不同功能的蛋白質組分,從而參與硫的氧化還原過程[25]。在這些基因產物中,DsrAB蛋白復合物是硫循環代謝途徑中不可或缺的功能物質,不僅是催化異化型亞硫酸鹽還原過程的關鍵酶,也是硫化物氧化過程的重要參與者[29]。而其他蛋白物質,如DsrC,可作為氧化還原中心調控硫的代謝過程,協助DsrAB發揮相關作用,并介導DsrMKJOP蛋白復合物行使與DsrAB相似的功能特性[25,30]。除此之外,微生物中的其他多種蛋白組分也在硫酸鹽還原過程中發揮著舉足輕重的作用。由qmoABC基因編碼的QmoABC蛋白復合物可作為電子傳遞通道從細胞膜向AprAB輸送電子;Hmc、Tmc和Qrc蛋白參與細胞質與周質間的電子傳遞過程,與脫氫酶等功能物質的代謝過程息息相關;同時,由相關基因編碼的Nuo、Rnf和Hdr等蛋白組分也在電子傳遞、能量轉化、過程耦合等方面扮演著重要角色[17]。
除了異化硫酸鹽還原過程,微生物還可以在同化型亞硫酸鹽還原酶(sulfite reductases,SirA)的作用下吸收硫酸鹽形成有機硫化物[31-32]。目前,依托基因敲除、代謝篩選、酶組學和宏基因組學等分子生物學的相關技術,針對大腸桿菌、枯草桿菌、棒狀桿菌、分枝桿菌、鏈霉菌以及放線菌等微生物已經進行了較為詳盡的代謝途徑的闡述[31-33]。sirA、aprBA、fpr2-cysIXHDNYZ基因簇等功能基因編碼了同化硫酸鹽還原過程的大量相關的蛋白酶,如同化型亞硫酸鹽還原、APS還原酶和半胱氨酸合成酶等,從而參與了無機硫化物還原中的重要步驟[32-33]。
硫酸鹽還原過程常常伴隨著土壤環境中多種氧化還原反應,如鐵循環、污染物的還原脫鹵、產甲烷及其厭氧氧化等過程,不同的蛋白質復合體(Qmo、Apr、Hdr等)及其功能基因也常常被用于共同研究硫酸鹽還原菌的豐度及功能特性。

圖1 異化硫酸鹽還原代謝途徑[17]Fig.1 Metabolic schemes associated with dissimilatory sulfate reduction[17]
3 鐵還原過程
在自然環境中,鐵作為最豐富、最活躍的變價金屬元素,以多種化合價形式(如0、+2、+3、+6等)存在于各種氧化還原反應中,廣泛參與地球生物化學循環。由微生物介導的異化鐵還原過程作為最主要的鐵還原途徑之一,其本質是一種電子傳遞過程[34-37]。在厭氧條件下,異化鐵還原菌能夠以Fe(Ⅲ)作為外在電子受體,將其還原成Fe(Ⅱ),并通過該過程獲取能量,滿足自身生長發育需要[37]。
現有的研究重點針對微生物的Fe(Ⅲ)呼吸代謝途徑展開,發現多種細胞色素蛋白質和相關功能酶等物質參與了電子轉移和呼吸調節等過程。SAFFARINI等[38]研究發現甲基萘醌(menaquinones,MQ)是介導Fe(Ⅲ)呼吸的一個必要構件,承擔著從脫氫酶中傳遞電子的重要角色,并在其他多種功能蛋白的作用下依次完成電子向周質細胞色素、細胞膜及Fe(Ⅲ)氧化物的傳遞過程。由cymA基因編碼的CymA是錨定在細胞質膜上的可驅動電子從醌類(MQ)中間體到周質細胞色素的電子傳遞過程的四血紅素周質細胞色素[39-40]。而四血紅素黃素細胞色素Ifc3和小分子質量的酸性低電勢的四血紅素細胞色素Cct也可作為輔助途徑從CymA中接遞電子[41-43]。隨后,由mtrA基因編碼的具有非特異電子傳遞作用和完整細胞Fe(Ⅲ)末端還原酶本質的MtrA(周質十血紅素細胞色素),可完成電子從周質向細胞外膜的傳遞過程[44-45],并最終在Fe(Ⅲ)末端還原酶的催化作用下,將位于細胞外膜的電子傳遞給 Fe(Ⅲ)[46]。
在整個Fe(Ⅲ)呼吸代謝過程中,位于細胞外膜(少量在周質)的多血紅素細胞色素大多具有非特異的Fe(Ⅲ)還原酶的特性,主要依賴與Fe(Ⅲ)之間存在的電勢差,發揮調控與催化Fe(Ⅲ)還原、構建電子傳遞網絡的重要作用[47-48]。從Shewanella oneidensis SR-21中獲取的mtrDEF-omcA-mtrCAB基因簇[45-46],可編碼3個外膜十血紅素細胞色素(MtrC、MtrF和OmcA),2個周質十血紅素細胞色素(MtrD和MtrA),以及2個可能的β-barre蛋白(MtrE和MtrB),參與Fe(Ⅲ)還原呼吸的多條代謝途徑[49]。其中,mtrB作為最早發現的Fe(Ⅲ)還原相關的功能基因之一,其編碼產物MtrB(十血紅素c型細胞色素)在Fe(Ⅲ)還原蛋白的正確定位和插入外膜等方面起著重要作用[44]。此外,由ferE基因編碼的鐵吸收調節蛋白(ferric uptake regulator,Fur)是鐵捕獲系統的負調控子,在細菌Fe(Ⅲ)呼吸代謝途徑中起到了關鍵的調控作用,ferE基因缺失將導致還原過程所需的分子質量為91 kDa的鐵卟啉蛋白無法正常分泌到細胞外膜,從而抑制了Fe(Ⅲ)的還原代謝過程[48-49]。
近年來,隨著相關技術手段的持續發展,針對微生物Fe(Ⅲ)還原過程的代謝機制已經得到了進一步的完善。然而從目前已分離得到的鐵還原菌中尚未發現1個通用的功能基因。典型的鐵還原菌[如希瓦式菌(Shewanella)、地桿菌(Geobacter)等]中雖然都以細胞色素c作為主要的電子傳遞蛋白,但關鍵功能物質的同源性并不強[50],因此,將某個功能基因作為分子標志物來表征異化型鐵還原菌的微生物代謝特性和分布規律等方面的系統發育分析仍需要進行深入的研究和探索。
4 產甲烷過程
甲烷(CH4)是重要的溫室氣體之一,其溫室效應是二氧化碳(CO2)的20余倍,凈釋放量大約500 Tg/a,并且以每年約1%的速度增長[51-53]。因此,研究甲烷的產生途徑、減少甲烷氣體排放對于緩解全球氣候變暖、維持生態環境平衡具有至關重要的作用。
目前已知的產甲烷菌主要來自廣古菌門,根據碳代謝過程的不同,主要包括CO2還原、甲基裂解和乙酸發酵3種產甲烷途徑,其本質是一種微生物介導、多種功能蛋白質參與的電子傳遞過程。其中,甲烷呋喃(methanofuran,MFR)、鐵氧化還原蛋白(ferredoxin,Fd)、細胞色素(cytochrome)和輔酶F420等是電子傳遞的重要載體。
甲基輔酶M還原酶(methyl coenzyme M reductase,Mcr)可以在碳代謝的最后一步以輔酶B(HS-CoB)為直接的電子供體還原甲基輔酶M(CH3-S-CoM)產生甲烷,是3種代謝過程中共有的關鍵功能酶(圖2),由3個不同的亞基(α2β2γ2)構成的六聚體結構和含鎳輔酶F430組成[54-56]。編碼Mcr中α亞基的功能基因(mcrA)作為分子標志物,被廣泛應用于產甲烷古菌群落結構及多樣性等方面的表征研究[57]。而隨著近年來技術的發展和對產甲烷微生物研究的深入,在非廣古菌門的微生物中也發現了編碼Mcr以及其他與產甲烷相關的功能基因[58]。EVANS等[58]通過宏基因組測序,從地下含水層中篩選得到了2株屬于深古菌門的產甲烷古菌,并獲得了編碼甲基輔酶M還原酶的功能基因。其中:BA1菌株中還包含有 mtsA、mtbA、mtaA、mttBC、mtbBC 和mtrH等與產甲烷途徑相關的功能基因;BA2菌株中也存在編碼甲基裂解途徑相關的基因,但是其能量儲存和電子傳遞機制可能不同于BA1。該發現對于深入了解產甲烷微生物的分布與代謝機制,拓展對功能基因調控的甲烷產生機制的研究具有重要意義。同時,Mcr及其同系物也是甲烷厭氧氧化過程中的關鍵功能酶,在全球甲烷代謝過程中發揮著重要作用。異質二硫化物還原酶(heterodisulfide reductase,Hdr)同樣是產甲烷最后關鍵步驟中共有的功能物質,它能以CoB-S-S-CoM作為產甲烷過程的末端電子受體,將其還原為HS-CoB和HS-CoM(圖2)。Hdr是一種膜相結合的、具有電子傳遞功能的蛋白復合物,由hdrABCDE基因編碼產生。根據微生物細胞生長條件的不同,不同Hdr功能酶(HdrED和HdrABC)在利用傳遞電子、ATP合成及還原代謝過程中發揮著特有的生理功能[59-60]。
輔酶M甲基轉移酶(coenzyme M methyltransferase,Mtr)是CO2還原代謝過程中的重要功能酶,可以催化甲基基團轉移到輔酶M(HS-CoM)的硫醇基上形成CH3-S-CoM的過程,同時充當鈉離子泵的角色,用于合成ATP[61-62](圖2)。Mtr是由8個亞基組成的嵌膜蛋白復合體,由mtrECDBAFGH基因所編碼[63]。其中:含有1個鈷胺素(Ⅰ)輔基的MtrA亞基能夠在催化循環中不斷進行甲基化和去甲基化的循環,并在該過程中產生鈉離子梯度[62,64];亞基MtrE能夠將MtrA中類咕啉輔基上的甲基基團轉移到輔酶M的巰基上[62-63];而MtrH亞基則能夠利用CH3-H4MPT催化自由鈷胺素(Ⅰ)輔基轉變為甲基化的鈷胺素(Ⅰ)輔基[62,65]。
輔酶M甲基轉移酶復合體(coenzyme M methyl transfer,MT)是催化和激活甲基裂解途徑的酶系統,根據底物利用和代謝途徑不同,主要包括MT1(MtaB、MtmB、MtbB和MttB)和MT2(MtaA、MtbA和MtsA)2個轉移酶,分別由相應的轉移酶基因(mta、mtm、mtb、mtt和mts等)編碼表達[66]。MT1能夠將底物上的甲基轉移到MT1上與類咕啉蛋白結合,MT2再轉移甲基與HS-CoM連接產生CH3-S-CoM(圖2)。MT2蛋白序列中都含有高度保守的結構,可以完成甲基化合物的激活和甲基的轉移,并同時伴隨著電子傳遞和能量代謝等過程[66-67]。
一氧化碳脫氫酶/乙酰輔酶A合成酶復合體(CO dehydrogenase/acetyl-CoA synthase,Codh/Acs)是解乙酸途徑中的關鍵酶,用于剪切乙酰輔酶A上的C-C和C-S鍵(圖2)。它能以Fdox作為電子受體,催化產生CO2和CH3-H4SPT。同時,甲烷呋喃(MFR)、輔酶F420和F430、鐵氧化還原蛋白(Fd)等也可作為氫化酶系統的電子載體,參與到產甲烷的呼吸代謝過程中[62,68]。
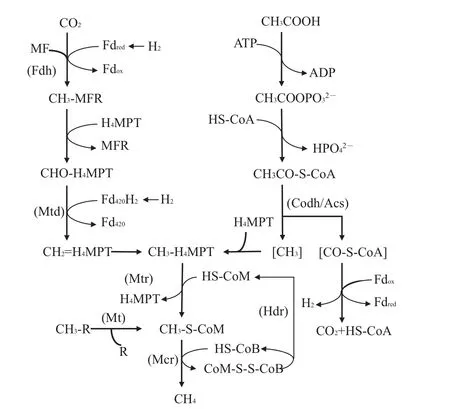
圖2 產甲烷的生化代謝途徑[62,68]Fig.2 Biochemical pathways of methanogenesis[62,68]
甲烷產生的代謝過程體系復雜,并伴隨著多種功能蛋白的偶聯,因而不同的輔酶系統以及電子傳遞載體相互交叉,共同促成了甲烷的產生。現如今各國對于產甲烷過程相關的研究愈發深入,隨著基因組學和生物信息學等新技術的發展,以功能基因作為分子靶標的研究,對人們深入理解產甲烷過程相關的代謝途徑和過程調控機制具有重要意義。
5 鹵代有機污染物的還原脫鹵過程
鹵代有機污染物是全球范圍內普遍使用的,具有難降解、高致癌性的芳香族或脂肪族化合物[69]。在厭氧條件下,許多微生物可以在還原脫鹵酶的催化作用下以鹵代有機污染物作為電子受體進行還原,自身獲得能量進行生長[70]。不同的還原脫鹵酶具有相同的脫鹵功能,但由于鹵代有機污染物種類繁多以及相關功能微生物的多樣性,編碼相關還原脫鹵酶的具體功能基因也不盡相同。基于現代分子生物學技術,以還原脫鹵功能基因作為高效的生物標志物,可以用來詳細研究環境中微生物的脫鹵呼吸過程[71-72]。
目前,各國學者已經通過對Sulfurospirillum、Desulfitobacterium、 Dehalobacte、 Dehalococcoides、Anaeromyxobacter和其他多種鹵呼吸微生物進行了詳盡的全基因組測序和宏基因組學研究,發現了大量具有還原脫鹵功能的基因(rdh)。rdh基因簇中包含可編碼具有催化活性的還原脫鹵酶的rdhA基因,編碼膜固蛋白的rdhB基因,以及其他與還原脫鹵相關的rdhTKZECD基因等[70,73-74]。由rdh編碼的RdhA蛋白具有幾個特殊的保守特征,包括2個Fe-S簇結合基序和可用于易位或跨越細胞膜的雙精氨酸信號基序[74]。大多數RdhA蛋白還包含著類咕啉的輔酶因子[75]。根據可利用的污染底物不同,還原脫鹵酶RdhA也包含了多種具有催化脫鹵功能的不同的蛋白組分,如可利用氯乙烯的VcrA、BvcA、TceA、MbrA、PceA和PrdA[76-81],還原氯乙烷的CfrA、DcrA和DcaA[82-84],催化氯代芳烴的CbrA、CprA和CrdA[85-87],以及可利用氯苯的PcbA[88]等,不同的還原脫鹵酶均由相關特定的功能基因所編碼。
最早發現的rdhA基因序列是Sulfurospirillum multivorans中編碼三氯乙烯還原酶的pceA基因[73],Desulfitobacterium dehalogenans中編碼氯酚還原酶的cprA基因[88],及其相鄰的pceB和cprB基因。隨著現代分子生物學技術的發展,經過長期的研究總結發現,在 Desulfitobacterium dehalogenans、D.hafniense、Desulfitobacterium chlororespirans、D.dichloroeliminans、Sulfurospirillum multivorans、Dehalobacter restrictus和Dehalococcoides mccartyi等微生物中包含著cprA、crdA、pceA、tmrA、vcrA、cbrA、mbrA和pcbA等與轉錄調節相關的功能基因簇,可編碼特定鹵代有機污染物的還原脫鹵過程中的蛋白酶[69,89-93]。
通過轉錄組和蛋白組學等技術方法可基于特定的功能基因分析對應的還原脫鹵酶,從而研究鹵代有機污染物的逐級還原過程。BISAILLON等[85]發現:在Desulfitobacterium hafniense PCP-1中的CprA3還原脫氯酶可以通過鄰位脫氯,將PCP依次還原為四氯或三氯的物質;而另一種CprA5還原脫氯酶,則可以完成間位或者對位脫氯,將3,4,5-TCP還原為3,5-DCP,并繼續將其脫氯轉化為3-CP[94]。而編碼CprA的cprA基因同樣也可在Desulfitobacterium hafniense TCP-A和DCB-2菌株中發現[95]。5種在Dehalococcoides得到表征的還原脫鹵酶可廣泛參與到氯乙烯、氯乙烷和氯苯等的呼吸代謝過程中。其中:PceA可將PCE脫氯為TCE[74];TceA能夠還原TCE為乙烯[96];VcrA可催化TCE、DCE異構體和氯乙烯(VC)轉化為乙烯[97]。Dehalococcoidesides mccartyi CBDB1中的CbrA可催化完成1,2,3,4-四氯苯和1,2,3-三氯苯的還原脫氯過程[86];而BvcA也可進行TCE、DCE的一種異構體,以及VC和1,2-DCA的還原脫氯過程[83]。由此可知,在特定的微生物基因組中可能不僅包含一種還原脫鹵功能基因,而且由某種基因編碼的還原脫鹵酶也可能會參與多種底物的代謝過程。因而,基于測序技術研究微生物的還原脫鹵過程,不僅能夠從基因水平豐富污染物的降解代謝體系,而且對于鑒定污染地區具有還原脫鹵作用的微生物種類、促進有機污染的消減具有重要意義。
6 厭氧土壤中各氧化還原過程的耦合關系
厭氧微生物催化完成了土壤環境中大量的氧化還原反應,而在此過程中產生的能量又可以驅動多種生源要素的轉化。其中,C、N、Fe、S等重要生命元素的轉化及污染物的還原脫鹵過程之間互相耦合,彼此影響,從而共同構成了土水界面復雜的生物地球化學循環體系。
由厭氧微生物驅動的反硝化過程能夠偶聯多種有機和無機化合物的氧化還原反應,并顯著促進CN,Fe-N和S-N等的耦合關系[98-100]。ROBERTSON等[101]通過化學計量統計和同位素標記的方法證明了自養型鐵氧化及硝酸鹽還原微生物的存在,該類微生物在催化完成NO3-還原過程的同時,可以耦合Fe(Ⅱ)氧化。盡管如此,相關佐證催化Fe(Ⅱ)氧化NO3-還原途徑的遺傳和酶學證據仍然缺乏[102]。此外,發現硝酸鹽/亞硝酸鹽還原菌在厭氧條件下還能夠以硫化物作為電子供體,耦合N/S氧化還原反應。研究表明,硝酸鹽/亞硝酸鹽還原耦合硫的氧化還原過程在淡水、沉積物以及厭氧土壤等各種環境中起著重要作用[103-105]。
土壤中異化型鐵還原菌還原Fe(Ⅲ)的過程可以偶聯多種有機物的氧化還原反應。在厭氧環境中,Fe(Ⅲ)和硫酸鹽還原微生物常以互惠共生的關系存在,共同介導Fe/S的氧化還原過程,而其反應的熱力學順序取決于底物的可利用性、產物濃度和pH條件等。還原產物,如游離Fe2+和HS-等的濃度可能會因為形成FeS沉淀而降低,從而推動Fe(Ⅲ)和硫酸鹽的還原[106]。此外,在堿性環境下,由硫酸鹽還原產生的S0可以作為一種電子穿梭體介導微生物 Fe(Ⅲ)的還原轉化[107]。
除此之外,在厭氧條件下的還原脫氯過程也可以耦合其他多種元素,如NO3-、Fe(Ⅲ)、SO42-、CH4等的還原代謝反應。以嚴格厭氧的非專性脫氯菌Desulfitobacterium為例,它可以利用硝酸鹽、亞硫酸鹽、金屬、腐殖酸、鹵代有機化合物等多種物質作為電子受體,促進自身生長并參與其他元素的氧化還原反應[108]。其中,cprBA和pceAB基因是轉錄還原脫氯蛋白的重要物質,同時cprCDEKTZ,pceCT,prdAB等基因也在多株微生物中介導脫氯代謝的關鍵過程。有研究表明,D.hafniense TCE1可以和硫酸鹽還原菌Desulfovibrio fructosivorans在低硫酸鹽濃度和果糖存在的條件下形成互養共生體系,并通過種間氫傳遞來獲取電子,從而促進PCE的還原脫氯[109-110],并推測其同樣適用于與產甲烷菌的共生模式。NIGGEMYER等[111]從富鐵沉積物中分離并鑒定得到的D.hafniense GBFH能夠以甲酸鹽作為電子供體和碳源,還原Fe(Ⅲ)、SO42-及其他多種物質。同時,D.dehalogenans、D.chlororespirans、D.hafniense PCP-1、DCB-2、TCE1和G2等多株菌在特定條件下也可以促進 Fe(Ⅲ)還原[108]。此外,Anaeromyxobacter dehalogenans 2CP-C可以利用Fe(Ⅲ)作為電子受體,并通過鄰位脫氯過程獲得能量生長[112]。Desulfuromonas chlorethenic可以同時完成Fe(Ⅲ)還原和四氯乙烯及三氯乙烯的還原脫氯過程[113]。典型的異化鐵還原菌Shewanella和Geobacter能耦合Fe(Ⅲ)還原和氯代有機化合物的還原脫氯過程[114-116]。而一些具有硫酸鹽還原功能的微生物如Desulfovibrio、Desulfotomaculum、Clostridium等也可能具有還原脫氯功能,這些微生物能夠通過呼吸作用代謝多氯聯苯(PCBs)、二氯二苯三氯乙烷(DDT)等氯代有機污染物,并從中獲取自身生長所需能量[117-119]。
與此同時,多種功能酶或細胞色素等蛋白組分在能量代謝和電子轉移等過程中均發揮著重要的調控作用,如mtr基因簇的編碼產物在產甲烷過程、Fe(Ⅲ)還原過程的能量合成與電子傳遞過程中具有關鍵作用;cys基因簇在介導硝酸鹽還原、硫酸鹽還原等循環代謝過程中扮演著關鍵角色;hdr基因能夠耦合硫酸鹽還原過程與產甲烷過程;dsr基因簇參與硫酸鹽還原等過程,其產物介導的還原過程與氮的還原轉化、產甲烷、有機污染物的還原脫鹵以及Fe(Ⅲ)還原過程等相互耦合,在電子競爭與物質代謝方面關系密切;此外,涉及ATP合成及轉化的功能基因,如sat、apr、mtr等,以及在細胞周質和細胞膜等成分間進行電子傳遞的功能基因,如qmo、hmc、cym等,均在微生物的多條呼吸代謝途徑中起著重要作用[27-28]。因此,通過標記功能基因的方法,可以更加直觀地探究和分析特定氧化還原過程中微生物的功能特性和分布特征。
7 結論與展望
微生物介導著厭氧環境中的各種氧化還原反應,一方面從反應中獲得能量促進自身生長,另一方面又驅動著生源要素的生物地球化學循環過程,這些過程由于微生物的參與而相互耦合,共同構成了復雜而又活躍的土壤體系。盡管我們綜述了土壤各典型還原過程中的功能基因、物質轉化間的耦合關系,以及與鹵代有機污染物的還原脫鹵過程的結合等,但不可否認土壤環境中的實際情況遠遠超出現有的研究對其功能和復雜程度的理解。
目前關于C、N、S、Fe等生源要素代謝過程及其對環境影響的研究依然是國際熱點之一。隨著分子生物學技術的發展,由微生物主導的多介質、多界面、多要素、多過程、多尺度的耦合研究是探究生物地球化學循環過程的最新的科學前沿[120]。編碼環境中NO3-、Fe(Ⅲ)、SO42-和鹵代有機污染物的還原和產甲烷過程中特定功能酶基因在不斷地被完善,大大促進了對于元素生物地球化學循環過程的認知,推動了環境微生物生態學的縱深發展。因此,借助生物化學和生物信息學的蓬勃發展,廣泛而深入地解析不同代謝過程中的功能蛋白及其編碼基因,完善微生物的環境功能基因多樣性,加強對元素循環機制的認識,促進污染修復和環境改善是該領域未來研究的重要內容。
[1] YERGEAU E,KANG S,HE Z L,et al.Functional microarray analysis of nitrogen and carbon cycling genes across an Antarctic latitudinal transect.The ISME Journal,2007,1(2):163-179.
[2] 張晶,林先貴,尹睿.參與土壤氮素循環的微生物功能基因多樣性研究進展.中國生態農業學報,2009,17(5):1029-1034.ZHANG J,LIN X G,YIN R.Advances in functional gene diversity of microorganism in relation to soil nitrogen cycling.Chinese Journal of Eco-Agriculture,2009,17(5):1029-1034.(in Chinese with English abstract)
[3] LEVY-BOOTH D J,PRESCOTT C E,GRAYSTON S J.Microbial functional genes involved in nitrogen fixation,nitrification and denitrification in forest ecosystems.Soil Biology&Biochemistry,2014,75:11-25.
[4] ZEHR J P,JENKINS B D,SHORT S M,et al.Nitrogenase gene diversity and microbial community structure:A cross-system comparison.Environmental Microbiology,2003,5(7):539-554.
[5] 侯海軍,秦紅靈,陳春蘭,等.土壤氮循環微生物過程的分子生態學研究進展.農業現代化研究,2014,35(5):588-594.HOU H J,QIN H L,CHEN C L,et al.Research progress of the molecular ecology on microbiological processes in soil nitrogen cycling.Research of Agricultural Modernization,2014,35(5):588-594.(in Chinese with English abstract)
[6] PHILIPPOT L.Denitrifying genes in bacterial and Archaeal genomes.Biochimica et Biophysica Acta-Gene Structure and Expression,2002,1577(3):355-376.
[7] 王瑩,胡春勝.環境中的反硝化微生物種群結構和功能研究進展.中國生態農業學報,2010,18(6):1378-1384.WANG Y,HU C S.Research advances on community structure and function of denitrifiers.Chinese Journal of Eco-Agriculture,2010,18(6):1378-1384.(in Chinese with English abstract)
[8] 楊杉,吳勝軍,蔡延江,等.硝態氮異化還原機制及其主導因素研究進展.生態學報,2016,36(5):1224-1232.YANG S,WU S J,CAI Y J,et al.The synergetic and competitive mechanism and the dominant factors of dissimilatory nitrate reduction processes:A review.Acta Ecologica Sinica,2016,36(5):1224-1232.(in Chinese with English abstract)
[9] BRAKER G,FESEFELDT A,WITZEL K P.Development of PCR primer systems for amplification of nitrite reductase genes(nirK and nirS)to detect denitrifying bacteria in environmental samples.Applied and Environmental Microbiology,1998,64(10):3769-3775.
[10]BRAKER G,TIEDJE J M.Nitric oxide reductase(norB)genes from pure cultures and environmental samples.Applied and Environmental Microbiology,2003,69(6):3476-3483.
[11]SCALA D J,KERKHOF L J.Nitrous oxide reductase(nosZ)genespecific PCR primers for detection of denitrifiers and three nosZ genes from marine sediments.FEMS Microbiology Letters,1998,162(1):61-68.
[12]STEVENS R J,LAUGHLIN R J.Measurement of nitrous oxide and di-nitrogen emissions from agricultural soils.Nutrient Cycling in Agroecosystems,1998,52(2/3):131-139.
[13]STEVENS R J,LAUGHLIN R J,MALONE J P.Soil pH affects the processes reducing nitrate to nitrous oxide and di-nitrogen.Soil Biology and Biochemistry,1998,30(8/9):1119-1126.
[14]殷士學.淹水土壤中硝態氮異化還原成銨過程的研究.南京:南京農業大學,2000:55-56.YIN S X.Dissimilatory nitrate reduction to ammonium in submerged soils.Nanjing:Nanjing Agricultural University,2000:55-56.(in Chinese with English abstract)
[15]COLE J A.Physiology,biochemistry and genetics of nitrate dissimilation to ammonia//REVSBECH N P,SORENSEN J.Denitrification in Soil and Sediment.USA:Springer,1990:57-76.
[16]劉新展,賀紀正,張麗梅.水稻土中硫酸鹽還原微生物研究進展.生態學報,2009,29(8):4455-4463.LIU X Z,HE J Z,ZHANG L M.The sulfate-reducing bacteria and surfer cycle in paddy soil.Acta Ecologica Sinica,2009,29(8):4455-4463.
[17]D?RRIES M,W?HLBRAND L,KUBE M,et al.Genome and catabolic subproteomes of the marine,nutritionally versatile,sulfate-reducing bacterium Desulfococcus multivorans DSM 2059.BMC Genomics,2016,17:918.
[18]PéREZ-JIMéNEZ J R,YOUNG L Y,KERKHOF L J.Molecular characterization ofsulfate-reducing bacteria in anaerobic hydrocarbon-degrading consortia and pure cultures using the dissimilatory sulfite reductase(dsrAB)genes.FEMS Microbiology Ecology,2001,35(2):145-150.
[19]WAGNER M,ROGER A J,FLAX J L,et al.Phylogeny of dissimilatory sulfite reductases supports,an early origin of sulfate respiration.Journal of Bacteriology,1998,180(11):2975-2982.
[20]CHIN K J,SHARMA M L,RUSSELL L A,et al.Quantifying expression of a dissimilatory(bi)sulfite reductase gene in petroleumcontaminated marine harbor sediments.Microbial Ecology,2008,55(3):489-499.
[21]NERETIN L N,SCHIPPERS A,PERNTHALER A,et al.Quantification of dissimilatory(bi)sulphite reductase gene expression in Desulfobacterium autotrophicum using real-time RT-PCR.Environmental Microbiology,2003,5(8):660-671.
[22]PETRI R,PODGORSEK L,IMHOFF J F.Phylogeny and distribution of the soxB gene among thiosulfate-oxidizing bacteria.FEMS Microbiology Letters,2001,197(2):171-178.
[23]TOUROVA T P,SLOBODOVA N V,BUMAZHKIN B K,et al.Analysis of community composition of sulfur-oxidizing bacteria in hypersaline and soda lakes using soxB as a functional molecular marker.FEMS Microbiology Ecology,2013,84(2):280-289.
[24]ZHENG Y,BU N S,LONG X E,et al.Sulfate reducer and sulfur oxidizer respond differentially to the invasion of Spartina alterniflora,in estuarine salt marsh of China.Ecological Engineering,2017,99:182-190.
[25]GHOSH S,BAGCHI A.Comparative analysis of the mechanisms of sulfur anion oxidation and reduction by dsr operon to maintain environmental sulfur balance.Computational Biology and Chemistry,2015,59:177-184.
[26]STRITTMATTER A W,LIESEGANG H,RALF R,et al.Genome sequence of Desulfobacterium autotrophicum HRM2,a marine sulfate reducer oxidizing organic carbon completely to carbon dioxide.Environmental Microbiology,2009,11(5):1038-1055.
[27]W?HLBRAND L,JACOB J H,KUBE M,et al.Complete genome,catabolic sub-proteomes and key-metabolites of Desulfobacula toluolica Tol2,a marine,aromatic compound-degrading,sulfatereducing bacterium.Environmental Microbiology,2013,15(5):1334-1355.
[28]CALLAGHAN A V,MORRIS B E L,PEREIRA I A,et al.The genome sequence of Desulfatibacillum alkenivorans AK-01:A blueprint for anaerobic alkane oxidation.Environmental Microbiology,2012,14(1):101-113.
[29]LOY A,DULLER S,BARANYI C,et al.Reverse dissimilatory sulfite reductase as phylogenetic marker for a subgroup of sulfuroxidizing prokaryotes.Environmental Microbiology,2009,11(2):289-299.
[30]VENCESLAU S S,STOCKDREHER Y,DAHL C,et al.The“bacterial heterodisulfide”DsrC is a key protein in dissimilatory sulfurmetabolism.Biochimica etBiophysica Acta (BBA)-Bioenergetics,2014,1837(7):1148-1164.
[31]FISCHER M,SCHMIDT C,FALKE D,et al.Terminal reduction reactions of nitrate and sulfate assimilation in Streptomyces coelicolor,A3(2):Identification of genes encoding nitrite and sulfite reductases.Research in Microbiology,2012,163(5):340-348.
[32]PINTO R,HARRISON J S,HSU T,et al.Sulfite reduction in mycobacteria.Journal of Bacteriology,2007,189(18):6714-6722.
[33]RüCKERT C,KOCH D J,REY D A,et al.Functional genomics and expression analysis of the Corynebacterium glutamicum fpr2-cysIXHDNYZ,gene cluster involved in assimilatory sulphate reduction.BMC Genomics,2005,6:121.
[34]FAUQUE G D,BARTON L L.Hemoproteins in dissimilatory sulfate-and sulfur-reducing prokaryotes.Advances in Microbial Physiology,2012,60:1-90.
[35]DING L J,SU J Q,XU H J,et al.Long-term nitrogen fertilization of paddy soil shifts iron-reducing microbial community revealed by RNA-13C-acetate probing coupled with pyrosequencing.The ISME Journal,2015,9(3):721-734.
[36]LI H J,PENG J J,WEBER K A,et al.Phylogenetic diversity of Fe(Ⅲ)-reducing microorganisms in rice paddy soil:Enrichment cultures with different short-chain fatty acids as electron donors.Journal of Soils and Sediments,2011,11:1234.
[37]WEBER K A,ACHENBACH L A,COATES J D.Microorganisms pumping iron:Anaerobic microbial iron oxidation and reduction.Nature Reviews Microbiology,2006,4(10):752-764.
[38]SAFFARINI D A,BLUMERMAN S L,MANSOORABADI K J.Role of menaquinones in Fe(Ⅲ)reduction by membrane fractions of Shewanella putrefaciens.Journal of Bacteriology,2002,184(3):846-848.
[39]MYERS C R,MYERS J M.Cloning and sequence of cymA,a gene encoding a tetraheme cytochrome c required for reduction of iron(Ⅲ),fumarate,and nitrate by Shewanella putrefaciens MR-1.Journal of Bacteriology,1997,179(4):1143-1152.
[40]MYERS J M,MYERS C R.Role of the tetraheme cytochrome CymA in anaerobic electron transport in cells of Shewanella putrefaciens MR-1 with normal levels of menaquinone.Journal of Bacteriology,2000,182(1):67-75.
[41]DOBBIN P S,BUTT J N,POWELL A K,et al.Characterization of a flavocytochrome that is induced during the anaerobic respiration of Fe3+by Shewanella frigidimarina NCIMB400.Biochemical Journal,1999,342(2):439-448.
[42]REYES-RAMIREZ F,DOBBIN P,SAWERSG,etal.Characterization oftranscriptionalregulation ofShewanella frigidimarina Fe(Ⅲ)-induced flavocytochrome c reveals a novel iron-responsive gene regulation system.Journal of Bacteriology,2003,185(15):4561-4571.
[43]GORDON E J,PIKE A D,HILL A E,et al.Identification and characterization of a novel cytochrome(c3)from Shewanella frigidimarina that is involved in Fe(Ⅲ)respiration.Biochemical Journal,2000,349(Pt1):153-158.
[44]MYERS J M,MYERS C R.MtrB is required for proper incorporation of the cytochromes omcA and omcB into the outer membraneofShewanellaputrefaciensMR-1.Appliedand Environmental Microbiology,2002,68(11):5585-5594.
[45]PITTSK E,DOBBIN PS,REYESRAMIREZF,etal.Characterization of the Shewanella oneidensis MR-1 decaheme cytochrome MtrA.Journal of Biological Chemistry,2003,278(30):27758-27765.
[46]BELIAEV A S,SAFFARINI D A,MCLAUGHLIN J L,et al.MtrC,an outer membrane decahaem c,cytochrome required for metal reduction in Shewanella putrefaciens,MR-1.Molecular Microbiology,2001,39(3):722-730.
[47]GASPARD S,VAZQUEZ F,HOLLIGER C.Localization and solubilization of the iron(Ⅲ)reductase of Geobacter sulfurreducens.Applied and Environmental Microbiology,1998,64(9):3188-3194.
[48]DICHRISTINA T J,MOORE C M,HALLER C A.Dissimilatory Fe(Ⅲ)and Mn(Ⅳ)reduction by Shewanella putrefaciens requires ferE,a homolog of the pulE(gspE)typeⅡprotein secretion gene.Journal of Bacteriology,2002,184(1):142-151.
[49] 洪義國,許玫英,郭俊,等.細菌的Fe(Ⅲ)還原.微生物學報,2005,45(4):653-656.HONG Y G,XU M Y,GUO J,et al.Bacterial Fe(Ⅲ)reduction.Acta Microbiologica Sinica,2005,45(4):653-656.(in Chinese with English abstract)
[50]黎慧娟,彭靜靜.異化Fe(Ⅲ)還原微生物研究進展.生態學報,2012,32(5):1633-1642.LI H J,PENG J J.Recent advances in studies on dissimilatory Fe(Ⅲ)-reducing microorganisms.Acta Ecologica Sinica,2012,32(5):1633-1642.(in Chinese with English abstract)
[51]CALDWELL S L,LAIDLER J R,BREWER E A,et al.Anaerobic oxidation of methane:Mechanisms,bioenergetics,and the ecology of associated microorganisms.Environmental Science&Technology,2008,42(18):6791-6799.
[52]REEBURGH W S.Oceanic methane biogeochemistry.Chemical Reviews,2007,107(2):486-513.
[53]SIMPSON I J,CHEN T Y,BLAKE D R,et al.Implications of the recent fluctuations in the growth rate of tropospheric methane.Geophysical Research Letters,2002,29(10):1479.
[54]ERMLER U,GRABARSE W,SHIMA S,et al.Crystal structure of methyl-coenzyme M reductase:The key enzyme of biological methane formation.Science,1997,278(5342):1457-1462.
[55]PRAKASH D,WU Y,SUH S J,et al.Elucidating the process of activation of methyl-coenzyme M reductase.Journal of Bacteriology,2014,196(13):2491-2498.
[56]FRIEDRICH M W.Methyl-coenzyme M reductase genes:Unique functional markers for methanogenic and anaerobic methaneoxidizing Archaea.Methods in Enzymology,2005,397:428-442.
[57]LUEDERS T,CHIN K J,CONRAD R,et al.Molecular analyses of methyl-coenzyme M reductase alpha-subunit(mcrA)genes in rice field soil and enrichment cultures reveal the methanogenic phenotype of a novel archaeal lineage.Environmental Microbiology,2001,3(3):194-204.
[58]EVANS P N,PARKS D H,CHADWICK G L,et al.Methane metabolism in the archaeal phylum Bathyarchaeota revealed by genome-centric metagenomics.Science,2015,350(6259):434-438.
[59]THAUER R K,KASTER A K,SEEDORF H,et al.Methanogenic archaea:Ecologically relevant differences in energy conservation.Nature Reviews Microbiology,2008,6(8):579-591.
[60]BUAN N R,METCALF W W.Methanogenesis by Methanosarcina acetivorans involves two structurally and functionally distinct classes of heterodisulfide reductase.Molecular Microbiology,2010,75(4):843-853.
[61]WEISS D S,G?RTNER P,THAUER R K.The energetics and sodium-ion dependence of N5-methyltetrahydromethanopterin:Coenzyme M methyltransferase studied with cob(Ⅰ)alamin as methyl acceptor and methylcob(Ⅲ)alamin as methyl donor.European Journal of Biochemistry,1994,226(3):799-809.
[62]方曉瑜,李家寶,芮俊鵬,等.產甲烷生化代謝途徑研究進展.應用與環境生物學報,2015,21(1):1-9.FANG X Y,LI J B,RUI J P,et al.Research progress in biochemical pathways of methanogenesis.Chinese Journal of Applied and Environmental Biology,2015,21(1):1-9.
[63]GOTTSCHALK G,THAUER R K.The Na+-translocating methyltransferase complex from methanogenic archaea.Biochimica et Biophysica Acta,2001,1505(1):28-36.
[64]G?RTNER P,ECKER A,FISCHER R,et al.Purification and properties of N5-methyltetrahydromethanopterin:Coenzyme M methyltransferase from Methanobacterium thermoautotrophicum.European Journal of Biochemistry,1993,213(1):537-545.
[65]HIPPLER B,THAUER R K.The energy conserving methyltetrahydromethanopterin:coenzyme M methyltransferase complex from methanogenic archaea:Function of the subunit MtrH.FEBS Letters,1999,449(2/3):165-168.
[66]承磊,鄭珍珍,王聰,等.產甲烷古菌研究進展.微生物學通報,2016,43(5):1143-1164.CHENG L,ZHENG Z Z,WANG C,et al.Recent advances in methanogens.Microbiology China,2016,43(5):1143-1164.
[67]HAGEMEIER C H,KRER M,THAUER R K,et al.Insight into the mechanism of biological methanol activation based on the crystal structure of the methanol-cobalamin methyltransferase complex.Proceedings of the National Academy of Sciences of the USA,2006,103(50):18917-18922.
[68]LIU Y,WHITMAN W B.Metabolic,phylogenetic,and ecological diversity of the methanogenic archaea.Annals of the New York Academy of Sciences,2008,1125(1):171-189.
[69]JUGDER B E,ERTAN H,LEE M,et al.Reductive dehalogenases come of age in biological destruction of organohalides.Trends in Biotechnology,2015,33(10):595-610.
[70]HUG L A,MAPHOSA F,LEYS D,et al.Overview of organohalide-respiring bacteria and a proposal for a classification system for reductive dehalogenases.Philosophical Transactions of theRoyalSociety B:BiologicalSciences,2013,368(1616):20120322.
[71]BAELUM J,NICOLAISEN M H,HOLBEN W E,et al.Direct analysis of tfdA gene expression by indigenous bacteria in phenoxy acid amended agricultural soil.The ISME Journal,2008,2(6):677-687.
[72]KOUTINAS M,KIPARISSIDES A,SILVA-ROCHA R,et al.Linking genes to microbial growth kinetics:An integrated biochemical systems engineering approach.Metabolic Engineering,2011,13(4):401-413.
[73]NEUMANN A,WOHLFARTH G G.Tetrachloroethene dehalogenase from Dehalospirillum multivorans:Cloning,sequencing of the encoding genes,and expression of the pceA gene in Escherichia coli.Journal of Bacteriology,1998,180(16):4140-4145.
[74]MAGNUSON J K,ROMINE M F,BURRIS D R,et al.Trichloroethene reductive dehalogenase from Dehalococcoides ethenogenes:Sequence of tceA and substrate range characterization.Applied and Environmental Microbiology,2000,66(12):5141-5147.
[75]KR?UTLER B,FIEBER W,OSTERMANN S,et al.The cofactor of tetrachloroethene reductive dehalogenase of Dehalospirillum multivorans is norpseudo-B12,a new type of a natural corrinoid.Helvetica Chimica Acta,2003,86(11):3698-3716.
[76]MAILLARDJ,SCHUMACHERW,VAZQUEZF,etal.Characterization of the corrinoid iron-sulfur protein tetrachloroethene reductive dehalogenase of Dehalobacter restrictus.Applied and Environmental Microbiology,2003,69(8):4628-4638.
[77]KRAJMALNIK-BROWN R,HOLSCHER T,THOMSON I N,et al.Genetic identification of a putative vinyl chloride reductase in Dehalococcoides sp.strain BAV1.Applied and Environmental Microbiology,2004,70(10):6347-6351.
[78]CHENG D,HE JZ.Isolation and characterization of‘Dehalococcoides’ sp. strain MB, which dechlorinates tetrachloroethene to trans-1,2-dichloroethene.Applied and Environmental Microbiology,2009,75(18):5910-5918.
[79]MüLLER J A,ROSNER B M,VON ABENDROTH G,et al.Molecular identification of the catabolic vinyl chloride reductase from Dehalococcoides sp.strain VS and its environmental distribution.Applied and Environmental Microbiology,2004,70(8):4880-4888.
[80]TSUKAGOSHI N,EZAKI S,UENAKA T,et al.Isolation and transcriptional analysis of novel tetrachloroethene reductive dehalogenase gene from Desulfitobacterium sp.strain KBC1.Applied and Environmental Microbiology,2006,69(5):543-553.
[81]NAKAMURA K,MIZUMOTO M,UENO T,et al.Cloning and analysis of trichloroethene reductive dehalogenase gene and its detection by quantitative real-time PCR.Environmental Engineering Research,2006,43:119-125.
[82]MARZORATI M,DE FERRA F,VAN RAEMDONCK H,et al.A novel reductive dehalogenase,identified in a contaminated groundwater enrichmentculture and in Desulfitobacterium dichloroeliminans strain DCA1,is linked to dehalogenation of 1,2-dichloroethane.Applied and Environmental Microbiology,2007,73(9):2990-2999.
[83]TANG S Q,EDWARDS E A.Identification of Dehalobacter reductive dehalogenases that catalyze dechlorination of chloroform,1,1,1-trichloroethane and 1,1-dichloroethane.Philosophical Transactions of the Royal Society B:Biological Sciences,2013,368(1616):20120318.
[84]GROSTERN A,EDWARDSE A.Characterizationofa Dehalobacter coculture that dechlorinates 1,2-dichloroethane to ethene and identification of the putative reductive dehalogenase gene.Applied and Environmental Microbiology,2009,75(9):2684-2693.
[85]BISAILLON A,BEAUDET R,LéPINE F,et al.Identification and characterization of a novel CprA reductive dehalogenase specific to highly chlorinated phenols from Desulfitobacterium hafniense strain PCP-1.Applied and Environmental Microbiology,2010,76(22):7536-7540.
[86]ADRIAN L,RAHNENFUHRER J,GOBOM J,etal.Identification of a chlorobenzene reductive dehalogenase in Dehalococcoides sp.strain CBDB1.Applied and Environmental Microbiology,2007,73(23):7717-7724.
[87]KRASOTKINAJ,WALTERST,MARUYAKA,etal.Characterization of the B12-and iron-sulfur-containing reductive dehalogenase from Desulfitobacterium chlororespirans.Journal of Biological Chemistry,2001,276(44):40991-40997.
[88]MATTURRO B,DI L M,UBALDI C,et al.First evidence on the occurrence and dynamics of Dehalococcoides mccartyi PCB-dechlorinase genes in marine sediment during Aroclor1254 reductive dechlorination.Marine Pollution Bulletin,2016,112(1/2):189-194.
[89]VAN DE B A,SMIDT H,HAGEN W R,et al.Purification and molecular characterization of ortho-chlorophenolreductive dehalogenase,a key enzyme of halorespiration in Desulfitobacterium dehalogenans.Journal of Biological Chemistry,1999,274(29):20287-20292.
[90]SMIDT H,DE VOS W M.Anaerobic microbial dehalogenation.Annual Review of Microbiology,2004,58:43-73.
[91]SMIDT H,VANLEEST M,VANDEROOST J,et al.Transcriptional regulation of the cpr gene cluster in ortho-chlorophenol-respiring Desulfitobacterium dehalogenans.Journal of Bacteriology,2000,182(20):5683-5691.
[92]FUTAGAMI T,YAMAGUCHI T,NAKAYAMA S,et al.Effects of chloromethanes on growth of and deletion of the pce gene cluster in dehalorespiring Desulfitobacterium hafniense strain Y51.Applied and Environmental Microbiology,2006,72(9):5998-6003.
[93]DESHPANDE N P,WONG Y K,MANEFIELD M,et al.Genome sequence of Dehalobacter UNSWDHB,a chloroform-dechlorinating bacterium.Genome Announcements,2013,1(5):e00720-13.
[94]VILLEMUR R.The pentachlorophenol-dehalogenating Desulfitobacterium hafniense strain PCP-1.Philosophical Transactions of the Royal Society B:Biological Sciences,2013,368(1616):20120319.
[95]GAUTHIER A,BEAUDET R F,JUTEAU P,et al.Occurrence and expression of crdA and cprA5 encoding chloroaromatic reductive dehalogenases in Desulfitobacterium strains.Canadian Journal of Microbiology,2006,52(1):47-55.
[96]MAGNUSON J K,STERN R V,GOSSETT J M,et al.Reductive dechlorination of tetrachloroethene to ethene by a two-component enzyme pathway.Applied and Environmental Microbiology,1998,64(4):1270-1275.
[97]MüLLER J A,ROSNER B M,ABENDROTH A V,et al.Molecular identification of the catabolic vinyl chloride reductase from Dehalococcoides sp.strain VS and its environmental distribution.Applied and Environmental Microbiology,2004,70(8):4880-4888.
[98]CANFIELD D E,JORGENSEN B B,FOSSING H,et al.Pathways of organic carbon oxidation in three continental margin sediments.Marine Geology,1993,113(1/2):27-40.
[99]FOSSING H,GALLARDO V A,J?RGENSEN B B,et al.Concentration and transport of nitrate by the mat-forming sulphur bacterium Thioploca.Nature,1995,374(6524):713-715.
[100]LAUFER K,R?Y H,J?RGENSEN B B,et al.Evidence for the existence of autotrophic nitrate-reducing Fe(Ⅱ)-oxidizing bacteria in marine coastal sediment.Applied and Environmental Microbiology,2016,82(20):6120-6131.
[101]ROBERTSON E K,ROBERTS K L,BURDORF L D W,et al.Dissimilatory nitrate reduction to ammonium coupled to Fe(Ⅱ)oxidation in sedimentsofa periodically hypoxic estuary.Limnology and Oceanography,2016,61(1):365-381.
[102]CHAKRABORTY A,RODEN E E,SCHIEBER J,et al.Enhanced growth of Acidovorax sp.strain 2AN during nitrate-dependent Fe(Ⅱ)oxidation in batch and continuous-flow systems.Applied and Environmental Microbiology,2011,77(24):8548-8556.
[103]BURGIN A J,HAMILTON S K.NO3?-driven SO42?production in freshwater ecosystems:Implications for N and S cycling.Ecosystems,2008,11(6):908-922.
[104]PAYNE E K,BURGIN A J,HAMILTON S K.Sediment nitrate manipulation using porewater equilibrators reveals potential for N and S coupling in freshwaters.Aquatic Microbial Ecology,2009,54(3):233-241.
[105]YANG X N,HUANG S,WU Q H,et al.Nitrate reduction coupled with microbial oxidation of sulfide in river sediment.Journal of Soils and Sediments,2012,12(9):1435-1444.
[106]NEAL A L,TECHKARNJANARUK S,DOHNALKOVA A,et al.Iron sulfides and sulfur species produced at hematite surfaces in the presence of sulfate-reducing bacteria.Geochimica et Cosmochimica Acta,2001,65(2):223-235.
[107]FLYNN T M,O’LOUGHLIN E J,MISHRA B,et al.Sulfurmediated electron shuttling during bacterial iron reduction.Science,2014,344(6187):1039-1042.
[108]VILLEMUR R,LANTHIER M,BEAUDET R,et al.The Desulfitobacterium genus.FEMS Microbiology Reviews,2006,30(5):706-733.
[109]DRZYZGA O,GOTTSCHAL J C.Tetrachloroethene dehalorespiration and growth of Desulfitobacterium frappieri TCE1 in strict dependence on the activity of Desulfovibrio fructosivorans.Applied and Environmental Microbiology,2002,68(2):642-649.
[110]DRZYZGA O,GERRITSE J,DIJK J A,et al.Coexistence of a sulphate-reducing Desulfovibrio species and the dehalorespiring Desulfitobacterium frappieri TCE1 in defined chemostat cultures grown with various combinations of sulphate and tetrachloroethene.Environmental Microbiology,2001,3(2):92-99.
[111]NIGGEMYER A,SPRING S,STACKEBRANDT E,et al.Isolation and characterization ofa novelAs(Ⅴ)-reducing bacterium:Implications for arsenic mobilization and the genus Desulfitobacterium.Applied and Environmental Microbiology,2001,67(12):5568-5580.
[112]HE Q,SANFORD R A.Characterization of Fe(Ⅲ)reduction by chlororespiring Anaeromxyobacter dehalogenans.Applied and Environmental Microbiology,2003,69(5):2712-2718.
[113]KRUMHOLZ L R,SHARP R,FISHBAIN S S.A freshwater anaerobe coupling acetate oxidation to tetrachloroethylene dehalogenation.Applied and Environmental Microbiology,1996,62(11):4108-4113.
[114]CHEN M J,CAO F,LI F B,et al.Anaerobic transformation of DDT related to iron(Ⅲ)reduction and microbial community structure in paddy soils.Journal of Agricultural and Food Chemistry,2013,61(9):2224-2233.
[115]HE Y T,WILSON J T,WILKIN R T.Transformation of reactive iron minerals in a permeable reactive barrier(biowall)used to treat TCE in groundwater.Environmental Science&Technology,2008,42(17):6690-6696.
[116]馮小莉.水稻土混合菌群耦合鐵氧化物對五氯酚還原轉化的作用與機制.杭州:浙江大學,2014:59-60.FENG X L.Reductivetransformationandmechanism of pentachlorophenol by anaerobic microbial communities enriched from paddy soil coupled with iron oxides.Hangzhou:Zhejiang University,2014:59-60.(in Chinese with English abstract)
[117]GUERRERO-BARAJAS C,GARCíA-PE?A E I.Evaluation of enrichments of sulfate reducing bacteria from pristine hydrothermal vents sediments as potential inoculum for reducing trichloroethylene.World Journal of Microbiology and Biotechnology,2010,26(1):21-32.
[118]BAO P,HU Z Y,WANG X J,et al.Dechlorination of p,p’-DDTs coupled with sulfate reduction by novelsulfate-reducing bacterium Clostridium sp.BXM.Environmental Pollution,2012,162:303-310.
[119]BARBOUR J P,SMITH J A,CHIOU C T.Sorption of aromatic organic pollutants to grasses from water.Environmental Science&Technology,2005,39(21):8369-8373.
[120]宋長青,吳金水,陸雅海,等.中國土壤微生物學研究十年回顧.地球科學進展,2013,28(10):1087-1105.SONG C Q,WU J S,LU Y H,et al.Advances of soil microbiology in the last decade in China.Advances in Earth Science,2013,28(10):1087-1105.(in Chinese with English abstract)

