遼寧爪鯢(Onychodactylus zhaoermii)與吉林爪鯢(O.zhangyapingi)的遺傳多樣性
欒東岳,原 野,徐 昕,董丙君,楊寶田
(1.沈陽師范大學生命科學學院,遼寧沈陽110034;2.鞍山市野生動植物保護管理工作站,遼寧鞍山114004)
[通訊作者]楊寶田(1963- ),男,副教授,碩士生導師,博士,從事野生動物保護與利用、分子生態學研究。
遼寧爪鯢(Onychodactyluszhaoermii)和吉林爪鯢(O.zhangyapingi)隸屬于兩棲綱(Amphibia)有尾目(Caudata)小鯢科(Hynobiidae)爪鯢屬(Onychodactylus)[1]。此兩種爪鯢是Poyarkov et al.(2012)[2]在對東北亞分布的爪鯢物種形態和分子數據研究基礎上,從原屬于Onychodactylusfischeri[3]中分離出的新物種,目前已知分布在遼寧省(岫巖)和吉林省(渾江、臨江、集安、白河、延吉)的長白山及其余脈[1],本研究在遼寧遼陽縣甜水滿族鄉發現有遼寧爪鯢分布。兩種爪鯢棲息地環境較為相似,均在山高林密,雜草叢生,常年流水的溪流中或其附近。爪鯢多以陸地棲息為主,晝伏夜出,黃昏和雨后活動較為頻繁[1,4],常見于土縫與溪中石縫間。以溪流內的小蝦及小昆蟲為食[1,5]。
爪鯢以其指、趾末端具有黑色角質爪而得名,與其他小鯢科動物的明顯區別是其無肺[1]。遼寧爪鯢與吉林爪鯢的形態結構差異在于遼寧爪鯢的犁骨齒明顯彎曲,齒數在16~17個且沒有縫隙,而吉林爪鯢的犁骨齒較為平整,齒數在13~14個且沒有明顯縫隙[1-2]。近些年國內有關爪鯢的研究多集中在形態與組織解剖學[6-9]、生理學[10-11]、保護生物學[5,12]和生態學[13]等方面。分子方面的相關文獻多來自國外學者或機構的研究,Yoshikawa et al.(2008)[14]對爪鯢系統發生關系和譜系地理學進行研究,利用線粒體Cytb基因序列對日本爪鯢種群系統關系和地理演化進行了分析,認為日本爪鯢存在4個支系,地質事件(日本海的開放)和氣候變化是日本爪鯢種群分化的主要影響因素。
生物多樣性的核心是遺傳多樣性,生物進化潛力由種內的變異程度決定,物種對環境變化適應能力的增強,是由于種內的遺傳多樣性豐富度的上升。保護生物遺傳多樣性的本質就是保護生物多樣性[15]。本研究采用Cytb基因序列分析方法對分布于我國的2種爪鯢進行種群遺傳學研究。通過對其遺傳多樣性的分析,評估遺傳多態性的程度和分布,為爪鯢物種保護提供理論依據。
1 材料和方法
1.1 樣本
本研究野外采集樣本共計73個(表1),其中吉林爪鯢43個,遼寧爪鯢30個。野外樣本分別來自吉林省集安市五女峰(8個)、通化市石湖鎮(18個)、通化市老嶺(17個),遼寧省岫巖縣三家子(26個)、遼陽縣大黑山(4個)等5個地理種群。采集爪鯢樣本取后尾部末端組織或肢趾尖,放置于無水乙醇中固定帶回實驗室保存于-25℃冰箱中,樣本采集后爪鯢個體放生。此外,在GenBank下載線粒體Cytb基因序列15條,來自遼寧爪鯢8條,其中2條來自吉林通化老嶺;來自吉林爪鯢序列7條,其中4條來自吉林臨江(JLLJ)。用以分析的線粒體Cytb基因序列共計88條。

表1 采集的樣本信息
1.2 總DNA提取、Cytb基因PCR擴增和測序
采用TIANamp Genomic DNA Kit試劑盒提取基因組DNA,具體操作方法按照試劑盒說明書進行。提取的DNA存放在-20℃冰箱內保存待用。線粒體Cytb基因DNA擴增所用引物如表2,引物由北京華大基因有限公司合成。線粒體Cytb基因PCR擴增反應體系為25μL,10×Buffer 2.5μL;MgCl22.5μL;dNTP 2μL(10mmol/L);引物 2μL(10pmol/μL),上下游引物各1μL(10pmol/μL);TaqDNA聚合酶1U,模版DNA 100ng/2μL;加雙蒸水至25μL。PCR反應程序如下:94℃預變性8min,之后94℃變性30sec,51~57℃退火30sec,72℃延伸45sec,運行35個循環后,72℃再延伸10min。PCR反應在Applied Biosystems Inc. 2720 PCR儀或Applied Biosystems Inc. 9902 PCR儀中進行。PCR產物經電泳檢測后由北京華大基因有限公司測序。

表2 用于爪鯢線粒體Cytb基因擴增的引物
1.3 數據處理與分析
將所測得的Cytb基因片段首先進行Blast搜索,驗證其可靠性;然后用Lasergene v.7.0對DNA片段進行拼接和手工編輯校正;再利用Clustal X[16]對拼接好的基因序列進行比對;最后采用脊椎動物線粒體遺傳密碼子進行序列翻譯,以排除假基因的存在并證明其準確性。用DAMBE[17]檢測Cytb基因序列的堿基替換模式,以檢驗它們是否發生堿基替換飽和效應。用DNAsp 5.10[18]軟件計算單倍型多態性和核苷酸多樣性等遺傳多樣性指數,同時進行中性(Tajima’s Test; Fu and Li’s Tests)檢測和連鎖不平衡(Linkage Disequilibrium)檢測。用MEGA 6.0[19]軟件分析不同樣本序列間的堿基組成和變異位點,并計算基于Kimura雙參數模型的遺傳距離、構建NJ系統樹。利用Arlequin 3.5[20]軟件進行遺傳結構分析和分子方差分析(Analysis of molecular variance, AMOVA)。單倍型網絡關系圖用TCS 1.20[21]軟件完成。
2 結果與分析
2.1 線粒體Cytb基因序列特征
兩種爪鯢線粒體DNA Cytb基因經PCR擴增后得到的序列長度有所不同,遼寧爪鯢Cytb基因長度為1146bp,吉林爪鯢Cytb基因長度為1149bp。相對于吉林爪鯢Cytb基因,遼寧爪鯢Cytb基因在1098-1100bp處存在“GTT”3個堿基的缺失。將所獲得的Ctyb基因序列通過進行Blast同源性搜索以確定其可靠性。Cytb基因為氨基酸編碼基因,我們采用脊椎動物線粒體遺傳密碼子進行序列翻譯,以排除假基因的存在并證明其準確性。在Cytb基因序列中,遼寧爪鯢樣本A+T含量為68.1%,吉林爪鯢樣本的A+T含量為67.7%,二者非常接近,都明顯高于G+C的含量,此數據符合脊椎動物線粒體堿基組成的基本特征。
2.2 線粒體Cytb基因遺傳變異
結合GenBank下載數據,遼寧爪鯢共38條Cytb基因序列,存在16個變異位點,其中有8個簡約信息位點。核苷酸多樣性(π)和平均核苷酸差異數(k)分別為0.00265和3.3037(表3)。定義單倍型(h)9個(單倍型多樣度Hd=0.721)(表4)。
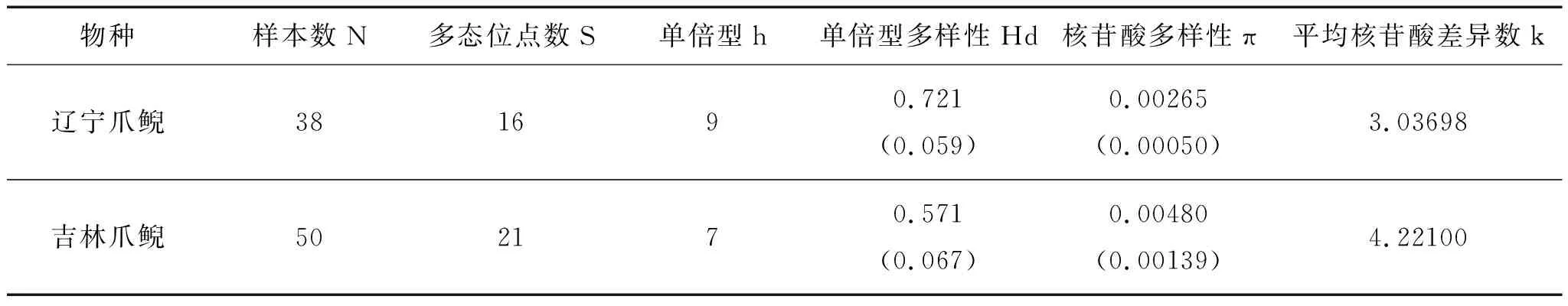
表3 兩種爪鯢Cytb基因遺傳多樣性參數
注:圓括號內數字為標準誤差。
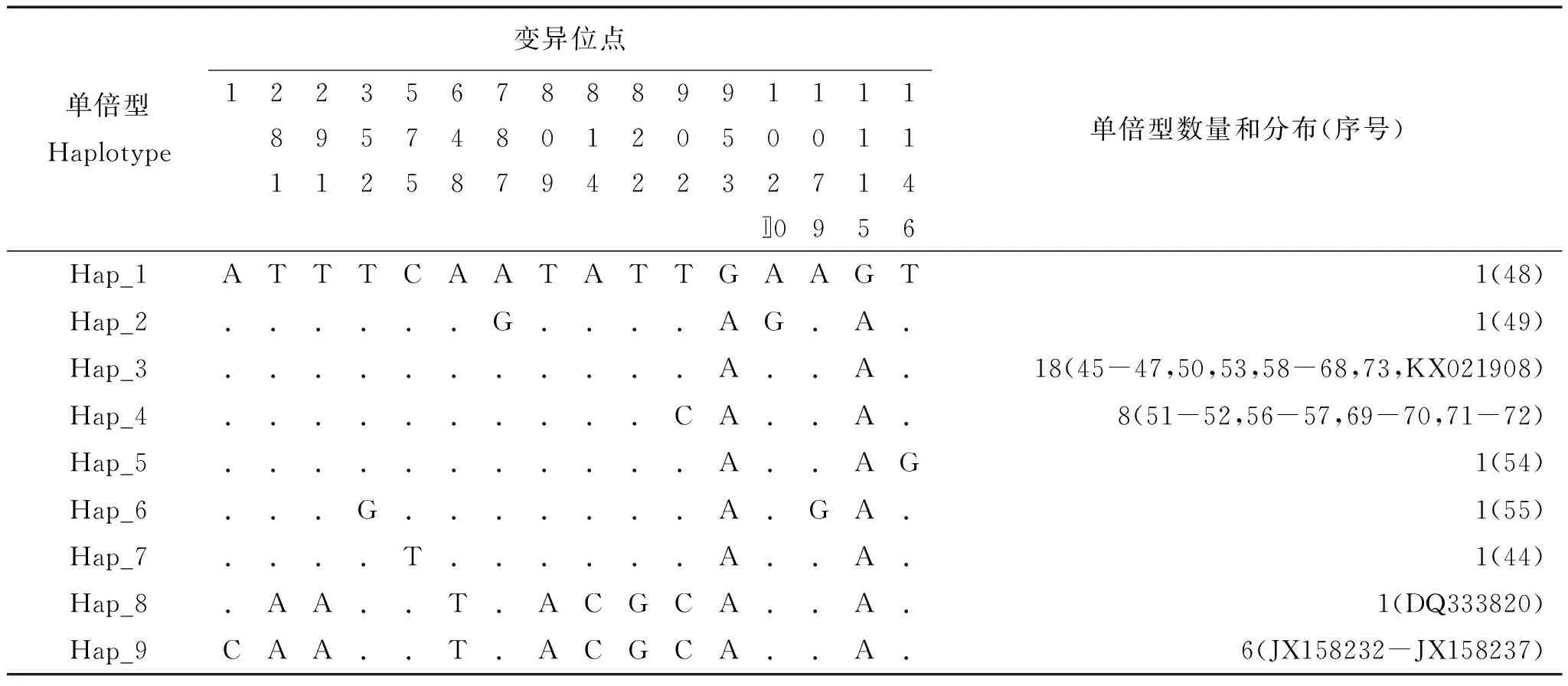
表4 遼寧爪鯢線粒體Cytb單倍型及變異位點分布
吉林爪鯢的50條Cytb基因序列存在21個變異位點,其中有19個簡約信息位點。核苷酸多樣性(π)和平均核苷酸差異數(k)分別為0.00480和4.2210(表3)。定義單倍型(h)7個(單倍型多樣度Hd=0.571)(表5)。

表5 吉林爪鯢線粒體Cytb單倍型及變異位點分布
采用Kimura-2計算的種群內平均遺傳距離(表6中對角線數據)表明種群內個體間的遺傳距離非常小,最大的為JLTH種群(0.0041),最小的為JLLJ和JLLL兩個種群(0.0000)。遼寧爪鯢種群內平均遺傳距離為0.00266±0.00254,吉林爪鯢為0.00486±0.00100。
單倍型網絡關系圖(圖1)顯示單倍型間無對應種群分支結構,單倍型間接近,突變步數較少。
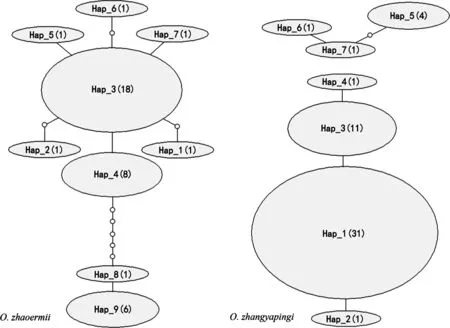
圖1 單倍型網絡關系
注:每個橢圓形代表一個定義的單倍型,橢圓的大小表示該單倍型的頻率。“○”表示理論分析得出、但沒有實際觀測到的單倍型。單倍型間的每一連線代表一個突變步驟。
2.3 種群分歧與遺傳結構分析
用MEGA軟件計算的線粒體Cytb基因序列種群內和種群間的遺傳距離結果(表6)顯示,兩個爪鯢物種種群間的遺傳距離非常小,大多數種群間的遺傳距離都小于1%,只有吉林爪鯢的臨江種群與其他種群的遺傳距離大于1%(1.9%~2.1%)。
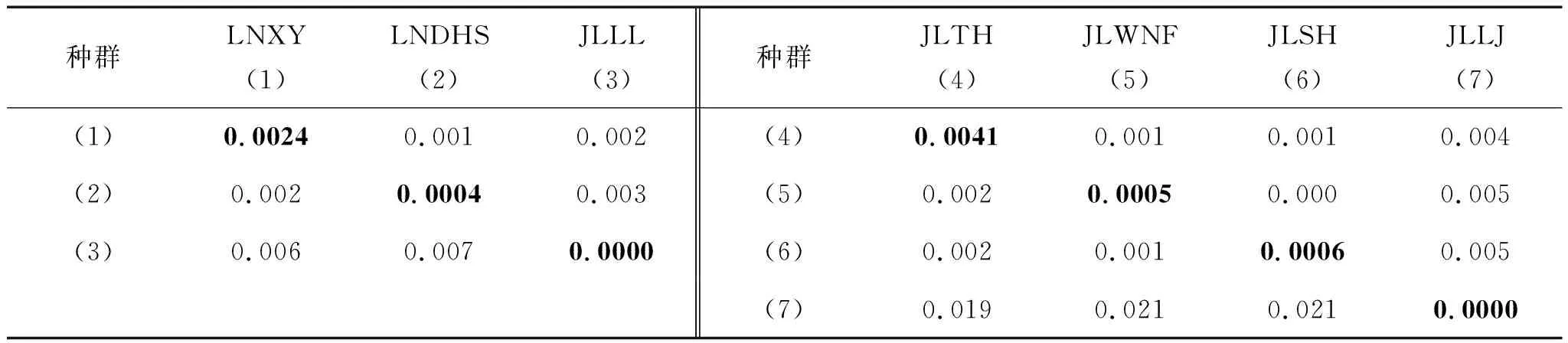
表6 種群內和種群間Cytb基因序列遺傳距離分析
注:下三角為遺傳距離,上三角為標準誤,對角線為種群內平均遺傳距離。
遺傳分化指數(FST)分析顯示(表7),遼寧爪鯢種群間分化狀況不平衡,JLLL種群與LNXY種群和LNDHS種群分化明顯(p<0.05),而LNXY種群與LNDHS種群則無明顯遺傳分化(p≥0.05)。對遼寧爪鯢種群間基因流(Nm=0.91,Nei 1982)分析可以看出種群間基因交流程度不高。

表7 遼寧爪鯢種群間FST
注:下三角為FST值;上三角為統計檢驗p值,*為p<0.05水平差異顯著。
對遼寧爪鯢進行分子方差分析(AMOVA),將遼寧的兩個種群劃為一組,吉林的遼寧爪鯢(JLLL)劃為另一組,分析結果顯示2個分組之間的變異在總變異中占到65.91%,而組內種群間的變異占比非常小,只有0.56%(表8)。結合種群間FST分析(表7)可以看出,這種占比特征主要是JLLL種群與其他種群差異引起。
FST=0.66478(p<0.05)。
對吉林爪鯢的遺傳分化指數(FST)分析,吉林爪鯢除吉林臨江種群與其他種群存在較大分化外(p<0.05),其余種群間均無明顯遺傳分化(p≥0.05)(表9)。吉林爪鯢種群間基因流Nm=0.18(Nei 1982)。

表9 吉林爪鯢種群間FST
注:下三角為FST值;上三角為統計檢驗p值,*為p<0.05水平差異顯著。
對吉林爪鯢進行分子方差分析(AMOVA),由于各樣本采集地相對比較集中,均屬于長白山南部山地,因此沒有將其進行分組設計,僅對種群與個體兩個階層進行分析。結果顯示種群之間的變異在總變異中占到67.43%,而種群內的變異占比相對較小(32.57%)(表10)。結合種群間FST分析(表9)可以看出,這種占比特征主要由JLLL種群與其他種群的差異引起。
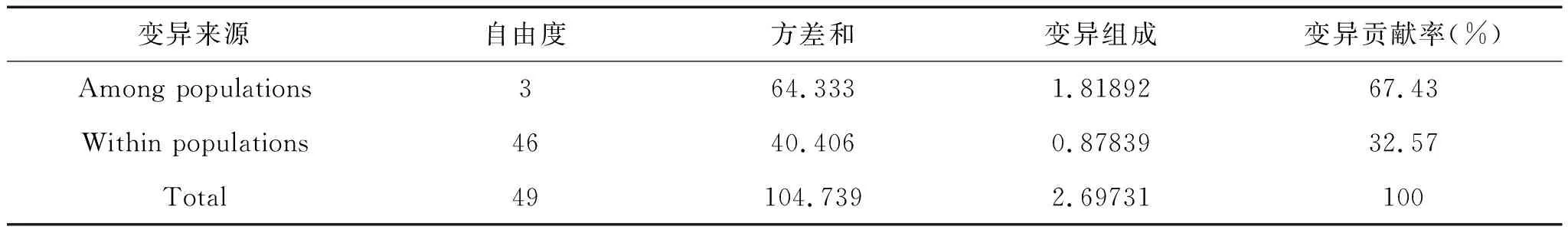
表10 吉林爪鯢AMOVA分析
FST=0.67435(p<0.05)。
鑒于上述分析中分化程度較大的爪鯢種群來自Nikolay et al.(2012)[2]的DNA序列,即作為遼寧爪鯢JLLL種群和吉林爪鯢JLLJ種群的樣本,本研究去掉這部分樣本,用自測的DNA數據重新做了遺傳結構分析。在所分析的2個遼寧爪鯢種群間(LNXY vs LNDHS)的分化指數(FST)為0.035,而吉林爪鯢的3個種群(JLSH、JLWNF和JLTH)之間的遺傳分化指數均為0.000。在沒有進一步對種群分組的情況下(因采樣種群自然地理區系相同,地理距離很近),遼寧爪鯢種群間遺傳變異只占總變異的3.000%,而吉林爪鯢種群間的遺傳變異占比為0.000%。
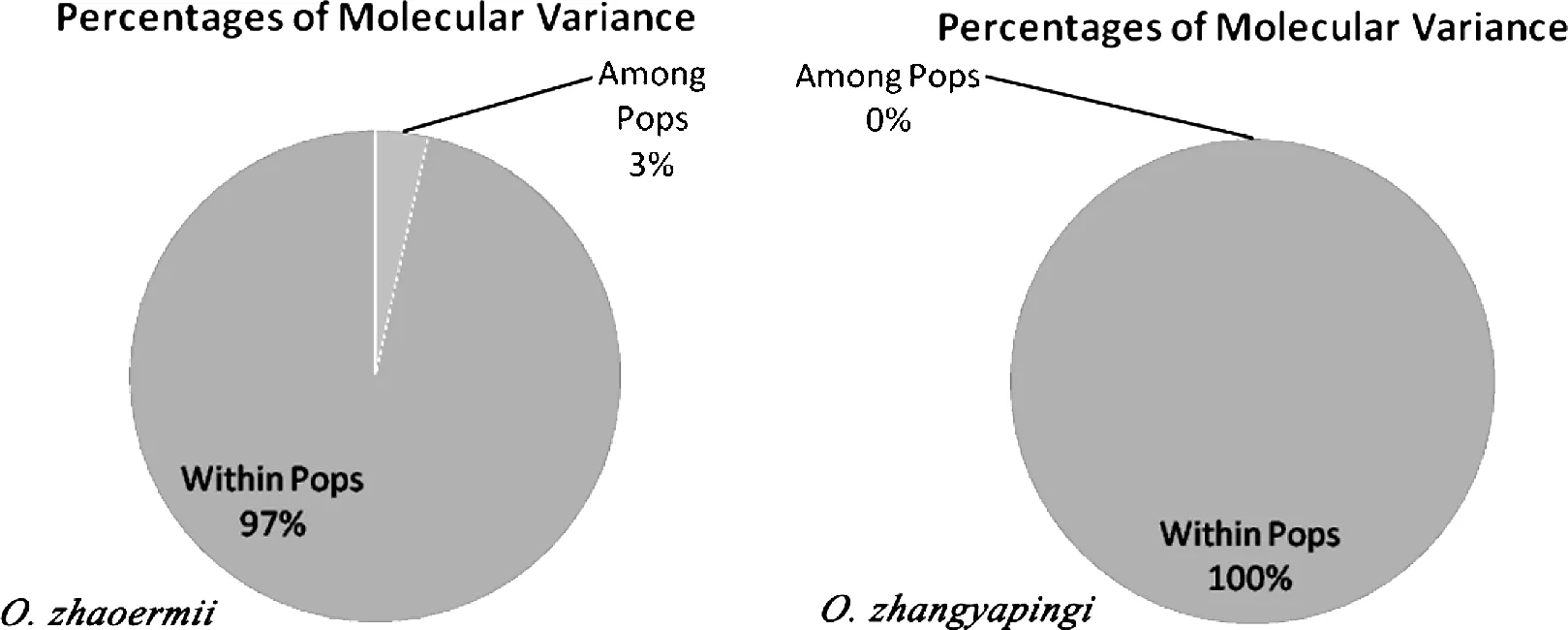
圖2 用自測DNA數據所做AMOVA分析圖
2.4 線粒體Cytb基因系統發生關系
在GenBank下載線粒體Cytb基因序列116條(Nikolay et al.,2012)[2],包括O.fischeri、O.koreanus、O.cf.japonicus、O.sp.、O.nipponoborealis等5個爪鯢物種(或新種)。結合本研究中自測的73條序列,共計189個爪鯢Cytb基因序列,采用鄰接法(Neighbor-Joining, NJ)用MEGA6.0軟件構建系統關系樹,結果如圖3。從圖中可以看出這些爪鯢物種(種群)可以分成3枝,O.cf.japonicus、O.nipponoborealis和O.zhangyapingi聚為一枝,O.koreanus、O.sp.和O.zhaoermii為一枝,O.fischeri單獨一枝。O.fischeri顯示為較原始類群(物種),處于系統樹的根部。
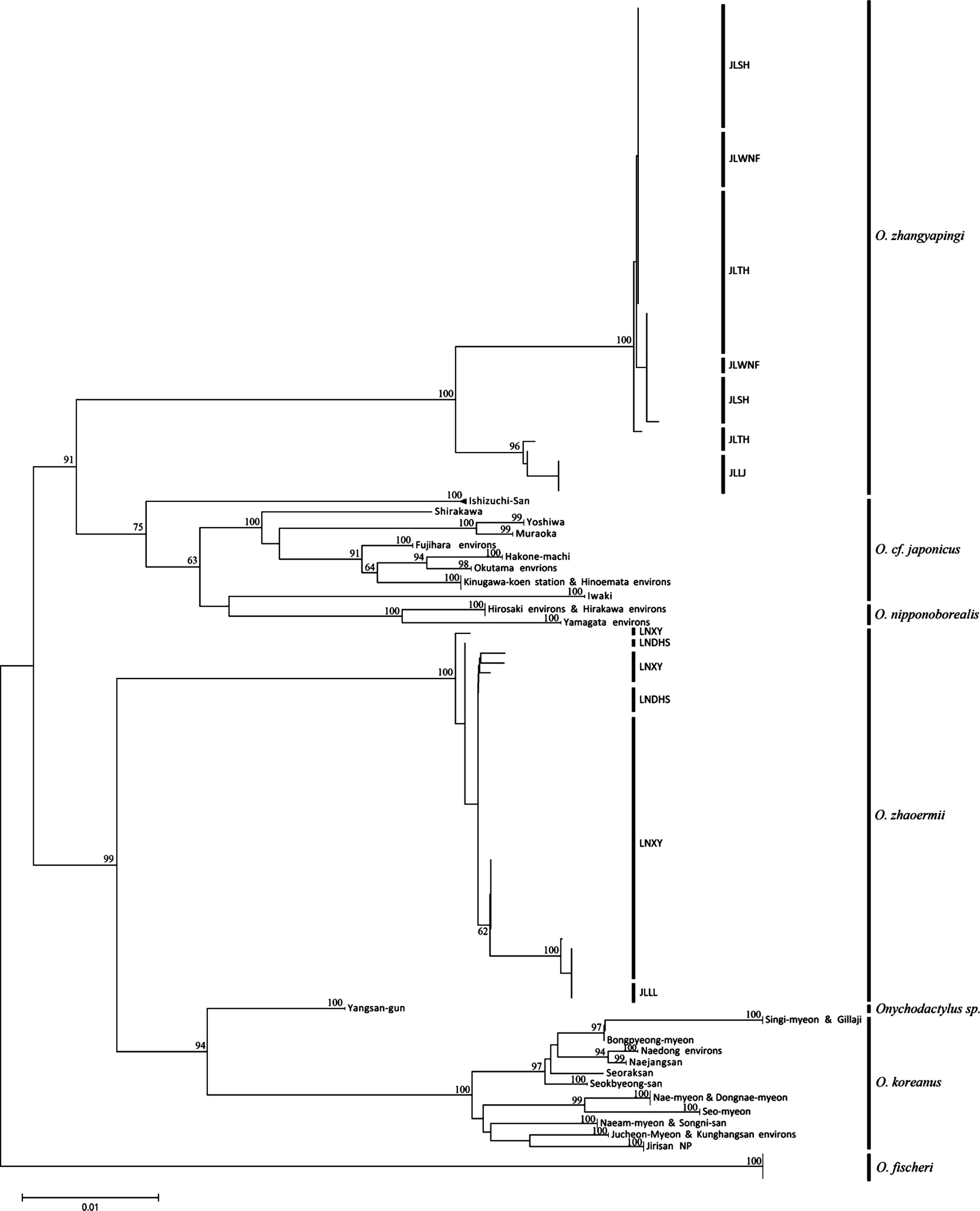
注:節點處數值為置信值,沒有標示數值的節點置信值低于60。圖3 用7個爪鯢物種189條Cytb序列構建的鄰接樹
3 討論
3.1 兩種爪鯢的遺傳多樣性比較
Cytb基因標記結果顯示遼寧爪鯢和吉林爪鯢遺傳多樣性較低。從變異位點、簡約信息位點、核苷酸多樣性(π)、平均核苷酸差異數(k)和單倍型多樣度等多個遺傳多樣性參數上可以得出這一結果。就2個爪鯢物種進行比較,吉林爪鯢在多態位點數、核苷酸多樣性和平均核苷酸差異數上略高于遼寧爪鯢,但其單倍型數和單倍型多樣性低于遼寧爪鯢。在種群遺傳多樣性變化趨勢上,吉林爪鯢的遺傳多樣性高于遼寧爪鯢。在遺傳多樣性研究中分子標記的選擇和應用對結果有十分重要的影響。線粒體DNA分子標記優點是不受環境和其他因素的影響而直接以DNA的形式出現,其具有母系遺傳、選擇中性、進化速率快且穩定等特點。一般來說,種群遺傳多樣性高低受種群自身特點的影響,但動物種群線粒體DNA所反映的遺傳多樣性基本和種群大小無關。Frentiu et al.(2001)[22]在對澳洲肺魚的研究中發現從控制區至ATPase 8的線粒體DNA序列有很少的遺傳變異。
3.2 遺傳分化與遺傳結構
基于線粒體Cytb序列的遼寧爪鯢種群間遺傳變異分析,種群遺傳距離(表6)和遺傳分化指數(FST,表7)均顯示了一致的結果,即分布于吉林的JLLL種群與分布于遼寧的2個種群(LNDHS & LNXY)分化程度很大,而遼寧分布LNDHS種群與LNXY種群具有中等程度分歧。對吉林爪鯢種群分析,遺傳距離(表6)和FST(表9)顯示JLLJ種群與其他3個種群(JLSH & JLWNF & JLTH)存在很大遺傳分化,而JLSH、JLWNF和JLTH種群間無分化或僅有很小分化。在上述利用Cytb基因所做分析中,兩種爪鯢顯示較大分化的種群(遼寧爪鯢JLLL種群和吉林爪鯢JLLJ種群)數據出自Nikolay et al.(2012),如果采用本研究自測的DNA數據重新分析,遼寧爪鯢LNXY種群與LNDHS種群的分化指數為0.035,而吉林爪鯢3個種群(JLSH & JLWNF & JLTH)之間的遺傳分化指數均為0.000,即遼寧爪鯢2個種群間存在較小遺傳分化(0 < FST< 0.05),吉林爪鯢3個種群間均無遺傳分化(FST=0.000)。
本研究從遼寧爪鯢、吉林爪鯢和遼寧爪鯢+吉林爪鯢三個側面進行了遺傳結構分析。將遼寧爪鯢LNXY種群和LNDHS種群劃為一組,JLLL種群為另一組的情況下,分子方差分析結果表明,組間的變異占65.91%,成為遺傳變異的主要來源。對吉林爪鯢4個種群的AMOVA分析,變異主要來自于種群間(67.43%)。用自測Cytb數據重新分析,將JLLL種群數據去掉,不進行組別劃分,則遼寧爪鯢種群間遺傳變異占總變異的份額為3%,FST=0.035;同樣,去掉JLLJ種群數據,吉林爪鯢的遺傳變異則全部來自于個體間(100%),FST=0.000,遼寧爪鯢和吉林爪鯢種群間的變異占比都非常小,遼寧爪鯢種群略大于吉林爪鯢。將遼寧爪鯢和吉林爪鯢合并AMOVA分析,則種間的變異占到98.82%,種群間和個體間的變異僅占1.18%。
交配系統和擴散機制對動物種群的遺傳分歧水平有顯著影響。爪鯢棲息于海拔500至1000米植被茂密的山區,水質清涼、多碎石的溪流、水溝或泉眼附近是其常棲之地。由于爪鯢依賴水環境且對水質條件要求苛刻,極大限制了爪鯢的遷徙擴散能力,因而分布地域狹窄。爪鯢只能集中在所處棲息地繁衍,從而限制了基因交流,進而導致了一定程度的遺傳分化。環境因子的差異所導致的選擇差異在爪鯢種群遺傳分化中起了重要作用。
3.3 系統發生關系與地理演化
線粒體DNA是動物親緣地理學最常用的遺傳標記,盡管隨著新一代測序技術的普遍應用而有所下降,但在已發表的有關譜系地理研究中仍占據較大份額。線粒體DNA具有高突變率、PCR擴增對模板要求低、在動物組織中拷貝數多等優點,特別是線粒體DNA在種內多態性為中性,不受選擇影響,因而能更好反映種群經歷的歷史事件。動物線粒體DNA極少重組,每一個單倍型對應一個前一世代祖先,所以很容易推斷單倍型間的遺傳關系,進而通過單倍型分布和相對頻率推斷種群歷史地理關系。本研究遼寧爪鯢單倍型網絡關系顯示出典型的星狀模式(圖1),新單倍型(Hap_1-2, Hap_4-7)的形成發生在出現頻率最高的單倍型(Hap_3),后續的核苷酸突變不可能發生在相同位點,因而這些稀少的新單倍型出現頻率和數目較低。盡管研究樣本采自分屬不同行政區域的兩個地點,但遼寧爪鯢地理分布的狹域性使其依然構成一個獨立分化中心。與遼寧爪鯢有所不同,吉林爪鯢顯示出相對復雜的單倍型網絡關系(圖1),單倍型Hap_1占有主導地位,但Hap_3頻率出現次級峰。Hap_1與Hap_3間單倍型距離很近(僅一步突變),可能是由于短暫的遺傳隔離因素形成,由二者衍生出來的稀少衛星單倍型(Hap_2, Hap_4)很少。
地理隔離和擴散能力是一個物種地理分布格局形成的決定因素,狹域分布的物種也很少有以單一的未分化的種群存在。基于Cytb基因的系統發生關系顯示(圖3),遼寧爪鯢種群呈現多系譜系關系,亦即來自不同種群的一些樣本攜帶來自不同進化枝的單倍型。這從另一個方面反映在遼寧爪鯢種群間很少地理隔離或無隔離因素存在,種群間基因交流頻繁。同樣,吉林爪鯢種群也表現為多系譜系關系,各種群沒有形成獨立進化枝。但遼寧爪鯢與吉林爪鯢卻分屬2個不同的進化枝,遼寧爪鯢與O.sp.和O.koreanus構成一支,而吉林爪鯢與O.cf.japonicus和O.nipponoborealis構成一支,O.fischeri位于樹的根部單獨一支。爪鯢屬在28.7MYA漸新世中期從其它小鯢中分離出來,分布于東北亞地區的山地森林中[14]。在第三紀中新世的早期,大概15.5 MYA,俄羅斯的O.fischeri和其他世系首先在爪鯢屬中分離出來。基于Yoshikawa et al.(2008)的分析,日本分布的爪鯢最早于8.1 MYA與東北亞大陸分布的爪鯢分離[14],而中國東北與朝鮮半島分布的爪鯢則在5.5 MYA分離,而在本研究中中國東北的2種爪鯢分屬日本和朝鮮半島2個不同的進化枝,這其中的矛盾和東北亞地區爪鯢物種系統發育和地理演化關系有待于進一步深入研究。
兩棲類動物種群的普遍衰退是當今普遍現象,兩棲動物遷移能力低,當種群變得小而隔離時,在遺傳問題方面變得特別脆弱。鑒于遼寧爪鯢和吉林爪鯢種群呈現多系的譜系結構,因而建議將2個物種分別作為一個管理單元加以保護。兩種爪鯢(特別是遼寧爪鯢)已知分布區域極其狹窄,棲息地多為山地,景色宜人,夏季涼爽,有部分爪鯢分布區為開發旅游資源而建立風景區,這在很大程度上對爪鯢生境造成了干擾和破壞。建議加強爪鯢物種監測和棲息地保護力度,對爪鯢分布區實施封閉管理,目前,遼寧鞍山市野生動植物保護站建立了爪鯢自然保護小區[23]。同時,需進一步加強爪鯢生物學、種群遺傳學以及保護生物學研究,為爪鯢保護提供科學的理論指導。
4 結論
Cytb分析兩種爪鯢種群遺傳變異水平很低,但吉林爪鯢的遺傳多樣性高于遼寧爪鯢。遼寧爪鯢種群間存在較小遺傳分化,而吉林爪鯢種群間均無遺傳分化。遼寧爪鯢和吉林爪鯢種群均呈現多系的譜系結構,建議將兩個物種分別作為一個管理單元加以保護。
[參考文獻]
[1]費梁, 葉昌媛, 江建平. 中國兩棲動物及其分布色彩圖鑒[M]. 成都: 四川科技出版社, 2012:64-65.
[2]Nikolay A, Poyarkov JR, Jing Che, et al. Review of the systematics, morphology and distribution of Asian Clawed Salamanders, genusOnychodactylus(Amphibia, Caudata: Hynobiidae), with the description of four new species[J]. Zootaxa, 2012(3465): 1-106.
[3]Boulenger GA. First report on additions to the Batrachian collection in the Natural History Museum[J]. Proceedings of the Zoological Society of London, 1886(29):411-416.
[4]趙文閣.黑龍江省兩棲爬行動物志[M].北京:科學出版社,2008.
[5]李建立.岫巖縣爪鯢生物學及其棲息地的保護[J].四川動物,2004(3):169-171.
[6]王麗文,趙艷艷,梁傳成.爪鯢骨骼系統的研究[J].四川動物,2004(3):172-177.
[7]陸宇燕,王建,劉新海,等.爪鯢皮膚的顯微結構和呼吸作用[J].四川動物,2004(3):178-182.
[8]WANG Huan-Huan. Morphological and histological studies on the telencephalon of the salamanderOnychodactylusfischeri[J]. Neuroscience Bulletin, 2007(3): 170-174.
[9]趙艷艷,王麗文,梁傳成,等.爪鯢消化系統的解剖學和組織學初步研究[J].四川動物,2004(3):290-293.
[10]王麗文,梁傳成,黃薇,等.環境溫度對爪鯢體溫及能量代謝的影響[J].動物學報,2008(4):640-644.
[11]晁賀,張勇岡,王勇,等.爪鯢生長與耗氧率的胚后發育[J].安徽農業科學,2013(5):1966-1968.
[12]孟翔宇.鞍山市爪鯢保護區現狀分析[J].遼寧農業科學,2013(3):68-69.
[13]樸正吉,張永虎.長白山爪鯢生態的初步觀察[J].吉林農業科技,1990(1):1005-7129.
[14]Yoshikawa N, Matsui M, Nishikawa K, et al. Phylogenetic relationships and biogeography of the Japanese clawed salamander,Onychodactylusjaponicus(Amphibia: Caudata: Hynobiidae), and its congener inferred from the mitochondrial cytochrome b gene[J]. Molecular Phylogenetics and Evolution, 2008(49): 249-259.
[15]馬靜,安永平,王彩芬,等.遺傳多樣性研究進展[J].陜西農業科學,2010(1):126-130.
[16]Thompson JD, Gibson TJ, Plewniak F, et al. The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research, 1997(25):4876-4882.
[17]Allendorf FW, Leary RF. Heterozygosity and fitness in natural populations of animals[M].In: Soulé ME ed. Conservation biology: the science of scarcity and diversity,Sunderland: Sinauer Associates, 1986.
[18]Librado P, Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009(25):1451-1452.
[19]Tamura K, Dunley J, Nei M, et al. MEGA4: Molecular evolutionary genetics analysis(MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007(24): 1596-1599.
[20]Excoffier L, Laval G, Schneider S. Arlequin ver. 3.0: An integrated software package for population genetics data analysis[J].Evolutionary Bioinformatics Online, 2005(1): 47-50.
[21]Clement M, Posada D, Crandall K A. TCS: acomputer program to estimate gene genealogies [J]. Molecular Ecology, 2000(10): 1657-1660.
[22]Lesser MR, Parchman TL, Jackson ST. Development of genetic diversity, differentiation and structure over 500 years in four ponderosa pine populations[J]. Molecular Ecology,2013(22): 2640-2652.
[23]韓樹才.爪鯢自然保護小區建設與資源現狀[J].黑龍江科技信息,2015(14):270.

