基于DNA條形碼對南極Yelcho站周邊海域魚類的種類鑒定
李淵 張然 宋普慶 李海 陳堅 林龍山
(國家海洋局第三海洋研究所, 福建 廈門 361005)
0 引言
南大洋具有獨特的海洋生態環境, 常年低溫、海冰漂浮、初級生產力波動劇烈等是其主要的特征, 而南極魚類則是該海域具有代表性的種類之一, 在食物網中具有重要的位置, 在經歷了顯著的輻射進化后已適應了南極大尺度極端的生態系統[1]。南極魚類大部分為具有區域性特征的底棲魚類, 主要隸屬于鱸形目(Perciform)、南極魚亞目(suborder Notothenioidei), 其中常見的5個科為阿氏龍科(Artedidraconidae)、淵龍科(Bathydraconidae)、冰魚科(Channichthyidae)、裸南極魚科(Harpagiferidae)和南極魚科(Nototheniidae),而南極魚科種類占南極海域魚類種類的70%以上,生物量占91%以上[2]。
以線粒體COI基因部分序列作為DNA條形碼, 該技術一經提出就被廣泛應用[3], 因為它實現了物種鑒定的標準化, 不再完全依賴于樣品的完整性和鑒定者的經驗[4-5], 如今該標記在物種鑒定方面的優勢越來越明顯。在物種間的條形碼比較方面, 同種內的差異較小, 而種間的差異顯著, 同時該標記還被成功用于隱存多樣性發掘[6-7]、新紀錄種或新種的發現[8-9]及魚類浮游生物鑒定等方面的研究[2,5,10]。
Yelcho站(64°62′S, 63°35′W)是智利南極度夏科考站, 位于帕默群島(Palmer Islands), 有關該站周邊海域海洋生物的研究較少, 僅見智利南極研究所(Chilean Antarctic Institute, INACH)內部研究報道, 而南極不同海域魚類的地域性較強, 種類組成差異較大, 因此, 有必要采用DNA條形碼技術對該海域的魚類樣品進行準確的種類鑒定,積累南極魚類分子遺傳學信息, 也為今后不同海域南極魚類的比較研究提供基礎數據。
1 材料與方法
1.1 樣品采集與鑒定
基于中國第32次南極科學考察暨中智第2次聯合南極半島綜合科學考察, 于2016年2月在帕默群島Yelcho站周邊海域, 利用地籠網捕獲8尾南極魚類成魚。依據Fischer和Hureau[11]的分類標準對所有魚類樣品進行形態學鑒定, 并對其進行編號,測量其體長和體重, 體長范圍為234—309 mm, 體重范圍為155.2—332.1 g。所有南極魚類中文名稱參考《拉漢世界魚類系統名典》[12]。所有樣品帶回實驗室進行后續研究, 目前樣品保存在國家海洋局第三海洋研究所海洋生物與生態實驗室。
1.2 實驗方法
用剪刀剪取南極魚類適量背部肌肉組織進行酒精固定, 采用DNA提取試劑盒提取總基因組DNA, 4℃保存備用。用于擴增COI片段的引物為[3]:F1: 5′-TCAACCAACCACAAAGACATTGGCAC-3′;R1: 5′-TAGACTTCTGGGTGGCCAAAGAATCA-3′。PCR反應體系為25 mL[13]: 0.15 μLTaqDNA聚合酶, 2.5 μL dNTP(2 mmol?L–1), 2 μL 10×Taq buffer(含Mg2+), 正反引物各1 μL(2 mmol?L–1), DNA模板1 μL, 其余dd H2O補足。反應條件為: 95℃預變性5 min; 95℃變性30 s, 50℃退火30 s, 72℃延伸30 s, 30個循環; 72℃延伸10 min。對符合測序要求的純化PCR產物送生物有限公司進行雙向測序。
結合形態學的鑒定結果, 下載GenBank中革首南極魚(Notothenia coriiceps)、花紋南極魚(Notothenia rossii)、喬奇裸南極魚(Harpagifer georgianus)和伯氏肩孔南極魚(Trematomus bernacchii)的同源序列用于分子遺傳學比較分析(表1)。
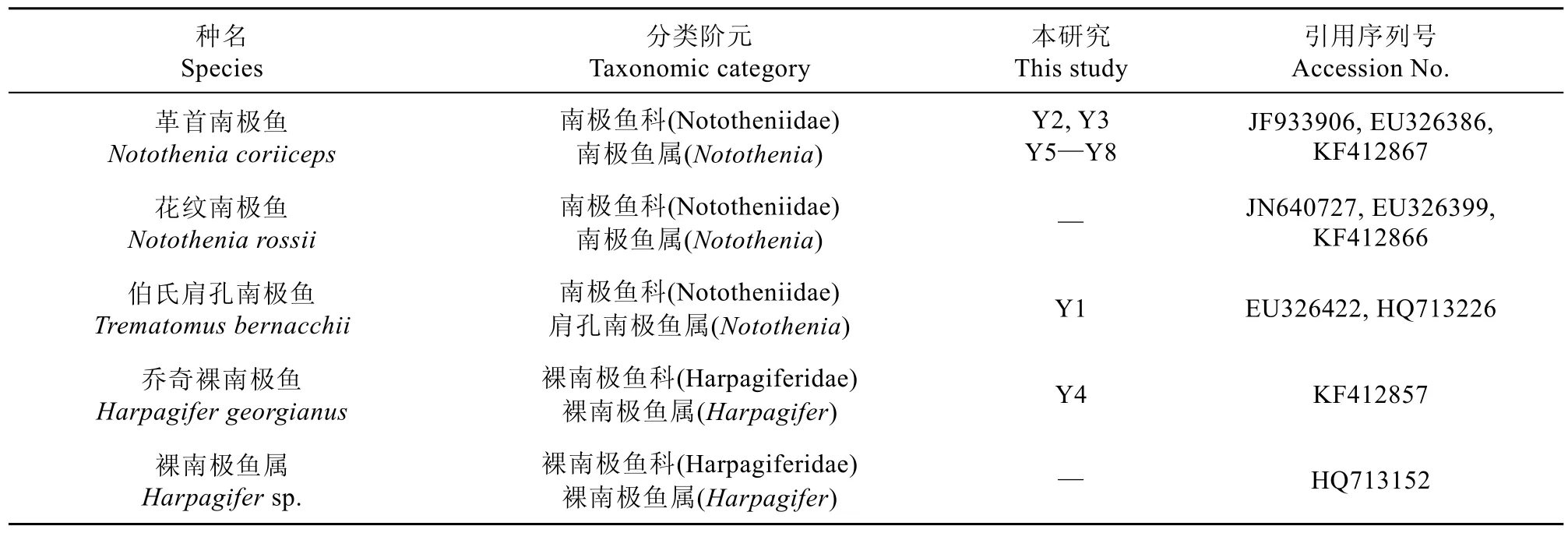
表1 研究所用南極魚類樣品和序列信息Table1. Information of Antarctic fishes’ samples and investigated sequences
1.3 數據處理
對測得的原始序列進行比對及校正, 獲得目的片段用于后續分析。將所有序列在NCBI數據庫中進行BLAST序列相似性比對, 采用兩兩序列間遺傳相似度>98%的為同一物種, 92%—98%為同一屬, 85%—91%為同一科的標準對采集的南極魚類進行比對和種類鑒定, 選取最佳匹配值(the best match)和種間最佳匹配值(interspecific best match)[5-6]。選取2%為種間的遺傳分化界限, 對所有比對結果劃分為3種模式[5,10]。模式Ⅰ是鑒定到種(match to species): 最佳比對值(the best match)低于遺傳分化界限, 最佳鄰接比對值(the nearest neighbor best match)高于遺傳分化界限, 該模式下能鑒定到種; 模式Ⅱ是模糊種(uncertain match):最佳比對值和最佳鄰接比對值均低于遺傳分化界限, 該模式下鑒定到的種是不確定的或者錯誤的;模式Ⅲ是無法匹配種(unmatched): 最佳比對值和最佳鄰接比對值均高于遺傳分化界限, 該模式下由于條形碼參考數據庫(reference library)的限制導致物種無法鑒定。基于K2P模型利用Mega 4.0軟件計算各物種間的變異位點、簡約信息為點、種內遺傳距離、種間遺傳距離、條形碼間隙(barcoding gap)等參數, 并構建鄰接關系樹[14-15]。
2 結果
2.1 分子多態性
經形態學鑒定后, 發現8尾南極魚類中存在3個種, 分別為革首南極魚(Notothenia coriiceps)、伯氏肩孔南極魚(Trematomus ber-nacchii)和喬奇裸南極魚(Harpagifer georgianus)(表1)。為了進一步確認形態鑒定結果的準確性,利用COI基因片段通用引物對8尾南極魚類進行擴增并雙向測序, 去除引物得到目的片段長度655 bp, 結合GenBank中的同源序列最終截取608 bp用于后續分析。8尾樣品中保守位點484個, 變異位點124個(圖1), 簡約信息位點123, 單一信息位點1個, 無插入/缺失位點。A+T堿基含量(53.5%)明顯高于G+C堿基含量(46.5%)(圖2)。

圖1 8尾南極魚類部分COI基因序列的變異位點Fig.1. Variable sites of partly sequences of COI gene among eight Antarctic fishes
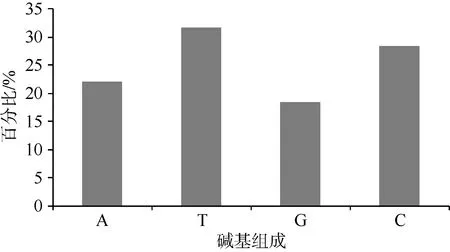
圖2 8尾南極魚類部分COI基因序列的堿基組成比例Fig.2. Percentage of base composition of partly sequences of COI gene among eight Antarctic fishes
對所有8條目的序列進行相似性比對發現,序列相似度(最佳匹配值)均在98%以上, 為了檢測比對結果的準確性, 同時對每一個體的種間最佳匹配值進行統計。以相似度98%作為同一物種的界限(2%為種間的遺傳分化界限)對所有個體進行分析, 結果顯示有2個個體屬于模式Ⅰ(25%),能準確鑒定到種; 有6個個體屬于模式Ⅱ(75%),屬于模糊種, 個體Y2、Y3、Y5—Y8對應兩種相似度在99%以上的物種(N. coriiceps和N. rossii);沒有個體出現在模式Ⅲ中(圖3)。
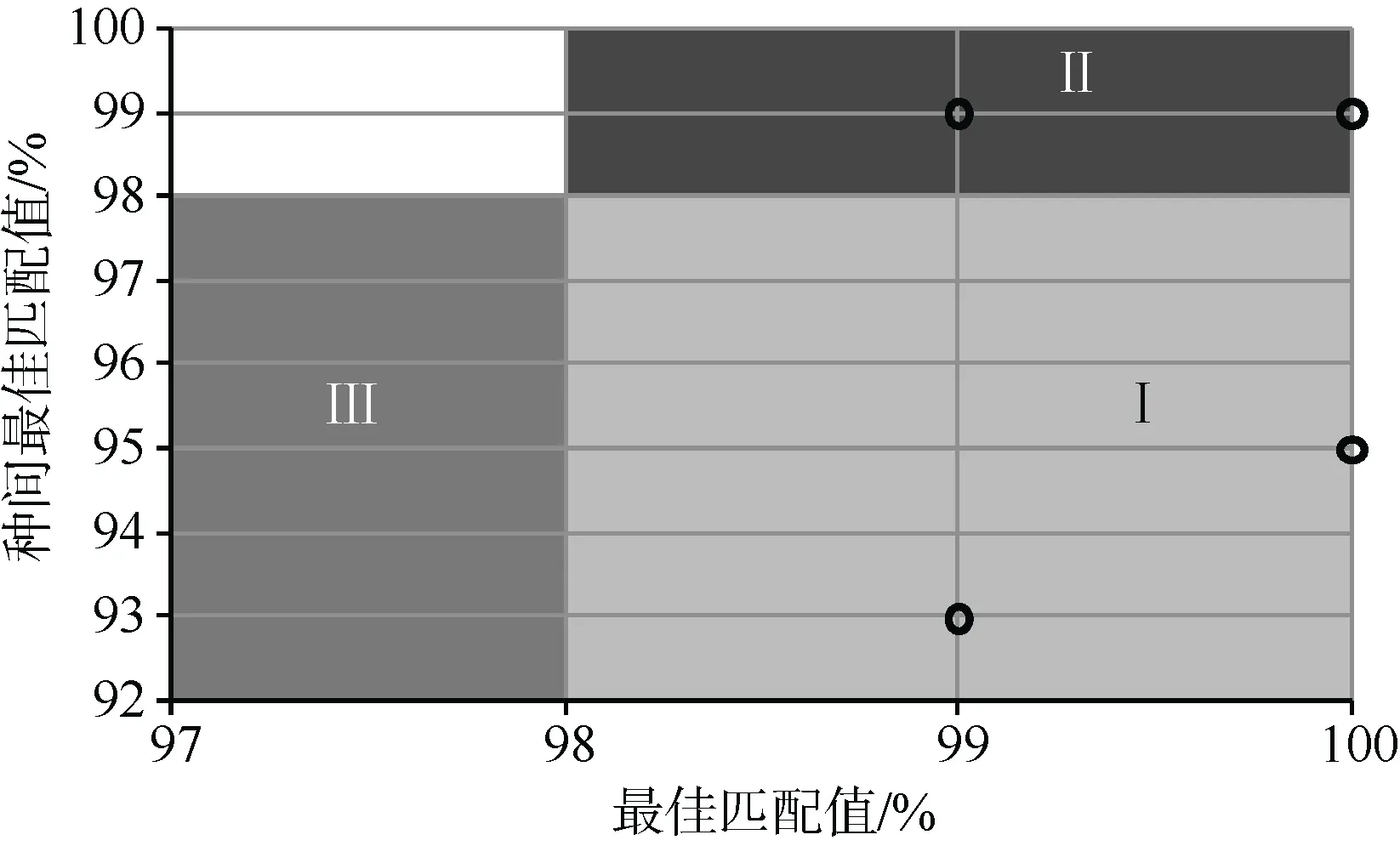
圖3 每個個體最佳匹配值和種間最佳匹配值之間的比較, 黑色線代表物種間分化的界限. Ⅰ-鑒定到種;Ⅱ-模糊種; Ⅲ-無法匹配種)Fig.3. Best match compared with nearest neighbor (similarity percentage) for each specimen. Dotted lines correspond to 2% divergence threshold for species boundaries. Ⅰ- match to species, Ⅱ- ambiguous species, Ⅲ- unmatched species
2.2 遺傳結構
結合GenBank中下載的同源序列構建NJ系統發育樹, 結果顯示所有序列明顯分為4個組群,其中組群1有9條序列組成, 組群2、組群3和組群4均有3條序列組成(圖4)。結合形態學鑒定結果可以發現, 組群1為革首南極魚, 但其中KF412866是以N. rossii(花紋南極魚)為學名提交;組群2為花紋南極魚, 但KF412867是以N.coriiceps(革首南極魚)為學名提交; 組群3為喬奇裸南極魚, 其中HQ713152以Harpagifersp.學為名提交; 組群4為伯氏肩孔南極魚。
2.3 DNA條形碼間隙
基于核苷酸最佳替換模型K2P模型, 分別計算4個組群間和組群內的遺傳距離(表2), 結果顯示各種間的遺傳距離范圍為0.039—0.176, 均高于2%的種間遺傳分化界限, 明顯為4個有效種;各組群內的遺傳距離范圍為0—0.004, 均低于2%的種間的遺傳分化界限, 明顯屬于種內差異。種間遺傳距離和種內遺傳距離基本符合種屬間的“10倍法則”[16]。4種南極魚類的最大種內遺傳距均低于該物種的最小種間遺傳距離, 圖5顯示種間和種內的遺傳距離間存在明顯的間隙, 即DNA條形碼間隙, 進一步證實各物種的有效性。

圖4 基于所有個體構建鄰接關系樹. 魚類圖片引自文獻[11]Fig.4. Neighbor-joining tree for all fish individuals based on COI sequences. Pictures of Antarctic fishes cited from reference [11]
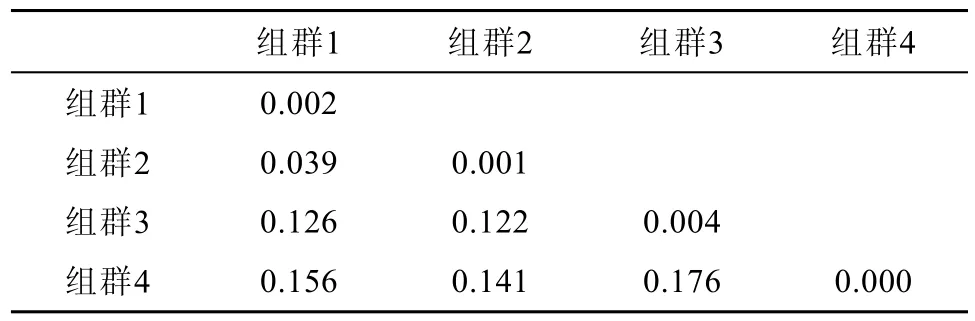
表2 4個組群基于COI基因序列的組內(對角線)和組間(下三角)的遺傳距離Table 2. Genetic distances of COI within (on the diagonal)and among (below the diagonal) four groups
3 討論
南極魚類不同海域間物種組成差異顯著[1-2],尤其在雪龍號科考船無法到達的Yelcho站海域采集的魚類樣品更顯得彌足珍貴, 因此有必要對采集到的魚類樣品進行準確物種鑒定, 為后續的研究提供科學參考。

圖5 基于K2P模型計算各物種的DNA條形碼間隙. 種間遺傳距離的最大值和最小值分別用上下橫杠進行表示, 中間橫杠為中位值; 藍線代表最大種內遺傳距離; 紅線代表平均種內遺傳距離Fig.5. DNA barcoding gaps for all of the species based on the K2P model. Median interspecific distances with maximum and minimum values are represented by the upper and lower bars, respectively; Blue line:Maximum intraspecific distance; Red line: Mean intraspecific distance
由序列相似性比對結果看出, 8尾魚中僅2尾能準確鑒定到種, 有6尾屬于模糊種。為了進一步確認物種的準確性, 本研究結合GenBank中的同源序列進行系統發育分析發現, 所有序列明顯分為4個組群, 其中8尾樣品明顯分為3個組群。結合形態學鑒定結果可以確定組群1為革首南極魚,組群2為花紋南極魚, 組群3為喬奇裸南極魚, 組群4為伯氏肩孔南極魚。由系統發育樹中可以看出KF412866和KF412867均以錯誤的學名進行提交, 但筆者認為造成這種錯誤的原因是序列提交者在序列提交時混淆了序列編號, 而非最初將兩種魚類進行錯誤鑒定, 畢竟這兩種魚類在胸鰭鰭條數上差異明顯(革首南極魚16—17 VS 花紋南極魚21—24)。Harpagifersp.(HQ713152)實際為喬奇裸南極魚。由此可以看出本研究不但對條形碼參考數據庫中的錯誤序列進行糾正, 還能對以前研究中的未定種類進行種類鑒定。4個組群間的遺傳距離明顯高于種間遺傳分化界限, 而各物種的種內遺傳距離均低于種間的遺傳分化界限, 基本符合種屬間的“10倍法則”[16], 且各物種內均存在明顯的DNA條形碼間隙, 進一步驗證了各物種的有效性。
綜合上述結果可以看出, 以COI基因片段作為DNA條形碼與形態學相結合可成功用于南極海域魚類物種準確鑒定, 8尾樣品內存在3個有效種, 其中組群1可以作為革首南極魚鑒別的DNA條形碼,組群3可以作為喬奇裸南極魚鑒別的DNA條形碼,組群4可以作為伯氏肩孔南極魚鑒別的DNA條形碼; 獲取的樣品中有7尾南極魚科魚類, 1尾裸南極魚科魚類, 該結果也在一定程度上反映出Yelcho站周邊海域在物種組成和數量分布方面均以南極魚科魚類為主[2]。
因此, 將傳統的形態學和分子遺傳學相結合對物種進行分類才能確保物種鑒定的準確性, 正確的形態鑒定能保證條形碼參考數據庫的準確性,準確的條形碼又能指導后續的物種鑒定[5], 二者相輔相成, 形成良好的物種鑒定循環, 有助于今后的種類鑒定、隱存種的發現、多樣性調查及系統發育等研究。
4 結論
利用形態學和DNA條形碼技術能成功鑒定出Yelcho站附近海域采集到的8尾魚類樣品, 3種魚類的種內和種間遺傳界限明顯, 基本符合種屬間的“10倍法則”, 且各物種內均存在明顯的DNA條形碼間隙, 其中南極魚科魚類有7尾, 在一定程度上反映出Yelcho站周邊海域魚類在物種組成和生物量上均以南極魚科魚類為主。
1 BARGELLONI L, MARCATO S, ZANE L, et al. Mitochondrial phylogeny of Notothenioids: a molecular approach to Antarctic fish evolution and biogeography[J]. Systematic Biology, 2000, 49(1): 114—129.
2 MURPHY K R, KALMANEK E A, CHENG C H C. Diversity and biogeography of larval and juvenile notothenioid fishes in McMurdo Sound, Antarctica[J]. Polar Biology, 2017, 40(1): 161—176.
3 HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Soci-ety B: Biological Sciences, 2003, 270(1512): 313—321.
4 李海濤, 張保學, 高陽, 等. DNA條形碼技術在海洋貝類鑒定中的實踐: 以大亞灣生態監控區為例[J]. 生物多樣性, 2015, 23(3):299—305.
5 李淵, 張麗艷, 張然, 等. 基于DNA條形碼技術對蒼南海域仔稚魚的物種鑒定[J]. 中國海洋大學學報, 2017, 47(12): 72—79.
6 CHEN W T, MA X H, SHEN Y J, et al. The fish diversity in the upper reaches of the Salween River, Nujiang River, revealed by DNA barcoding[J]. Scientific Reports, 2015, 5: 17437, doi: 10.1038/srep17437.
7 DIVYA P R, MOHITHA C, RAHUL G K, et al. Molecular based phylogenetic species recognition in the genusPampus(Perciformes:Stromateidae) reveals hidden diversity in the Indian Ocean[J]. Molecular Phylogenetics and Evolution, 2017, 109: 240—245.
8 TORNABENE L, VAN TASSELL J L, GILMORE R G, et al. Molecular phylogeny, analysis of character evolution, and submersible collections enable a new classification of a diverse group of gobies (Teleostei: Gobiidae:Nessubgroup), including nine new species and four new genera[J]. Zoological Journal of the Linnean Society, 2016, 177(4): 764—812.
9 XIAO J G, SONG N, HAN Z Q, et al. Description and DNA barcoding of a newSillagospecies,Sillago shaoi(Perciformes: Sillaginidae), in the Taiwan Strait[J]. Zoological Studies, 2016, 55: 47, doi: 10.66201ZS. 2016. 55—47.
10 HUBERT N, ESPIAU B, MEYER C, et al. Identifying the ichthyoplankton of a coral reef using DNA barcodes[J]. Molecular Ecology Resources, 2015, 15(1): 57—67.
11 FISCHER W, HUREAU J C. FAO Species Identification Sheets for Fishery Purposes: Southern Ocean: Fishing Areas 48, 58 and 88(CCAMLR Convention Area) (Volume II)[M]. Rome: Food and Agriculture Organization of the United Nations, 1985: 282-284, 323—385.
12 伍漢霖, 邵廣昭, 賴春福, 等. 拉漢世界魚類系統名典[M]. 青島: 中國海洋大學出版社, 2017: 281—283.
13 李淵, 宋娜, KHAN F S, 等. 銀鯧形態特征與DNA條形碼研究[J]. 水產學報, 2013, 37(11): 1601—1608.
14 HEBERT P D N, STOECKLE M Y, ZEMLAK T S, et al. Identification of birds through DNA barcodes[J]. PLoS Biology, 2004, 2(10):e312, doi: 10.1371/journal.pbio.0020312.
15 SHEN Y J, GUAN L H, WANG D Q, et al. DNA barcoding and evaluation of genetic diversity in Cyprinidae fish in the midstream of the Yangtze River[J]. Ecology and Evolution, 2016, 6(9): 2702—2713.
16 WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia’s fish species[J]. Philosophical Transactions of the Royal Society B: Biological Sciences, 2005, 360(1462): 1847—1857.

