乙醇脫氫核酶ribox02與NAD+/NADH結合位點的探測
李丹彬 張 靜 陳東戎
(復旦大學生物醫學研究院RNA生物學實驗室 上海 200032 )
NAD+和NADH是細胞能量代謝所必需的輔酶[1]。細胞內氧化還原狀態特別是 NAD+/NADH比值直接影響著細胞的節律、衰老、癌變和死亡。近年來,細胞內與NAD+和NADH相關的物質代謝研究倍受關注。
乙醇脫氫酶(alcohol dehydrogenase,ADH)能以煙酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NAD+)為輔酶介導伯醇和醛之間的可逆反應。2003年,Tsukiji等[2]發現,具有與ADH相似催化功能的核酶ribox02與反應底物(苯甲醇/苯甲醛)之間的空間距離足夠接近且在NAD+與Zn2+同時存在時ribox02能催化苯甲醇反應生成苯甲醛,同時NAD+被還原為NADH;之后又發現核酶ribox02能夠在NADH和Zn2+同時存在時催化苯甲醇生成苯甲醛,而NADH被氧化生成NAD+,即ribox02 RNA 能以NAD+和NADH為輔酶催化苯甲醛與苯甲醇之間的可逆反應[3],且以NADH為輔酶生成醇的速度比以NAD+為輔酶催化生成醛的速度快[2-3]。研究人員通過觀察突變后ribox02的活性變化來推測酶活性位點[2-4],間接驗證法所得結果與mfold預測的結構相結合獲得ribox02的二級結構。但該二級結構來自于間接推測,真實結果有待驗證。通過突變實驗獲得ribox02全部活性的最小功能單位,去除66U~79G區域序列可使酶活性全部喪失,敲除L4序列也能大大降低酶活性,而去除或突變該RNA其他位點并未對酶活性產生明顯影響,因此預測 ribox02 與輔酶小分子的結合位點可能位于GU序列較為豐富的J1/4區,而ribox02與NAD+和NADH的作用位點及結合機制尚不明確。
選擇性2-OH酰基化學探測法(selective 2-hydroxyl acylation analyzed by primer extension,SHAPE)是基于FAM 引物引導的逆轉錄反應的RNA二級結構探測技術[5],非配對的核苷酸的 2’-OH 相比于配對的或其他限制性的核苷酸更容易受到親核攻擊,而NMIA 作為一種羥基選擇性化學試劑能更迅速地與非配對的 RNA 核苷酸形成穩定的復合物。逆轉錄酶可在酰基復合物形成位點特異性終止延伸反應,從而產生特異性長度的 FAM-cDNA 熒光信號,而未經NMIA修飾的核苷酸位點FAM-cDNA的信號則處于本底水平。根據cDNA熒光信號的強度,可推測出 RNA各個核苷酸的配對情況,從而直接精確探測RNA的二級結構。
等溫滴定量熱法(isothermal titratio calorimetry,ITC)是通過檢測樣品池補償功率的變化來擬合計算結合常數及結合熱力學參數反應的熱力學技術,在類似于化學滴定的過程中跟蹤化學反應的熱流隨時間的變化[6-7],可用于檢測NAD+、NADH與ribox02的特異性結合。
紫外交聯技術(UV-crosslinking)的原理是基于紫外照射能激發核酸上的堿基,處于激發態的堿基能與數埃范圍內含有不飽和雙鍵的抗生素發生交聯結合,且這種結合是不可逆的共價交聯[8],通過帶FAM熒光引物引導的逆轉錄反應可得到抗生素結合在RNA上的位置信息,從而精準定位NAD+、NADH與核酶ribox02的特異性結合位點。
本研究旨在揭示核酶ribox02與NAD+、NADH特異性結合的位點及其結合力性質,以期為核酶ribox02的應用提供一定的理論基礎。
資 料 和 方 法
質粒和菌株質粒pGEx-4T-1由本實驗室構建,大腸埃希菌JM109購于上海生工生物工程有限公司。
試劑氨芐青霉素;LB培養基;LB固體培養基;1%胰蛋白酶;0.5%酵母提取液;1%氯化鈉;1.5%和1%瓊脂糖凝膠;1 mol/L三羥甲基氨基甲烷鹽酸鹽(pH=8.0);0.5 mol/L乙二胺四乙酸;3 mol/L醋酸鈉;1 mol/L 二硫蘇糖醇;5-溴-4-氯-3-吲哚-β-D-半乳糖苷;異丙基-β-D-硫代半乳糖苷;40%(w/v)丙烯酰胺;5×體外轉錄緩沖液;200 mmol/L 3-羥甲基氨基甲烷鹽酸鹽;0.1%(v/v)聚乙二醇辛基苯基;100 mmol/L氯化鎂;10 mmol/L亞精胺;5×TBE電泳緩沖液(pH=8.3):500 mmol/L 3-羥甲基氨基甲烷,400 mmol/L H3BO3,10 mmol/L乙二胺四乙酸;EK 緩沖液(pH=8.0):50 mmol/L 4-羥乙基哌嗪丙磺酸,500 mmol/L 氯化鉀,100 mmol/L氯化鎂,0.5 mmol/L氯化鋅。
分子克隆ribox02 RNA 序列:3’-GGG-AUCGUCAGUGCAUUGAGAUGUUUCUCAGA-CUGAUUAUCGUGAGACAGUGAUCUCGUGG-UCAGUCCUAGUUUGGGGGCUGGUCCUCACG-GUGGUAUCCCCAAGGGGUA-5’;ribox02-linke RNA序列:3’-GGGAUCGUCAGUGCAUUGAG-AUGUUUCUCAGACUGAUUAUCGUGAGACA-GUGAUCUCGUGGUCAGUCCUAGUUUGGGG-GCUGGUCCUCACGGUGGUAUCCCCAAGGGG-UAUCGAUCCGGUUCGCCGGAUCCAAAUCGG-GCUUCGGUCCGGUUC-5’。
ribox02是經過隨機組合篩選出來的RNA序列,無模板,故通過引物退火延伸獲得該序列,ribox02的DNA序列加上保護堿基,酶切位點以及T7序列后所得序列如下:3’-CTAGTCTAGATA-ATACGACTCACTATAGGGGGGATCGTCAGT-GCATTGAGATGTTTCTCAGACTGATTATCG-TGAGACAGTGATCTCGTGGTCAGTCCTAGT-TTGGGGGCTGGTCCTCACGGTGGTATCCCCA-AGGGGTAGGATCCGCG-5’ (保護堿基+XbaⅠ+T7 seq+ribox02 seq+BamHⅠ+保護堿基)。
將待研究序列放入Helix Systems的DNA Works中,得到合成ribox02序列所需的寡核苷酸片段:Oligos 1:3’-CTAGTCTAGATAATACGACTCA-CTATAGG-5’;Oligos 2:3’-CAATGCACTGAC-GATCCCCCCTATAGTGAGTCGTATTATCTAG-ACT-5’;Oligos 3:3’-GGGGATCGTCAGTGCA-TTGAGATGTTTCTCAGACTGATTATCG-5’;Oligos 4:3’-AGGACTGACCACGAGATCACT-GTCTCACGATAATCAGTCTGAGAAACATCT-5’;Oligos 5:3’-TGATCTCGTGGTCAGTCCTA-GTTTGGGGGCTGGTCCTCACGGTGGTATCCC-CA-5’;Oligos 6:3’-CGCGGATCCTACCCCTTG-GGGATACCACCGTGA-5’。
退火延伸得到的DNA片段與載體質粒pGEx-4T-1中加入內切酶XbaⅠ和BamHⅠ,37 ℃反應1.5 h。分別加入4 μL 10×上樣緩沖液,將酶切產物通過1%瓊脂糖凝膠電泳分離,割膠回收;0.3 pmol DNA片段與0.03 pmol載體在T4 DNA 連接酶作用下進行連接,連接產物轉化到感受態大腸埃希菌JM109中,涂板,37 ℃培養過夜,第2天挑取單菌落測序,測序正確的單克隆菌株體內所含質粒即可作為體外轉錄RNA的DNA模板。
體外轉錄設計含有T7RNA聚合酶啟動子的引物以及帶有連接片段linker的引物,通過PCR擴增獲得轉錄模板。PCR體系:2 μL ribox02上游引物,2 μL linker下游引物,0.5 μL質粒模板,25 μL 2×Phanta SF反應緩沖液,4 μL dNTP混合物,1 μL Phanta聚合酶,18.5 μL H2O。反應過程:95 ℃變性10 s,56 ℃退火15 s,72 ℃延伸45 s,30個循環,72 ℃延伸10 min。乙醇沉淀PCR產物,適量水溶解PCR產物作為轉錄體系的模板。500 μL轉錄體系:50 μL 10×轉錄緩沖液,5 μL二硫蘇糖醇(1 mol/L),112.5 μL rNTP混合物(25 mmol/L),25 μL DNA 模板(700 ng/μL),12.5 μL RNA核酶抑制劑,50 μL T7 RNA 聚合酶,245 μL超純水。37 ℃反應過夜,加1 μL脫氧核酶Ⅰ(500 U/μL),37 ℃反應1 h;加入等體積甲酰胺(含溴酚藍和二甲苯氰),95 ℃變性5 min,冰上保存待用。
SHAPE和紫外交聯實驗結果都通過逆轉錄引物延伸反應來展示,而引物延伸會使RNA的5’端的引物結合部位信息缺失,因此SHAPE和紫外交聯實驗所用的ribox02-linker RNA與ITC實驗所用的ribox02 RNA不一樣,ribox02-linker RNA的下游引物前面加上一段不會影響RNA折疊的linker 序列。ribox02上游引物:3’-TAATACGA-CTCACTATAGGGGGGA-5’;ribox02下游引物:3’-TACCCCTTGGG-GATACCACCG-5’;ribox02-linker下游引物:3’-GAACCGGACCGAAGCCC-GATTTGGATCCGGCGAACCGGATCGATACC-CCTTGGGGATACCACCGTGAGG-5’。ribox02上游引物作為通用引物,可用于2種RNA的合成體外轉錄體系中。ribox02上游引物與ribox02下游引物作為一對引物,合成的ribox02為后續ITC實驗所用;ribox02上游引物與ribox02-linker下游引物作為一對引物,合成的ribox02-linker為后續SHAPE實驗及紫外交聯實驗所用。
純化RNA配置12%的變性聚丙烯酰胺凝膠,電壓1 200 V,功率70 W,預電泳30 min直至玻璃膠板高于50 ℃,上樣,電泳6 h;脫膠置于保鮮膜上,以熒光板為背景,暗室操作,紫外燈照射發光,254 nm下核酸條帶呈黑色;切下條帶,切碎后放入50 mL康寧管中,加入約20 mL含醋酸鈉(0.3 mol/L,pH=5.2)和EDTA(1 mmol/L,pH=8.0)的透析緩沖液,4 ℃震蕩4 h,析出RNA;取溶液,用0.22 μm濾膜過濾除去聚丙烯酰胺膠顆粒,剩余膠帶進行第2次透析;透析后的溶液用乙醇沉淀,加入1/10體積的醋酸鈉(3 mol/L,pH=5.2)和2.5倍體積的無水乙醇,-80 ℃放置30 min,4 ℃下9 000×g離心15 min,棄上清;加入600 μL預冷的75%乙醇,4 ℃下9 000×g離心5 min,棄上清(重復上述步驟),室溫晾干制成RNA干粉,-80 ℃保存備用。
SHAPE法用EK緩沖液溶解RNA干粉,根據260 nm處的吸光度(D)值來配置10 pmol/μL的ribox02-linker RNA,取1 μL RNA(10 pmol/μL)加入去核糖核酸酶的EP管中,加入17 μL EK緩沖液;4 ℃離心30 s,使溶液到達1.5 mL EP管底部,95 ℃變性5 min,自然冷卻至室溫,加入1 μL NMIA,37 ℃反應1 h;75%乙醇洗2次,晾干后用于后續逆轉錄。沉淀RNA中依次加入8 μL miliQ-H2O,2 μL特定的引物(10 μmol/L),5×逆轉錄緩沖液(4 μL),RNA marker反應體系還需加2 μL ddNTP (10 mmol/L),4 ℃離心30 s,65 ℃加熱5 min,取出后置于冰上,4 ℃離心30 s,EP管內加入1 μL二硫蘇糖醇(0.1 mol/L)、0.5 μL逆轉錄酶Ⅲ、0.5 μL核酶抑制劑(40 U/μL),混勻,55 ℃反應1 h;加入1 μL NaOH(4 mol/L),95 ℃變性5 min,冰上快速冷卻,乙醇沉淀。用19 μL H2O溶解RNA沉淀,取1 μL用于ABI自動測序儀3730進行測序,采用Peak Scanner軟件V1.0分析結果。
ITC法配制合適濃度的小分子和RNA:電子天平稱取NAD+和NADH各0.066 g分別溶于1 mL的EK緩沖液中,配制成母液(100 mmol/L),根據實驗需要進行稀釋。500 μL 轉錄體系獲得的干粉 RNA 用 500 μL EK緩沖液溶解,根據 RNA 在260 nm處的D值計算 RNA 濃度,根據實驗需要用EK緩沖液進行稀釋,稀釋后的RNA在95 ℃變性5 min,退火復性。小分子和RNA在4 ℃下9 000×g離心1 min,去氣泡。至少設置另外3個空白滴定實驗:配體滴緩沖液、緩沖液滴大分子溶液和緩沖液滴緩沖液。取60 μL樣品加入0.2 mL離心管中進行滴定實驗,加樣時應避免產生氣泡,設置樣品池溫度為25 ℃,間隔時間為120 s,滴定針數為20,實驗完成后采用Origin 7.0處理數據。
紫外交聯技術根據260 nm處的D值來配置10 pmol/μL RNA,取1 μL RNA于去核糖核酸酶的EP管中,加入EK緩沖液(去鎂離子),使體積達到18 μL;短暫離心,使溶液到達1.5 mL EP管底部,95 ℃加熱5 min,無干擾退火至室溫,加入1 μL MgCl2(2 mmol/L),25 ℃反應30 min;加入1 μL各濃度梯度的NAD+和NADH,25 ℃反應30 min;在冰盒中打開EP管,254 nm紫外光下照射5 min;反應結束后用75%乙醇洗2次,晾干,沉淀RNA用于后續逆轉錄。逆轉錄與SAHPE逆轉錄操作步驟相同。
結 果
ribox02RNA二級結構的探測在ribox02序列的SHAPE實驗結果中,未經NMIA修飾與經過NMIA修飾的RNA所產生的二級結構信號明顯不同。未經修飾的RNA經逆轉錄后在各個核苷酸位點不呈現FAM-cDNA的熒光信號差異,圖1A中紅色測序信號為ribox02 RNA經過NMIA修飾后的逆轉錄信號,黑色測序信號(作為對照樣本)為RNA未經化學試劑修飾所產生的信號圖,可見紅色測序信號有7個信號明顯較高的區域,即非配對單鏈區域。我們根據SHAPE數據結果繪測出ribox02 RNA的二級結構(圖1B)。
NAD+、NADH與ribox02RNA的相互作用因NADP+和NADPH的化學性質、吸收光譜、氧化還原形式等均與NAD+和NADH相似,故選取NADP+和NADPH作為陰性對照(實驗結果未公布)。一定濃度的NAD+和NADH分別滴定ribox02,數據經Origin 7.0軟件處理。由ITC反應中補償功率隨時間變化的曲線圖以及反應熱隨摩爾比變化的曲線圖中可看出,NADH能與ribox02 RNA進行迅速而有效的特異性結合(圖2A),平衡解離常數高達2.92×107±8.35×106(表1),而NAD+(圖2B)與NADH相比,呈現出較弱的親和力。由此可見,NADH與ribox02能更快、更有效地識別結合并催化反應。
對ITC原始數據反應熱函數用One Set of Sites模型進行擬合,這些結合參數包括結合反應的結合常數、焓變、熵變和化學計量比。此外,根據Ross等[9]的熱力學規律:ΔH0≥0且ΔS0>0,為疏水作用力;ΔH0<0且ΔS0>0,為靜電作用力;ΔH0<0且ΔS0<0,為范德華力或氫鍵作用,可以推斷NAD+、NADH與ribox02 RNA結合的作用力都為范德華力或氫鍵作用。
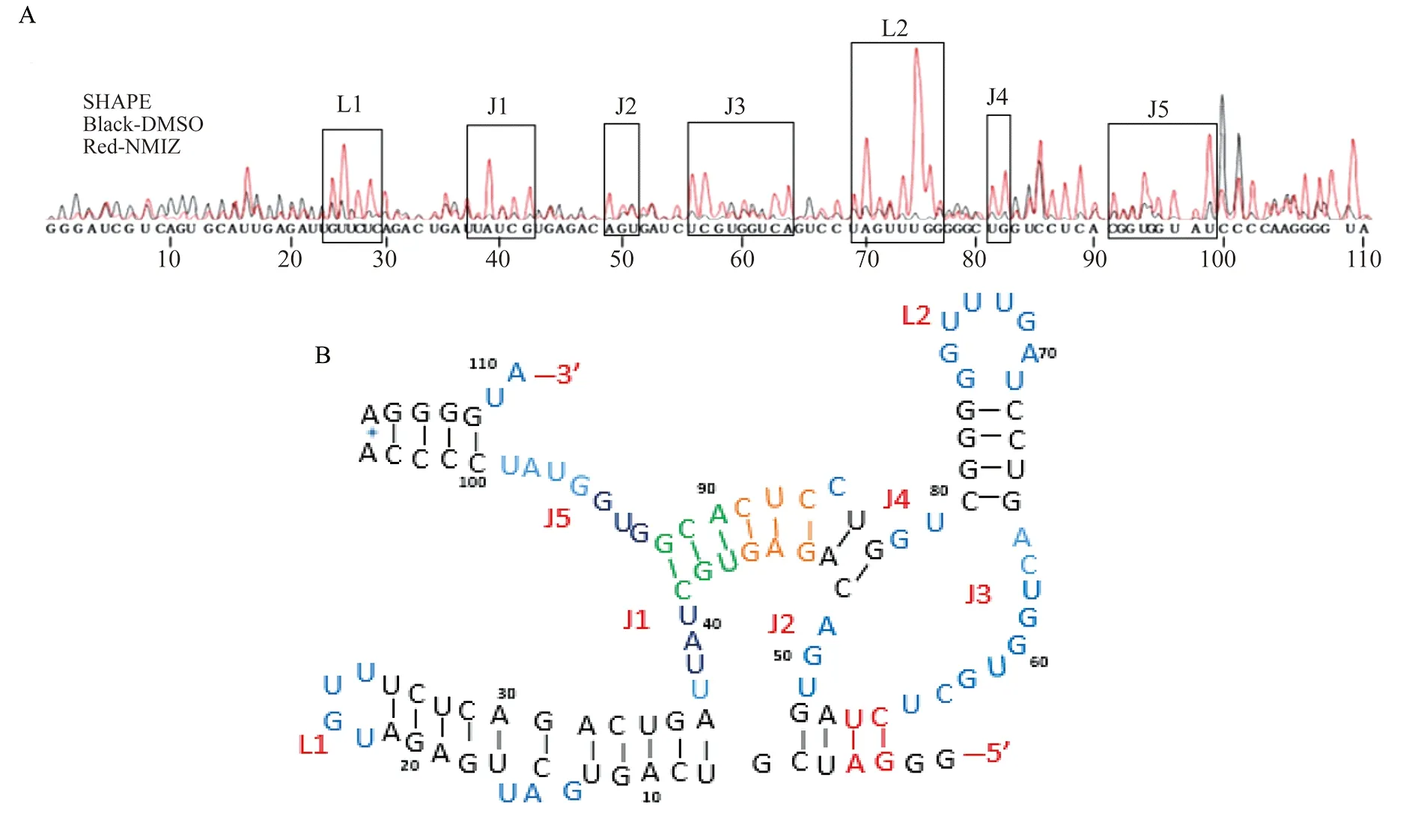
圖1 ribox02 RNA的SHAPE實驗結果(A)及其二級結構預測(B)Fig 1 SHAPE result of ribox02 RNA (A) and its secondary structure probed by SHAPE (B)

MicromoleculeKd (/mol)ΔH (cal/mol)ΔS(cal/deg) NADH2.92×107± 8.35×106-2.05×104±274.2-36.4NAD+9.15×103±2.18×104-1.472×104±1.681×104-32.4
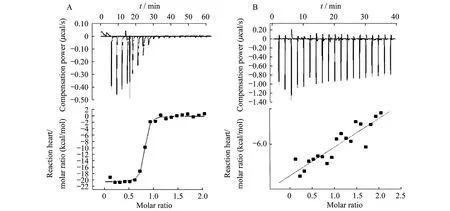
The upper curver showed compensation power changed with time in isthermal titration reaction,and the lower curver showed reaction heart changed with molar ratio.A:160 μmol/L NADH titraced 16 μmol/L ribox02 RNA;B:400 μmol/L NAD+tritraced 40 μmol/L ribox02 RNA.Reaction condition:25 ℃,a total of 20 needle,2 μL/needle,intervel of 120 s.
圖2ITC所得數據經Origin7.0處理后的曲線圖
Fig2GraphofITCdateprocessedbyOrigin7.0
紫外交聯技術探測NAD+、NADH與ribox02RNA的特異性結合位點ribox02 RNA分別與NADH、NAD+、NADP+和NADPH進行紫外交聯(圖3),逆轉錄后測序得到信號峰;其中藍色信號為 RNA 自我交聯的背景值,紅色信號為 RNA 與小分子交聯產生的信號峰。由圖3A、3B可見23G~25U、38U、72U~75G和95G這9個位點的信號值在加入小分子NAD+、NADH后均有明顯上升,這說明NAD+、NADH與RNA在上述位點結合并被交聯,對于NADP+、NADPH 并未檢測到交聯位點,這與ITC的實驗結果相一致。對比圖3A和圖3B,NADH所引起的信明號的改變比NAD+所引起的信號改變更為明顯,這也從側面驗證了ITC實驗所預測的結論,即相較于NAD+,NADH與ribox02能更快速而有效地識別結合并催化反應。在RNA二級結構上NAD+、NADH與ribox02的紫外交聯位點如圖4所示。
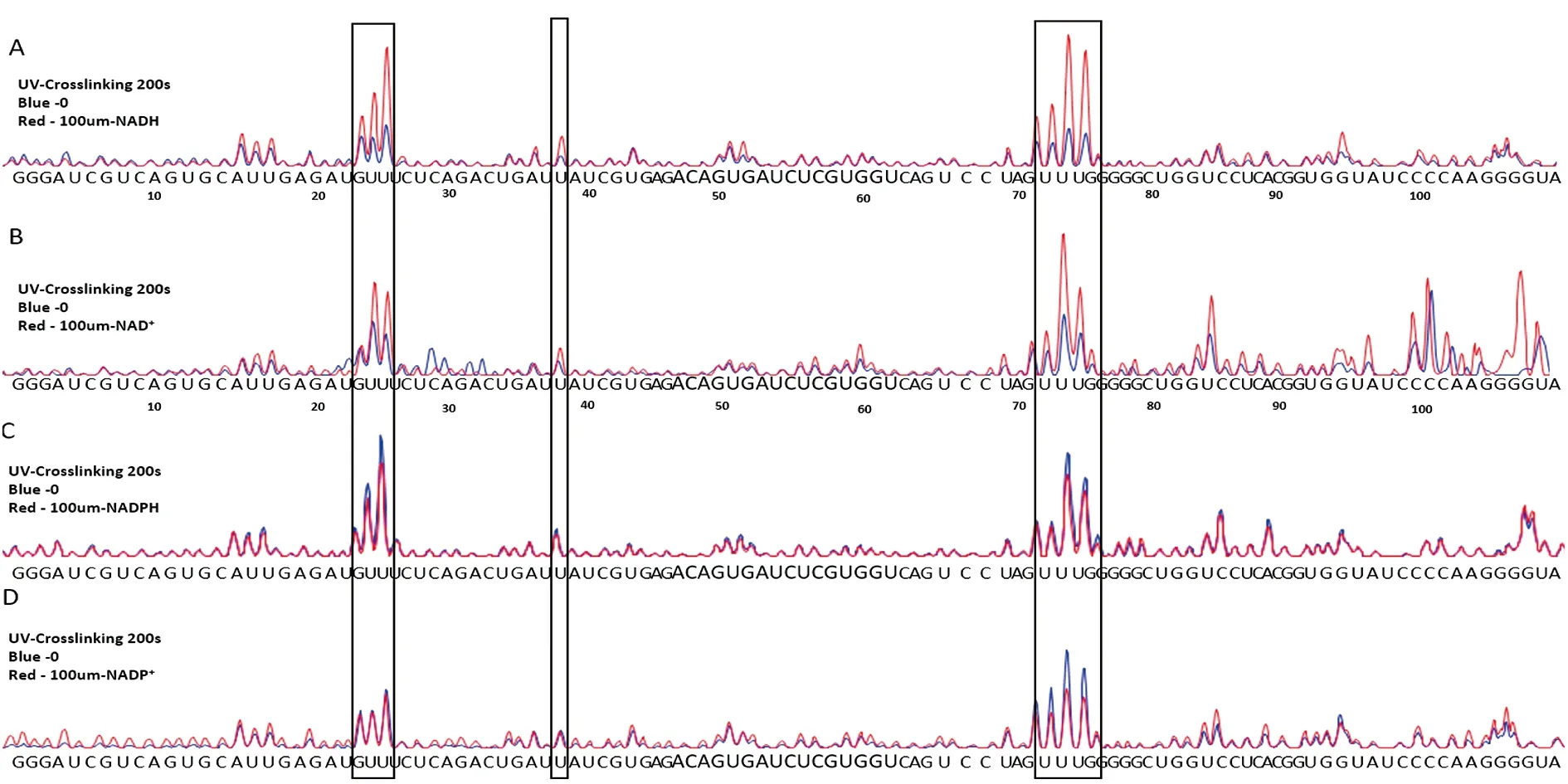
Blue signal:Background of RNA self crosslinking;Red signal:RNA crosslinking with the small molecules.
圖3ribox02RNA與NADH、NAD+、NADPH和NADP+的紫外交聯實驗結果
Fig3Theresultofribox02RNAcrosslinkedwithNADH,NAD+,NADPHandNADP+
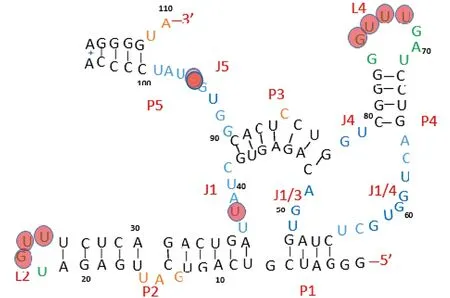
圖4NAD+/NADH與ribox02的紫外交聯位點在RNA二級結構上的圖示
Fig4UV-crosslinkingbindingsitesofNAD+/NADHonthesecondarystructureofribox02RNA
討 論
ITC實驗結果證實小分子NADH和NAD+能與ribox02 RNA結合,即在空間位置上小分子能夠與ribox02 RNA處于無限靠近的距離,而NADH、NAD+、NADP+和NADPH分子結構上都含有不飽和雙鍵,紫外交聯反應能夠使得小分子不可逆地以共價鍵的形式交聯在RNA上,經交聯的RNA通過FAM熒光引物引導逆轉錄反應,在交聯位點逆轉錄會終止,通過測序可知交聯位點就是NAD+、NADH與ribox02 RNA的結合位點。結合紫外交聯數據和SHPEA所探測到的ribox02 RNA二級結構(圖4)分析發現:(1)小分子與RNA的9個結合位點23G、24U、25U、38U、72U、73U、74U、75G和95G均為G/U,并未出現A/C,這是因為NAD+和NADH分子內含有腺嘌呤殘基,根據搖擺堿基對原則,NAD+和NADH更容易與G、U結合。(2)所有的結合位點均位于RNA的頸環結構,這是因為頸環結構的堿基在空間上更有利于與小分子進行結合。(3) L2區域中的72U~75G位點是NAD+、NADH的主要結合位點之一,這一結果也解釋了P4/L2區域之所以能夠顯著影響ribox02的酶活性,但主要結合位點并非位于Tsukiji等[2]預測的J1/4,而是位于L2區域72U~75G。(4) 這9個結合位點都存在于最小功能單位compact ribox02里,因此compact ribox02具有核酶ribox02的所有功能。(5)38U和95G是NAD+和NADH的重要結合位點。Tsukiji等[2]發現突變38U~40U和92G~94G對ribox02酶活性的影響很大,解釋為38U~40U和92G~94G應處于配對狀態,但這一推測并不嚴謹,結合SHAPE的結果,我們發現38U~40U和92G~94G處于非配對狀態,而該區域之所以能影響ribox02的活性,是因為38U與95G是NAD+和NADH的重要結合位點。(6) 輔酶小分子NAD+、NADH與RNA的結合位點主要分布在二級結構上,并不十分靠近L1、L2和J1這3個區域。這是因為任何RNA尤其是功能性RNA,在溶液中的結構絕不止二級結構這樣簡單,RNA會折疊為復雜的三維結構形式以行使其生物學功能。由小分子與RNA的結合位點可以推測,P4和L2借助J3面向L1彎曲折疊,與J1部位形成了一個三維空間“口袋”,從而為NADH和NAD+的特異性結合提供了空間可能性。
核酶可特異性與靶分子mRNA結合并促進mRNA裂解,且裂解完畢后自行脫離,再與其他靶分子RNA結合,因此可在不影響宿主細胞RNA的條件下,特異性結合并切割病毒RNA。利用核苷酸序列特異的酶切活性不僅可以研究特定基因的功能,也可利用此特點來尋找疾病治療的新靶標。鑒于核酶具有分子結構簡單、分子量小和易于合成并設計的優點,可望開發出具有治療作用的核酶制劑。目前已有不少核酶已經進入臨床試驗階段(如腫瘤治療和AIDS治療等)。

