miRNA調(diào)控植物抗逆機制的研究現(xiàn)狀
李昕晏 崔杰 李俊良 王琮玉 羅成飛
摘要:逆境脅迫會對植物的生長發(fā)育產(chǎn)生負面影響,在漫長的進化中,植物擁有了一套復(fù)雜的調(diào)控網(wǎng)絡(luò)以應(yīng)對不良環(huán)境,在這一套調(diào)控網(wǎng)絡(luò)中miRNA起到了重要作用。本文對近幾年關(guān)于miRNA及其靶基因調(diào)控作用的研究進行了總結(jié),并對miRNA的形成過程和作用機制進行了概括,介紹了與抗逆相關(guān)的miRNA及其靶基因,包括以靶基因為抗逆相關(guān)轉(zhuǎn)錄因子的miRNA(miR160/miR167、miR159、miR156和miR164)以及以靶基因為抗逆相關(guān)結(jié)構(gòu)基因的miRNA(miR398和miR395),可為利用基因工程手段增強植物的抗逆性提供更有效的改良途徑。
關(guān)鍵詞:miRNA;逆境脅迫;轉(zhuǎn)錄因子;結(jié)構(gòu)基因;靶基因
中圖分類號: S332.1;S184文獻標志碼: A
文章編號:1002-1302(2019)21-0063-04
收稿日期:2018-09-28
基金項目:國家自然科學(xué)基金(編號:31571731)。
作者簡介:李昕晏(1995—),女,湖南長沙人,碩士研究生,主要從事植物抗逆分子生物學(xué)研究。E-mail:496349958@qq.com。
通信作者:崔杰,博士,副教授,主要從事植物抗逆分子生物學(xué)研究。E-mail:cuijie2006@163.com。
1miRNA概述
microRNA(miRNA)是小的非編碼內(nèi)源性RNA(18~24個核苷酸),首先在線蟲(Caenorhabditis elegans)中被發(fā)現(xiàn),可在突變的秀麗隱桿線蟲中暫時表達,所以miRNA最初被稱為短時間RNA(stRNA)[1]。植物miRNA是一類內(nèi)源性非編碼小RNA,對植物生長發(fā)育至關(guān)重要。植物miRNA主要經(jīng)過以下幾個過程產(chǎn)生:首先編碼miRNA的基因依賴RNA聚合酶Pol Ⅱ復(fù)合體形成長約1 000 bp的初級轉(zhuǎn)錄物,初級轉(zhuǎn)錄物呈現(xiàn)不完全配對的莖環(huán)結(jié)構(gòu),隨后初級轉(zhuǎn)錄物轉(zhuǎn)變?yōu)槌墒斓膍iRNA,該過程需要經(jīng)過2個步驟,第1步在細胞核中進行,蛋白酶DCL1識別莖環(huán)結(jié)構(gòu)并對其進行切割,形成前體miRNA,前體miRNA通過HASTY轉(zhuǎn)運蛋白轉(zhuǎn)運到細胞質(zhì)中;第2步在細胞質(zhì)中進行,ATP依賴性RNase Ⅲ蛋白Dicer識別前體miRNA的2 nt 3′突出端,并從其末端去除約21 nt序列形成成熟的miRNA-miRNA*雙鏈體[2]。接著成熟的miRNA雙鏈體展開形成22 nt的單鏈成熟miRNA,其與Argonaute蛋白-1(AGO-1)以及其他調(diào)節(jié)蛋白如GW182和聚A結(jié)合蛋白(PABP)一起形成RNA誘導(dǎo)沉默復(fù)合物(RISC)。最后,成熟的miRNA沉默復(fù)合體(RISC)通過與靶mRNA轉(zhuǎn)錄物3′端非翻譯區(qū)(3′UTR)中的區(qū)域進行堿基互補配對導(dǎo)致靶mRNA翻譯抑制或降解。靶mRNA被抑制還是被降解取決于其與同源miRNA序列的互補程度。在動物中miRNA與靶mRNA的相互作用是部分互補,會導(dǎo)致蛋白質(zhì)翻譯被抑制,而在植物中是完全互補,會導(dǎo)致靶基因完全降解[3-4]。
2miRNA在植物抗逆過程中的作用方式
2.1通過靶向應(yīng)激相關(guān)轉(zhuǎn)錄因子抗逆
研究表明,一些miRNA的靶基因可翻譯為與應(yīng)激相關(guān)的轉(zhuǎn)錄因子[5-7]。轉(zhuǎn)錄因子是一種蛋白質(zhì),它能夠與下游靶基因啟動子區(qū)域中的順式作用元件發(fā)生特異性結(jié)合,激活或抑制基因的表達。轉(zhuǎn)錄因子一般有4個保守的功能區(qū),分別為DNA結(jié)合域、轉(zhuǎn)錄激活或抑制結(jié)構(gòu)域(轉(zhuǎn)錄調(diào)控域)、寡聚化位點和核定位信號。但并不是所有的轉(zhuǎn)錄因子都具有以上所有的功能區(qū),有些轉(zhuǎn)錄因子沒有寡聚化位點。DNA結(jié)合域能夠識別并結(jié)合特定的順式作用元件,其氨基酸序列的特異性決定了轉(zhuǎn)錄因子能夠結(jié)合的順式作用元件的特異性。轉(zhuǎn)錄調(diào)控域是轉(zhuǎn)錄因子家族的分類依據(jù),同一類轉(zhuǎn)錄因子家族擁有相似的轉(zhuǎn)錄調(diào)控區(qū)域,根據(jù)對靶基因作用的差異可將其分為轉(zhuǎn)錄激活域和轉(zhuǎn)錄抑制域。寡聚化位點的存在是轉(zhuǎn)錄因子發(fā)生相互作用形成同源或異源二聚體的條件。核定位信號是富含精氨酸和賴氨酸殘基的核定區(qū)域,轉(zhuǎn)錄因子通過該區(qū)域的調(diào)控進入細胞核[8]。植物中的轉(zhuǎn)錄因子數(shù)量眾多,且廣泛地參與植物生長發(fā)育過程,其中一部分轉(zhuǎn)錄因子與抗逆有關(guān),能夠在植物受到脅迫時被脅迫信號激發(fā),與特定基因的啟動子結(jié)合,調(diào)控其表達從而引起植物發(fā)生生理生化變化,進而提高植物抵抗外界環(huán)境壓力的能力。
2.1.1miR160/miR167與轉(zhuǎn)錄因子ARF互作研究
生長素可調(diào)節(jié)許多基因的表達,生長素響應(yīng)因子(ARF)是調(diào)節(jié)其下游靶基因表達的基因。大多數(shù)ARF蛋白由3個特征結(jié)構(gòu)域組成,即B3 DNA結(jié)合結(jié)構(gòu)域(DBD),位于N末端區(qū)域;C-末端二聚化結(jié)構(gòu)域(CTD),位于C-末端區(qū)域,是一個蛋白質(zhì)-蛋白質(zhì)相互作用結(jié)構(gòu)域,其氨基酸序列與Aux/IAA蛋白質(zhì)C-末端的結(jié)構(gòu)域Ⅲ和Ⅳ相關(guān),允許ARF或ARF和Aux/IAA蛋白的二聚化;賦予活化劑或阻遏物活性的可變中間區(qū)域(MR)。miR160和miR164被發(fā)現(xiàn)靶向ARF轉(zhuǎn)錄因子[9-10]。
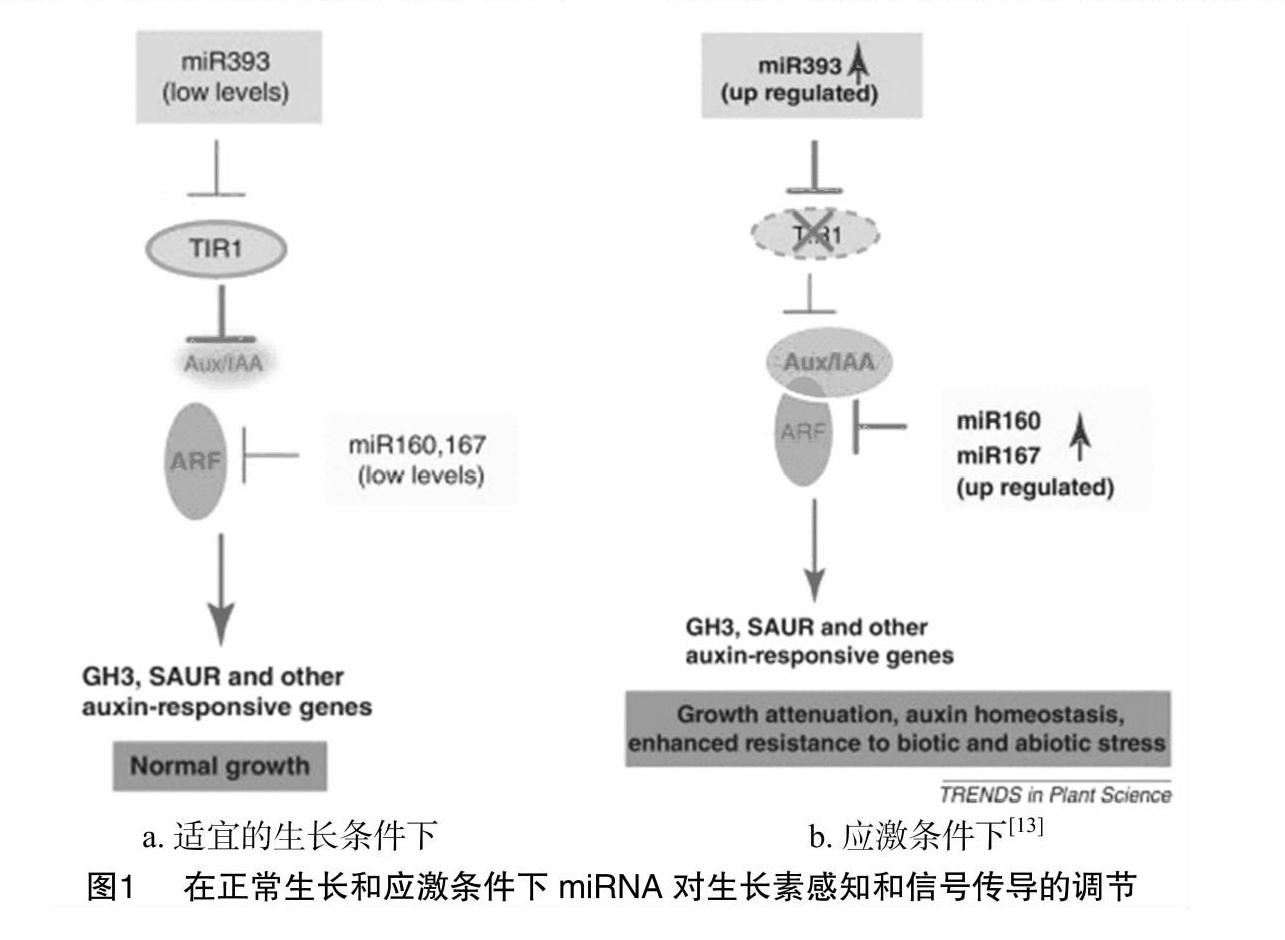
在幾種植物物種中觀察到干旱和鹽脅迫條件下,miR393、miR160和miR167的表達上調(diào),已有研究證明,鹽度觸發(fā)miR393表達,導(dǎo)致TIR1和AFB2受體的結(jié)合被抑制,進而影響生長素信號轉(zhuǎn)導(dǎo)途徑[11]。而miR160和miR164通過負調(diào)節(jié)生長素響應(yīng)因子ARF10、ARF16、ARF17等在植物發(fā)育中起關(guān)鍵作用[12]。miR393/(miR160,miR167)與ARF轉(zhuǎn)錄因子之間存在一個應(yīng)對脅迫的調(diào)控途徑,如圖1所示。生長素受體TIR1通過釋放AUX/IAA與生長素響應(yīng)因子形成異二聚體的方式調(diào)控植物生長和發(fā)育所必需的生長素響應(yīng)基因的表達。當生長素水平低時,ARF與AUX/IAA因子異二聚化。在結(jié)合生長素時,TIR1和AFB等受體被激活并使AUX/IAA成員泛素化,使原本因與AUX/IAA結(jié)合而被抑制的ARF得到釋放。這幾種保守的miRNA在生長素感知和信號傳導(dǎo)中起重要作用,miR393下調(diào)TIR1和AFB轉(zhuǎn)錄物的含量,miR160和miR167通過指導(dǎo)mRNA的切割來下調(diào)5種不同的ARF轉(zhuǎn)錄物的含量。在適宜的生長條件下,低水平的miR393以及miR160和miR167即可微調(diào)轉(zhuǎn)錄生長素響應(yīng)基因所需的ARF水平。在應(yīng)激期間,miR393的上調(diào)表達將TIR1保持在較低的水平,有助于抑制生長素信號傳導(dǎo),從而增加AUX/IAA-ARF異二聚化。此外,miR160和miR167的表達量在應(yīng)激期間也被上調(diào),它們的上調(diào)引起ARF的下調(diào),從而下調(diào)ARF介導(dǎo)的基因表達。總的來說,ARF介導(dǎo)的基因表達受到miR393、miR160和miR167的抑制,導(dǎo)致植物生長和脅迫下的發(fā)育減弱,促進植物對脅迫的耐受性。
2.1.2miR159與轉(zhuǎn)錄因子MYB互作研究
MYB轉(zhuǎn)錄因子廣泛分布于真核生物中,是植物界最大的轉(zhuǎn)錄因子家族之一。MYB蛋白具有高度保守的MYB結(jié)構(gòu)域,其由在N末端的1~4個不完整的串聯(lián)重復(fù)組成。每個MYB重復(fù)序列編碼3個α-螺旋,其中第2個和第3個螺旋形成螺旋-轉(zhuǎn)角-螺旋結(jié)構(gòu),識別并結(jié)合特定識別位點C/TAACG/TG處的DNA主溝。前人研究證明,MYB轉(zhuǎn)錄因子被miR159、miR828和miR858等miRNA靶向,其主要調(diào)節(jié)植物在各種脅迫條件下的信號轉(zhuǎn)導(dǎo)和發(fā)育[14]。Gupta等研究表明,在不同脅迫條件下miR159的表達量有不同的表現(xiàn),例如在20%聚乙二醇6000滲透脅迫下miR159表達量被顯著上調(diào),在鹽脅迫和冷脅迫下miR159表達量呈現(xiàn)下調(diào)趨勢[15],miR159可以通過控制ABI3的表達來調(diào)控MYB33轉(zhuǎn)錄因子的含量,從而參與ABA信號通路響應(yīng)干旱[16]。
2.1.3miR156與轉(zhuǎn)錄因子SPL互作研究
SPL(SQUAMOSA promoter-binding protein-like)是植物特有的一類轉(zhuǎn)錄因子,廣泛存在于植物中。最初從金魚草(Antirrhinum majus)中篩選到2個SPL基因,分別命名為SBP1和SBP2,其因能夠識別并結(jié)合到SQUAMOSA的啟動子上而得名。SPL轉(zhuǎn)錄因子是植物發(fā)育時間的主要調(diào)節(jié)因子,由miR156在轉(zhuǎn)錄后調(diào)節(jié)。最近在擬南芥中的一項研究中發(fā)現(xiàn),miR156介導(dǎo)的SPL2和SPL11表達量的下調(diào)增強了植物對環(huán)境脅迫的反應(yīng),包括植物的耐熱性[17]。研究發(fā)現(xiàn),通過miR156過表達和SPL13 RNA含量降低能夠使紫花苜蓿顯示出對熱應(yīng)激耐受性的增加,在熱應(yīng)激下表現(xiàn)為水勢升高,非酶抗氧化劑含量、花青素含量和葉綠素豐度增加[18]。miRNA156同時被發(fā)現(xiàn)對植物耐鹽機制也有作用,在鹽處理條件下,miR156過表達基因型中苜蓿中記錄到總氮含量的增加和離子穩(wěn)態(tài)的改變;在嚴重的鹽度脅迫下,miR156下調(diào)SPL轉(zhuǎn)錄因子家族基因的表達量,間接調(diào)控其他重要轉(zhuǎn)錄因子以及下游鹽脅迫應(yīng)答基因的表達[19]。另有研究表明,miR156通過沉默SPL13減少水分損失和增加氣孔導(dǎo)度、增加葉綠素含量和增強光合同化作用,從而達到抗旱的作用[20]。
2.1.4miR164與轉(zhuǎn)錄因子NAC互作研究
作為植物中最大的轉(zhuǎn)錄因子家族之一,NAC包含復(fù)雜的植物特異性超家族,并且存在于多種物種中。NAC的首字母縮寫詞源自3種最早發(fā)現(xiàn)NAC結(jié)構(gòu)域的蛋白質(zhì),分別為矮牽牛NAM、擬南芥ATAF1/2和CUC2。NAC被證明在植物應(yīng)激反應(yīng)期間在復(fù)雜的信號傳導(dǎo)網(wǎng)絡(luò)中發(fā)揮重要作用,尤其是在非生物脅迫下,NAC在植物中的表達會發(fā)生明顯變化[21]。NAC轉(zhuǎn)錄因子被miRNA廣泛靶向。幾項關(guān)于擬南芥和水稻的研究已經(jīng)顯示,miR164通過切割NAC mRNA調(diào)節(jié)植物發(fā)育過程,并且能夠響應(yīng)非生物脅迫[22-24]。在水稻中測試了6個miR164靶向NAC基因(OMTN1~OMTN6),結(jié)果發(fā)現(xiàn),其中4個參與耐旱性的調(diào)控[22]。在胡楊中也發(fā)現(xiàn),miR164指導(dǎo)PeNAC070、PeNAC012和PeNAC028 mRNAs的切割,在干旱和鹽脅迫條件下,miR164及其靶基因NAC070在根、葉和莖中的表達情況不同,總體而言增加了植物的抗旱性和耐鹽性[25]。孫宗艷在對甜菜抗逆性的研究中發(fā)現(xiàn),甜菜根中的miR164在鹽處理12 h時表達量下降劇烈,葉中的miR164表達量在處理12 h 時下降,在處理24 h時呈現(xiàn)上升趨勢,隨后又下降,在根和葉中的miR164表達量都分別與根和葉中的NAC21/22呈負相關(guān)關(guān)系[26]。
2.2通過靶向應(yīng)激相關(guān)結(jié)構(gòu)基因抗逆
2.2.1miR398與CSD基因互作研究
逆境會引起植物體內(nèi)自由基的積累,超氧化物歧化酶(SOD)是一種重要的活性氧(ROS)清除酶,它催化超氧化物自由基向H2O2和O2轉(zhuǎn)化,該反應(yīng)構(gòu)成了對氧化應(yīng)激的第1層細胞防御,植物已經(jīng)進化出3種具有不同金屬配體的SOD,即鐵SOD(Fe-SOD)、錳SOD(Mn-SOD)和銅/鋅SOD(Cu/Zn-SOD,也稱為CSD),其中Cu/Zn-SOD是植物中的主要銅酶。擬南芥基因組編碼3種CSD同工酶:細胞質(zhì)中的CSD1、葉綠體中的CSD2和過氧化物酶體中的CSD3。在擬南芥中,miR398具有4個靶基因,分別為CSD1、CSD2、CCS和COX5b-1。當植物暴露于高光、重金屬(Cu2+和Fe3+)或甲基紫精(MV)條件下時,miR398的表達量下調(diào),引起CSD2表達量的上調(diào)[27],進而在氧化應(yīng)激下提高植物性能。在高溫脅迫下,miR398表達量呈現(xiàn)上調(diào)趨勢,在miR398的4個靶標中除了COX5b-1之外,其余3個靶標的表達量都呈現(xiàn)下降趨勢,通過定點誘變CSD1、CSD2、CCS而產(chǎn)生的3種miR398特性轉(zhuǎn)基因擬南芥與野生型相比,在37 ℃中對熱應(yīng)激更加敏感,可積累更高水平的超氧化物自由基[28]。
2.2.2miR395與APS基因互作研究
硫元素參與許多生物功能,并且是多種次級代謝物的必需元素,包括硫脂、硫酸葡萄糖胺、谷胱甘肽和輔酶。缺硫環(huán)境會嚴重影響植物的生長,因此,硫營養(yǎng)對植物生長和發(fā)育至關(guān)重要。植物可從土壤中獲得無機硫酸鹽形式的硫,從大氣中獲得二氧化硫和硫化氫氣體形式的硫。硫酸鹽在同化過程首先由ATP硫酸化酶催化無機硫酸鹽和ATP形成腺苷5′-磷酸硫酸鹽和無機焦磷酸鹽。隨后,腺苷5′磷酸硫酸鹽被2種不同的硫酸鹽同化途徑利用。前人對擬南芥中的4種ATP硫酸化酶編碼基因(APS基因)進行定量分析,結(jié)果發(fā)現(xiàn),miR395靶向APS基因,在硫酸鹽限制期間,3種APS基因在轉(zhuǎn)錄后水平受miR395調(diào)節(jié),miR395過表達植物中的硫酸鹽濃度增加[29-30]。更有研究證明,硫酸鹽濃度與miR395的豐度呈正相關(guān)關(guān)系,即miR395水平升高導(dǎo)致硫酸鹽濃度增加,而miR395豐度的下調(diào)導(dǎo)致硫酸鹽濃度降低。相應(yīng)地,miR395靶向的APS基因的過表達導(dǎo)致硫酸鹽濃度的下降[31]。有研究在鎘脅迫下鑒定了過表達miR395的轉(zhuǎn)基因油菜,結(jié)果表明,過表達miR395的植物表現(xiàn)出比野生型更低程度的鎘誘導(dǎo)氧化應(yīng)激,相比之下,轉(zhuǎn)化體中的葉綠素、谷胱甘肽和非蛋白質(zhì)硫醇含量高于野生型[32]。
3展望
非生物脅迫對植物生長發(fā)育影響很大[33],植物進化出了一套復(fù)雜的調(diào)控機制來應(yīng)對不良環(huán)境。非生物脅迫誘導(dǎo)miRNA的異常表達,miRNA位于調(diào)節(jié)植物對非生物脅迫反應(yīng)的基因網(wǎng)絡(luò)中心,使得miRNA成為改良農(nóng)作物遺傳性狀的重要新靶標,包括植物對環(huán)境脅迫的反應(yīng)。前人研究已經(jīng)確定,miRNA的調(diào)控是通過與靶基因完全或不完全結(jié)合的方式來完全降解靶基因或抑制靶基因的翻譯過程,進而起到對靶基因不同程度的抑制作用。前人鑒定了多種植物中的miRNA,并且研究了各個miRNA的靶基因以及在不同脅迫下miRNA和其靶基因的表達量變化,分析其在植物抗逆中的作用。但總的來說,對于miRNA的研究大多數(shù)還是以不同物種中miRNA的鑒定為主,對于其在整體逆境調(diào)控網(wǎng)絡(luò)中作用的研究較少,根據(jù)現(xiàn)有的研究可以總結(jié)出,植物中的miRNA既有靶向應(yīng)激相關(guān)轉(zhuǎn)錄因子的,也有靶向應(yīng)激相關(guān)結(jié)構(gòu)基因的,最終使植物在生理生化上作出應(yīng)對脅迫的改變。今后應(yīng)當加強對調(diào)控網(wǎng)絡(luò)的研究,更深入地探討miRNA在抗逆調(diào)控網(wǎng)絡(luò)的功能,進而更好地指導(dǎo)農(nóng)作物的改良,增加作物產(chǎn)量,優(yōu)化作物質(zhì)量。
參考文獻:
[1]Lee R C,F(xiàn)einbaum R L,Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell,1993,75(5):843-854.
[2]Kurihara Y,Watanabe Y. Arabidopsis micro-RNA biogenesis through Dicer-like 1 protein functions[J]. Proceedings of the National Academy of Sciences,2004,101(34):12753-12758.
[3]Hck J,Meister G. The Argonaute protein family[J]. Genome Biology,2008,9(2):210.
[4]Kumar R. Role of microRNAs in biotic and abiotic stress responses in crop plants[J]. Applied Biochemistry & Biotechnology,2014,174(1):93-115.
[5]Wang J W,Czech B,Weigel D. miR156-regulated SPL transcription factors define an endogenous flowering pathway in Arabidopsis thaliana[J]. Cell,2009,138(4):738-749.
[6]Palatnik J F,Wollmann H,Schommer C,et al. Sequence and expression differences underlie functional specialization of Arabidopsis microRNAs miR159 and miR319[J]. Developmental Cell,2007,13(1):115-125.
[7]Rodriguez R E,Mecchia M A,Debemardi J M,et al. Control of cell proliferation in Arabidopsis thaliana by microRNA miR396[J]. Development,2010,137(1):103-112.
[8]Imagawa M,Sakaue R,Tanabe A,et al. Two nuclear localization signals are required for nuclear translocation of nuclear factor 1-A[J]. FEBS Letters,2000,484(2):118-124.
[9]Huang J,Li Z Y,Zhao D Z. Deregulation of the OsmiR160 target gene OsARF18 causes growth and developmental defects with an alteration of auxin signaling in rice[J]. Scientific Reports,2016,6:29938.
[10]Glazińska P,Wojciechowski W,Wilmowicz E,et al. The involvement of InMIR167 in the regulation of expression of its target gene InARF8,and their participation in the vegetative and generative development of Ipomoea nil plants[J]. Journal of Plant Physiology,2014,171(3/4):225-234.
[11]Iglesias M J,Terrile M C,Windels D,et al. MiR393 regulation of auxin signaling and redox-related components during acclimation to salinity in Arabidopsis[J]. PLoS One,2014,9(9):e107678.
[12]Lin Y L,Lai Z X,Tian Q L,et al. Endogenous target mimics down-regulate miR160 mediation of ARF10,-16,and-17 cleavage during somatic embryogenesis in Dimocarpus longan Lour[J]. Frontiers in Plant Science,2015,6:956.
[13]Sunkar R,Li Y F,Jagadeeswaran G. Functions of microRNAs in plant stress responses[J]. Trends in Plant Science,2012,17(4):196-203.
[14]Sun G L. MicroRNAs and their diverse functions in plants[J]. Plant Molecular Biology,2012,80(1):17-36.
[15]Gupta O P,Meena N L,Sharma I,et al. Differential regulation of microRNAs in response to osmotic,salt and cold stresses in wheat[J]. Molecular Biology Reports,2014,41(7):4623-4629.
[16]Lopez-Molina L,Mongrand S,McLachlin D T,et al. ABI5 acts downstream of ABI3 to execute an ABA-dependent growth arrest during germination[J]. Plant Journal,2002,32(3):317-328.
[17]Matthews C,Arshad M,Hannoufa A. Alfalfa response to heat stress is modulated by microRNA156[J]. Physiologia Plantarum,2018,165(4):830-842.
[18]Arshad M,Gruber M Y,Wall K,et al. An insight into microRNA156 role in salinity stress responses of alfalfa[J]. Frontiers in Plant Science,2017,8:356.
[19]Arshad M,F(xiàn)eyissa B A,Amyot L,et al. MicroRNA156 improves drought stress tolerance in alfalfa (Medicago sativa) by silencing SPL13[J]. Plant Science,2017,258:122-136.
[20]Le D T,Nishiyama R,Watanabe Y,et al. Genome-wide survey and expression analysis of the plant-specific NAC transcription factor family in soybean during development and dehydration stress[J]. DNA Research,2011,18(4):263-276.
[21]Huang H,Wang Y,Wang S L,et al. Transcriptome-wide survey and expression analysis of stress-responsive NAC genes in Chrysanthemum lavandulifolium[J]. Plant Science,2012,193-194:18-27.
[22]Fang Y,Xie K,Xiong L. Conserved miR164-targeted NAC genes negatively regulate drought resistance in rice[J]. Journal of Experimental Botany,2014,65(8):2119-2135.
[23]Guo H S,Xie Q,F(xiàn)ei J F,et al. MicroRNA164 directs NAC1 mRNA cleavage of the transcription factor NAC1 to downregulate auxin signals for Arabidopsis lateral root development[J]. Plant Cell,2005,17:1376-1386.
[24]Nakashima K,Takasaki H,Mizoi J,et al. NAC transcription factors in plant abiotic stress responses[J]. Biochimica et Biophysica Acta(Gene Regulatory Mechanisms),2012,1819(2):97-103.
[25]Lu X,Dun H,Lian C L,et al. The role of peu-miR164 and its target PeNAC genes in response to abiotic stress in Populus euphratica[J]. Plant Physiology and Biochemistry,2017,115:418-438.
[26]孫宗艷. 鹽/干旱脅迫下甜菜幼苗中miR160/164及其靶基因的表達與分析[D]. 哈爾濱:哈爾濱工業(yè)大學(xué),2017.
[27]Sunkar R,Kapoor A,Zhu J K. Posttranscriptional induction of two Cu/Zn superoxide dismutase genes in Arabidopsis is mediated by downregulation of miR398 and important for oxidative stress tolerance[J]. Plant Cell,2006,18(8):2051-2065.
[28]Guan Q M,Lu X Y,Zeng H T,et al. Heat stress induction of miR398 triggers a regulatory loop that is critical for thermotolerance in Arabidopsis[J]. Plant Journal,2013,74(5):840-851.
[29]Kawashima C G,Yoshimoto N,Maruyama-Nakashita A,et al. Sulphur starvation induces the expression of microRNA-395 and one of its target genes but in different cell types[J]. Plant Journal,2009,57(2):313-321.
[30]Liang G,Yang F X,Yu D Q. MicroRNA395 mediates regulation of sulfate accumulation and allocation in Arabidopsis thaliana[J]. Plant Journal,2010,62(6):1046-1057.
[31]Ai Q,Liang G,Zhang H M,et al. Control of sulfate concentration by miR395-targeted APS genes in Arabidopsis thaliana[J]. Plant Diversity,2016,38(2):92-100.
[32]Zhang L W,Song J B,Shu X X,et al. miR395 is involved in detoxification of cadmium in Brassica napus[J]. Journal of Hazardous Materials,2013,250-251:204-211.
[33]李曉剛,李慧,楊青松,等. 杜梨bHLH轉(zhuǎn)錄因子家族兩成員的序列特征及對非生物脅迫的轉(zhuǎn)錄響應(yīng)[J]. 江蘇農(nóng)業(yè)科學(xué),2017,45(22):40-45.

