食管鱗癌組織中miRNAs差異表達譜的篩選及研究
楊雅娟 李靜 朱琳 石俊杰 喬冠恩

摘要:目的 ?篩選并鑒定在食管鱗癌(ESCC)及癌旁組織中miRNAs的差異表達,為進一步闡明其在ESCC發病機制中的作用奠定基礎。方法 ?選取在邯鄲市第一醫院行食管癌切除術患者新鮮的鱗癌組織及癌旁正常組織,各3例,抽提總RNA,利用miRNAs芯片篩選其中差異表達的miRNAs,并對miR-106b-3p通過進一步RT-qPCR 技術進行驗證。結果 ?miRNAs芯片從配對組織中共篩檢出62個差異表達的miRNAs,其中41個miRNAs表達上調,21個miRNAs表達下調,鱗癌組織與癌旁正常組織中差異表達的miRNAs比較,差異有統計學意義(P<0.05)。miR-106b-3p在ESCC組織中的表達高于癌旁組織,差異有統計學意義(P<0.05),與芯片結果一致。結論 ?食管鱗癌組織中miRNAs的差異表達為進一步研究miRNAs在ESCC發病中的作用奠定了基礎;miR-106b-3p的高表達可能與ESCC的發生、發展有關。
關鍵詞:食管鱗癌;miRNAs芯片;microRNAs
中圖分類號:R735.1 ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? 文獻標識碼:A ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? ? DOI:10.3969/j.issn.1006-1959.2019.20.018
文章編號:1006-1959(2019)20-0063-04
Screening and Study of Differential Expression Profiles of miRNAs in
Esophageal Squamous Cell Carcinoma
YANG Ya-juan1,LI Jing1,ZHU Lin2,SHI Jun-jie3,QIAO Guan-en1
Abstract:Objective ?To screen and identify differential expression of miRNAs in esophageal squamous cell carcinoma (ESCC) and adjacent tissues, and to lay the foundation for further elucidation of its role in the pathogenesis of ESCC.Methods ?Fresh squamous cell carcinoma and adjacent normal tissues were obtained from patients with esophageal cancer resection in the First Hospital of Handan City, 3 cases were extracted, total RNA was extracted, and miRNAs were used to screen differentially expressed miRNAs, and miR-106b-3p was used. Verification by further RT-qPCR technology.Results ?A total of 62 differentially expressed miRNAs were screened from the paired tissues by miRNAs. Among them, 41 miRNAs were up-regulated, 21 miRNAs were down-regulated, and miRNAs were differentially expressed in squamous cell carcinoma and adjacent normal tissues,the difference was statistically significant (P<0.05). The expression of miR-106b-3p in ESCC tissues was higher than that in adjacent tissues,the difference was statistically significant (P<0.05), which was consistent with the chip results.Conclusion ?The differential expression of miRNAs in esophageal squamous cell carcinoma lays a foundation for further study of the role of miRNAs in the pathogenesis of ESCC. The high expression of miR-106b-3p may be related to the occurrence and development of ESCC.
Key words:Esophageal squamous cell carcinoma;miRNAs chip;microRNAs
食管鱗癌(esophageal squamous cell carcinoma,ESCC)是我國常見的消化道惡性腫瘤,其發病的機制尚不清楚。microRNAs(miRNAs)是一類長度在22 nt左右的非編碼小RNAs,參與了人體內大部分生物學調節途徑[1,2]。miRNAs能夠通過其堿基序列區域與其相應靶基因mRNA的3'-非翻譯區(3'-untranslated region,3'-UTR)中的互補堿基序列進行完全或不完全的互補結合,在基因的轉錄后階段調控其相應靶基因mRNA的表達水平[3-5]。miRNAs芯片又稱microRNAs微陣列,可同時檢測數千個miRNAs的表達量,適用于miRNAs表達譜分析。本研究采用安捷倫miRNAs芯片技術篩選ESCC組織與癌旁正常組織差異表達的miRNAs,并對部分miRNA采用反轉錄熒光定量PCR(RT-qPCR)進行驗證,為進一步研究miRNAs在ESCC發生、發展過程中的作用提供參考。
1材料與方法
1.1材料來源 ?選取在邯鄲市第一醫院行食管癌切除術患者新鮮的鱗癌組織及癌旁正常組織,各3例。所有樣本均經病理組織學證實,癌組織和癌旁組織一并保存,所有患者術前均無放、化療史。
1.2主要試劑 ?miRNAs芯片購自美國安捷倫公司,總RNA提取試劑盒RNAprep Pure Tissue Kit購自北京TIANGEN公司,熒光定量PCR試劑盒購自Roche Applied Science公司,反轉錄試劑盒購自美國Applied Biosystems公司,引物由南京金斯瑞生物公司合成。
1.3方法
1.3.1組織總RNA的提取 ?從液氮中取出樣本,稱取約0.5 g組織放入缽中研磨(先用液氮預冷),邊研磨邊加液氮,待組織成粉末狀后轉到1.5 ml離心管中,按照RNA提取試劑盒操作說明書提取總RNA。
1.3.2芯片雜交及數據分析 ?采用安捷倫miRNAs芯片(V21.0)及配套的檢測儀器及試劑盒檢測,該芯片覆蓋2549個人源miRNAs。步驟如下:取100 ng 總RNA 經Cy3 熒光標記后,在雜交儀中與miRNAs芯片于55℃、20 r/min雜交 20 h,隨后用GE洗脫液1和2各洗5 min,最后用芯片掃描儀掃描。掃描完成后通過Feature Extraction軟件進行數據的提取,原始數據文件經Genespring標準化后進行數據分析。篩選差異表達的miRNAs,并對其進行非監督層次聚類,用熱圖的形式顯示差異miRNAs在不同樣本間的表達情況。
1.3.3反轉錄反應 ?按照試劑盒操作說明進行反轉錄反應,RNA量為200 ng,加反轉錄引物1 μl,加雙蒸水至體積為11 μl,65℃孵育5 min,冰上預冷 ? ? ? ?5 min,加入反轉錄酶1.0 μl,RNA酶抑制劑1 μl,dNTPs 2.0 μl,5× Reaction緩沖液 4.0 μl,至總體積為19 μl,42°C孵育60 min,70°C反應10 min后立即取出冰浴冷卻,合成cDNA。
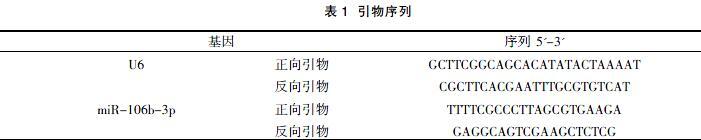
1.3.4 RT-qPCR ?合成cDNA 后,以Fast Start Universal SYBR Green Master (Rox)PCR試劑盒擴增目的基因,U6為內參基因,miR-106b-3p、U6 引物序列見表1。反應體系為: cDNA 產物 1.0 μl,qPCR Mix10 μl,上游引物、下游引物各0.4 μl,余用雙蒸水補齊至20 μl。擴增條件: 94 ℃預變性5 min,94℃30 s、60℃30 s、72℃30 s,40 個循環,設立 3 個復孔。根據擴增曲線得到Ct值,miRNAs含量采用相對表達量 2- △△Ct表示。
1.4統計學方法 ?采用GraphPad 5.0軟件分析數據,計量資料用(x±s)表示,兩組間比較采用t檢驗,P<0.05表示差異有統計學意義。
2結果
2.1組織樣本總RNA質量控制 ?3對送檢組織樣本檢測發現,各樣本RNA在18、28 s處見明顯峰值,A260/A280在1.9~2.1,表明RNA質量和純度符合miRNAs芯片的檢測要求。
2.2芯片中差異表達的miRNAs ?芯片從配對組織中共篩檢出62個差異表達的miRNAs,其中表達上調有:miR-106b-3p、miR-193a-5p、miR-342-5p、miR-21-3p、miR-18a-5p、miR-500a-3p、miR-186-5p、miR-148b-5p、miR-123-5p、miR-3651、miR-128、miR-29b-1-5p、miR-523-3p、miR-505-3p、miR-18b-5p、miR-361-3p、miR-664b-3、miR-26a-5p、miR-181a-3p、miR-4286、let-7d-5p、miR-29c-3p、miR-107、miR-320b、miR-320d、miR-425-5p、miR-320e、miR-92a-3p、let-7f-5p、miR-181a-5p、miR-25-3p、miR-17-5p、miR-26b-5p、miR-155-5p、miR-16-5p、miR-30b-5p、miR-15b-5p、let-7g-5p、let-7a-5p、miR-30d-5p、miR-93-5p;表達下調的有:miR-3910-5p、miR-5787、miR-6126、miR-6722-3p、miR-4631、miR-4270、miR-150-3p、miR-4327、miR-4459、miR-6090、miR-30d-5p、miR-4166、miR-1290、miR-3196、miR-6087、miR-4530、miR-4687-3p、miR-1216、miR-1275、miR-1234-5p、miR-4665-3p,對差異表達的miRNAs進行聚類分析,差異無統計學意義(P>0.05);鱗癌組織與癌旁正常組織差異表達的miRNAs比較,差異有統計學意義(P<0.05),見圖1。
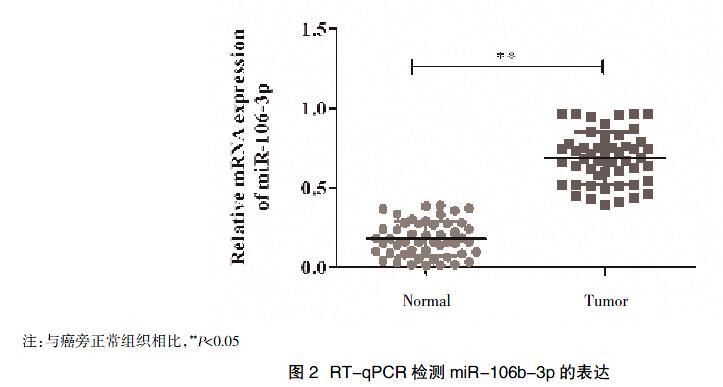
2.3 RT-qPCR技術驗證 ?對表達上調的miR-106b-3p進行進一步驗證,結果顯示miR-106b-3p在ESCC組織中的表達高于癌旁組織,差異有統計學意義(P<0.05),與芯片結果一致,見圖2。
3討論
miRNAs在腫瘤中的表達與在正常組織或細胞的表達存在差異,疾病的不同階段以及不同組織或細胞中,miRNAs的表達和作用均不同。一個miRNA能調控多個靶mRNAs的表達,多個miRNAs也能共同調控一個靶mRNA發揮作用[6]。有研究顯示,通過PCR、芯片及測序等方法可發現腫瘤相關的miRNAs[7-9]。安捷倫miRNAs芯片為目前較為先進的miRNAs表達譜芯片,具有高特異性、高靈敏性及可重復性,能與高通量測序相媲美,安捷倫 HumanmiRNAs V21.0芯片能檢測2549個成熟的miRNAs。
本研究通過對ESCC組織和癌旁組織中的miRNAs檢測,篩選出62個差異表達的miRNAs,其中上調的有41個,下調的有21個。進一步對表達異常的miR-106b-3p采用RT-qPCR技術進行驗證,結果顯示miR-106b-3p在ESCC組織的表達要高于癌旁組織,與芯片結果一致,說明miRNAs 芯片能夠較準確的反映各miRNA 在腫瘤樣本中的表達情況。
利用miRNAs芯片技術篩選癌組織miRNAs表達譜是發現異常miRNAs的常見手段。周萍[10]的研究選取了10例ESCC新鮮凍存組織及對應的正常食管粘膜組織,采用上海康成生物有限公司代理的Exiqon公司的miRNAs表達譜芯片檢測,結果顯示有淋巴結轉移組與無淋巴結轉移組ESCC患者相比較,差異表達的miRNAs共16個,其中表達上調的miRNAs有9個,表達下調的miRNAs有7個,經RT-PCR驗證,與正常黏膜組織相比,ESCC組織中miR-612表達上調,且淋巴結轉移組高于無淋巴結轉移組。姚麗[11]的研究對ESCC組織及正常食管黏膜組織的miRNAs表達譜統計分析,結果則顯示miRNAs差異表達共43個,其中27個miRNAs表達下調,16個miRNAs表達上調;Okumura T[12]等通過芯片技術檢測了多種miRNAs的差異表達,確定了miR-574-3p和miR-106b的高表達與腫瘤無復發及良好的總生存率有關,miR-106b被認為具有腫瘤抑制作用。本研究顯示有41個表達上調的miRNAs,其中miR-106b-3p表達明顯上調,說明通過miRNAs芯片能較好的篩選出有意義的差異表達miRNA,與上述研究結果不完全一致,分析原因可能與取材方法、疾病分期、試劑產地、異常表達標準及miRNAs的時序性等有關。
總之,食管鱗癌組織中miRNAs的差異表達為進一步研究miRNAs在食管鱗癌發病中的作用奠定了基礎,miR-106b-3p的高表達可能與食管鱗癌的發生、發展有關。
參考文獻:
[1]Liu B,Li J,Cairns MJ.Identifying miRNAs,targets and functions[J].Briefings in Bioinformatics,2014,15(1):1-19.
[2]Ameres SL,Zamore PD.Diversifying microRNA sequence and function[J].Nature Reviews Molecular Cell Biology,2013,14(8):475-488.
[3]Orom UA,Nielsen FC,Lund AH.MicroRNA-10a binds the 5′MUTR of ribosomal protein mRNAs and enhances their translation[J].Molecular cell,2008,30(4):460-471.
[4]Qin W,Shi Y,Zhao B,et al.miR-24 Regulates Apoptosis by Targeting the Open Reading Frame (ORF)Region of FAF1 in Cancer Cells[J].PLoS One,2010,5(2):e9429.
[5]Singh NK.miRNAs target databases:developmental methods and target identification techniques with functional annotations[J].Cellular and Molecular Life Sciences,2017,74(12):2239-2261.
[6]Fassan M,Baffa R,Palazzo JP,et al.MicroRNA expression profiling of male breast cancer[J].Breast Cancer Research,2009,11(4):R58.
[7]Syed P,Gidwani K,Kekki H,et al.Role of lectin microarrays in cancer diagnosis[J].Proteomics,2016,16(8):1257-1265.
[8]Hu B,Niu X,Cheng L,et al.Discovering cancer biomarkers from clinical samples by protein microarrays[J].Proteomics Clin Appl,2015,9(1-2):98-110.
[9]Willier S,Butt E,Grunewald TGP.Lysophosphatidic acid(LPA)signalling in cell migration and cancer invasion:A focussed review and analysis of LPA receptor gene expression on the basis of more than 1700 cancer microarrays[J].Biology of the Cell,2013,105(8):317-333.
[10]周萍.食管鱗狀細胞癌轉移相關miRNa-612調控P53的功能和機制研究[D].山東大學,2017.
[11]姚麗.miR-1在食管鱗狀細胞癌發生中的作用及其機制的初步研究[D].第四軍醫大學,2015.
[12]Okumura T,Kojima H,Miwa T,et al.The expression of microRNA 574-3p as a predictor of postoperative outcome in patients with esophageal squamous cell carcinoma[J].World Journal of Surgical Oncology,2016,14(1):228.
收稿日期:2019-8-4;修回日期:2019-8-12
編輯/肖婷婷

