基于生長和抗逆功能基因SNP分子標記的凡納濱對蝦野生及選育群體遺傳多樣性分析
申淑慧 戴習林
摘要:【目的】分析凡納濱對蝦野生群體與選育群體的遺傳多樣性及遺傳差異,為進一步改良優化凡納濱對蝦種質提供參考依據。【方法】通過凡納濱對蝦轉錄組測序數據挖掘候選SNP位點,設計擴增引物,采用10尾野生凡納濱對蝦進行直接測序驗證,篩選獲得11對有效引物及24個位于生長和抗逆功能基因上的SNPs位點,并用于分析2個野生群體(Y1和Y2)和2個選育群體(F1和F2)的遺傳多樣性。【結果】凡納濱對蝦野生群體的觀測雜合度(Ho)、期望雜合度(He)及多態信息含量(PIC)等遺傳參數平均值明顯高于選育群體。在2個野生群體中,Y2的各項遺傳參數平均值均高于Y1;在2個選育群體中,F2的各項遺傳參數平均值均高于F1,且在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位點上,2個選育群體的各項遺傳參數平均值為0。4個凡納濱對蝦群體中均存在SNP位點不同程度偏離Hardy-Weinberg平衡現象。野生群體Y1與選育群體F1間的近交系數(Fis)最高(0.0875),且基因流(Nm)也較高(4.2728);野生群體Y2與選育群體F1間的遺傳分化指數(Fst)最高(0.0947),但Nm最低(2.3886),表明二者間的遺傳背景較遠。4個凡納濱對蝦群體的遺傳變異主要來源于群體內(85.15%),僅有14.85%變異來源于群體間。4個凡納濱對蝦群體的遺傳相似性系數為0.9351~0.9747,遺傳距離為0.0256~0.0671;基于凡納濱對蝦群體遺傳距離的UPGMA聚類分析結果顯示,Y1、F1和F2聚為一支,而Y2單獨聚為一支。【結論】凡納濱對蝦野生群體在抗逆功能基因SNP分子標記中的遺傳多樣性顯著高于選育群體,且野生群體與選育群體間的遺傳距離較遠,具有較高的遺傳改良潛力。在實際生產中,可通過雜交措施將野生群體的優良抗逆基因引入選育群體,提高凡納濱對蝦選育群體的免疫及抗病能力。
關鍵詞: 凡納濱對蝦;野生群體;選育群體;SNP分子標記;生長功能基因;抗逆功能基因;遺傳多樣性
中圖分類號: S945.49 ? ? ? ? ? ? ? ? ? ? ? ? ? ? ?文獻標志碼: A 文章編號:2095-1191(2020)11-2836-10
Genetic diversity analysis in wild and selected populations of Penaeus vannamei based on SNP markers in growth
and stress resistance genes
SHEN Shu-hui1,2,3, DAI Xi-lin1,2,3*
(1National Demonstration Center for Experimental Fisheries Science Education(Shanghai Ocean University), Shanghai 201306, China; 2Key Laboratory of Freshwater Aquatic Genetic Resources, Ministry of Agriculture and Rural Affairs (Shanghai Ocean University), Shanghai ?201306, China; 3Shanghai Collaborative Innovation for Aquatic Animal
Genetics and Breeding(Shanghai Ocean University), Shanghai ?201306, China)
Abstract:【Objective】The genetic diversity and genetic differences between wild populations and selected populations of Litopenaeus vannamei were analyzed to provide reference for improvement and optimization of germplasm resources. 【Method】Single nucleotide polymorphisms(SNPs) found from amplification primers, which designed with high-throughput sequencing of L. vannamei, were verified using ten wild L. vannamei by sequencing. Finally, 11 primers and 24 identified SNPs in growth and stress resistance genes, were selected to analyze genetic diversity of Y1, Y2, which were wild populations, and F1, F2, which were selected populations. 【Result】The results showed that the genetic parameters such as ?observed heterozygosity(Ho), expected ?heterozygosity(He) and polymorphism information content(PIC) in Y1 and Y2 were higher than those in F1 and F2. Generally, all the genetic parameters in Y2 were higher than Y1. Similarly, in two selected families, genetic parameters of F2 were higher than F1. In addition, the genetic parameters in the two selected populations were 0 at some SNP loci of functional genes such as CATL, ANT, Tryptase, Crustin-like and Penaeidin4c. Deviation from Hardy-Weinberg equilibrium existed in all four populations differently. The coefficient of inbreeding (Fis) was the highest(0.0875) and the gene flow(Nm) was also high (4.2728) between wild population Y1 and selected population F1. The genetic differentiation index(Fst) between Y2 and F1 was the highest(0.0947), but Nm was the lowest (2.3886), indicating a distant genetic relationship between the two populations. AMOVA analysis found that 85.15% genetic variation of four populations was mainly within the population, and only 14.85% was found between populations.The genetic similarity coefficients of the four populations ranged from 0.9351 to 0.9747, and the genetic distance varied from 0.0256 to 0.0671. UPGMA clustering analysis showed that Y1, F1 and F2 clustered into a cluster, while Y2 clustered into a single cluster. 【Conclusion】Generally, in wild populations, genetic diversity of SNP markers in stress resistance function gene is significantly higher than that of selected populations. And wild populations are far from the selected populations, which shows a high potential for breeding. Therefore, it is necessary that the excellent stress resistance genes from wild populations should be introduced into the selected population through hybridization in production process, to improve the immunity and disease resistance of the selected population.
Key words: Litopenaeus vannamei; wild population; ?selected populations; SNP markers; growth functional genes; ?stress resistance genes;genetic diversity
Foundation item: Special Project of Shanghai Shrimp Industry Technology System Construction (HNKGZ〔2014〕7-1-11)
0 引言
【研究意義】凡納濱對蝦(Litopenaeus vannamei)又稱南美白對蝦,原產于太平洋沿岸,因具有含肉率高、抗逆性強、生長快、餌料需求低、便于活蝦運輸及耐高密度和低鹽度養殖等優點,已發展成為世界上最主要的蝦類養殖品種,約占蝦類總產量的85%(楊銘等,2017;李強勇等,2020)。隨著對蝦養殖業的不斷發展,因病害頻發和種質退化造成的對蝦抗病力下降、生產周期延長及產量下降等問題日益凸顯,給對蝦養殖帶來巨大經濟損失,嚴重制約了凡納濱對蝦養殖業的持續健康發展(戴習林等,2018;蔣葛等,2019)。因此,培育抗逆性強且生長快的優良對蝦養殖品種,是對蝦養殖業今后發展的重要任務(楊銘等,2017;葉寧等,2017)。其中,引進原種或改良現有養殖品種是恢復和優化凡納濱對蝦種質的有效途徑,但在遺傳改良過程中必須掌握已有群體的種質資源現狀及遺傳背景。【前人研究進展】對蝦的遺傳育種工作起步相對較晚,但隨著PCR和分子標記技術的不斷成熟及推廣應用,近年來對蝦遺傳育種研究取得了長足進展。于洋(2014)、郝敏(2016)、李東宇等(2016)、王全超(2017)、Yu等(2019)先后從凡納濱對蝦遺傳圖譜構建、數量性狀定位、遺傳多樣性分析、親權鑒定及分子輔助育種等方面開展研究,研究結果為凡納濱對蝦的良種選育提供了參考依據。目前,針對凡納濱對蝦的遺傳學研究主要是利用等位酶、單拷貝核DNA、線粒體DNA、限制性片段長度多態性(RFLP)、擴增片段長度多態性(AFLP)及微衛星(SSR)等分子標記進行群體遺傳背景分析(張靈俠等,2008;葉寧等,2017;陳錦豪等,2019),其中又以SSR分子標記在凡納濱對蝦遺傳多樣性研究中的應用最廣泛。Rezaee等(2016)、劉洪濤等(2018)、方振朋等(2019)、黃小帥等(2019)分別研發出SSR分子標記并應用于凡納濱對蝦群體遺傳多樣性研究,但SSR分子標記也存在一些缺陷,包括可能存在無效等位基因、無法存在于功能基因中及費時費力等。單核苷酸多態性標記(Single nucleotide polymorphism,SNP)作為第三代分子標記,屬于共顯性標記,雖然每個標記的基礎信息量較少,但能穩定、均勻、廣泛地分布于整個基因組中,且可能出現在功能基因內部,更有利于開展生長、抗逆及繁殖等相關功能基因的研究,是分析遺傳變異的有效分子工具(張曉萌等,2013;王全超,2017;陳靜等,2018)。有關SNP分子標記在水產動物群體遺傳學方面的應用研究已有較多報道,Agarwal等(2016)通過高通量測序獲得大量羅氏沼蝦SNP分子標記,并用于分析印度不同區域羅氏沼蝦群體的遺傳背景;于磊等(2018)基于酶切測序技術開發獲得38個多態性較高的SNP分子標記,并以此分析遼寧丹東和福建三沙2個日本鰻鱺群體的遺傳結構及其本地適應性;Thongda等(2018)研究表明,開發獲得的58個東方牡蠣SNP分子標記均可用于遺傳分化、群體結構分析及親緣關系鑒定等相關研究。【本研究切入點】SNP分子標記是一種研究物種遺傳變異的理想分子標記,但至今鮮見應用于凡納濱對蝦遺傳多樣性分析的研究報道。【擬解決的關鍵問題】基于凡納濱對蝦轉錄組數據,挖掘與生長和抗逆相關候選基因中的SNP位點,并以此分析凡納濱對蝦野生群體與選育群體的遺傳多樣性及遺傳差異,為進一步改良優化凡納濱對蝦種質提供參考依據。
1 材料與方法
1. 1 樣品采集及DNA提取
凡納濱對蝦樣本為2個厄瓜多爾野生群體和2個選育群體。其中,厄瓜多爾野生群體為2016和2019年從厄瓜多爾引進的野生凡納濱對蝦,捕撈自瓜亞基樂灣水域,分別命名為Y1和Y2;選育群體是以美國關島大學引進家系選育獲得的子二代,分別命名為F1和F2。從各群體中隨機取30尾樣本,無水酒精固定后以天根海洋動物組織基因組DNA提取試劑盒提取其DNA,-20 ℃冰箱中保存備用。
1. 2 SNP位點發掘與引物篩選
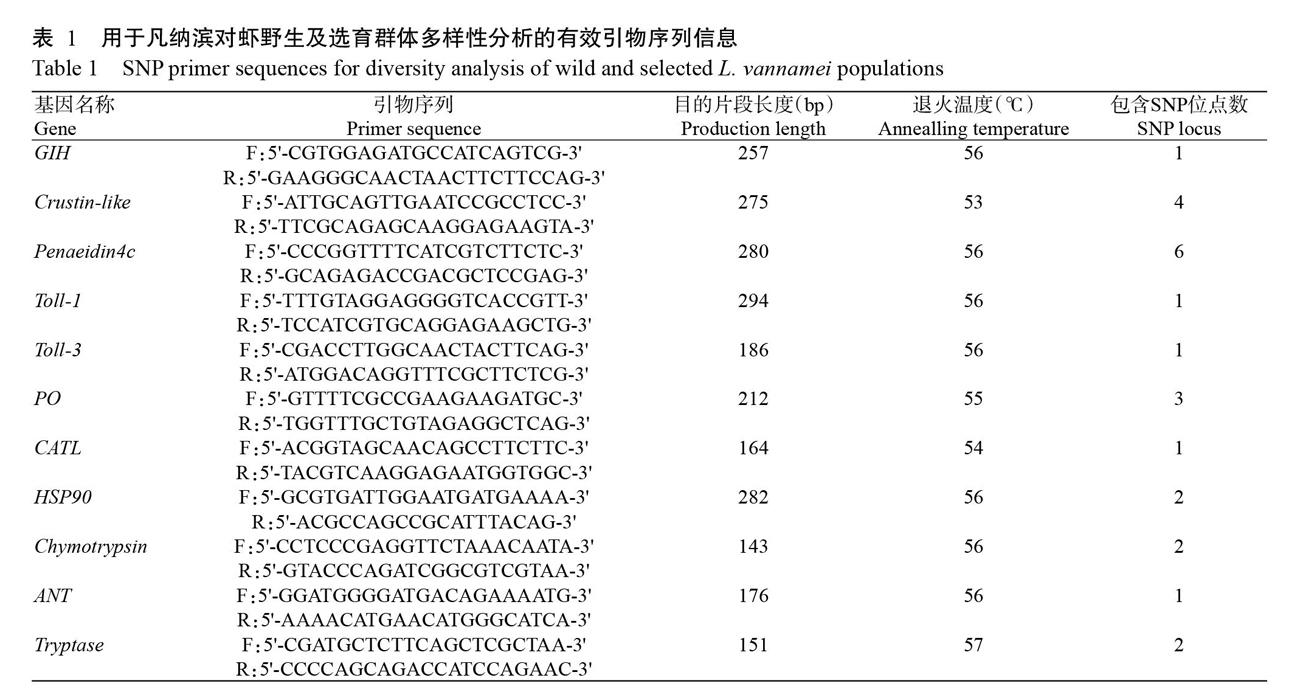
根據農業農村部淡水水產種質資源重點實驗室已有轉錄組數據中的基因注釋,挖掘與凡納濱對蝦生長和抗逆相關候選基因的預測SNP位點,包括胰蛋白酶(Tryptase)、糜蛋白酶(Chymotrypsin)、保幼激素(JH)(杜育哲等,2001)、腺苷酸轉移酶(ANT)、性腺抑制激素(GIH)(邱慶慶,2019)、組織蛋白酶L(CATL)、有絲分裂原活化蛋白激酶(MAPK)、Alpha淀粉酶(AMY)(劉立鶴等,2006)、熱休克蛋白(HSP90)(夏西超等,2012)、ATP轉運酶、對蝦素(Penaeidin)(杜志強,2012,2013)、酚氧化酶(Phenoloxidase,PO)(樊廷俊,2002)、β-葡聚糖結合蛋白(BGBP) (胡世康等,2018)、Toll受體(黃旭雄等,2012)和Crustin-like(張艷艷等,2012)等功能基因。然后選用10尾野生凡納濱對蝦樣本進行驗證,在挑選基因序列中的預測SNP位點兩端設計引物,PCR擴增結果送至上海邁浦生物科技有限公司測序。測序結果使用BioEdit進行分析,讀取轉錄本預測位置的基因型,在10尾野生凡納濱對蝦中其雜合基因型概率達20%的SNP位點所對應的引物即為有效引物,可用于后續的凡納濱對蝦群體基因型檢測。
1. 3 統計分析
獲得4個凡納濱對蝦群體的基因分型數據后,使用PopGen32計算群體的觀測雜合度(Ho)、期望雜合度(He)、Nei基因多樣性指數(H)、Hardy-Weinberg平衡偏離指數(D)、近交系數(Fis)、遺傳分化指數(Fst)、遺傳相似性系數及遺傳距離等(戴習林等,2017);利用Cervus 3.0計算SNP位點的多態信息含量(PIC),剔除多態性過低的SNP位點;采用Arlequin V3.5分析Fst和基因流(Nm),并以MEGA 5.0進行UPGMA聚類分析。
2 結果與分析
2. 1 SNP位點發掘及引物篩選結果
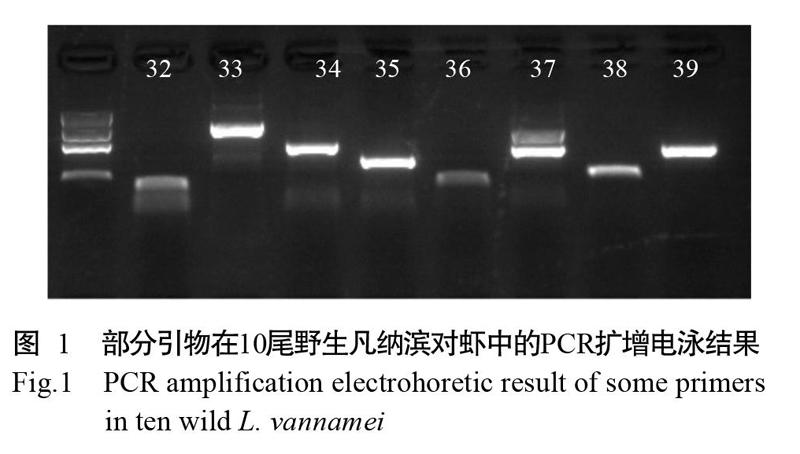
從轉錄組中挖掘獲得70個SNPs位點,設計對應的引物并在10尾野生蝦中進行PCR擴增,結果顯示有46對引物能成功擴增出目的片段。圖1為部分擴增結果的凝膠電泳圖,將擴增獲得且片段大小在預期范圍內的單一明亮條帶(編號33、34、35和39)送至上海邁浦生物科技有限公司測序。根據測序結果讀取峰圖,并統計基因型,發現SNP雜合基因型概率達20%的有效引物共計15對,包含33個SNPs位點。采用15對篩選出的有效引物對4個凡納濱對蝦群體進行PCR擴增,分別統計33個SNPs位點的基因型,并剔除在4個凡納濱對蝦群體中突變率均低于15%的SNP位點,最終獲得11對有效引物(表1),包含24個SNPs位點。其中,HSP90、GIH、CATL、ANT、Tryptase和Chymotrypsin為生長功能基因,含有9個SNPs位點;Toll、Crustin-like、Penaeidin4c和PO為抗逆功能基因,含有15個SNPs位點。
2. 2 遺傳多態性分析結果
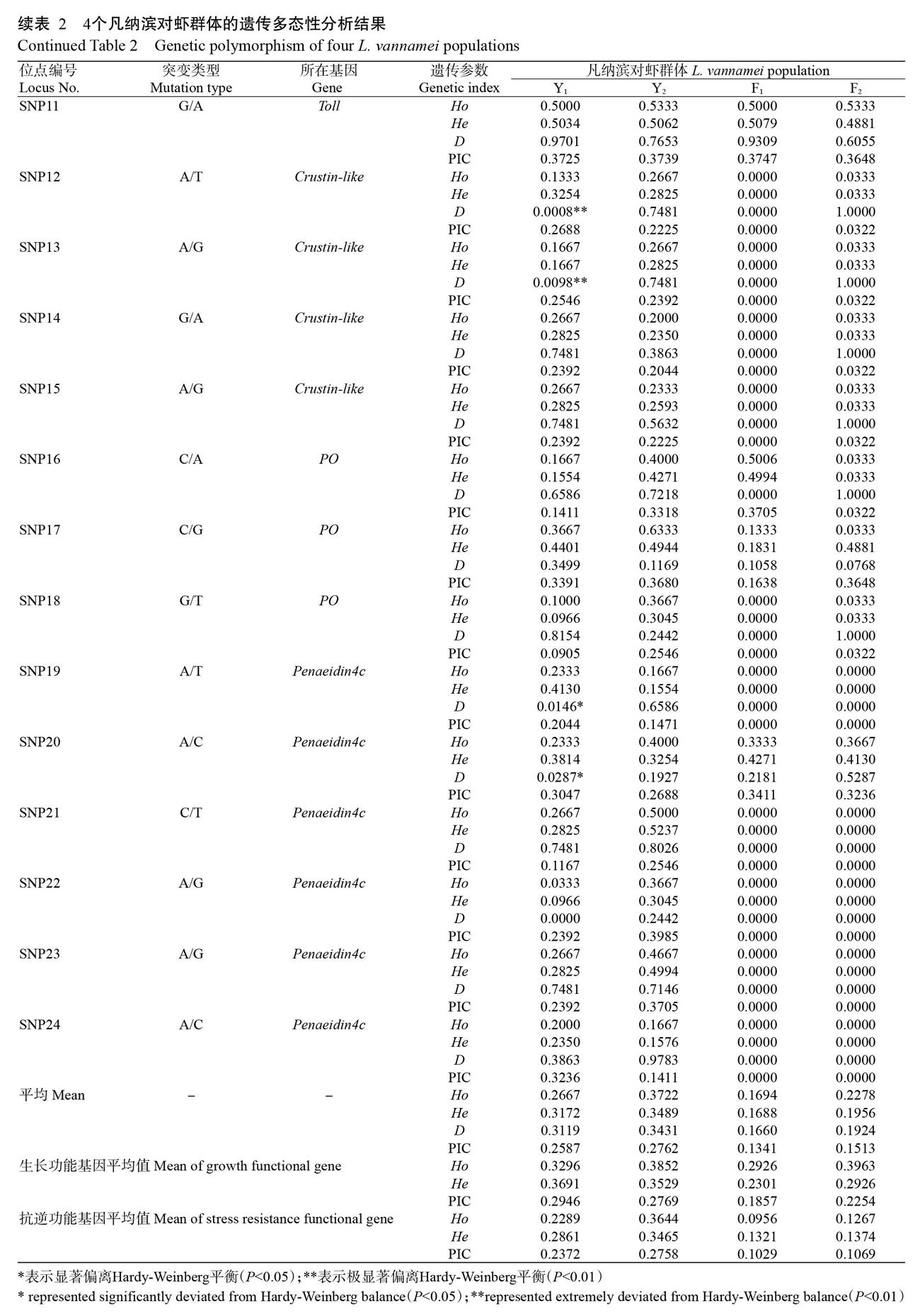
基于9個生長功能基因SNPs位點和15個抗逆功能基因SNPs位點,研究4個凡納濱對蝦群體(2個野生群體和2個選育群體)的遺傳多樣性。結果(表2)顯示,24個SNP分子標記在4個凡納濱對蝦群體中的PIC平均值分別為Y1:0.2587、Y2:0.2762、F1:0.1341和F2:0.1513,2個野生群體明顯高于2個選育群體。其中,Y1和Y2屬于中度多態水平(0.25 總體上,2個野生群體(Y1和Y2)的各項遺傳參數平均值明顯高于2個選育群體(F1和F2)。在2個野生群體中,Y2的各項遺傳參數平均值均高于Y1,在生長功能基因中Y1的PIC和He平均值高于Y2,但在抗逆功能基因中Y2高于Y1。在2個選育群體中,F2的各項遺傳參數平均值均高于F1,在抗逆功能基因中F1和F2基本相同,但在生長功能基因中二者存在明顯差異,表現為F2高于F1。在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位點上,2個選育群體(F1和F2)的各項遺傳參數平均值為0,可能是雜合子缺失所引起。4個凡納濱對蝦群體中均存在SNP位點不同程度偏離Hardy-Weinberg平衡現象(P<0.05為顯著偏離,P<0.01為極顯著偏離)。其中,Y1群體的SNP19和SNP20位點顯著偏離Hardy-Weinberg平衡,處于Penaeidin4c基因上;SNP12和SNP13位點極顯著偏離Hardy-Weinberg平衡,位于Crustin-like基因上;Y2群體的SNP3和SNP7位點顯著偏離Hardy-Weinberg平衡,分別位于GIH和Chymotrypsin基因上;F1群體中除突變缺失位點外,僅SNP3位點顯著偏離Hardy-Weinberg平衡,位于GIH基因上;F2群體中除突變缺失位點外,僅SNP6和SNP7位點顯著偏離Hardy-Weinberg平衡,均位于Chymotrypsin基因上。 2. 3 遺傳分化分析結果 4個凡納濱對蝦群體在24個SNPs位點上的Fis平均值為-0.0224,且負值較多(表3),表明不同群體間的近交程度較低。不同群體間各SNP位點的Fst變化范圍為0.0050~0.2754,說明4個凡納濱對蝦群體在24個SNPs位點上表現出明顯的遺傳分化。4個凡納濱對蝦群體相互間的Fis、Fst和Nm詳見表4。其中,Y1與F1間的Fis最高(0.0875),且Nm也較高(4.2728);Y2與F1間的Fst最高(0.0947),但其Nm最低(2.3886),表明Y2與F1間的遺傳背景較遠。基于SNP分子標記的凡納濱對蝦群體遺傳變異分子生物學方差分析結果(表5)顯示,4個凡納濱對蝦群體的遺傳變異大部分來源于群體內(85.15%),僅有14.85%變異來源于群體間。 2. 4 遺傳距離及聚類分析結果 由表6可知,4個凡納濱對蝦群體的遺傳相似性系數為0.9351~0.9747,遺傳距離為0.0256~0.0671。其中,F1與F2的遺傳相似性系數最大(0.9747),Y2與F1的遺傳相似性系數最小(0.9351);F1與F2的遺傳距離最小(0.0256),Y2與F1的遺傳距離最大(0.0671)。基于凡納濱對蝦群體間的遺傳距離,采用MEGA 5.0進行UPGMA聚類分析,結果(圖2)顯示,Y1、F1和F2聚為一支,而Y2單獨聚為一支。 3 討論 SNP可通過改變堿基引起所處基因的表達變化,導致其編碼蛋白發生結構功能變異,進而影響表型性狀。目前,開展水產動物遺傳多樣性分析時有關分子標記的選擇尚存在隨機性,且主要針對相關功能基因的分子標記進行研究(崔蕾等,2012;孫成飛等,2015;武菲等,2016),利用SNP分子標記進行遺傳多樣性的研究相對較少。本研究基于凡納濱對蝦的生長和抗逆相關功能基因,并通過轉錄組注釋篩選其中的候選SNP位點,最終篩選出24個SNPs位點(9個SNPs位點位于生長功能基因上,15個SNPs位點位于抗逆功能基因上)用于凡納濱對蝦野生和選育群體遺傳多樣性分析。生長和抗逆相關功能基因中的SNP位點差異會影響其所處功能基因表達,相對于其他中性位點更容易造成機體生長性狀及抗病能力的差異,若將其應用到群體遺傳背景分析中,則有望從DNA水平了解群體生長性狀及抗病能力的遺傳差異,更有利于后續將分子標記與性狀聯系起來,輔助良種選育工作的開展。 本研究通過轉錄組數據結合直接測序挖掘出24個功能基因相關SNPs位點(9個生長功能基因SNPs位點,15個抗逆功能基因SNPs位點),并應用于凡納濱對蝦野生和選育群體遺傳多樣性分析,結果表明,野生群體Y1和Y2的遺傳多樣性水平顯著高于選育群體F1和F2,與董在杰等(2018)在鯉魚、王軍等(2018)在中國明對蝦、王沈同等(2018)在草魚中的研究結果相似,可能是由于選育過程中封閉群體的親本數量限制、近親交配、遺傳漂變及對養殖環境適應等因素所引起。2個野生群體具有更豐富的遺傳多樣性,說明其進化潛力更高,即具有較高的遺傳育種改良潛力和適應生存能力。此外,在2個野生群體中,Y2的各項遺傳參數平均值均高于Y1,在生長功能基因中Y1的PIC和He平均值高于Y2,在抗逆功能基因中則Y2高于Y1,究其原因可能是不同野生種群在自然環境中遷移或變異所造成。在2個選育群體中,F2的各項遺傳參數平均值均高于F1,在抗逆功能基因中F1和F2基本相同,但在生長功能基因中二者存在明顯差異,表現為F2高于F1,說明在選擇育種中F2較F1更具優勢。在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位點上,2個選育群體(F1和F2)的各項遺傳參數平均值為0,推測F1和F2存在雜合子缺失現象,與頡曉勇等(2008)、葉寧等(2017)的研究結果相似,是由于人工選育過程中的近親交配現象或對某一特定基因型個體的篩選,造成某些基因從該基因庫中流失。凡納濱對蝦遺傳多樣性降低,極易造成種質資源退化、生長滯緩,進而嚴重影響其養殖業的健康發展(李強勇等,2020)。因此,在凡納濱對蝦選育過程中,需及時了解其遺傳多樣性水平,并在此基礎上進行科學管理,才能在遺傳改良的同時維持選育群體具有較高的遺傳多樣性水平,或從野生群體引種,進一步豐富凡納濱對蝦選育群體的遺傳多樣性。 從本研究的遺傳分化及UPGMA聚類分析可看出,凡納濱對蝦野生群體Y1與2個選育群體(F1和F2)聚為一支,且Nm和Fis均較高,與三者間遺傳距離較近的結果相對應;而凡納濱對蝦野生群體Y2與2個選育群體(F1和F2)間的Fst較高,且Nm較低,與三者間遺傳距離最遠的結果相對應,表明野生群體Y2在家系選擇育種中更具引種優勢和選育潛力。分子生物學方差分析結果也顯示,4個凡納濱對蝦群體的遺傳變異主要來源于群體內(85.15%),僅有14.85%變異來源于群體間,與童馨等(2009)、Rezaee等(2016)的研究結果類似,說明不同凡納濱對蝦群體間相似性較小且基因交流較少,仍具有較高的選育潛力。本研究還發現,在生長功能基因SNP位點遺傳多樣性分析中,F2的遺傳多樣性水平明顯高于F1,且在相同養殖條件下F2的生長性能也優于F1,說明F2無論是在遺傳上還是生長性狀上均優于F1,在后續執行選育計劃時應優先考慮。但從抗逆性狀的遺傳背景來看,2個選育群體(F1和F2)均不具備抗逆優勢,在傳染病頻發的養殖現狀中極易被淘汰。因此,在實際生產中非常有必要通過選育群體與野生群體雜交以提高凡納濱對蝦后代的抗逆能力,可采取抗逆性狀高的野生群體Y2與選育群體相雜交的措施,將Y2的優良抗逆基因引入選育群體,優化其種質資源,提高凡納濱對蝦選育群體的免疫及抗病能力。 4 結論 凡納濱對蝦野生群體在抗逆功能基因SNP分子標記中的遺傳多樣性顯著高于選育群體,且野生群體與選育群體間的遺傳距離較遠,具有較高的遺傳改良潛力。在實際生產中,可通過雜交措施將野生群體的優良抗逆基因引入家系選育群體,優化其種質資源,以提高凡納濱對蝦選育群體的免疫及抗病能力。 參考文獻: 陳錦豪,鄭錦濱,王攀攀,李天驕,毛勇,蘇永全,王軍. 2019. 凡納濱對蝦養殖親本群體遺傳多樣性分析[J]. 漁業研究,41(1):1-10. [Chen J H,Zheng J B,Wang P P,Li T J,Mao Y,Su Y Q,Wang J. 2019. Analysis for genetic diversity of broodstock populations of breeding Litopenaeus vannamei[J]. Journal of Fisheries Research,41(1):1-10.] 陳靜,何吉祥,樊佳佳,黃龍,吳本麗,宋光同,汪翔,武松. 2018. 草魚MyoD基因SNP和Indel標記的篩選及其與生長性狀的關聯分析[J]. 江蘇農業學報,34(3):612-616. [Chen J,He J X,Fan J J,Huang L,Wu B L,Song G T,Wang X,Wu S. 2018. Screening of SNP and Indel mar-ker of MyoD gene and its association with growth traits in Ctenopharyngodon idella[J]. Jiangsu Journal of Agricultural Sciences,34(3):612-616.] 崔蕾,謝從新,李艷和,于永耀,劉肖蓮,張杰,周小云. 2012. 斑點叉尾鮰4個群體遺傳多樣性的微衛星分析[J]. 華中農業大學學報,31(6):744-751. [Cui L,Xie C X,Li Y H,Yu Y Y,Liu X L,Zhang J,Zhou X Y. 2012. Analysis of genetic diversity among four different channel catfish populations by using microsatellite markers[J]. Journal of Huazhong Agricultural University,31(6):744-751.] 戴習林,劉潔,李晶晶,江宗冰,明磊,蔣飛. 2017. 羅氏沼蝦種群SSR分析中樣本量及標記量對遺傳多樣性指標的影響[J]. 水產學報,41(7):1083-1095. [Dai X L,Liu J,Li J J,Jiang Z B,Ming L,Jiang F. 2017. Effects of sample size and loci number on genetic diversity index in Macrobrachium rosenbergii with microsatellite markers[J]. Journal of Fisheries of China,41(7):1083-1095.] 戴習林,楊展昆,朱其建. 2018. 凡納濱對蝦室內養殖密度和簡易水質調控措施對水質及養殖效果的影響[J]. 上海海洋大學學報,27(6):894-906. [Dai X L,Yang Z K,Zhu Q J. 2018. Effects of indoor aquaculture density and simple water quality control measures on water quality and culture efficiency of Litopenaeus vannamei[J]. Journal of Shanghai Ocean University,27(6):894-906.] 董在杰,劉念,傅建軍,朱文彬,王蘭梅,蘇勝彥. 2018. 6個野生與選育鯉群體的微衛星遺傳分析[J]. 南方水產科學,14(4):48-57. [Dong Z J,Liu N,Fu J J,Zhu W B,Wang L M,Su S Y. 2018. Genetic analysis for six wild and selection populations of common carp(Cyprinus carpio) using microsatellites[J]. South China Fisheries Science,14(4):48-57.] 杜育哲,郭世宜,劉麗. 2001. 甲殼動物保幼激素的研究[J]. 天津農學院學報,8(3):34-36. [Du Y Z,Guo S Y,Liu L. 2001. Juvenile hormone in crustacean[J]. Journal of Tianjin Agricultural College,8(3):34-36.] 杜志強. 2012. 先天免疫系統中抗菌肽的研究進展[J]. 廣東農業科學,39(10):231-233. [Du Z Q. 2012. Research progress in antimicrobial peptides of innate immune system[J]. Guangdong Agricultural Sciences,39(10):231-233.] 杜志強. 2013. 凡納濱對蝦對蝦素3a基因的克隆與序列分析[J]. 云南農業大學學報(自然科學),28(1):79-82. [Du Z Q. 2013. Gene cloning and sequence analysis of Penaeidin3a in Litopenaeus vannamei[J]. Journal of Yunnan Agricultural University(Natural Science),28(1):79-82.] 樊廷俊. 2002. 對蝦非特異性免疫與對蝦疾病監控的研究進展[J]. 海洋科學,26(4):26-31. [Fan T J. 2002. Resear-ching Progresses in shrimp non-specific immunity and di-sease control[J]. Marine Sciences,26(4):26-31.] 方振朋,孟憲紅,李旭鵬,欒生,曹家旺,陳寶龍,孔杰,閆茂倉,胡利華. 2019. 基于微衛星分子標記的凡納濱對蝦商業苗種遺傳多樣性分析[J].漁業科學進展,41(5):101-109. [Fang Z P,Meng X H,Li X P,Luan S,Cao J W,Chen B L,Kong J,Yan M C,Hu L H. 2019. Genetic diversity analysis of domestic commercial brands seedlings of Litopenaeus vannamei based on microsatellite molecular markers[J]. Progress in Fishery Sciences,41(5):101-109.] 郝敏. 2016. 凡納濱對與生長繁殖相關的遺傳標記的篩選[D]. 上海:上海海洋大學. [Hao M. 2016. The genetic marker screening for growth and reproduction properties in Litopenaeus vannamei[D]. Shanghai:Shanghai Ocean University.] 胡世康,王博,秦海鵬,廖栩崢,孫成波,劉永勝,曲朋. 2018. 抗菌肽對凡納濱對蝦抗病性和免疫指標的影響[J]. 熱帶生物學報,9(3):281-286. [Hu S K,Wang B,Qin H P,Liao X Z,Sun C B,Liu Y S,Qu P. 2018. Effect of microbial antimicrobial peptides on disease resistance and immune indices of Litopenaeus vannamei[J]. Journal of Tropical Biology,9(3):281-286.] 黃小帥,徐煜,胡曉娟,徐武杰,蘇浩昌,文國樑,楊鏗,曹煜成. 2019. 利用微衛星標記分析7個凡納濱對蝦引進群體子一代的遺傳多樣性[J]. 南方水產科學,15(1):54-62. [Huang X S,Xu Y,Hu X J,Xu W J,Su H C,Wen G L,Yang K,Cao Y C. 2019. Genetic diversity analysis of first filial generation of seven introduced Litopenaeus vannamei populations using microsatellite DNA markers[J]. South China Fisheries Science,15(1):54-62.] 黃旭雄,羅詞興,郭騰飛,溫文,華雪銘,周洪琪. 2012. 凡納濱對蝦Toll受體和溶菌酶mRNA組織表達的差異研究[J]. 上海海洋大學學報,21(1):28-32. [Huang X X,Luo C X,Guo T F,Wen W,Hua X M,Zhou H Q. 2012. Expre-ssions of Toll receptor and lysozyme mRNA among ti-ssues of white shrimp,Litopenaeus vannamei[J]. Journal of Shanghai Ocean University,21(1):28-32.] 蔣葛,沈輝,萬夕和,喬毅,黎慧,王李寶,史文軍. 2019. 凡納濱對蝦急性肝胰腺壞死綜合癥病蝦與健康蝦腸道優勢菌群比較分析[J]. 江蘇農業學報,35(1):142-148. [Jiang G,Shen H,Wan X H,Qiao Y,Li H,Wang L B,Shi W J. 2019. Difference analysis of intestinal flora between healthy Litopenaeus vannamei and acute hepatopancreatic necrosis syndrome L. vannamei[J]. Jiangsu Journal of Agricultural Sciences,35(1):142-148.] 李東宇,孔杰,孟憲紅,欒生,羅坤,盧霞,曹寶祥. 2016. 凡納濱對蝦(Litopenaeus vannamei)微衛星多重PCR體系的建立及其在家系親權鑒定中的應用[J]. 漁業科學進展,37(3):58-67. [Li D Y,Kong J,Meng X H,Luan S,Luo K,Lu X,Cao B X. 2016. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp(Litopenaeus vannamei) and its application for parentage identification[J]. Progress in Fishery Sciences,37(3):58-67.] 李強勇,李旻,曾地剛,朱威霖,彭敏,楊春玲,劉青云,趙永貞,陳秀荔,陳曉漢. 2020. 凡納濱對蝦微衛星分子標記的開發及不同養殖家系遺傳多態性分析[J]. 南方農業學報,51(2):429-436. [Li Q Y,Li M,Zeng D G,Zhu W L,Peng M,Yang C L,Liu Q Y,Zhao Y Z,Chen X L,Chen X H. 2020. Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism ana-lysis of different cultured families[J]. Journal of Southern Agriculture,51(2):429-436. 劉洪濤,楊明秋,何玉貴,唐賢明. 2018. 凡納濱對蝦八個地理群體遺傳多樣性的微衛星分析[J]. 海南大學學報(自然科學版),36(2):146-152. [Liu H T,Yang M Q,He Y G,Tang X M. 2018. Microsatellite analysis of genetic diversity in eight geographical populations of Litopenaeus vannamei[J]. Natural Science Journal of Hainan University,36(2):146-152.] 劉立鶴,陳立僑,周永奎,江洪波. 2006. 甲殼動物消化酶的研究[J]. 飼料工業,27(18):56-62. [Liu L H,Chen L Q,Zhou Y K,Jiang H B. 2006. Studies on digestive enzymes of crustaceans[J]. Feed Industry,27(18):56-62.] 邱慶慶. 2019. 羅氏沼蝦生長性狀候選基因的克隆及mRNA表達規律研究[D]. 南寧:廣西大學. [Qiu Q Q. 2019. Cloning and mRNA expression of candidate genes for growth performance of Macrobrachium rosenbergii[D]. Nanning:Guangxi University.] 孫成飛,葉星,董浚鍵,田園園,梁健輝. 2015. 羅氏沼蝦6個養殖群體遺傳多樣性的微衛星分析[J]. 南方水產科學,11(2):20-26. [Sun C F,Ye X,Dong J J,Tian Y Y,Liang J H. 2015. Genetic diversity analysis of six cultured populations of Macrobrachium rosenbergii using microsate-llite markers[J]. South China Fisherise Science,11(2):20-26.] 童馨,龔世圓,喻達輝,黃桂菊,杜博,李色東. 2009. 凡納濱對蝦(Litopenaeus vannamei)不同世代養殖群體的遺傳多樣性分析[J]. 海洋與湖沼,40(2):214-220. [Tong X,Gong S Y,Yu D H,Huang G J,Du B,Li S D. 2009. Genetic diversity of cultured Pacific white shrimp(Litopenaeus vannamei) stocks of different generations in China[J]. Oceanologia et Limnologia Sinica,40(2):214-220] 王軍,王清印,孔杰,孟憲紅,曹家旺,王明珠,馮亞萍,呂丁. 2018. 中國明對蝦人工選育群體與野生群體遺傳多樣性的SSR分析[J]. 漁業科學進展,39(2):104-111. [Wang J,Wang Q Y,Kong J,Meng X H,Cao J W,Wang M Z,Feng Y P,Lü D. 2018. SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis[J]. Progress in Fishery Sciences,39(2):104-111.] 王全超. 2017. 凡納濱對蝦生長和抗病性狀的全基因組關聯分析與基因組選擇育種研究[D]. 北京:中國科學院大學. [Wang Q C. 2017. Genome-wide association study and genomic selection of growth and disease resistance traits in Litopenaeus vannamei[D]. Beijing:University of Chinese Academy of Sciences.] 王沈同,沈玉幫,孟新展,王榮泉,李家樂. 2018. 草魚野生與選育群體遺傳變異微衛星分析[J]. 水產學報,42(8):1273-1284. [Wang S T,Shen Y B,Meng X Z,Wang R Q,Li J L. 2018. Genetic variability in wild and selected populations of Ctenopharyngodon idella using microsatellite markers[J]. Journal of Fisheries of China,42(8):1273-1284.] 王帥豪. 2012. 淅川烏骨雞群體遺傳多樣性及綠殼蛋性狀相關基因SNP分析[D]. 鄭州:河南農業大學. [Wang S H. 2012. SNP analysis about green egg shell correlation gene and population genetic diversity of Xichuan silky[D]. Zhengzhou:Henan Agricultural University.] 武菲,胡文革,王翠華,郭飏,莫超. 2016. 河鱸養殖與野生群體遺傳多樣性比較分析[J]. 水生生物學報,40(1):181-188. [Wu F,Hu W G,Wang C H,Guo Y,Mo C. 2016. Genetic diversity of the cultivated and natural Perca fluviatilis[J]. Acta Hydrobiologica Sinica,40(1):181-188.] 夏西超,楊洪,王文鋒,李昕,寧黔冀. 2012. 凡納濱對蝦熱休克蛋白90基因cDNA全長克隆及表達分析[J]. 水產科學,31(11):673-678. [Xia X C,Yang H,Wang W F,Li X,Ning Q J. 2012. Molecular cloning and expression of heat shock protein 90(HSP90) gene in Pacific white leg shrimp Litopenaeus vannamei[J]. Fisheries Science,31(11):673-678.] 頡曉勇,蘇天鳳,陳文,張志,顏遠義,江世貴. 2008. 凡納濱對蝦6個養殖群體遺傳多樣性的比較分析[J]. 南方水產,4(6):42-49. [Xie X Y,Su T F,Chen W,Zhang Z,Yan Y Y,Jiang S G. 2008. Analysis on genetic diversity of six cultured stocks of Litopenaeus vannamei[J]. South China Fisheries Science,4(6):42-49.] 楊銘,于洋,張曉軍,王全超,劉敬文,李富花,相建海. 2017. 基于轉錄組數據的凡納濱對蝦微衛星標記開發[J]. 海洋科學,41(2):96-102. [Yang M,Yu Y,Zhang X J,Wang Q C,Liu J W,Li F H,Xiang J H. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp(Litopenaeus vannamei)[J]. Marine Sciences,41(2):96-102.] 葉寧,包秀鳳,劉建勇. 2017. 凡納濱對蝦引進群體與養殖群體遺傳多樣性的AFLP分析[J]. 水產學報,41(3):339-346. [Ye N,Bao X F,Liu J Y. 2017. AFLP analysis of genetic diversity of imported Litopenaeus vannamei popu-lations and cultured stocks[J]. Journal of Fisheries of China,41(3):339-346.] 于磊,劉炳艦,劉進賢. 2018. 日本鰻鱺離散SNP標記篩選及群體遺傳多樣性分析[J]. 中國海洋大學學報(自然科學版),48(4):37-47. [Yu L,Liu B J,Liu J X. 2018. Outlier SNPs development and preliminary genetic diversity ana-lysis of Japanese eel(Anguilla japonica)[J]. Periodical of Ocean University of China,48(4):37-47.] 于洋. 2014. 凡納濱對蝦分子標記的開發及其在遺傳育種中的應用[D]. 北京:中國科學院大學. [Yu Y. 2014. Deve-lopment of molecular markers and their applications in selective breeding of the Pacific white shrimp,Litopenaeus vannamei[D]. Beijing:University of Chinese Academy of Sciences.] 張靈俠,沈琪,胡超群. 2008. 凡納濱對蝦的遺傳育種研究現狀[J]. 海洋科學,32(2):91-95. [Zhang L X,Shen Q,Hu C Q. 2008. A review of genetics and breeding of Litopenaeus vannamei[J]. Marine Sciences,32(2):91-95.] 張曉萌,馬普,王洪迪,王秀利. 2013. SNPs在水產動物中的研究進展[J]. 生物技術通報,(8):7-11. [Zhang X M,Ma P,Wang H D,Wang X L. 2013. Progresses of SNPs studies in aquaculture animals[J]. Biotechnology Bulletin,(8):7-11.] 張艷艷,劉小林,黃海洪,錢昭英,王憲宗,相建海,李義軍. 2012. 凡納濱對蝦Crustin-like基因的克隆及在副溶血弧菌感染條件下的表達分析[J]. 西北農林科技大學學報(自然科學版),40(12):41-48. [Zhang Y Y,Liu X L,Huang H H,Qian Z Y,Wang X Z,Xiang J H,Li Y J. 2012. Molecular cloning of Crustin-like gene in the white shrimp(Litopenaeus vannamei) and its mRNA expression with Vibrio parahaemolyticus challenge[J]. Journal of Nor-thwest A & F University(Natural Science Edition),40(12):41-48.] Agarwal D,Aich N,Pavan-Kumar A,Kumar S,Sabnis S,Joshi C G,Koringa P,Pandya D,Patel D,Karnik T,Bhingarde R,Gireesh-Babu P,Chaudhari A. 2016. SNP mi-ning in transcripts and concomitant estimation of genetic variation in Macrobrachium rosenbergii stocks[J]. Conservation Genetics Resources,8(2):159-168. Rezaee S,Farahmand H,Nematollahi M A. 2016. Genetic diversity status of Pacific white shrimp(Litopenaeus vannamei) using SSR markers in Iran[J]. Aquaculture International,24(2):479-489. Thongda W,Zhao H G,Zhang D D,Jescovitch L N,Liu M,Guo X M,Schrandt M,Powers S P,Peatman E. 2018. Development of SNP panels as a new tool to assess the genetic diversity,population structure,and parentage ana-lysis of the eastern oyster(Crassostrea virginica)[J]. Marine Biotechnology(New York),20(3):385-395. Yu Y,Wang Q C,Zhang Q,Luo Z,Wang Y,Zhang X J,Huang H,Xiang J H,Li F H. 2019. Genome scan for genomic regions and genes associated with growth trait in Pacific white shrimp Litopenaeus vannamei[J]. Marine Biotechnology(New York),21(3):374-383. (責任編輯 蘭宗寶) 收稿日期:2020-02-15 基金項目:上海市蝦類產業技術體系建設專項(滬農科攻字〔2014〕第7-1-11號) 作者簡介:*為通訊作者,戴習林(1969-),教授,主要從事水產養殖與海洋生物學研究工作,E-mail:xldai@shou.edu.cn。申淑慧(1995-),研究方向為羅氏沼蝦遺傳育種,E-mail:18994535472@163.com

