特異性檢測珍珠龍膽石斑魚虹彩病毒病的核酸適配體的篩選與鑒定
余慶 劉明珠 肖賀賀 易弋 程昊 Putra Dedi-Fazriansyah 黎思巧 李鵬飛

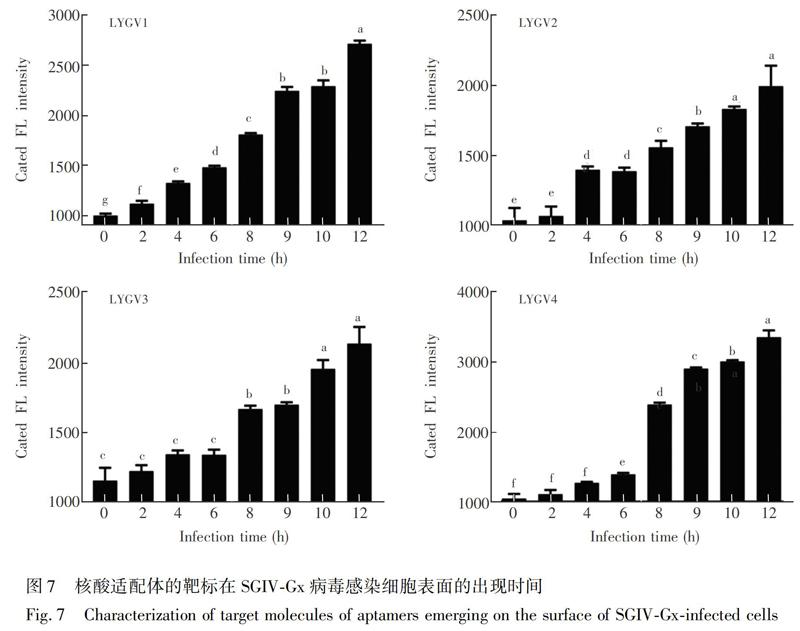
摘?要?石斑魚虹彩病毒是一種核質DNA病毒,不僅給海水養殖造成巨大的經濟損失,而且嚴重威脅全球生物多樣性。為了進一步闡明石斑魚虹彩病毒侵染致病的分子機理,同時為石斑魚虹彩病毒病的預防、診斷和治療提供新的理論依據,本研究以珍珠龍膽石斑魚虹彩病毒(SGIV-Gx)感染的宿主細胞為靶標,采用指數富集配體系統進化技術(SELEX)篩選獲得4條核酸適配體(LYGV1、 LYGV2、 LYGV3、 LYGV4),并對核酸適配體的性質進行了系統的分析研究。各條核酸適配體識別SGIV-Gx病毒感染的宿主細胞具有高特異性和高親和性,篩選的核酸適配體無毒副作用,而且核酸適配體LYGV3具有一定的抗病毒效果。對核酸適配體的靶標性質進行了分析,結果表明,核酸適配體(LYGV1、 LYGV2、 LYGV3、 LYGV4)的靶標可能是細胞膜蛋白或膜蛋白相關成分。其中,核酸適配體LYGV1的靶位點在SGIV-Gx病毒感染細胞2 h后出現在宿主細胞表面,核酸適配體LYGV2、LYGV3和LYGV4的靶位點的出現時間分別為病毒感染細胞后的4、8和6 h。
關鍵詞?石斑魚虹彩病毒;指數富集配體系統進化技術;核酸適配體;特異性;抗病毒;靶蛋白
1?引 言
石斑魚肉質細膩,營養豐富,為大宗名貴海水養殖魚類,目前國內養殖年產量已達15萬噸以上,直接產值超過百億元,經濟價值極高[1,2]。然而,隨著石斑魚養殖密度增加、養殖規模擴大,養殖環境日益惡化,進而導致了魚類疫病頻繁暴發,經濟損失巨大[3]。其中,石斑魚虹彩病毒危害嚴重的石斑魚疫病病原,其所導致的魚病致死率極高[4]。目前,研究者已對石斑魚虹彩病毒開展了系統研究,包括石斑魚虹彩病毒的流行病學調查、病原分離鑒定、石斑魚細胞系構建、病毒粒子的超微結構觀察、檢測診斷技術開發等,在一定程度上揭示了石斑魚虹彩病毒的侵染致病機理[5~7]。在目前高密度、集約化的養殖環境下,石斑魚虹彩病毒通常表現為暴發式感染,會在較短時間內導致人工養殖石斑魚大批死亡,因此,必須著力研發能夠快速、便捷檢測石斑魚虹彩病毒病的檢測技術,實現對病毒的及時診斷,保障養殖業健康持續發展。Qin等[8]制備了虹彩病毒SGIV的高特異性抗體,研發出可以特異性檢測石斑魚虹彩病毒感染的流式檢測技術。
核酸適配體是利用指數富集的配體系統進化技術(Systematic evolution of ligands by exponential enrichment,SELEX)在體外經過多輪嚴格篩選得到的高特異性識別靶物質的單鏈寡核苷酸。核酸適配體結構穩定,對溫度等外界環境的耐受性高,具有易于化學合成和化學修飾等諸多優點[9,10],廣泛應用于病原或疾病診斷治療等生命科學領域[11~14]。基于核酸適配體能夠識別病原微生物或病變細胞的高特異性高親和性等特性,核酸適配體被用于生物傳感器的構建,通過信號放大,實現對病原微生物或疾病的精確檢測診斷。Duan等[15]利用高特異性識別食源性致病菌的核酸適配體,結合上轉換熒光納米標記技術和免標記傳感器等構建了一系列靈敏、便捷的檢測新方法,成功實現了對樣品中目標致病菌的精確檢測。Zhou等[16]利用特異性識別石斑魚神經壞死病毒RGNNV衣殼蛋白CP的核酸適配體A10,開發了用于RGNNV精確快速檢測的Sandwich ELASA方法,該方法靈敏度高、穩定性強,可用于養殖現場對病原的快速檢測。此外,核酸適配體能夠高特異性地結合在病原的特定部位,發揮功能阻斷劑的作用,抑制病原的感染。Gao等[17]篩選了特異性結合Hepatitis C病毒NS2蛋白和包膜蛋白的ssDNA核酸適配體,對相關核酸適配體的抗病毒效果分析后發現,它們可有效抑制Hepatitis C病毒在細胞中的復制和裝配。綜上,核酸適配體在基礎研究和應用研究中具有重要的理論意義、實際價值和應用前景。
本研究以珍珠龍膽石斑魚虹彩病毒(SGIV-Gx)感染的石斑魚脾細胞為靶標,采用SELEX技術篩選獲得了可高特異性、高親和性識別SGIV-Gx感染細胞的核酸適配體,并對核酸適配體的性質進行系統的研究,包括核酸適配體的特異性、親和性、抗病毒活性、特異性主動,以及內吞能力和靶位點的性質等。
2?實驗部分
2.1?儀器與試劑
Nikon C2激光共聚焦顯微鏡、TS2倒置光學顯微鏡(日本Nikon公司);FACSAria II流式細胞儀(美國BD公司);Multiskan Ascent多功能酶標儀、Nano Drop one超微量分光光度計;Mastercycler nexus gradient PCR儀(德國Eppendorf公司);qTOWER3G touch熒光定量PCR儀(德國Analytikjena公司);AC2-4S1生物安全柜(新加坡ESCO公司);Centrifuge 5417R離心機(德國Eppendorf公司);Gel Doc XR+凝膠成像系統(美國BIO-RAD公司);DK-8D水浴鍋(博訊公司)。
珍珠龍膽石斑魚虹彩病毒廣西株(Grouper iridovirus guangxi strain,SGIV-Gx)、中華鱉虹彩病毒(Soft shell turtle iridovirus,STIV)、石斑魚脾細胞(Grouper spleen cell,GS)和胖頭鯉細胞(Fathead minnow cell,FHM)保存于本實驗室[18,19];細胞培養瓶、細胞培養皿、波底皿、細胞6孔板和細胞12孔板(美國Corning公司);胎牛血清(Fetal bovine serum,FBS,美國GIBCO公司);鏈霉親和素標記的磁珠(美國Thermo公司);PCR純化回收試劑盒、膠回收試劑盒、DL2000 Marker、DL50 bp Marker(日本TakaRa公司)。磷酸鹽緩沖溶液(PBS): 2 mmol/L KH2PO4,10 mmol/L Na2HPO4,137 mmol/L NaCl,pH 7.2。
2.2?實驗方法
2.2.1?隨機單鏈DNA文庫和引物的設計構建?用于核酸適配體篩選的隨機起始單鏈ssDNA文庫、引物和核酸適配體均由上海生工公司合成。隨機起始單鏈ssDNA文庫由中間50 bp的隨機核苷酸序列(N50)和兩端的固定核苷酸序列構成(5-GACGCTTACTCAGGTGTGACTCG-N50-CGAAGGACGCAGAGAAGTCTC-3)。5端引物序列為5-GACGCTTACTCAGGTGTGACTCG-3;3端引物序列為5-GAGACTTCATCTGCGTCCTTCG-3。其中,5端引物有兩種,分別標記羥基熒光素(6-Carboxy-fluorescein labeled forward primer,FAM-FP)和無熒光標簽的普通引物(FP);3端引物標記生物素(Biotin labeled reverse primer,biotin-RP)。
2.2.2?SGIV-Gx病毒感染細胞(靶標細胞)的準備與處理?將石斑魚脾細胞(GS)接入細胞培養瓶,瓶底基本鋪滿細胞后,細胞中接入20 μL SGIV-Gx(107 TCID50/mL)(TCID50(Tissue culture infective dose)是指病毒能使50%的組織培養細胞發生細胞病變(Cytopathic effect,CPE)的最小量),繼續培養24 h后,移除細胞培養基,將細胞用PBS緩沖液輕輕洗滌2次,備用。
2.2.3?SELEX技術篩選流程?首輪篩選流程如下所述:將10 nmol隨機起始單鏈DNA文庫溶解在500 μL PBS緩沖液中,92℃高溫變性8 min,迅速插入冰中,靜置15 min。起始文庫與靶標細胞在4℃孵育結合40 min后,離心,移除上清液,并且將靶標細胞用PBS緩沖液清洗。收集靶標細胞,在92℃孵育處理5 min,結合的單鏈DNA從靶細胞上解離,1000 r/min離心5 min,收集上清液中的單鏈DNA,將其作為模板進行PCR擴增。PCR程序為:94℃ 2 min,94℃ 1 min,60℃ 30 s,72℃ 1 min,20輪循環;72℃ 5 min,獲得雙鏈dsDNA。
利用“鏈霉親和素-生物素”結合系統分離得到單鏈核酸文庫,用于次輪篩選。將100 μL包被有鏈霉親和素的磁珠與PCR擴增所得的dsDNA室溫孵育30 min,利用dsDNA中反向鏈上的生物素與磁珠上鏈霉親和素的親和作用,使dsDNA結合到磁珠上;磁性分離,將磁珠用PBS緩沖液清洗2次后,加入200 μL 200 mmol/L NaOH,室溫孵育15 min,利用堿變性的方法將dsDNA的雙鏈分離,正向核酸單鏈游離在上清液中,利用磁性分離器分離磁珠并收集上清液,用HCl溶液調節上清液pH值。將上清液中正向單鏈核酸用PCR純化回收試劑盒純化回收,收集單鏈核酸文庫用于次輪篩選。為了提高后續篩選中核酸單鏈識別靶細胞的特異性和親和力,在隨后的篩選過程中逐步縮短核酸單鏈文庫與靶標細胞的結合時間,同時減少核酸單鏈和靶標細胞的使用量,增加PBS緩沖液清洗靶標細胞的次數。
2.2.4?陰性篩選?為了提高篩選效率和成功率,在第3輪及第3輪以后篩選中,進行了陰性篩選,包括“陰性前篩選”和“陰性后篩選”。“陰性前篩選”是在每輪正式篩選前,即ssDNA單鏈文庫與靶標細胞孵育結合前,先將ssDNA單鏈文庫與正常GS細胞在4℃孵育1 h,離心,上清液即為經過“陰性前篩選”的單鏈核酸文庫,將上清液與靶標細胞孵育結合。“陰性后篩選”是指每輪正式篩選后,收集靶標細胞上結合的ssDNA單鏈文庫,然后將此文庫與正常GS細胞在4℃孵育1 h,1000 r/min離心5 min,上清液即為經過“陰性后篩選”的單鏈核酸文庫,以此文庫作為模板進行PCR擴增。
2.2.5?核酸適配體序列的確定?將最終得到的單鏈核酸文庫經PCR擴增為dsDNA,切膠回收后,連接到pUC-T 載體并轉化到大腸桿菌DH5α中,菌液均勻涂布在LB 固體培養基(氨芐抗性),在28℃生化培養箱倒置培養過夜,挑取單克隆由上海生工公司測序,得到核酸適配體序列。使用MFOLD軟件預測核酸適配體的二級結構。
2.2.6?激光共聚焦顯微鏡分析核酸適配體識別靶標細胞的特異性?將GS細胞接入玻底皿中,28℃培養18 h。將20 μL SGIV-Gx病毒(107 TCID50/mL)接入細胞中,對照組共兩組,分別為不接入SGIV-Gx病毒的正常GS細胞和接入STIV病毒的FHM細胞。各組細胞在28℃繼續培養24 h,在光鏡下觀察細胞的病變情況。FAM熒光標記的核酸適配體(500 nmol/L)經過92℃恒溫水浴、冰浴處理后加入細胞中,4℃孵育40 min后,移除上清液,并用細胞培養基清洗,略干燥后,用抗熒光淬滅劑封片,使用激光共聚焦顯微鏡對各組細胞進行熒光觀察。
2.2.7?流式細胞術定量分析核酸適配體識別靶標細胞的特異性?GS細胞接入24孔板中,28℃培養18 h。將20 μL SGIV-Gx病毒(107 TCID50/mL)接入24孔板的細胞中,28℃繼續培養24 h。對照組共兩組,分別為不接入SGIV-Gx病毒的正常GS細胞和接入STIV病毒的FHM細胞。在光鏡下觀察細胞的病變情況,然后收集24孔板中的細胞。FAM熒光標記的核酸適配體(500 nmol/L)經過92℃恒溫水浴、冰浴的變復性處理,與病毒感染的細胞于4℃結合40 min。離心清洗3次,用500 μL PBS緩沖液重懸,使用流式細胞儀檢測。每個反應均重復3次。
2.2.8?核酸適配體的細胞毒性分析?在96孔板中接種GS細胞,28℃培養18 h。將核酸適配體在細胞培養基中稀釋至2000 和1000 nmol/L,與細胞于28℃孵育48 h。在各孔中加入20 μL CCK-8溶液,室溫孵育4 h后,利用酶標儀檢測450 nm的吸光值,以檢測細胞活性,以未與核酸適配體孵育的正常GS細胞作為對照組,做3個平行實驗。將測得的吸光值代入公式(1),計算各組細胞的存活率(Survival rate,SR)[20,21]:
2.2.9?核酸適配體的親和常數計算?FAM熒光標記的核酸適配體經過92℃恒溫水浴、冰浴的變復性處理后,稀釋至不同濃度(0~2000 nmol/L),與SGIV-Gx病毒感染的GS細胞在4℃避光結合40 min。離心清洗3次,重懸在500 μL PBS中,用流式細胞儀檢測,以FAM熒光標記的核酸適配體與正常細胞的結合作為對照。不同濃度核酸適配體與靶標細胞結合的熒光強度平均值減去相應對照細胞結合平均值,按公式(2)計算:
其中,Bmax表示核酸適配體與靶標細胞結合的最大平均熒光強度值。Y表示在核酸適配體在相應濃度下與靶標細胞結合的平均熒光強度值;X表示核酸適配體在相應濃度;Kd表示核酸適配體的親和常數,采用Sigmaplot軟件進行計算。每個反應均做3個重復。
2.2.10?核酸適配體的靶標性質分析?將SGIV-Gx病毒感染的GS細胞用胰酶消化處理2 min,將細胞離心清洗3次,與FAM熒光標記的核酸適配體(500 nmol/L)在4℃孵育40 min。離心清洗3次,然后重懸在500 μL PBS中,使用流式細胞儀檢測。對照組中,FAM熒光標記的核酸適配體與SGIV-Gx病毒感染的GS細胞孵育結合。每個反應均做3個重復。
2.2.11?核酸適配體的靶標出現在SGIV-Gx病毒感染的GS細胞表面的時間?GS細胞在24孔板中28℃培養18 h,將20 μL SGIV-Gx(107 TCID50/mL)病毒接入24孔板的細胞中,28℃繼續培養。分別在病毒感染的第2、4、6、8、10和12 h觀察細胞的病變情況,收集細胞與FAM熒光標記的核酸適配體(500 nmol/L) 在4℃避光結合40 min。離心清洗3次,重懸在500 μL PBS中,用流式細胞儀檢測。對照組將FAM熒光標記的核酸適配體(500 nmol/L)與正常GS細胞孵育結合。每個反應均做3個重復。
2.2.12?核酸適配體的抗病毒活性分析?核酸適配體對SGIV-Gx病毒的抑制效果分析實驗參照文獻[21,22]方法。GS細胞在24孔板于28℃培養18 h,然后將核酸適配體(500 nmol/L)與1 μL SGIV-Gx(107 TCID50/mL)病毒冰浴結合后接種入細胞。僅接入病毒、不接入核酸適配體的細胞作為對照組。然后分別在12和24 h收集24孔板中的細胞和培養基,提取RNA,反轉錄擴增獲得cDNA,以cDNA為模板,β-actin基因作為內參基因,利用RT-qPCR技術檢測石斑魚虹彩病毒主要衣殼蛋白MCP基因和病毒囊膜蛋白VP19基因的表達量,每個反應均做3個重復。結果用于分析核酸適配體的抑制SGIV-Gx病毒的抗病毒活性。RT-qPCR技術檢測MCP基因和囊膜蛋白VP19基因所使用的引物見表1。
3?結果與討論
3.1?SELEX技術篩選SGIV-Gx病毒感染細胞(靶標細胞)的特異性核酸適配體
細胞被病毒感染時,細胞膜成分將發生修飾或改變,這些修飾和改變是識別病毒感染細胞和病毒感染治療的重要生物標志物[23]。本研究以SGIV-Gx病毒感染的石斑魚脾細胞(GS)為靶標細胞,運用SELEX技術進行靶標細胞的核酸適配體篩選。以FAM熒光標記的隨機ssDNA文庫(500 nmol/L)與靶標細胞的結合為對照,利用流式細胞術檢測發現,隨著篩選輪數增加,單鏈核酸文庫的特異性逐步升高,經過12輪篩選,第11輪的單鏈核酸文庫對靶標細胞的特異性識別能力最強(圖1)。因此對第11輪的篩選文庫進行克隆測序,共得到4條核酸適配體LYGV1、LYGV2、LYGV3、LYGV4(表2)。已報道了一些針對病毒性或細菌性病的特異性核酸適配體[13,20~30]等。目前,核酸適配體的技術難點仍然主要在于篩選流程,不同物質作為靶標進行核酸適配體的篩選,所需的SELEX技術篩選輪數和參數不同,SELEX技術仍未能實現自動化和標準化,因此篩選出高特異性核酸適配體的難度較高。例如,Liang等[22]以狂犬病病毒感染細胞為靶標,經過35輪的篩選才最終得到高特異性的核酸適配體T14和 F34;而Zhou等[20]僅經過11輪的篩選就獲得了特異性識別赤點神經壞死癥病毒的核酸適配體。本研究為了提高篩選效率,參考文獻報道,對陰性篩選流程進行了優化改進,分別設置了“陰性前篩選”和“陰性后篩選”,因此,經過11輪篩選就成功獲得了高特異性識別SGIV-Gx病毒感染的石斑魚脾細胞的核酸適配體庫。
3.2?核酸適配體二級結構預測與分析
利用MFOLD對篩選獲得的核酸適配體LYGV1、LYGV2、LYGV3、LYGV4的二級結構和吉布斯自由能(ΔG)進行預測分析。4條核酸適配體的二級結構預測結果表明,它們均會形成獨特復雜的莖環結構,其中LYGV3的吉普斯自由能(ΔG)最低,為26.80 kJ/mol,提示其結構最穩定(圖2)。在二級結構的基礎上,核酸適配體通過氫鍵、堿基堆積和范德華力等自折疊形成更復雜的立體結構,當靶物質存在時,核酸適配體可以通過自適應變化、空間構型互補,特異性識別和結合到靶物質的結構位點。因此這些二級莖環結構構成了核酸適配體與靶物質特異性結合的結構基礎[11,31]。據報道,核酸適配體的部分核苷酸序列或結構域對于其特異性是非必需的,在未來的研究中,有必要對核酸適配體的高特異性識別靶物質的核酸序列或結構域進行分析鑒定,這對于優化核酸適配體結構、制備具有更高親和力和特異性的核酸適配體用于相關功能產品的開發具有重要意義[31]。
3.3?適配體的特異性分析
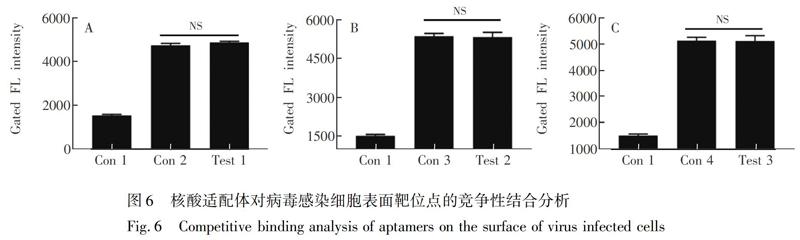
可用于核酸適配體篩選的靶標種類廣泛,包括金屬離子、無機和有機小分子、肽和蛋白質等單一靶標分子,以及病毒、寄生蟲、細胞、組織等復雜生物靶標體系[32]。但是,針對細胞復雜靶標體系的篩選,最終獲得的特異性核酸適配體庫可能會更復雜,這不僅取決于復雜靶標體系中目標分子的數量和豐度,還取決于核酸適配體自身針對不同靶標分子的特異性。對本研究篩選獲得的核酸適配體的特異性進行了系統分析(圖3)。流式細胞術檢測結果表明,實驗組SGIV-Gx病毒感染的細胞熒光值高,對照組細胞的熒光值較低,而且實驗組與對照組的數據差異極顯著,說明核酸適配體LYGV1、LYGV2、LYGV3、LYGV4識別SGIV-Gx病毒感染的細胞具有高特異性(圖3A)。
9?Ellington A D,Szostak J W. Nature,??1990,?346: 818-822
10?ZOU Xue-Mei,ZHOU Jia-Wei,SONG Shang-Hong,CHEN Guan-Hua. Chinese J. Anal. Chem.,??2019,?47(4): 488-499
鄒雪梅,周佳偉,宋尚紅,陳冠華. ?分析化學,?2019,?47(4): 488-499
11?Duclair S,Gautam A,Ellington A,Prasad V R. Mol. Ther. Nucleic Acids,??2015,?4: e228
12?Li P,Yan Y,Wei S,Wei J,Gao R,Huang X,Huang Y,Jiang G,Qin Q. Virus Res.,??2014,?188: 146-154
13?Yu Q,Liu M,Su H,Xiao H,Wu S,Qin X,Li S,Mi H,Lu Z,Shi D,Li P. J. Fish Dis.,???2019,?42(6): 851-858
14?Zhou J,Rossi J J. BioDrugs,??2012,?26(6): 393-400
15?Duan N,Wu S,Dai S,Gu H,Hao L,Ye H,Wang Z. Analyst,??2016,?141(13): 3942-3961
16?Zhou L,Li P,Ni S,Yu Y,Yang M,Wei S,Qin Q. J. Fish Dis.,??2017,?40(12): 1831-1838
17?Gao Y,Yu X,Xue B,Zhou F,Wang X,Yang D,Liu N,Xu L,Fang X,Zhu H. PLoS One,??2014,?9(2): e90333
18?Yu Q,Liu M,Wei S,Xiao H,Wu S,Qin X,Shi D,Li S,Wang T,Li P. Fish. Pathol.,??2019,?54(1): 16-19
19?Xiao H,Liu M,Li S,Shi D,Zhu D,Ke K,Xu Y,Dong D,Zhu L,Yu Q,Li P. J. Aquat. Anim. Health,??2019,?31(4): 364-370
20?Yu Q,Liu M,Wei S,Wu S,Xiao H,Qin X,Su H,Li P. J. Gen. Virol.,??2019,?100(3): 380-391
21??Li P,Wei S,Zhou L,Yang M,Yu Y,Wei J,Jiang G,Qin Q. J. Gen. Virol.,??2015,?96: 3348-3359
22?Liang H R,Hu G Q,Zhang T,Yang Y J,Zhao L L,Qi Y L,Wang H L,Gao Y W,Yang S T,Xia X Z. Int. Immunopharmacol.,??2012,?14(3): 341-347
23?Tang Z,Parekh P,Turner P,Moyer R W,Tan W. Clin. Chem.,??2009,?55(4): 813-822
24?Bai C,Lu Z,Jiang H,Yang Z,Liu X,Ding H,Li H,Dong J,Huang A,Fang T,Jiang Y,Zhu L,Lou X,Li S,Shao N. Biosens. Bioelectron.,??2018,?110: 162-167
25?Lu T,Ma Q,Yan W,Wang Y,Zhang Y,Zhao L,Chen H. Talanta,??2018,?176: 214-220
26?Li P,Zhou L,Yu Y,Yang M,Ni S,Wei S,Qin Q. BMC Vet. Res.,??2015,?11: 245
27?Hwang S D,Midorikawa N,Punnarak P,Kikuchi Y,Kondo H,Hirono I,Aoki T. J. Fish Dis.,??2012,?35(12): 927-934
28?Zhou L,Li P,Yang M,Yu Y,Huang Y,Wei J,Wei S,Qin Q. Antiviral. Res.,??2016,?129: 104-114
29?Tang X,Zheng J,Yan Q,Li Z,Li Y. Biotechnol. Lett.,??2013,?35(6): 909-914
30?Duan N,Wu S,Yu Y,Ma X,Xia Y,Chen X,Huang Y,Wang Z. Anal. Chim. Acta,??2013,?804: 151-158
31?Li P,Yu Q,Zhou L,Dong D,Wei S,Ya H,Chen B,Qin Q. Virus. Res.,??2018,?246: 46-54
32?WANG Wei,JIA Ling-Yun. Chinese J. Anal. Chem.,??2009,?37(3): 454-460
王 巍,賈凌云. ?分析化學,2009,?37(3): 454-460
33?WANG Yong,ZHAO Xin-Ying,SHI Dong-Dong,YANG Ge,QU Feng. Chinese Journal of Chromatography,??2016,?34(4): 361-369
王 勇,趙新穎,石冬冬,楊 歌,屈 鋒. ?色譜,?2016,?34(4): 361-369
34?LIU Pin-Duo,QU Feng. Chinese Journal of Chromatography,??2016,?34(4): 382-388
劉品多,屈 鋒. ?色譜,?2016,?34(4): 382-388
35?Shangguan D H,Meng L,Cao Z C,Xiao Z,Fang X,Li Y,Cardona D,Witek R P,Liu C,Tan W H. Anal. Chem.,??2008,?80(3): 721-728
36?Jin C,Qiu L,Li J,Fu T,Zhang X,Tan W. Analyst,??2016,?141(2): 461-466
37?Van S,Turek D,Champanhac C,Vaizer J,Sefah K,Zhen J,Sutphen R,Tan W. Anal. Chem.,??2014,?86(9): 4521-4527
38?Yu Q,Liu M,Wei S,Xiao H,Wu S,Ke K,Huang X,Qin Q,Li P. Front. Microbiol.,??2019,?10: 2684
39?Simaeys D V,Turek D,Champanhac C,Vaizer J,Sefah K,Zheng J,Sutphen R,Tan W H. Anal. Chem.,??2014,?86(9): 4521-4527
40?Porntep P,Mudjekeewis D S,Seong D H,Hidehiro K,Ikuo H,Yo K,Takashi A. Mar. Biotechnol.,??2012,?14: 752-761
41?Jeon S H,Kayhan B,Ben-Yedidia T,Arnon R. J. Biol. Chem.,??2004,?279: 48410-48419

