轉錄組測序分析糖尿病對小鼠AOM/DSS誘導結直腸癌發生的影響
田曠怡?林志玲?許稷豪?于濤?陳其奎?黎潔瑤

【摘要】目的 探討糖尿病(DM)對小鼠化學誘導結直腸癌發生的影響。方法 采用氧化偶氮甲烷/葡聚糖硫酸鈉誘導db/db小鼠和野生型(WT)小鼠產生結直腸癌,通過Illumina高通量測序技術篩選DM組與非DM組小鼠結腸腫瘤癌旁組織之間的差異表達基因,并對差異表達基因進行基因本體(GO)、京都基因與基因組百科全書(KEGG)功能注釋與富集分析。結果 與WT小鼠相比,DM小鼠結直腸的平均腫瘤體積增加(P < 0.05)。DM小鼠與WT小鼠共有1656個差異表達基因(q < 0.05),其中有70個mRNA在整個實驗過程中均差異表達,主要組織相容性復合體(MHC) Ⅱ類基因包括H2-Ea-ps、H2-Q1、H2-DMb2、H2-Q4均在DM小鼠中表達上調(q < 0.05)。GO富集分析發現免疫反應的調控和激活途徑、抗原加工與提呈途徑、抗原結合是顯著富集的類型(q < 0.05)。KEGG富集分析發現與免疫反應相關的抗原加工與呈遞是顯著富集的類型(q < 0.05)。結論 DM狀態下,小鼠免疫反應激活,MHC Ⅱ類基因表達存在顯著差異,可能參與促進結直腸癌的發生發展。
【關鍵詞】糖尿病;結直腸癌;小鼠模型;高通量測序
Transcriptomic high-throughput sequencing of AOM/DSS-induced colorectal carcinogenesis in mouse models with diabetes mellitus Tian Kuangyi, Lin Zhiling, Xu Jihao, Yu Tao, Chen Qikui, Li Jieyao. Department of Gastroenterology, Sun Yat-sen Memorial Hospital, Sun Yat-sen University, Guangzhou 510120, China
Corresponding author, Li Jieyao, E-mail: lijieyaoxh@ qq. com
【Abstract】Objective To investigate the effect of diabetes mellitus (DM) on the chemically-induced colorectal carcinogenesis in mouse modsels.? Methods Both db/db (DM) and wild-type (WT) mice were treated with AOM/DSS to induce colorectal carcinogenesis. The differentially expressed genes between the DM-CRC group and the WT-CRC group were identified using Illumina Hiseq sequencing systems. The functional enrichment analysis was mapped in gene ontology (GO) and Kyoto encyclopedia of genes and genomes (KEGG) pathway databases. Results Compared with the WT mice, DM mice presented with significantly enhanced average tumor volume (P < 0.05). A total of 1656 mRNAs were differentially expressed (all q < 0.05) between DM and WT mice, and 70 mRNAs were differentially expressed throughout the whole experiment. Major histocompatibility complex (MHC) classⅡ genes including H2-Ea-ps, H2-Q1, H2-DMb2 and H2-Q4 were all significantly up-regulated in DM mice (all q < 0.05). GO enrichment analysis demonstrated that? regulation and activation of immune response, antigen processing and presentation, and antigen binding were significantly enriched (all q < 0.05). The KEGG pathway enrichment analysis revealed that the DEGs were significantly associated with antigen processing and presentation (q < 0.05). Conclusion Immune response can be activated in DM mice.? The MHC classⅡ genes are significantly differentially expressed, which probably promotes the incidence and progression of colorectal cancer? under DM state.
【Key words】Diabetes mellitus;Colorectal cancer;Mouse model;High-throughput sequencing
中國約有1.164億人口患有糖尿病(DM),居世界首位[1]。近年來,大量研究認為DM與結直腸癌(CRC)之間有著密切的聯系,DM患者尤其是2型DM(T2DM)患者較非DM人群的CRC發病風險大幅升高[2-3]。癌旁組織常被用作腫瘤研究中的正常對照,也有研究表明,毗鄰腫瘤的癌旁組織在轉錄組和表觀遺傳學等方面亦有畸變,可被用于研究腫瘤發生發展及機制變化[4-5]。本課題組前期實驗對致癌劑氧化偶氮甲烷(AOM)合并促炎劑葡聚糖硫酸鈉(DSS)在小鼠模型上誘導CRC形成進行探索,并發現2循環的DSS飲水可得到成本/效益較佳的實驗結果;此外,我們發現與人類相似,T2DM小鼠的CRC成瘤效率高于非DM小鼠[6]。然而,DM與CRC之間的協同機制尚不完全清楚,深入探索DM并發CRC的機制有助于早期防治,改善患者預后,減輕社會負擔。本研究擬通過Illumina高通量測序技術檢測DM組及正常組小鼠的結腸癌旁組織中轉錄組的表達,篩選差異表達基因并進行差異表達基因的基因本體(GO)、京都基因與基因組百科全書(KEGG)功能注釋與富集分析,以期為DM狀態下AOM/DSS誘導的小鼠CRC發生發展的機制或潛在藥物治療靶點提供科學研究基礎。
材料與方法
一、主要試劑和動物
AOM(Sigma,美國),DSS(MP Biomedicals,美國)。C57BLKS/J-Leprem2Cd479/Nju(db/db)小鼠及其野生型對照C57BLKS/JNju(wild type,WT)小鼠,雄性,7周齡,各36只。小鼠均由南京集萃藥康生物科技公司(南京,中國)提供,常規飼養于中山大學東校園實驗動物中心,實驗室環境溫度為(23±3)℃,相對濕度為(55±15)%,晝夜明暗交替。所有有關動物的實驗過程均在中山大學實驗動物倫理委員會的監督指導下完成,符合實驗動物倫理學要求。
二、DM小鼠CRC模型的建立及處理
1.建模及分組
db/db小鼠隨機分為DM-CRC組及DM-Con組,WT小鼠隨機分為WT-CRC組及WT-Con組,每組各18只小鼠。所有小鼠經適應性飼養1周后開始實驗。CRC組均于第1周(W1)皮下注射10
mg/kg AOM,W2予2% DSS溶液自由飲用7 d,隨后予正常飲用水2周。每1周DSS與2周正常飲用水為1個循環周期,共循環2次。Con組均于W1皮下注射0.1 ml/10 g生理鹽水。各組分別于W7、W9、W11隨機處死6只小鼠。
2.標本采集及腫瘤計數
小鼠經頸椎脫臼法處死,沿腹中線打開腹腔,剪取自回盲部至直腸的腸段,使用預冷的磷酸鹽緩沖液(PBS)清洗腸道,沿長軸打開腸腔,仔細觀察并觸摸腸腔表面有無腫瘤形成。若發現腫瘤形成,觀察記錄腫瘤的數量、大小、位置,測量并記錄每個腫瘤的最長徑和垂直短徑。留取毗鄰腫瘤的癌旁組織保存于液氮中,以便后續實驗使用。
按下述公式計算腫瘤體積、腫瘤發生率、平均成瘤數量和荷瘤小鼠平均成瘤數量:腫瘤體積 =(最長徑×垂直短徑2) / 2;成瘤小鼠占比= 荷瘤小鼠數量/實驗小鼠數量;平均成瘤數量 = 腫瘤數量/實驗小鼠數量;荷瘤小鼠平均成瘤數量 = 腫瘤數量/荷瘤小鼠數量。
三、Illumina高通量測序
DM-CRC組及WT-CRC組分別于W7、W9、W11各隨機抽取3只小鼠。取小鼠結腸癌旁組織提取得到RNA,進行RNA質檢、完整性等分析合格后,進行文庫構建。去除rRNA、進行mRNA純化后將RNA片段化;一鏈、二鏈cDNA合成;3末端腺苷酸化、連接測序接頭;PCR擴增;Agilent 2100生物分析儀(Agilent Technogies,美國)定量cDNA文庫;DNA成簇擴增;于HiSeq2500測序儀(Illumina, 美國)進行高通量測序。
四、測序數據分析
測序得到的原始圖像文件經堿基識別、誤差過濾、除接頭序列、低質量片段等過濾方法,得到可用于數據分析的clean reads。應用Hisat2(version:2.0.4)的spliced mapping算法對clean reads進行轉錄組測序數據的基因組比對,估計基因表達的水平[7]。但由于reads數除與基因表達水平成正比外,還與基因本身的長度、測序的數據量有關。為了使不同基因、不同樣本間的基因表達水平具有可比性,應用Stringtie(version:1.3.0)對Hisat2比對后每個基因的片段數進行計數,再使用M值的加權截尾均值(TMM)法進行數據的歸一化,最后利用perl腳本計算出每個基因的每千個堿基的轉錄每百萬映射讀取的片段(FPKM)值,進行基因表達量的標準化[8-11]。
使用FPKM計算轉錄本表達量,并計算不同基因在不同組間的差異表達倍數。應用edgeR進行樣本間的差異基因分析,對P值進行多重假設檢驗校正,通過控制錯誤發現率以決定P值的閾值,得到校正后的P值,亦即q值[12-14]。根據q < 0.05篩選出不同組之間的差異表達基因。得到差異表達基因后,采用R語言、cluster、Cytoscape、David等軟件進行聚類分析,得到聚類分析熱圖、火山圖。
GO是基因本體聯合會建立的數據庫(http://geneontology.org),使用了規范化的術語描述基因和基因產物的特性。對細胞組分、分子功能和生物過程3個層次上對應的差異基因數目進行統計,并把所挑選的差異表達基因向GO數據庫的各個條目映射,計算每個條目的基因數目。應用超幾何分布進行假設檢驗,得到富集結果的P值,P值越低則富集結果越顯著,進而篩選出富集程度高的基因,從而對差異表達基因進行GO功能分析。同理,使用整合分子互動網絡的KEGG Pathway數據庫(https://www.genome.jp/kegg/pathway.html)對各個信號通路上的差異表達基因數目和富集情況進行KEGG Pathway分析。
五、統計學處理
采用SPSS 20.0分析。正態分布的定量資料用表示,非正態分布的定量資料用中位數(四分位數間距)表示;成瘤小鼠占比的比較使用Fisher確切概率法。2組非正態分布的定量資料的比較用Mann-Whitney U檢驗,多組非正態分布的定量資料的比較用Kruskal-Wallis法。α=0.05,兩兩比較用Bonferroni法,即P < 0.05 /比較次數為差異有統計學意義。
結果
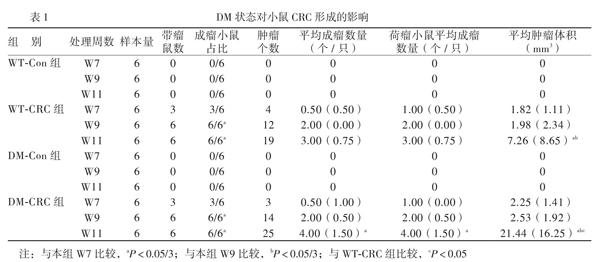
一、DM狀態對小鼠CRC形成的影響
WT-CRC組小鼠的成瘤小鼠占比均隨暴露時間的增加而增加(P < 0.05/3,表1);WT-Con組小鼠均未形成腫瘤。在W7和W11之間,DM-CRC
組小鼠的平均成瘤數量和荷瘤小鼠平均成瘤數量比較差異均有統計學意義(P < 0.05/3,表1);WT-CRC組差異則未見統計學意義(P > 0.05/3,表1)。WT-CRC組與DM-CRC組小鼠的平均腫瘤體積在W7和W11、W9和W11比較差異均有統計學意義(P < 0.05/3,表1)。此外,在W11,DM小鼠的平均腫瘤體積遠遠大于WT小鼠(P < 0.05,表1)。
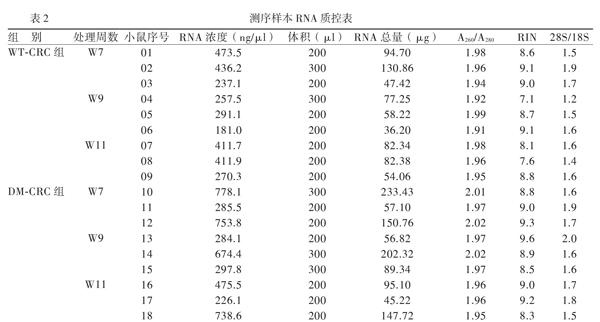
二、測序數據質控
各組小鼠結腸癌旁組織的RNA電泳條帶明亮清晰,泳道無彌散區,未見蛋白和DNA污染(圖1A);RNA完整值(RIN)接近10,28S/18S≥1.5(表2);檢測峰圖基線較平整(圖1B)。提示樣本質量和純度較好,質檢結果合格,進行Illumina高通量測序。
三、差異表達基因篩選與聚類
根據q≤0.05篩選差異表達基因,進行聚類分析,并繪制熱圖及火山圖。可見組內表達相似的基因聚類后出現在同一簇中,DM-CRC組與WT-CRC組共有1656個差異基因(圖2)。DM-CRC組與WT-CRC組相比,在W7有210個mRNA差異表達,其中112個上調,98個下調(圖3A);在W9有604個mRNA差異表達,其中136個上調,468個下調(圖3B);在W11有1237個mRNA差異表達,其中522個上調,715個下調(圖3C)。綜合上述所有差異表達基因,共有70個mRNA在整個實驗過程中差異表達(圖3D),其中43個上調,包括MHCⅡ類基因:H2-Ea-ps(qW7 = 2.50×10-59,
qW9 = 3.31×10-38,qW11 = 3.53×10-59)、H2-Q1(qW7 = 5.68×10-11,qW9 = 2.91×10-14,q W11 = 2.00×10-6)、H2-DMb2(qW7 = 1.01×10-2,qW9= 3.12×10-2,qW11 = 1.65×10-6)、 H2-Q4(qW7 =6.17×10-5,qW9 = 4.78× 10-3,qW11 = 3.26×10-5);另有24個下調;3個有波動,在部分時間點上調,部分時間點下調。
四、GO功能顯著性富集分析
對1656個差異表達基因的GO功能進行注釋、分類并統計(圖4)。結果顯示,在生物過程分組,免疫反應的調控和激活途徑、肽或多糖通過MHC Ⅱ類分子的抗原加工與提呈途徑、包括多肽抗原在內的抗原加工與提呈途徑是顯著富集的類型(q < 0.05);在分子功能分組,MHC Ⅰ類蛋白復合物和血漿膜蛋白復合物是顯著富集的類型(q < 0.05);在細胞組分分組,包括多肽在內的抗原結合、絲氨酸類型的肽酶、內肽酶及水解酶活性、酰胺類物質的結合是顯著富集的類型(q < 0.05)。
五、KEGG Pathway顯著富集分析
結合1656個差異表達基因的KEGG功能注釋結果進行KEGG Pathway顯著性富集分析,并選取富集度最高的前30個條目進行分類統計。結果顯示與免疫反應相關的抗原加工與呈遞、移植排斥反應、移植物抗宿主病、自身免疫性甲狀腺疾病等方向顯著富集(q < 0.05,圖5)。
討論
在T2DM患者中,約有10%的患者合并CRC,且初次診斷時大多處于中晚期,患者預后不夠理想[15-16]。因此,我們需要更多的研究來尋找DM狀態下CRC發生發展前期的分子機制變化。AOM可通過誘導基因突變損傷,進而促進嚙齒類動物CRC的形成;在其基礎上加用DSS,可誘導腸上皮的慢性炎癥狀態,加快建模效率[17]。db/db小鼠先天具有高血糖和高脂血癥,是一種常用的T2DM小鼠模型[18]。參考本課題組的前期實驗,我們本次使用AOM聯合2循環DSS,誘導DM小鼠及WT小鼠形成CRC[6]。通過對小鼠成瘤情況的評估,發現在W11,DM小鼠的平均腫瘤體積遠遠大于WT小鼠,提示DM狀態可促進小鼠CRC的發生,與人類的DM與CRC之間的協同機制相一致,對該模型進一步的研究或可為DM合并CRC的機制及潛在的藥物治療靶點提供參考依據。
癌旁組織在臨床實驗中常被用作腫瘤的正常對照,然而近年來已有多項研究發現,盡管毗鄰腫瘤的癌旁組織在大體上未見異常,病理觀察下可能已發生組織異常增生,并在分子水平較無腫瘤的正常組織發生改變,可用于研究腫瘤發生發展早期的分子變化[4-5, 19-21]。轉錄組測序是指利用以Illumina高通量測序為代表的二代測序技術,對組織或者細胞中所有mRNA反轉錄生成的cDNA序列進行測序,全面快速獲得細胞或組織在特定時期內的幾乎所有轉錄本序列信息,進而篩選差異表達基因,并對基因進行預測,為在分子水平尋找可能的生物標志物提供了極大的便利[22]。本實驗通過獲取成瘤小鼠毗鄰腫瘤的癌旁組織,利用Illumina高通量測序分析,探討DM對小鼠CRC發生的潛在病理機制。
我們通過實驗得到差異表達mRNA共1656條,并發現70個mRNA在整個實驗過程中均差異表達,其中MHC Ⅱ類基因包括H2-Ea-ps、H2-Q1、H2-DMb2、H2-Q4均在DM小鼠中表達顯著上調。在GO生物過程分組上,免疫反應的調控和激活途徑、肽或多糖通過MHC Ⅱ類分子的抗原加工與提呈途徑、包括多肽抗原在內的抗原加工與提呈途徑是顯著富集的類型;在分子功能分組上,MHC Ⅰ類蛋白復合物是顯著富集的類型;在細胞組分分組上,包括多肽在內的抗原結合是顯著富集的類型。在KEGG Pathway顯著性富集分析中,與免疫反應相關的抗原加工與呈遞顯著富集。
MHC Ⅱ類分子是免疫反應的關鍵介質之一,起著向CD4+ T淋巴細胞提呈經過加工的外源性抗原和誘發免疫反應的作用[23]。這些基因均具有高度的多態性,可影響功能性氨基酸的變化,并導致個體對抗原反應的差異[24]。結直腸由于持續暴露于外源性抗原,如食物、化學物質、細菌等,導致該處相對恒定的炎癥狀態,與其它器官相比,在MHC Ⅱ類基因表達方面具有顯著差異[25]。此外,有學者發現,MHC Ⅱ類等位基因的高表達可提示結腸炎患者患CRC的風險增高[26]。
在對DM易感的個體,其先天性免疫系統被激活,炎癥因子分泌增加,進而引起胰島素抵抗、胰島素分泌功能障礙和代謝綜合征的發生[27]。而DM患者在持續高血糖的狀態下,可損傷線粒體功能,增加活性氧產生,激活炎癥介質與機體的免疫反應[28]。上述研究提示免疫炎癥反應在DM和CRC的發病中均起著重要的作用。綜合本次實驗結果,我們認為,DM狀態下CRC的發病風險增高,可能與生物體以MHC Ⅱ類基因為主的免疫反應激活有關;H2-Ea-ps、H2-Q1、H2-DMb2、H2-Q4這4個基因有望成為DM患者CRC診治的靶點。
綜上所述,我們探討了AOM/DSS誘導小鼠產生CRC,發現DM可促進CRC的形成,顯著增加小鼠的平均腫瘤體積;比較DM與WT狀態下癌旁組織的差異表達基因,并對結果進行GO和KEGG分析,發現4個高表達的MHC Ⅱ類基因:H2-Ea-ps、H2-Q1、H2-DMb2、H2-Q4,為DM合并CRC的發病機制提供可靠的生物學標志物,并對其早期診治提供可能的新靶點。
參 考 文 獻
[1] International Diabetes Federation,IDF Diabetes Atlas, 9th edn. Brussels, Belgium: 2019.[2019-05-06] http://www.diabetesatlas.org.
[2] Tsilidis KK, Kasimis JC, Lopez DS, Ntzani EE, Ioannidis JP. Type 2 diabetes and cancer: umbrella review of meta-analyses of observational studies. BMJ,2015,350:g7607.
[3] Pearson-Stuttard J, Zhou B, Kontis V, Bentham J, Gunter MJ, Ezzati M. Worldwide burden of cancer attributable to diabetes and high body-mass index: a comparative risk assessment. Lancet Diabetes Endocrinol,2018,6(2):95-104.
[4] Heaphy CM, Griffith JK, Bisoffi M. Mammary field cance-rization: molecular evidence and clinical importance. Breast Cancer Res Treat,2009,118(2):229-239.
[5] Aran D, Camarda R, Odegaard J, Paik H, Oskotsky B, Krings G, Goga A, Sirota M, Butte AJ. Comprehensive analysis of normal adjacent to tumor transcriptomes. Nat Commun,2017,8(1):1077.
[6] 曾一文,黎潔瑤,田曠怡,于濤,許稷豪,陳其奎.通過AOM及AOM聯合DSS建立C57BL/6J小鼠結腸癌誘導模型的對比研究.新醫學,2018,49(10):720-725.
[7] Kim D, Langmead B, Salzberg SL. HISAT: a fast spliced aligner with low memory requirements. Nat Methods,2015,12(4):357-360.
[8] Pertea M, Pertea GM, Antonescu CM, Chang TC, Mendell JT, Salzberg SL. String Tie enables improved reconstruction of a transcriptome from RNA-seq reads. Nat Biotechnol, 2015,33(3):290-295.
[9] Pertea M, Kim D, Pertea GM, Leek JT, Salzberg SL. Transcript-level expression analysis of RNA-seq experiments with HISAT, String Tie and Ballgown. Nat Protoc,2016,11(9):1650-1667.
[10] Robinson MD, Oshlack A. A scaling normalization method for differential expression analysis of RNA-seq data. Genome Biol,2010,11(3):R25.
[11] Mortazavi A, Williams BA, McCue K, Schaeffer L, Wold B.Mapping and quantifying mammalian transcriptomes by RNA-Seq. Nat Methods,2008,5(7):621-628.
[12] Robinson MD, McCarthy DJ, Smyth GK. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data. Bioinformatics,2010,26(1):139-140.
[13] Benjamini Y, Hochberg Y. Controlling the false discovery rate: a practical and powerful approach to multiple testing. Royal Statistical Society, 1995,57(1):289-300.
[14] Benjamini Y , Yekutieli D . The control of the false discovery rate in multiple testing under dependency. The Annals of Stats, 2001, 29(4):1165-1188.
[15] 韓倩,楊巖,劉春水,王潤超,侯俊杰,白歐. 2型糖尿病伴發惡性腫瘤664例臨床分析.中國實驗診斷學,2012,16(1):121-123.
[16] 盧瑜, 方勇, 王青青,潘宏銘. 糖尿病與惡性腫瘤關系的回顧性臨床研究. 中華內分泌代謝雜志, 2010, 26(3):183-187.
[17] Neufert C, Becker C, Neurath MF. An inducible mouse model of colon carcinogenesis for the analysis of sporadic and inflam-mation-driven tumor progression. Nat Protoc,2007,2(8):1998-2004.
[18] Neelankal John A, Ram R, Jiang FX. RNA-seq analysis of islets to characterise the dedifferentiation in type 2 diabetes model mice db/db. Endocr Pathol,2018,29(3):207-221.
[19] Heaphy CM, Bisoffi M, Fordyce CA, Haaland CM, Hines WC, Joste NE, Griffith JK. Telomere DNA content and allelic imbalance demonstrate field cancerization in histologically normal tissue adjacent to breast tumors. Int J Cancer,2006,119(1):108-116.
[20] Trujillo KA, Heaphy CM, Mai M, Vargas KM, Jones AC, Vo P, Butler KS, Joste NE, Bisoffi M, Griffith JK. Markers of fibrosis and epithelial to mesenchymal transition demonstrate field cancerization in histologically normal tissue adjacent to breast tumors. Int J Cancer,2011,129(6):1310-1321.
[21] Gerweck LE, Seetharaman K. Cellular pH gradient in tumor versus normal tissue: potential exploitation for the treatment of cancer. Cancer Res,1996,56(6):1194-1198.
[22] 祁云霞,劉永斌,榮威恒.轉錄組研究新技術:RNA-Seq及其應用.遺傳,2011,33(11):1191-1202.
[23] Stokkers PC, Reitsma PH, Tytgat GN, van Deventer SJ. HLA-DR and -DQ phenotypes in inflammatory bowel disease: a meta-analysis. Gut,1999,45(3):395-401.
[24] Howell WM, Calder PC, Grimble RF. Gene polymorphisms, inflammatory diseases and cancer. Proc Nutr Soc,2002,61(4):447-456.
[25] Ferguson LR, Shelling AN, Browning BL, Huebner C, Peter-mann I. Genes, diet and inflammatory bowel disease. Mutat Res,2007,622(1-2):70-83.
[26] Garrity-Park MM, Loftus EV Jr, Sandborn WJ, Bryant SC, Smyrk TC. MHC Class Ⅱ alleles in ulcerative colitis-associated colorectal cancer. Gut,2009,58(9):1226-1233.
[27] 朱大龍.炎癥與2型糖尿病.中國糖尿病雜志,2006,14(1):73-74.
[28] 黃力,葛永純.免疫炎癥與糖尿病腎病.腎臟病與透析腎移植雜志,2019,28(3):247-251.
(收稿日期:2019-10-18)
(本文編輯:楊江瑜)

