澳洲堅果實時熒光定量PCR分析中內參基因的篩選
楊倩 楊子平 周婭麗 陳東泉 劉恒
摘? 要:澳洲堅果是重要的熱區經濟作物,目前國內外尚無關于澳洲堅果實時熒光定量PCR分析內參基因的報道。選擇合適的內參基因是提高實時熒光定量PCR分析準確性的先決條件。為篩選澳洲堅果實時定量PCR最適內參基因,以澳洲堅果的根、莖、葉、果皮、果仁為材料,利用實時熒光定量PCR技術,對18S rRNA,Actin,CYP,EF1a,EF1b,GAPDH,MDH,TUBa,TUBb,UBQ,UBC等11個常用的內參基因在澳洲堅果不同組織中的表達穩定性進行了分析。geNorm軟件分析的最適內參基因數目為2,最穩定內參組合為MDH和EF1b,TUBa基因的穩定性最差。BestKeeper分析結果認為,MDH基因表達最穩定,EF1b次之,與geNorm結果一致。NormFinder軟件穩定性分析顯示,GAPDH最穩定,其次是CYP基因;TUBa基因表達最不穩定。ΔCt算法結果表明,18S基因表達最穩定,其次是GAPDH基因,MDH和EF1b排第3和第4。RefFinder綜合排序為:MDH>18S>GADPH>EF1b>CYP>UBC>EF1a>Actin>TUBb>UBQ> TUBa。因此,MDH基因在澳洲堅果不同組織中表達最穩定,初步確認可以作為實時熒光定量PCR分析的校正內參基因,在澳洲堅果的基因表達模式分析中具有重要意義。
關鍵詞:澳洲堅果;內參基因;實時熒光定量PCR;穩定性分析
中圖分類號:S664.9? ? ? 文獻標識碼:A
Abstract: Macadamia nut is an important economics crop in subtropical areas of China. The selection of a suitable reference gene is an important prerequisite for successful gene expression analysis by real-time fluorescence quantitative PCR (RT-qPCR). In order to select the appropriate reference genes, we investigated the expression stability of 11 candidate genes (18S rRNA, Actin, CYP, EF1a, EF1b, GAPDH, MDH, TUBa, TUBb, UBQ, UBC) in RT-qPCR experiments in different tissues, including kernel, peel, roots, stem, leaf from Macadamia with geNorm, NormFinder, BestKeeper, ΔCt, RefFinder program software packages. As determined by geNorm, MDH/EF1b were the most stable reference genes, TUBa was the least stable gene. BestKeeper revealed that MDH was the most stables reference gene, and EF1b ranked the second. The rank in BestKeeper was similarly with that in geNorm. The result by NormFinder showed GAPDH was the most stable gene, CYP ranks the second, the least stable gene was TUBa. ΔCt algorithm demonstrated that18S was the most stable gene, GAPDH ranks the second, MDH and EF1b ranked the third and fourth. To obtain a consensus result of the most stable reference genes according to the RefFinder approach, the geometric mean of the four algorithms corresponding rankings for each candidate gene were calculated: MDH>18S>GADPH>EF1b>CYP>UBC>EF1a>Actin>TUBb>UBQ>TUBa. The result showed that MDH was the most suitable reference gene for macadamia in different tissues.
Keywords: Macadamia integrifolia; reference genes; real-time fluorescence quantitative PCR; stability analysis
DOI: 10.3969/j.issn.1000-2561.2020.08.001
澳洲堅果(Macadamia integrifolia),又稱夏威夷果、澳洲胡桃、昆士蘭栗,屬山龍眼科(Pro teaceae)澳洲堅果屬(Macadamia F. Muell)常綠喬木果樹,原產于澳大利亞昆士蘭州東南部和新南威爾士東北部沿岸的亞熱帶雨林地區[1]。澳洲堅果果實營養豐富,風味獨特,被譽為“堅果之王”,具有一定的經濟價值[2-3]。近年來,我國云南、廣西、廣東、貴州等南方各省(區)大面積推廣引種試種澳洲堅果優良品種,種植面積達16萬hm2,占世界種植面積的61.05%(數據來源:中國農業農村部南亞熱帶作物辦公室)。雖然我國澳洲堅果的果仁產量逐漸遞增,但是全球市場的需求量更大,因此澳洲堅果產業具有廣闊的發展前景[4]。
實時熒光定量PCR技術(real-time fluorescent quantitative polymerase chain reaction, qRT-PCR)的原理是在PCR反應體系中加入熒光化學物質,熒光物質能夠與PCR產物反應,隨著PCR產物不斷增加,熒光信號也成比例增強,所以PCR每個循環結束時的熒光值代表了PCR產物量的變化。利用qRT-PCR對目的基因的相對表達量進行計算時,需要以管家基因的表達量作為標準進行校正和均一化。理想狀態下,所選內參基因在不同的處理條件下、不同組織器官中、細胞的不同發育時期中都能穩定表達,而且其表達水平與目的基因表達水平相近[5]。然而,不斷有研究表明,實際實驗過程中并不存在任何一種內參基因能夠在任何條件、任何細胞類型和組織都能夠穩定表達[6],因此直接使用未經篩選的內參基因,會導致實驗數據出現偏差,影響目的基因表達水平結果的可靠性,所以有必要對內參基因進行篩選。
近年來,隨著分子生物學研究手段逐漸被應用到各個研究領域,分子生物學研究中的基因表達和調控機制研究成為熱點。基因的表達分析需要內參基因進行歸一化,而目前關于澳洲堅果內參基因的研究還未見報道。Actin是常見的內參基因,是微絲的結構成分,也是細胞骨架的主要成分。在魚的研究中發現,環境中的激素刺激會影響Actin基因的表達穩定性[7]。18S rRNA是真核生物體內含量最多的核糖體RNA,常被用來做內參基因,但是在楊樹的不同發育時期,18S rRNA的表達穩定性最差,并不適合作為內參基因[8]。GAPDH是糖酵解、糖異生及光合作用碳固定循環過程中的關鍵酶。但是在豬的不同組織中的表達水平和穩定性表達變化都很大,是最不穩定的內參基因[9]。蘋果酸脫氫酶(malate dehydrogenase, MDH)是一類廣泛存在動植物中的酶,參與植物體的多個代謝途徑,研究證實細胞質蘋果酸脫氫酶基因也可作內參基因[10]。延伸因子(elongation factor, eEF)是一類多功能調控蛋白,可以催化氨基酸鏈在核糖體上的延伸,從而調控相關蛋白的合成[11-13]。在植物中,真核生物延伸因子1(eEF1)家族包含eEF1A和eEF1B蛋白[14-16],延伸因子(elongation factor, eEF)家族基因常被選作內參基因[14,17]。因此,本研究以澳洲堅果根、莖、葉、果皮、果仁為材料,對18S rRNA,Actin,親環蛋白(CYP),EF1a和EF1b,GADPH,MDH,微管蛋白(TUBa和TUBb),多聚泛素酶(UBQ),泛素連接蛋白(UBC)等11個常用內參基因分別用geNorm[18]、NormFinder[19]、BestKeeper[20]和ΔCt法等不同算法進行表達穩定性分析,最后根據RefFinder[21]綜合分析篩選最適合的內參基因。本實驗目的是篩選出澳洲堅果不同組織器官中表達穩定的內參基因,以期為澳洲堅果的基因表達調控相關研究提供合適的內參基因。
1? 材料與方法
1.1? 植物材料
選取‘南亞1號澳洲堅果品種作為實驗材料,分別摘取成熟的果實、幼嫩葉片、枝條和根,清洗干凈后用液氮速凍,80 ℃保存備用。
1.2? 內參基因的選擇和引物設計
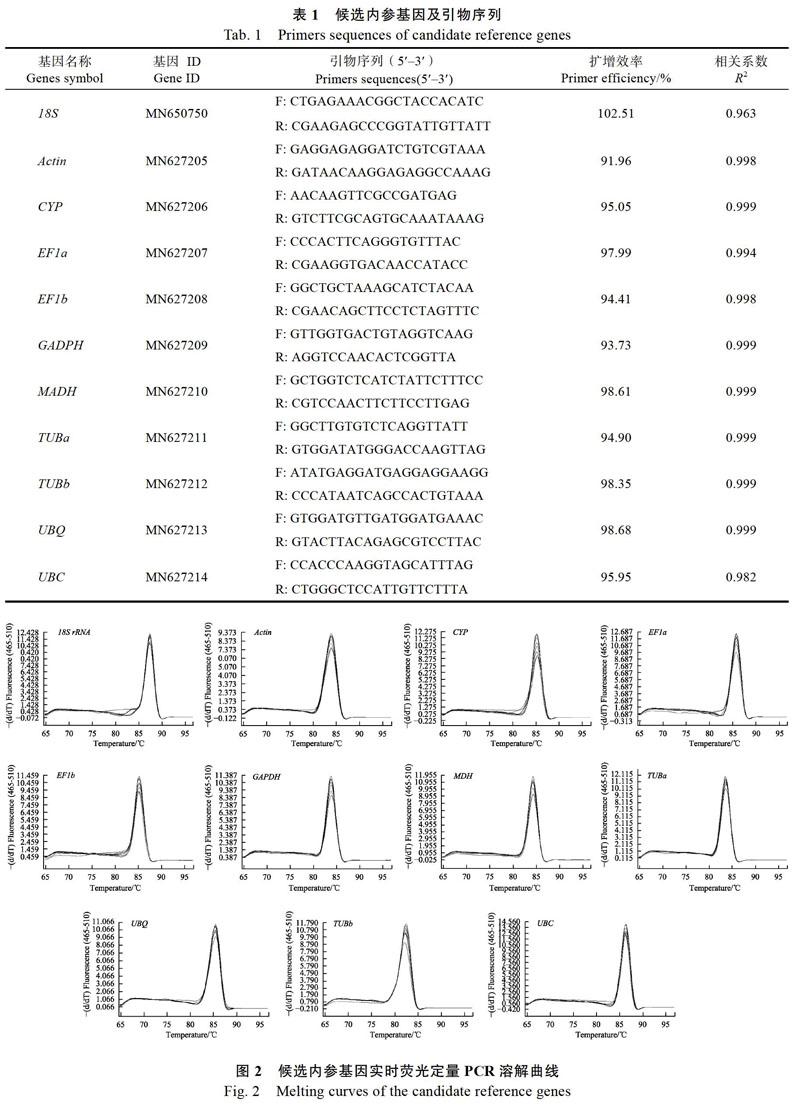
在植物實時熒光定量PCR研究中多采用肌動蛋白(Actin)、18S核糖體RNA(18S rRNA),甘油醛-3-磷酸脫氫酶(GADPH)等常用看家基因作為內參基因進行定量。本研究擬選擇11個常用內參基因(18S rRNA, Actin, CYP, EF1a, EF1b, GAPDH, MDH, TUBa, TUBb, UBC, UBQ)作為候選內參基因。這11個候選內參基因cDNA序列部分已經從澳洲堅果的轉錄組數據獲得。登錄實時定量引物設計網站,在候選內參基因的保守區域,使用在線軟件設計特異性熒光定量引物。網站地址:https://sg.idtdna.com/pages/products/qpcr-and- pcr/custom-primers,每個基因設計5對引物。引物由北京六合華大基因科技有限公司合成。
1.3? 不同組織RNA的提取和cDNA的合成
澳洲堅果葉片總RNA的提取按照Plant Total RNA Isolation Kit Plus(FOREGENE Biotech, Chengdu, China)方法進行。mRNA使用凝膠電泳檢測,Nanodrop測定濃度。反轉錄按照NOVA All-in-one First-Strand Synthesis Master Mix(Yugong Biolabs, Jiangsu, China)操作。反應體系20 μL,以1000 ng RNA為模板,反轉錄獲得單鏈cDNA。反轉錄程序為:55 ℃ 60 min;85 ℃ 1 min。
1.4? 標準曲線的制備
實時熒光定量PCR數據采集在羅氏(Roche)Light Cycler 480Ⅱ上完成,熒光試劑盒選用Taq SYBR Green qPCR Premix ROXⅠ(NOVA)。將根、莖、葉、果皮、果仁等5個不同組織的樣品等體積混合,5倍梯度稀釋,分別以50 、51 、52、53、54 、55作為模板制備標準曲線,每個樣品重復3次。3次生物學重復。
RT-PCR擴增體系:SYBR Premix ExTaqⅡ(2),10 L;PCR Forward Primer(10 mol/L),1 L;PCR Reverse Primer(10 mol/L),1 L;cDNA,1 L;dH2O,7 L;總體積20 L。標準曲線和斜率(slope)由稀釋倍數和熒光定量Cp值在Excel中計算得出。擴增效率計算公式:E=(5[1/slope]1)100%。
PCR擴增程序:94 ℃預變性3 min;然后94 ℃變性10 s,58 ℃退火30 s,72 ℃延伸10 s,45個循環;在每個循環結束后進行熒光信號的采集,所有循環結束后進行溶解曲線分析(65~95 ℃)。
1.5? 數據分析
實時熒光定量PCR數值可利用Light Cycler 480Ⅱ(Roche)的分析軟件直接獲得Cp值。分別采用geNorm、NormFinder、BestKeeper、ΔCt算法4個不同的程序,比較分析澳洲堅果中候選內參基因在不同組織中的表達穩定性,最后通過RefFinder計算出較為一致的穩定內參基因。
2? 結果與分析
2.1? 澳洲堅果RNA的分離與純化和內參引物的篩選
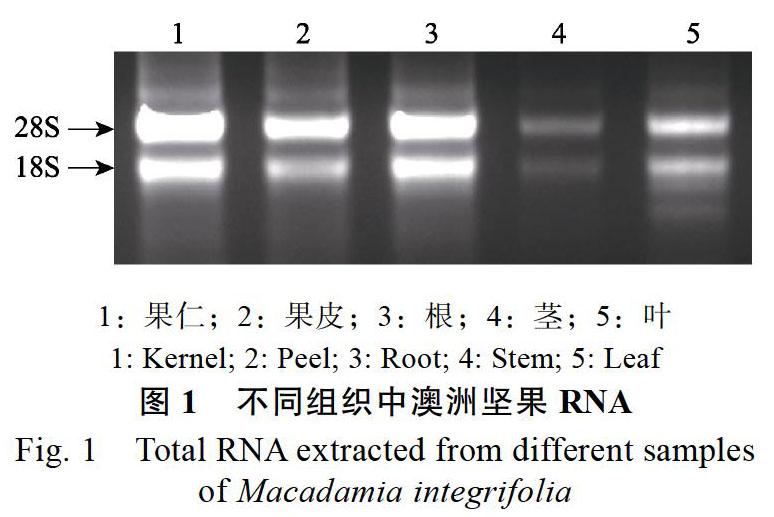
1.2%瓊脂糖凝膠電泳檢測提取RNA結果顯示,不同器官組織的RNA電泳條帶清晰,28S rRNA帶亮度約為18S rRNA的1.5~2倍(圖1),無拖尾,表明RNA的完整性良好,純度較高,可用于后續實驗。根據溶解曲線和熒光定量PCR的Cp值對每個候選內參基因的5對引物進行篩選,選取質量最好的一對常用內參基因引物(表1)用于后續實驗。
2.2? 內參基因的實時熒光定量PCR標準曲線和熔解曲線
將澳洲堅果根、莖、葉、果皮、果仁等樣品cDNA等量混合,依次連續5倍稀釋為50,51,52,53,54,55共6個梯度,隨后以6個不同濃度的混合樣品為模板分別對11個候選內參基因進行qRT-PCR。在Excel數據處理軟件中,以實時熒光定量PCR反應獲得的Cp值為縱坐標,5的稀釋倍數為橫坐標,自動生成標準曲線。結果顯示:除18S(R2=0.963)外,其余候選內參基因標準曲線的相關系數(R2)都在0.99附近(表1),擴增效率為91.9%~102.5%,說明引物、模板、實時熒光定量PCR反應體系和程序滿足實時熒光定量PCR分析的要求(90%~105%)。
實時熒光定量PCR產物具有熒光信號,隨著溫度升高雙鏈DNA降解,這個過程的熒光信號被采集生成熔解曲線,可以用來確定PCR的反應產物。熔解曲線分析通常是在qRT-PCR擴增程序的所有循環結束后,設置一個由65 ℃上升至95 ℃的反應程序,每升溫0.5 ℃測定1次熒光信號,共采集60次。熒光定量熔解曲線數據顯示,11個候選內參基因的都為單一銳鋒,不存在非特異擴增,Tm值為80~85 ℃(圖2),說明篩選的這些候選內參基因的引物特異性強,符合RT-qPCR分析的要求。
2.3? 不同組織內參基因的篩選
以澳洲堅果根、莖、葉、果仁、果皮不同組織的cDNA為模板,對11個候選內參基因進行實時熒光定量PCR分析。
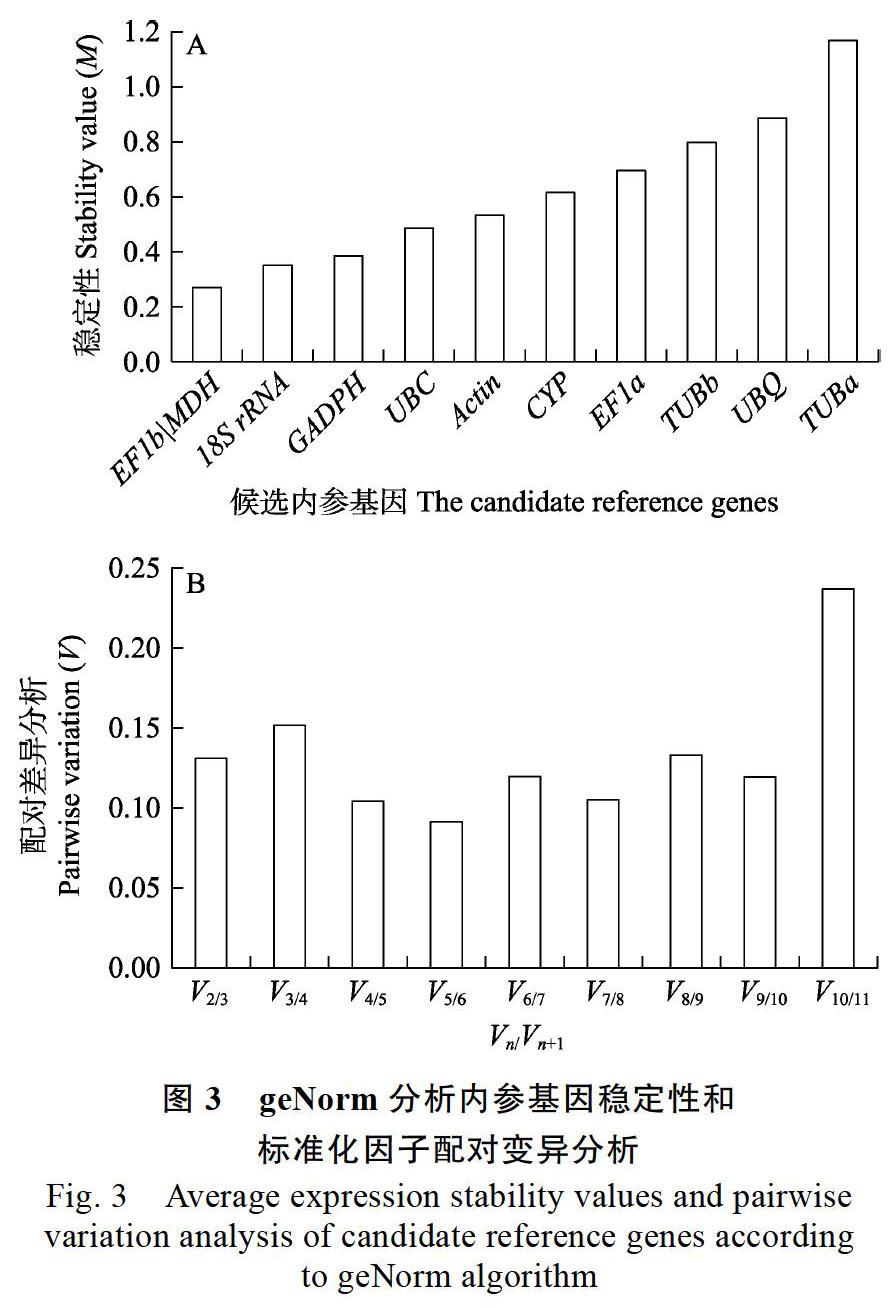
2.3.1? geNorm分析澳洲堅果不同組織中候選內參基因的穩定性? geNorm軟件算法利用M值和標準化因子的配對變異V值確定候選內參基因的穩定性和適合內參的數量。M值的閾值為1.5,小于1.5說明該基因適合做內參,反之則不適合做內參;且M值越小則內參基因的表達就越穩定。由圖3可見,本實驗中11個候選內參基因的M值均小于1.5,說明所有基因的表達都比較穩定。其中EF1b和MDH的M值最低(圖3A),其他基因的表達穩定性依次為18S>GAPDH>UBC> Actin>CYP>EF1a>TUBb>UBQ>TUBa,TUBa的M值最高。因此,geNorm軟件分析認為EF1b、MDH的穩定性最好,TUBa的穩定性最差。geNorm程序還可計算引入1個新基因后標準化因子的配對變異V值,并根據Vn/Vn+1值來確定所需最適內參基因的數目。實驗數據顯示,在11個檢測的候選內參基因中,V2/3的比值為0.131,小于推薦值0.15,說明本次實驗供試的澳洲堅果不同組織器官中,最適合的內參基因數目是2(圖3B)。因此,根據geNorm分析認為,EF1b和MDH為最穩定內參基因。
2.3.2? ΔCt算法分析澳洲堅果不同組織內參基因的穩定性? ΔCt算法根據計算的標準偏差數值(STDEV)確定基因表達的穩定性[22],標準偏差越小則基因越穩定。使用ΔCt分析結果顯示(表2),18S為最穩定內參基因,其次為GAPDH基因,MDH和EF1b基因排在中間,TUBa為最不穩定基因。
2.3.3? NormFinder分析澳洲堅果不同組織中候選內參基因的穩定性? NormFinder程序的穩定性分析同geNorm分析一樣,也是基于候選內參基因的相對定量表達數據,穩定性數值越小,基因表達越穩定[23]。NormFinder算法不但可以估計候選內參基因的整體表達差異,還可以計算樣品組間的變異,但是只能篩選出一個最合適的內參基因[24]。本研究使用NormFinder程序分析候選內參基因的表達穩定性排序見表2。分析認為,GAPDH為最穩定基因,其次是CYP、EF1a和18S,然后才是MDH和EF1b,TUBa為最不穩定基因。
2.3.4? BestKeeper分析澳洲堅果不同組織中內參基因的穩定性? BestKeeper程序是以每個樣品的原始Cp值進行配對相關分析。計算Cp值得到標準差(SD)、變異系數(CV)、配對相關系數(correlation coefficient),如果SD和CV越小,該內參基因越穩定,反之穩定性越差。一般認為,候選內參基因SD小于1的被認為是最穩定表達,由表3可見,除TUBa(2.38)和UBQ(1.108)基因外,本研究中其他的9個候選內參基因SD值都小于1,說明這些基因的表達都比較穩定。MDH和EF1b的SD分別為0.361和0.411,這個結果與geNorm的分析結果一致。
2.3.5? RefFinder綜合排序分析? 由于各個算法原理不同,得到最穩定基因的結果差異較大。本實驗使用RefFinder程序對上述4種算法的排序結果進行綜合分析[21],數值越小,則基因越穩定;反之穩定性越差。結果見表3,MDH表達最穩定,其次為18S rRNA,TUBa基因的穩定性最差。
2.4? 澳洲堅果SAD基因的表達分析和內參基因的驗證
植物硬脂酰-酰基載體蛋白脫飽和酶(SAD)是一種質體中存在的催化硬脂酸烴鏈脫飽和合成油酸的可溶性酶,是植物中形成不飽和脂肪酸的前提。已有研究表明,澳洲堅果SAD能以18∶0-ACP和16∶0-ACP為底物催化生成單不飽和脂肪酸——油酸和棕櫚油酸,并且對底物18∶0- ACP的酶反應速率高于底物16∶0-ACP[25]。為了分析SAD基因在澳洲堅果不同組織中的表達情況,分別用MDH和18S rRNA(最穩定內參)、TUBa基因(最不穩定內參)作為參照基因歸一化定量。結果顯示,以MDH和18S rRNA為參照時,基因在各個組中的表達量呈下降的趨勢;以TUBa基因作為參照時莖和葉片中基因表達上調,果皮和果仁中基因上調表達,而UBQ則相反;因此,如果采用UBQ和TUBa作為內參基因,那么則會得到完全相反的定量結果(圖4)。結果表明,選擇合適的內參基因直接影響基因的表達準確性,在本實驗中,MDH基因最適合作為澳洲堅果不同組織中進行實時熒光定量PCR分析的內參基因。
3? 討論與結論
使用實時熒光定量PCR技術分析基因的表達情況是理解生物學調控機制研究中常用的重要手段。但實際情況是,常用內參基因的表達在不同組織器官中、不同處理條件下、不同類型細胞中和不同發育階段中并不是恒定表達,也會隨著條件改變[26]。因此,為了獲得更為準確的定量結果,通常會選擇一個或多個持家基因作為內參基因對目的基因的表達量進行校準和標準化,故選擇合適的內參基因是獲得準確定量結果的關鍵[27]。
在本研究中,選擇了傳統的11個常用看家基因,包括18S rRNA,Actin,CYP,EF1a,EF1b,GAPDH,MDH,TUBa,TUBb,UBQ,UBC等基因,并利用geNorm、NormFinder、BestKeeper和ΔCt算法分析這些候選內參基因在不同組織器官中的表達穩定性。由于各個程序的計算方法原理不同,各軟件得出的穩定性結果基本一致,但仍舊有些差異。geNorm分析程序根據各個候選內參基因在每個樣品中表達情況的相似度進行排序,不考慮其他內參基因,故不適于區分表達模式類似的基因。而NormFinder是以組內方差與組間方差進行排序,綜合考慮其他基因之后,對候選內參基因進行打分。本試驗對4種算法數據比較發現:geNorm和BestKeeper軟件得到的內參基因的穩定性排序基本一致,MDH和EF1b表達最穩定,TUBa表達最不穩定。而NormFinder的分析認為:GAPDH的表達最穩定,TUBa表達最不穩定,MDH和EF1b的穩定性排第5和第6,與geNorm和BestKeeper分析結果差異較大。ΔCt算法分析結果表明,18S rRNA基因表達最穩定,其次是GAPDH,MDH和EF1b的穩定性排第3和第4。最后,我們通過幾何平均算法對上述4種結果綜合排序:MDH>18S rRNA>GADPH> EF1b>CYP>UBC>EF1a>Actin>TUBb>UBQ>TUBa。因此,在本研究中,可以初步選擇MDH作為澳洲堅果實根、莖、葉、果皮、果仁不同組織實時熒光定量PCR分析較為穩定的內參基因。
魏永贊等[28]認為,β-Actin在荔枝果實發育不同階段和外源生長調節劑處理下表達穩定性較好,但是,在114份香蕉品種中比較發現Actin表達最不穩定,在本實驗中,Actin基因的表達沒有MDH基因穩定。蘋果酸脫氫酶(malate dehydrogenase, MDH)是一類廣泛存在動植物中的酶,參與植物體的多個代謝途徑,根據輔酶專一性、亞細胞定位和生理功能。在小麥的研究中發現:TaMDH基因在不同物種間高度保守,推測cyMDH可能由一個共同的祖先進化而來,并且可能屬于管家基因[29-30]。在黃花大苞姜花粉母細胞不同的發育時期進行qRT-PCR研究發現:MDH的表達最穩定,可以作為內參基因[31]。本研究結果表明:MDH基因在澳洲堅果不同組織中的表達穩定性最好,適合做內參基因對澳洲堅果不同組織中的基因進行歸一化分析。
MiSAD基因是澳洲堅果的合成和積累不飽和脂肪酸的關鍵基因,本研究分別以最穩定基因MDH和最不穩定基因TUBa分別作為內參對MiSAD基因進行歸一化分析。研究結果表明:MDH和18S rRNA為參照時,基因在各個組中的表達量呈下降的趨勢;以TUBa基因作為參照時,在莖葉中呈上調表達,果皮和果仁中則表達量降低,而UBQ則相反;如果采用UBQ和TUBa作為內參基因,那么則會得到完全相反的定量結果。因此,如果在進行基因表達分析前,沒有對候選內參基因進行表達穩定性分析,盲目使用內參基因,會導致試驗結果不準確。由于不同的環境條件,如生物脅迫和非生物脅迫都會影響內參基因表達的穩定性,接下來將進一步對不同處理條件下,澳洲堅果中內參基因的表達穩定性進行深入研究。
參考文獻
Russ Stephenson. Macadamia: Domestication and commercialization[J]. Chronica Horticulture, 2005, 45(2): 11-15.
Manohar L G, Robert J B, Ron B H W. Macadamia nut consumption lowers plasma total and LDL cholesterol level in hypercholesterolemic men[J]. The Journal of Nutrition, 2003, 133(4): 1060-1063.
Duxbury D D. Lipid scientists shake healthy macadamia nut tree[J]. Food Processing, 2003(6): 83.
杜? 杉. 云南已成澳洲堅果種植老大——第八屆國際澳洲堅果大會將首次走進中國[J]. 時代金融, 2018(19): 52-54.
Dheda K, Huggett J F, Bustin S A, et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR[J]. Biotechniques, 2004, 37(4): 112-119.
孫美蓮, 王云生, 楊冬青, 等. 茶樹實時熒光定量PCR分析中內參基因的選擇[J]. 植物學報, 2010, 45(5): 579-587.
Amy L F, Charles R T. Appropriate “housekeeping” genes for use in expression profiling the effect of environmental estrogens in fish[J]. BMC Molecular Biology, 2007(8): 10.
Gutierrez L, Mauriat M, Guénin S, et al. The lack of a systematic validation of reference genes: a serious pitfall undervalued in reverse transcription polymerase chain reaction (RT-PCR) analysis in plants[J]. Plant Biotechnology Journal, 2008, 6(6): 609-618.
Nygard A B, J?rgensen C B, Cirera S, et al. Selection of reference genes for gene expression studies in pig tissues using SYBR green qPCR[J]. BMC Molecular Biology, 2007(8): 67.
Tomaz T, Bagard M, Pracharoenwattana I, et al. Mitochondrial malate dehydrogenase lowers leaf respiration and alters photorespiration and plant growth in Arabidopsis[J]. Plant Physiology, 2010, 154(3): 1143-1157.
楊彩霞, 雒? 軍, 王引權, 等. 當歸延伸因子AsEF-1β基因的克隆及脅迫應答分析[J]. 西北植物學報, 2019, 39(8): 1371-1378.
王? 芳, 董美玲, 董? 樂, 等. 蓖麻延伸因子基因的克隆與表達分析[J]. 核農學報, 2019, 33(3): 464-472.
JeeNa H, Chang-Sik O, Byoung-Cheorl K. Translation elongation factor 1B (eEF1B) is an essential host factor for Tobacco mosaic virus infection in plants[J]. Virology, 2013, 439(2): 105-114.
常鵬杰, 申亞梅, 董? 彬, 等. 玉蘭鹽脅迫下qRT-PCR分析中內參基因的篩選[J]. 農業生物技術學報, 2018, 26(9): 1611-1620.
朱海生, 劉建汀, 溫文旭, 等. 印度南瓜延伸因子基因CmEF1a的克隆與分析[J]. 核農學報, 2019, 33(6): 1096-1104.
陳曉榮, 陳昶旭, 施力銘, 等. 植物翻譯延伸因子eEF1A的研究進展[J]. 農技服務, 2019, 36(2): 64-67.
袁? 偉, 萬紅建, 楊悅儉. 植物實時熒光定量PCR內參基因的特點及選擇[J]. 植物學報, 2012, 47(4): 427-436.
Vandesompele J, De Preter K, Pattyn F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biology, 2002, 3(7): 1-11.
Andersen C L, Jensen J L, Rbtoft T F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Research, 2004, 64 (15): 5245-5250.
Pfaffl M W, Tichopad A, Prgomet C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper-Excel-based tool using pair-wise correlations[J]. Biotechnology Letters, 2004, 26(6): 509-515.
Xie F, Xiao P, Chen D, et al. miRDeepFinder: a miRNA analysis tool for deep sequencing of plant small RNAs[J]. Plant Molecular Biology, 2012, 80(1): 75-84.
Silver N, Best S, Jiang J. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Molecular Biology, 2006, 7(33): 1-9.
Fan C, Ma J, Guo Q. Selection of reference genes for quantitative real-time PCR in bamboo (Phyllostachys edulis)[J]. PLoS One, 2013(8): e56573.
Pérez S, Royo L J, Astudillo A, et al. Identifying the most suitable endogenous control for determining gene expression in hearts from organ donors[J]. BMC Molecular Biology, 2007, 8(1): 1-23.
Rodríguez M F, Sánchez-García A, Salas J J, et al. Characterization of soluble acyl-ACP desaturases from Camelina sativa, Macadamia tetraphylla and Dolichandra unguis-cati[J]. Journal of Plant Physiology, 2015, 178: 35-42.
Chervoneva I, Li Y, Schulz S, et al. Selection of optimal reference genes for normalization in quantitative RT-PCR[J]. BMC Bioinformatics, 2010, 11(1): 1-15.
Suzuki T, Higgins P J, Crawford D R. Reviews-control selection for RNA quantitation[J]. Biotechniques, 2000, 29(2): 332-337.
魏永贊, 賴? 彪, 胡福初, 等. 用于荔枝qPCR分析的內參基因克隆及穩定性分析[J]. 華南農業大學學報, 2012, 33(3): 301-306.
Chen L, Zhong H Y, Kuang J F, et al. Validation of reference genes for RT-qPCR studies of gene expression in banana fruit under different experimental conditions[J]. Planta, 2011, 234(2): 377-390.
Ding Y, Ma Q H. Characterization of a cytosolic malate dehydrogenase cDNA which encodes an isozyme toward oxaloacetate reduction in wheat[J]. Biochimie, 2004, 86(8): 509-518.
張俊平, 王英強. 黃花大苞姜花藥發育qRT-PCR內參基因篩選[J]. 廣西植物, 2018, 38(1): 76-83.

