基于線粒體12S rRNA基因的大鵬半島海域石珊瑚的系統發育分析
肖寶華,廖寶林,王 瓊,謝子強,2,謝勇琪,覃業曼
(1.廣東海洋大學 深圳研究院,廣東 深圳518000;2.深圳市碧海藍天海洋科技有限公司,廣東 深圳518000)
0 引言
造礁石珊瑚(刺胞動物門Cnidaria珊瑚綱Anthozoa六射珊瑚亞綱Hexocorallia石珊瑚目Scleractinia)組成的珊瑚礁生態系統是海洋環境中物種最豐富的生態群落之一[1],主要分布在地球南北緯30°以內的海域[2]2.以往的研究中,多采用珊瑚骨骼形態、顏色、觸手等依據構建珊瑚分類體系并對珊瑚進行分類[3].其中,石珊瑚(Scleractinian)多以珊瑚骨骼形態為依據進行分類[4],[5]1893.但是,珊瑚骨骼變異性和同源性[6]640等研究中尚未攻克的難關阻礙了人們對珊瑚分類進化的進一步認識.隨著分子生物學的快速發展與珊瑚多樣性研究的不斷深入,分子生物學方法逐漸應用于珊瑚的系統發育研究中,為珊瑚研究提供了新的思路.其中,基于不同算法模型建立的系統發育進化樹也為研究珊瑚進化關系提供新方法.20世紀90年代,科學家開始了石珊瑚的分子生物學研究[7]407.根據相關學者的分子生物學研究積累并結合已有的形態學研究,可以將造礁石珊瑚分為堅實型類群(Robust)和復合型類群(Complex)[6] 641.堅實型珊瑚被認為是鈣化程度較高,具備堅實的骨骼結構,珊瑚外觀主要以碟片狀和團塊狀為主的類群,包括蜂巢珊瑚科(Faviidae)、石芝珊瑚科(Fungiidae)和褶葉珊瑚科(Lobophyllidae)等[6] 641.復合型珊瑚骨骼鈣化程度較低,形態復雜多變,包括鹿角珊瑚科(Acroporidae)、菌珊瑚科(Agaricidae)和濱珊瑚科(Portidae)等[6] 641.同時,科學家在研究的過程中發現珊瑚蟲的線粒體基因進化速度慢,相對保守[8].線粒體的12S rRNA基因、16S rRNA基因和細胞色素b基因、細胞色素c氧化酶亞基I基因(COI)等通常作為研究珊瑚系統發育進化的標記基因,其中,rRNA基因結構保守,進化速度緩慢,是研究生物進化關系的典型特征基因[9] 460.
深圳市東部海域受亞熱帶季風氣候的影響,年平均海水表面溫度(SST)為21.6 ℃,平均鹽度32,地表徑流較少且短,呈放射狀入海,海岸曲折,灘涂面積少,海洋底質多為粘土和粉沙[10].優越理想的自然條件使大鵬半島海域成為石珊瑚生長發育的溫床.大鵬半島海域的石珊瑚不但種類豐富,并且珊瑚覆蓋率較高,由石珊瑚形成的珊瑚礁生態系統是深圳生態基石的重要組成.2007年對深圳海域珊瑚資源進行調查,共記錄了47種石珊瑚[11].近年來,由于受到氣候變暖、海洋酸化以及人類活動的影響,全球珊瑚資源呈現退化的趨勢[12-13],大鵬半島海域的石珊瑚覆蓋率也逐年下降.因此,本研究通過聚合酶鏈式反應技術(PCR),以石珊瑚線粒體12S rRNA基因為分子標記,對大鵬半島海域的16個石珊瑚樣品種間關系和系統進化關系進行研究,旨在為深圳東部海域珊瑚資源保護工作提供相應理論基礎.
1材料與方法
1.1 采樣站位及方法
供試的石珊瑚樣品采自深圳東部大鵬半島海域的6個站位(圖1),具體經緯度見表1.珊瑚樣品采于2018年7~8月,挑選風平浪靜,海水透明度高的時候出海采樣.實驗人員準備好潛水和水下拍攝設備后,在相應的站位下潛,剪取珊瑚邊緣斷枝(1~2 cm)并拍攝珊瑚樣品圖片和視頻,以便后期樣品的形態學鑒定.所采集的珊瑚樣品截取小部分于裝有無水乙醇的離心管中保存,用于后續提取樣品基因組總DNA.
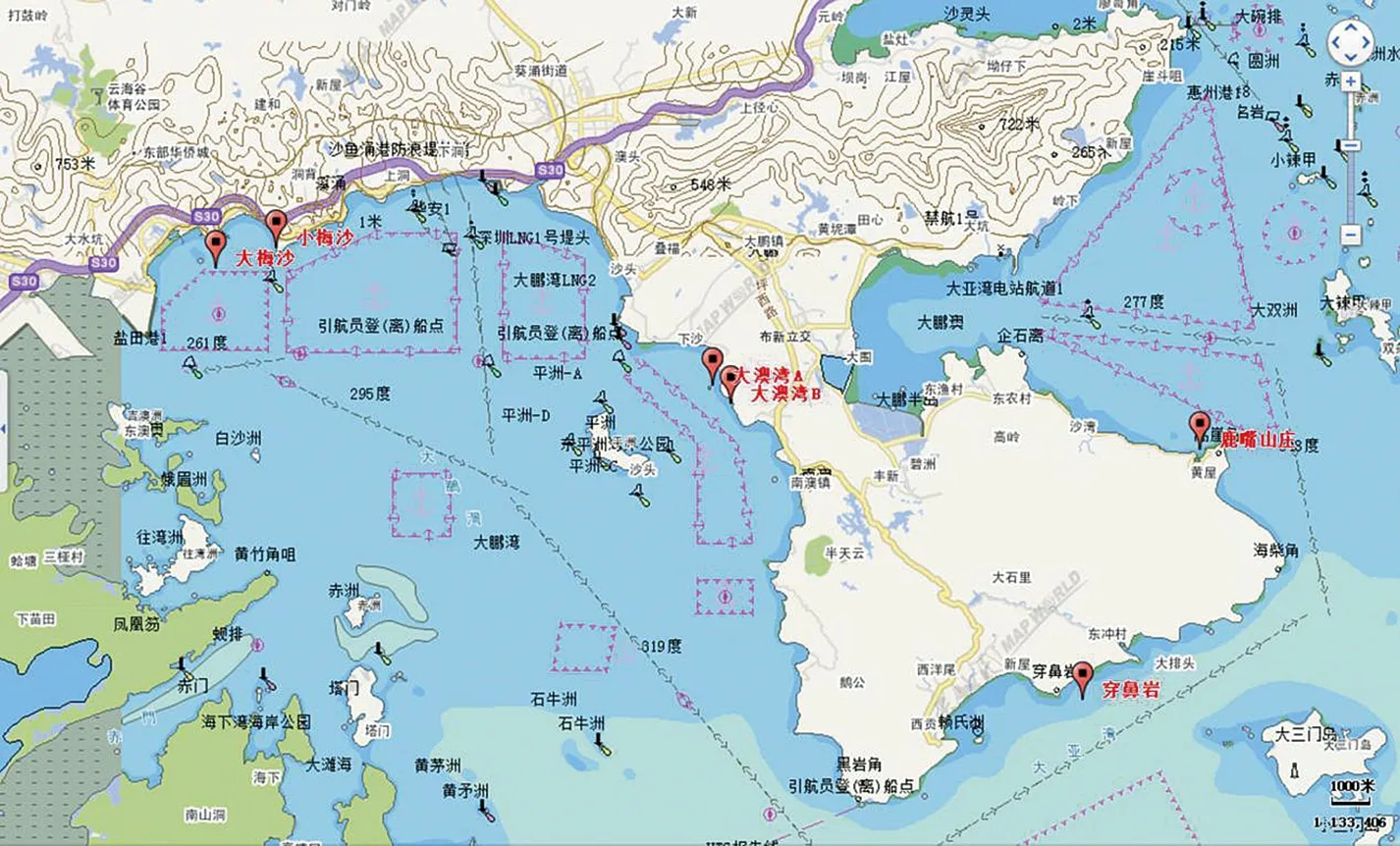
圖1 深圳東部大鵬半島海域采樣站位圖
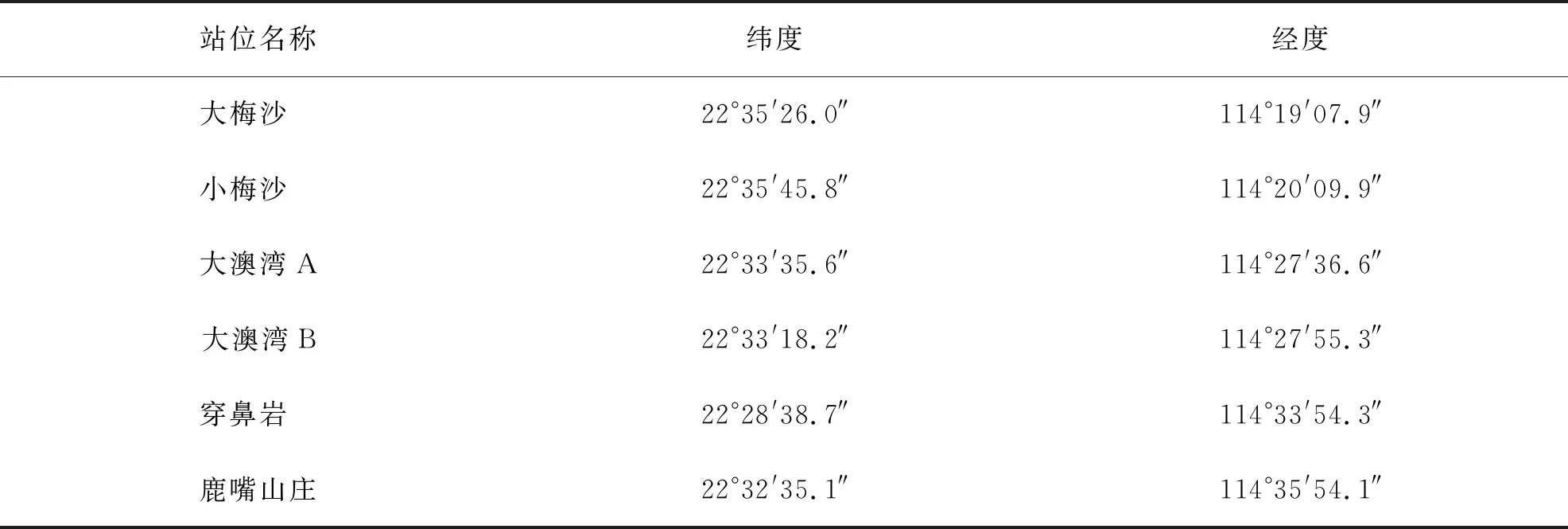
表1 深圳東部大鵬半島海域采樣站位信息表
1.2 樣品的形態學鑒定
樣品的形態學鑒定參照鄒仁林[2]19-235的《中國動物志-腔腸動物門·珊瑚蟲綱·石珊瑚目·石珊瑚》和Veron[5]1893-1894六射珊瑚骨骼形態特征分類系統,將所采集的珊瑚樣品依據珊瑚骨骼形態分為5科7屬9種,各樣品的形態學分類情況如表2所示.
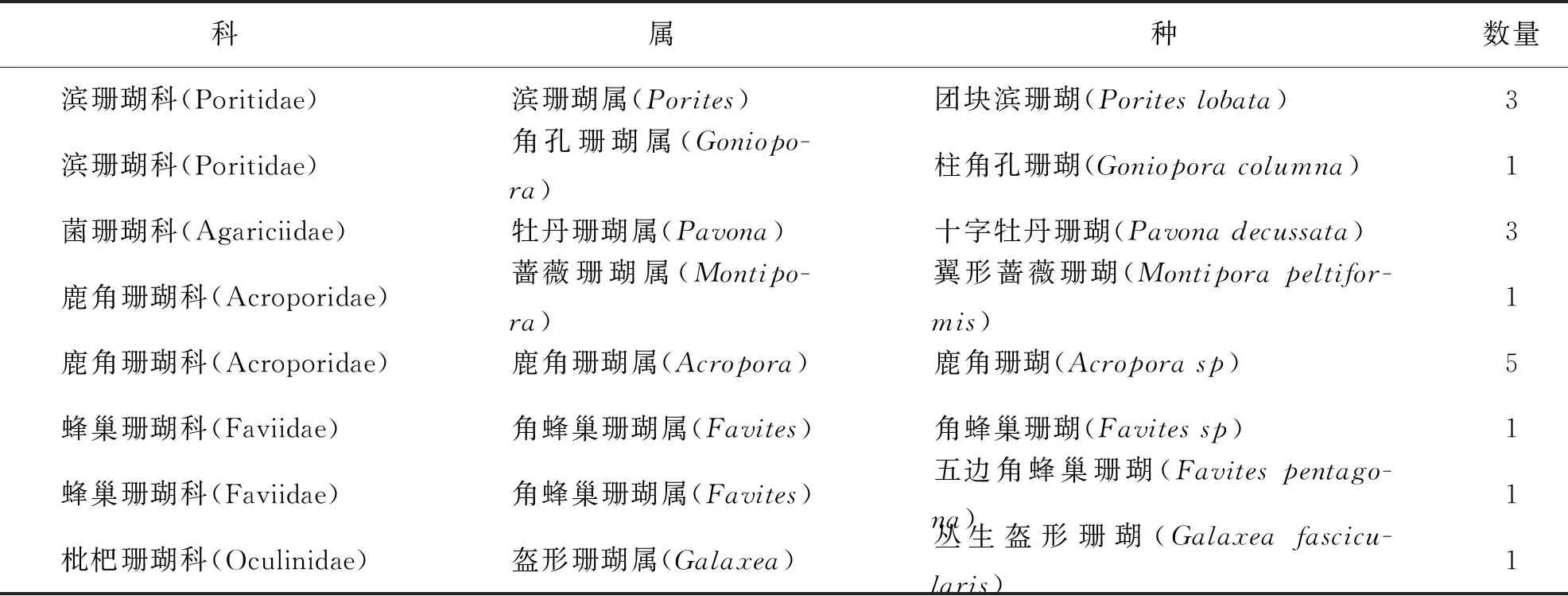
表2 珊瑚樣品的形態學分類
1.3 樣品的基因組總DNA提取
采集的樣品使用濾紙吸附表面黏液并用工具搗碎,搗碎后的樣品使用DNA提取試劑盒(北京天根生物有限公司)提取細胞組織基因組.提取獲得的DNA樣品在經過凝膠電泳檢測后,置于4 ℃冰箱中貯藏備用.
1.4引物設計合成
12S rRNA基因的PCR引物采用文獻[14]的12S基因通用引物,并委托上海生工生物技術服務有限公司合成.引物序列為:f-AGC CAC ACT TTC ACT GAA ACA AGG;r-GTT CCC YYW CYC TYA CYA TGT TAC GAC.采用升溫PCR擴增,先以50 ℃退火5個循環接56 ℃退火的30個循環,產物大小為703~887 bp.
1.5 PCR擴增和DNA測序
PCR反應體系反應(總體積為25 μL):0×Buffer 2.5 μL;dNTP 2 μL;上下游引物各1 μL;Taq DNA聚合酶1 U;DNA模板2 μL;其余用去離子水補至25 μL.
PCR反應條件:5 min預變性(95 ℃),46 s變性(95 ℃),46 s退火(50 ℃),1 min 30 s延伸(72 ℃),共35個循環,最后72 ℃延伸10 min.擴增產物在10 g·L-1的瓊脂糖凝膠中檢測,選取擴增條帶單一、濃度較高的PCR產物送至上海生工生物技術公司進行測序,獲取各實驗樣品的序列信息.
1.6 序列分析
測序所得序列用DNASTAR中的Editseq軟件進行編輯、剪切、校正,再將校正好的序列與NCBI非冗余數據庫進行同源性比對,以確保所獲得的序列為目標序列.
1.7 系統進化樹的構建
采用MEGA6.0軟件進行基因序列分析,獲取樣品12S rRNA基因組序列長度、堿基組成、簡約信息位點等相關信息,同時根據簡約信息位點,計算出種間遺傳距離.本研究中系統進化樹是使用MEGA6.0軟件基于Kimu-ra雙參數模型,采用鄰位連接法(Neighbor-Joinning,NJ)、最小進化法(Minimum-Evolution,ME)和最大似然法(Minimum-Likelihood,ML)構建出來的.系統進化樹中節點的自舉置信度水平由自引導值(Bootstrap value)估計,重復次數為1 000.
2 結果與分析
2.1 序列堿基組成分析
通過擴增測序,分別獲得16個石珊瑚樣品的12S rRNA的基因序列.從表3可知,本研究中的16條基因序列長度在692~830 bp之間.從堿基組成比例來看,16個石珊瑚樣品的12SrRNA序列的C含量在12.3%~15.5%之間,G含量在20.5%~24.3%之間,T含量在29%~31.8%之間,A含量最高,在31.8%~35.5%之間.所有序列堿基A、T的含量均高于G、C,具有明顯的AT偏倚.其中,Poriteslobata1與Poriteslobata2、Poriteslobat3序列堿基組成相同;Pavonadecussata1和Pavonadecussata3序列堿基組成相同;Acroporasp1、Acroporasp2、Acroporasp3、Acroporasp4和Acroporasp5序列堿基組成相同.
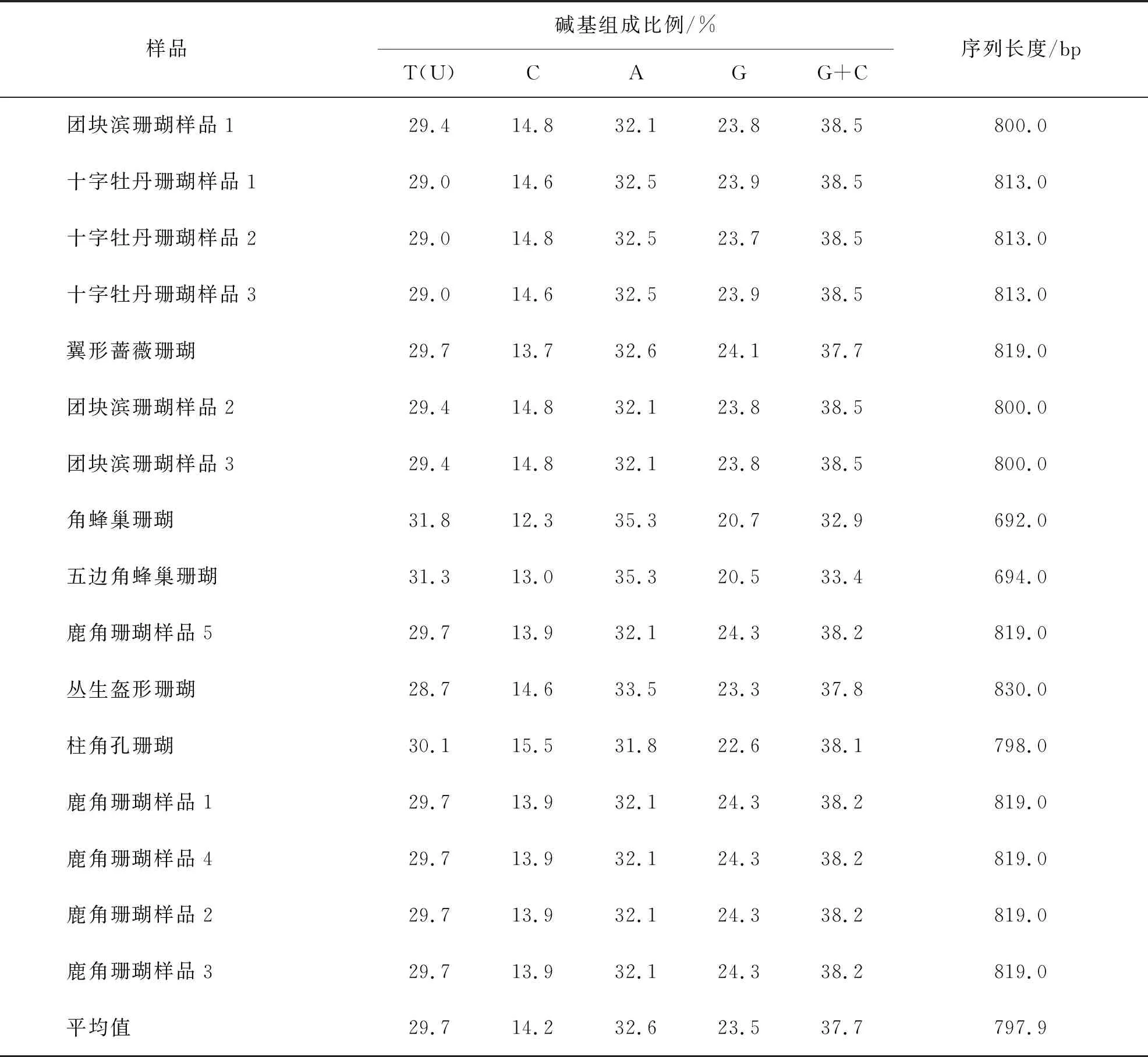
表3 16個石珊瑚樣品的12S rRNA基因片段的序列組成
2.2 序列變化情況及種間遺傳距離
通過MEGA6.0計算16個石珊瑚樣品的種間遺傳距離.從表4可知,16個樣品的平均距離為0.094 3.遺傳距離在科、屬、種間呈逐級遞減趨勢.其中,五邊角蜂巢珊瑚(Favitespentagona)和十字牡丹珊瑚第2個樣品之間(Pavonadecussata2)的種間遺傳距離最大,為0.256 8.團塊濱珊瑚的3個樣品(Poriteslobate1、Poriteslobate2、Poriteslobata3)間遺傳距離為0,根據形態學初步判定這3個樣品為同一種石珊瑚,序列堿基組成分析和種間遺傳距離分析數據也證實了這一點;十字牡丹珊瑚樣品1(Pavonadecussata1)和十字牡丹珊瑚樣品3(Pavonadecussata3)遺傳距離為0,結合序列堿基組成和形態學分析,初步判定兩個樣品為同一種十字牡丹珊瑚;鹿角珊瑚的5個樣品(Acroporasp1、Acroporasp2、Acroporasp3、Acroporasp4、Acroporasp5)之間遺傳距離為0,通過序列堿基組成和形態學分析也可以判定這5個樣品為同一種鹿角珊瑚.據此,判定本研究的16個石珊瑚樣品來源于9種石珊瑚.排除遺傳距離為0的樣品,十字牡丹珊瑚樣品1(Pavonadecussata1)和十字牡丹珊瑚樣品2(Pavonadecussata2)間的遺傳距離最小,為0.001 5.
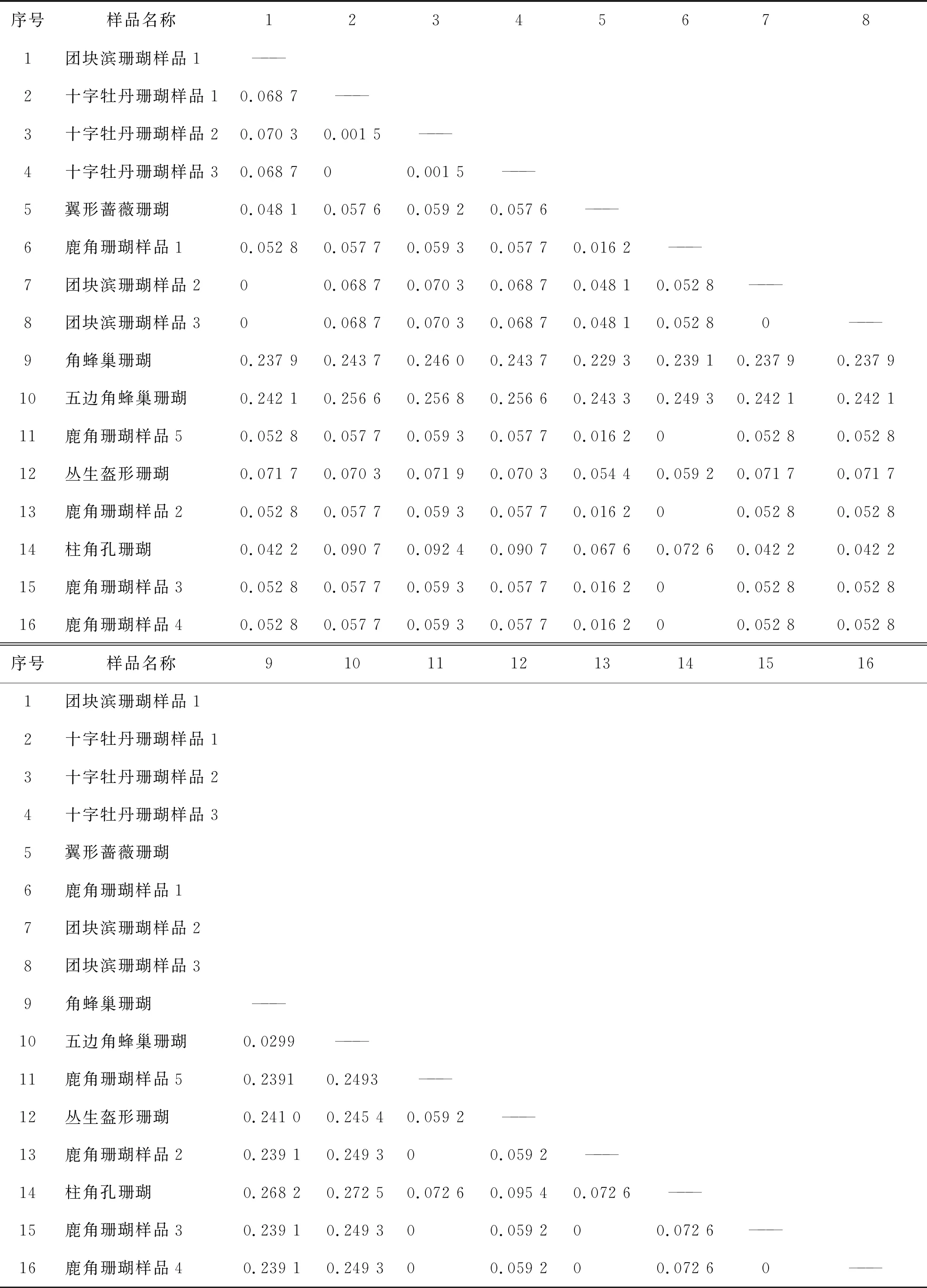
表4 基于12S rRNA基因的16個石珊瑚樣品種間遺傳距離
2.3 基于12S rRNA對九種石珊瑚的系統進化分析
使用MEGA6.0軟件將9種石珊瑚的12S rRNA通過鄰位連接法、最大似然法和最小進化法構建系統發育進化樹,結果顯示3種方法構建的系統發育進化樹拓撲結構基本一致,只是在節點置信度上略有差異.由系統發育進化樹(圖2)可知,這9種石珊瑚聚為2個大的分支:濱珊瑚科(Poritidae)、菌珊瑚科(Agariciidae)、鹿角珊瑚科(Acroporidae)和枇杷珊瑚科(Oculinidae)等復合型珊瑚聚為一簇;蜂巢珊瑚科(Faviidae)等堅實型珊瑚聚類為一簇,這個結果與已有的珊瑚分類學研究結果相符合[7]397,[15],上文種間遺傳距離分析中也能看出2個分支間的珊瑚種間距離值相較同一分支的科間、屬間、種間距離值較大.從聚類結果可見,根據傳統形態學方法劃分的各科親緣關系較近:同為濱珊瑚科(Poritidae)的柱角孔珊瑚(Gonioporacolumna)和團塊濱珊瑚(Poriteslobata)親緣關系較近,聚為一支;同為菌珊瑚(Agariciidae)的兩種十字牡丹珊瑚(Pavonadecussata1、Pavonadecussata2)的12SrRNA序列存在細微差異,聚為一支;同為蜂巢珊瑚科(Faviidae)的角蜂巢珊瑚(Favitessp)和五邊角蜂巢珊瑚(Favitespentagona)聚為一支.鹿角珊瑚(Acroporasp)與翼形薔薇珊瑚(Montiporapeltiformis)聚為一支,叢生盔形珊瑚(Galaxeafascicularis)和其他復合型珊瑚親緣關系較遠.
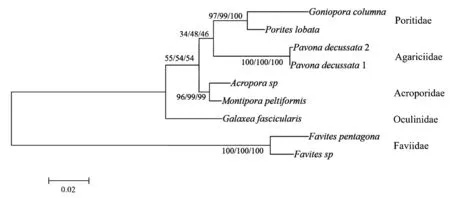
圖2 最大似然法構建九種石珊瑚12SrRNA基因序列系統發育進化樹注:分枝上數值表示最大似然法、鄰接法與最小進化法1 000次重復抽樣檢測的bootstrap值.
3 討論
本研究的石珊瑚樣品12S rRNA基因片段長度在692~830 bp之間,G+C含量在32.9%~38.9%之間,呈現明顯的AT偏倚.通過堿基組成分析、種間遺傳距離分析和形態學分析初步將本研究中的16個石珊瑚樣品分為9種,系統發育進化樹則將9種石珊瑚進一步分為堅實型珊瑚和復合型珊瑚兩簇.結合系統發育進化分析和堿基組成分析可見,本研究中的蜂巢珊瑚的12S rRNA序列長度在692~694 bp之間,G+C含量在32.9%~33.4%之間;而復合型珊瑚這一分支的12S rRNA序列長度在798~830 bp之間,G+C含量在37.7%~38.5%之間,因此,本研究中的堅實型珊瑚的蜂巢珊瑚和其余復合型珊瑚的12S rRNA在序列長度和G+C含量間有較為顯著的差別.但由于本研究中的實驗樣品較少,此結論還有待進一步驗證.由本文研究結果可知,不同種屬間的石珊瑚12S rRNA序列差異較為明顯,同一種屬的石珊瑚也存在細微差異.十字牡丹珊瑚的2個樣品(Pavonadecussata1、Pavonadecussata2)根據形態學鑒定為同一種,但兩者的12S rRNA基因存在這差異,說明石珊瑚12S rRNA基因具備用于石珊瑚種間分化研究的潛力.
在石珊瑚分子生物學研究中多采用線粒體COI基因和16S rRNA等作為分子標記基因.COI基因長度適宜、進化速率慢并且富含系統發育信號,能夠較好地進行遺傳進化分析[9]459.但也由于石珊瑚COI基因緩慢的進化速率,導致石珊瑚COI基因較低的種內分化,在種內鑒定方面存在不足[16].16S rRNA基因也具備較為保守、進化速率較慢的特點,適宜用作種以上分子分類的標記,但在部分珊瑚科屬內也存在著種間分化較低的問題[17].相較而言,12S rRNA的基因序列的長度較長、差異性較大并且能夠較好地鑒別珊瑚的種間的差異,能夠彌補COI基因和16S rRNA在分子鑒定方面的不足,可以較好地作為其他分子標記研究的補充和佐證.

