朝倉花椒DIR基因家族的鑒定及分析
陳炳竹 王潤英 趙懿琛 趙德剛
摘要:本研究通過NCBI的BLAST比對分析,最終從朝倉花椒轉錄組數據庫篩選出28條朝倉花椒Dirigent(DIR)蛋白家族的編碼基因序列。利用 MEGA、MEME、DNAMAN 等軟件對該蛋白家族進行生物信息學分析,結果顯示:從朝倉花椒轉錄組中獲得的28個DIR基因,經過鑒定,在進化上分
為6個小組,每個小組的基因數量分別為7、5、1、0、5、10。該家族的蛋白等電點均小于7,均偏酸性,且多數屬于穩定蛋白。多基因序列比對比發現,該家族蛋白均具有一個高保守性的DIR結構域。空間結構的分析預測ZpDIR 蛋白多為多鏈線性折疊卷曲蛋白,且以線性無鏈非規則多鏈卷曲蛋白為主。
關鍵詞:朝倉花椒;DIR基因家族;生信分析
中圖分類號:Q811.4
文獻標識碼:A
文章編號:1008-0457(2020)02-0021-06國際DOI編碼:10.15958/j.cnki.sdnyswxb.2020.02.003
Identification and analysis of DIR Gene Family in Zanthoxylum piperitum
DC.var.inerme Makino
CHEN Bingzhu1,WANG Runying1,ZHAO Yichen1*,ZHAO Degang1,2
(1.College of Life Sciences/College of Tea? Sciences, Guizhou University, Key Laboratory of Germplasm Innovation for
Conservation and Protection of Mountain Plant Resources, Guizhou Key Laboratory of Agricultural Biological Engineering, Guiyang, Guizhou 550025, China;2.Guizhou Academy of Agricultural Sciences/Guiyang Branch of National DUS Center,? Guiyang, Guizhou? 550006, China)
Abstract:
In this study, 28 coding gene sequences of the Zanthoxylum piperitum DC.var.
inerme Makino Dirigent (DIR) protein family from Z. piperitum DC.var.
inerme Makino transcriptome database were screened via GenBank National Center for Biotechnology Information (NCBI) Basic Local Alignment Search Tool (BLAST). Bioinformatics analysis of the protein family was carried out using MEGA, MEME, DNAMAN and other softwares. This research through the NCBI BLAST ratio analysis, finally from the Zanthoxylum? piperitum DC.var.inerme Makino transcriptome database selected article 28 at the Zanthoxylum piperitum DC.var.inerme Makino Dirigent(DIR) protein family encoding gene sequences using the MEGA, MEME, DNAMAN software such as bioinformatics analysis, carried out on the protein familyThe results showed that the 28 DIR genes obtained from Z. piperitum DC.var.inerme Makino transcriptome were identified and belonged to 6 subfamilies in evolution, according to the results of identification from the Zanthoxylum piperitum DC.var.inerme Makino transcriptome of 28 DIR genes in evolutionary points belong to 6 family, The number of genes in each subfamily was 1, 20, 3, 1, 2, and 1, respectively.each family of the number of genes of 20, 3, 1, 2, 1 respectively. The isoelectric points of the proteins ofin this family arewere all less than 7, all of themwhich are acidic, and most of them belong to? the stable proteins polygene sequence ratio. The results of multiple gene sequence comparisons showed that the family proteins all had a highly conserved DIR domain. The analysis of spatial structure predicts that most ZpDIR proteins were multi-chain linear folded coiled proteins, and mainly linear non-chain irregular multi-chain coiled proteinsIt was found that the proteins of this family all have a highly conserved DIR domain spatial structure.
Keywords:Zanthoxylum piperitum;
DIR gene family; bioinformatics analysis
木脂素激酶是分布廣泛的一類植物次生細胞代謝活性物質,具有一定抗菌抗病特性,參與很多植物機體抗病生長過程,Dirigent(DIR)蛋白參與木脂素的合成,是木脂素合成途徑的重要蛋白[12]。對小麥、楊樹、擬南芥和水稻等植物的
DIR基因家族進行鑒定發現,DIR蛋白相對比較保守,大多數DIR基因含有一個DIR結構域,該結構域是DIR蛋白的主要組成結構域[3]。DIR蛋白不僅在植物次生代謝產物合成過程中發揮作用,還參與植物抗逆性,在植物抵御非生物脅迫、生物防御應答中起重要作用。
花椒(Zanthoxylum bungeanum Maxim)作為我國常用的藥食兩用作物,具有較高的生態價值和經濟價值。除此之外,已有研究發現花椒還具有很好的抗菌能力,其對包括炭疽桿菌、金黃色葡萄球菌、枯草桿菌在內的革蘭氏陽性菌以及大腸桿菌、變形桿菌、霍亂弧菌等多種革蘭氏陰性菌有很好的抑制作用[4]。朝倉花椒(
Zanthoxylum piperitum DC.var.inerme Makino)也稱山椒,為蕓香科(Rutaceae)花椒屬
(Zanthoxylum L.)落葉灌木[5],具有高產、優質、精油含量高等特點。本實驗室前期針對朝倉花椒在抗菌方面的分子機制展開了相關研究,目前已成功克隆得到一個朝倉花椒幾丁質酶類似基因ZaCHIT1-like[6]。本研究主要對朝倉花椒中具有抗菌活性的DIR蛋白家族進行鑒定和生物信息學分析,以期對朝倉花椒抗菌調控機制的研究提供理論基礎。
1材料與方法
1.1材料
從朝倉花椒轉錄組數據庫中提取的31條朝倉花椒DIR蛋白家族的編碼基因序列作為本研究的實驗材料。
1.2朝倉花椒DIR基因家族的鑒定
本研究基于前期獲得的朝倉花椒轉錄組數據庫注釋信息,從朝倉花椒轉錄組中初步篩選獲得31條DIR蛋白的編碼序列,并通過與NCBI(http://www.ncbi.nlm.nih.gov/) 數據庫中BLAST比對和保守結構域分析[7],最終鑒定出28條朝倉花椒的DIR家族基因序列。
1.3朝倉花椒DIR蛋白基本性質
采用ProtParam(https://web.expasy.org/protparam/)在線分析工具[8]預測分析朝倉花椒DIR蛋白家族的理化性質。
1.4進化關系分析
利用朝倉花椒DIR家族序列在BLAST程序上比對出3個同源性較高的物種:蒺藜苜蓿(
Medicago truncatula)、擬南芥(
Arabidopsis thaliana L.)、甜橙(
Citrus sinensis)。并從基因序列下載網站(https://phytozome.jgi.doe.gov/pz/portal.html#)中下載蒺藜苜蓿、擬南芥和甜橙已經發表了的DIR蛋白氨基酸序列,利用MEME軟件構建DIR基因系統進化樹。
1.5ZpDIR蛋白結構分析
采用SOPMA在線工具完成蛋白質二級結構的預測分析[9],采用SWISS-MODEL在線分析軟件完成蛋白質空間結構的預測[10]。
1.6ZpDIR多序列比對和保守基序分析
利用DNAMAN序列分析軟件對得到的28條序列進行多序列比對[11],在線分析軟件MEME(http://meme-suite.org/tools/meme)定位各條DIR蛋白序列的保守基序[12]。
2結果與分析
2.1ZpDIR基因家族的鑒定
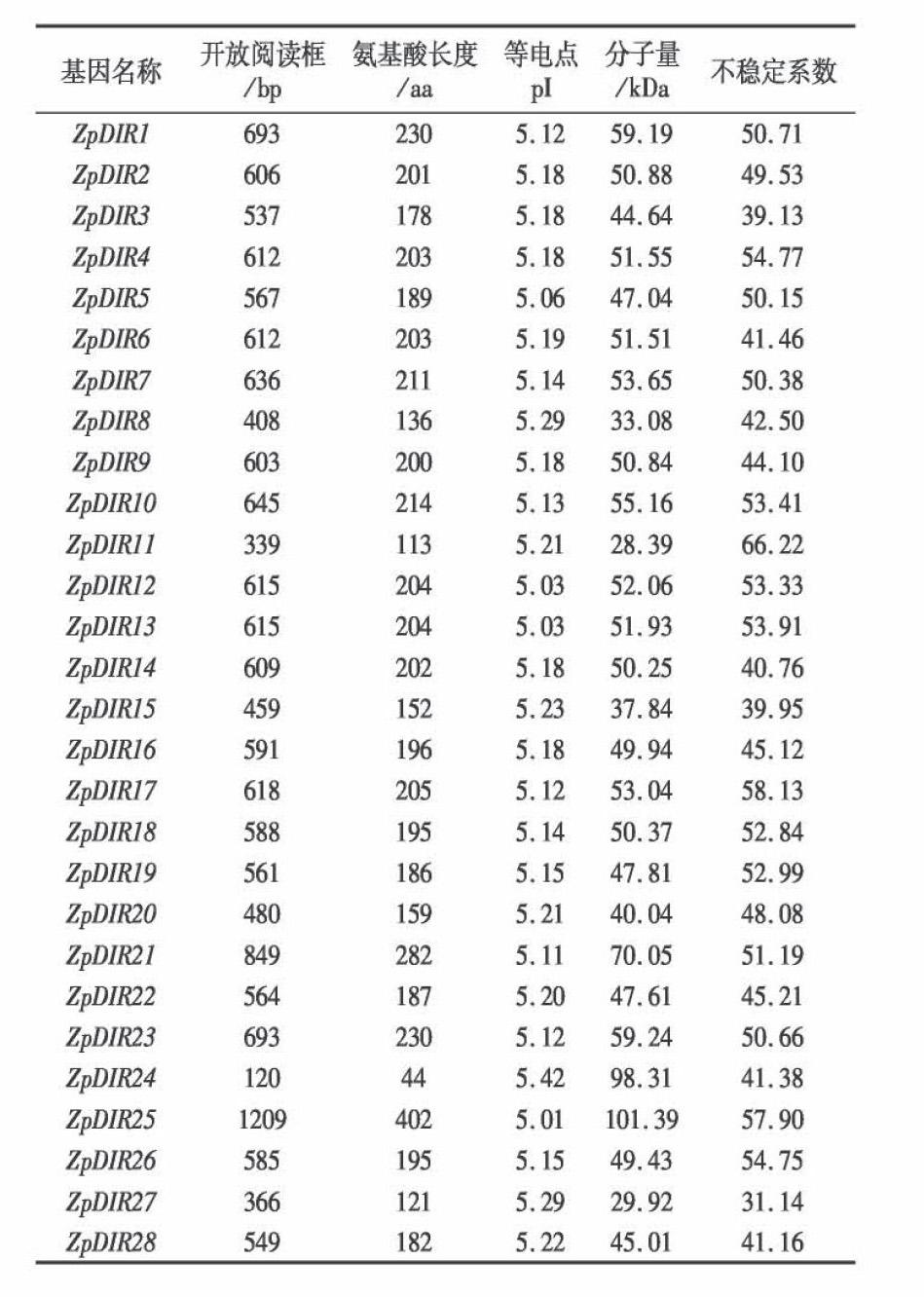
通過朝倉花椒的轉錄組數據的注釋信息和NCBI數據庫的比對結果,對初步篩選得到的31個DIR候選基因的蛋白保守結構域進行預測,最終鑒定獲得28條朝倉花椒DIR家族基因,并將其命名為
ZpDIR1~ZpDIR28(表1)。利用ExPASy在線軟件,對該家族的28個蛋白分別進行理化性質的分析,結果顯示,該家族基因在分子大小上具有顯著差距,其中基因編碼長度最短的為
ZpDIR27, 其長度為120 bp,基因編碼長度最長的基因為
ZpDIR28, 其編碼基因總長度為1209 bp,兩者差距近10倍。由此可知其對應的蛋白大小相差較大,28個蛋白的相對分子量在28.39 kDa~101.39 kDa之間,其中蛋白分子量最小的為
ZpDIR11, 最大的為ZpDIR28
。對其蛋白質理化性質預測顯示,該家族蛋白雖然在大小上差異顯著,但均為偏酸性蛋白,等電點范圍在
5.03~5.42。
2.2ZpDIR 蛋白進化關系分析
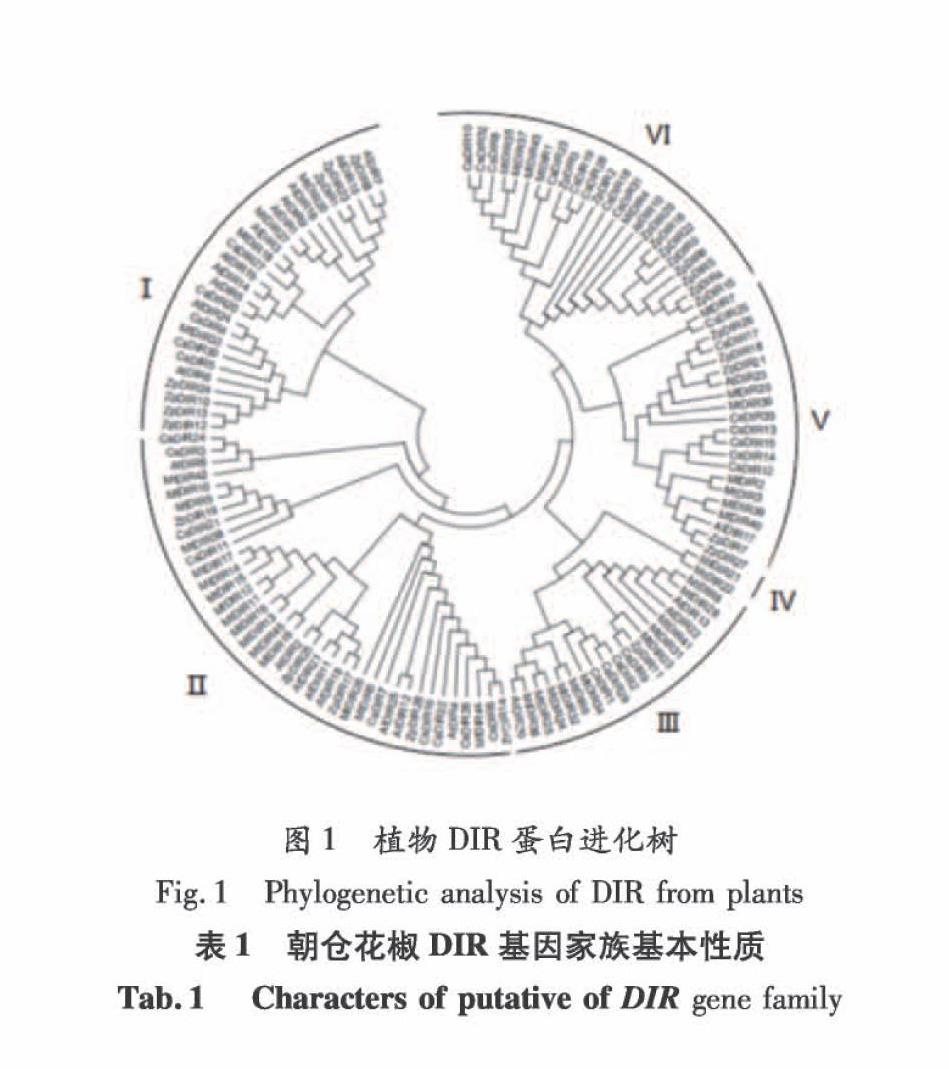
為研究ZpDIR的進化關系,使用MEGA軟件構建朝倉花椒與蒺藜苜蓿、擬南芥和甜橙的DIR家族系統進化樹,根據進化樹的分枝,可將朝倉花椒DIR家族(ZpDIR1-ZpDIR28)與蒺藜苜蓿DIR家族(MtDIR1~MtDIR45)、擬南芥DIR家族(TpDIR1~TpDIR25)和甜橙DIR家族(CsDIR1~CsDIR33)家族分成6個小組(I~VI),其中I組中包括7個ZpDIR成員:ZpDIR1、ZpDIR8、ZpDIR10、ZpDIR12、ZpDIR13、ZpDIR23、ZpDIR24,而ZpDIR14、ZpDIR17、ZpDIR19、ZpDIR25、ZpDIR28屬于II組,共5個,III組只包含1個ZpDIR成員,即ZpDIR11,VI組中不包含ZpDIR成員,V組中也包括了5個ZpDIR成員:ZpDIR7、ZpDIR18、ZpDIR21、ZpDIR26、ZpDIR27,VI組包含的ZpDIR最多,分別為ZpDIR2、ZpDIR3、ZpDIR4、ZpDIR5、ZpDIR6、ZpDIR9、ZpDIR15、ZpDIR16、ZpDIR20、ZpDIR22共10個成員(圖1) 。
2.3ZpDIR蛋白高級結構預測分析
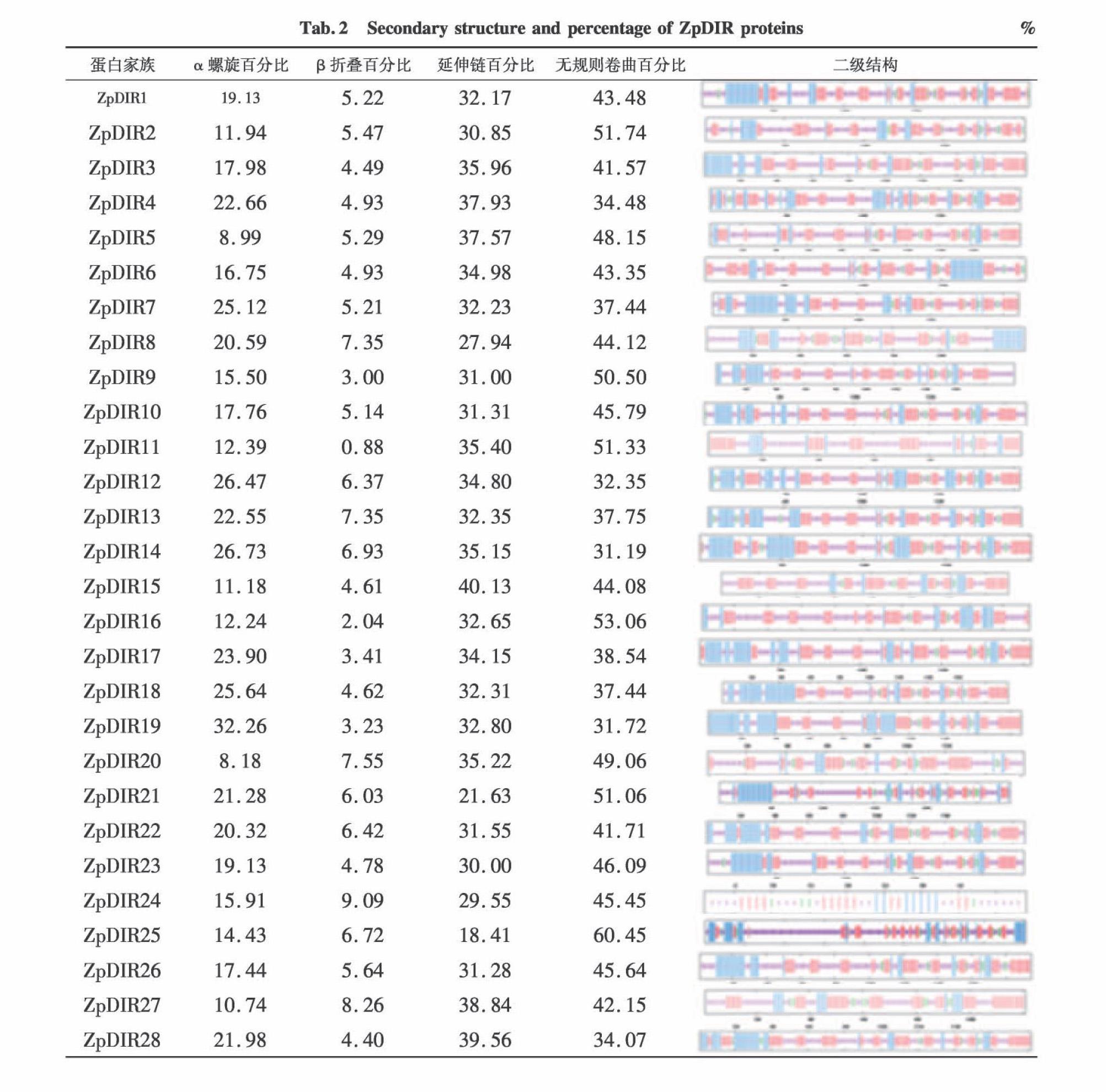
為深入研究ZpDIR蛋白空間結構上的相似性,對ZpDIR家族蛋白進行二級結構預測分析(表2)。結果顯示,該蛋白家族在二級結構上的特征略有差異,其中ZpDIR蛋白α螺旋的比例最大的值為32.26%,而最大值卻是8.18%,β折疊比例最大值為8.26%,最小值則是2.04%,由此可知ZpDIR蛋白的α螺旋與β折疊比例差異大,最大最小之間差距約為4倍。延伸鏈的比例范圍在18.41%~39.56%,無規則卷曲比例偏高,范圍是31.19~至61.45%,延伸鏈與無規則卷曲最大最小的比例差距較小一些,約為2倍。對ZpDIR蛋白三級(空間)結構進行預測,發現各蛋白三級結構模型大致相似(圖2),除ZpDIR11、ZpDIR24、ZpDIR25的三級結構較為簡單以外,其余蛋白在三級模型上相差不大。對蛋白空間模型的預測分析發現ZpDIR蛋白屬于多鏈線性折疊卷曲蛋白,絕大多數是線性無鏈非規則多鏈卷曲蛋白。
2.4ZpDIR家族蛋白結構域序列比對
利用DNAMAN軟件對篩選得到的28條朝倉花椒DIR蛋白的保守結構域序列進行多序列比對,發現朝倉花椒DIR家族DIR結構域多個氨基酸位點維持保守不變,因此具有較高保守性(圖3)。
2.5ZpDIR家族蛋白結構保守基序分析
使用在線軟件MEME對28條氨基酸序列的保守基序進行預測分析(見圖4),除ZpDIR27蛋白由于保守結構域序列過短,沒有找到保守基序以外,其余大多氨基酸保守結構域序列都擁有5種保守基序,這再一次證明了DIR結構域是高度保守的。
3結論與討論
DIR蛋白是植物木脂素生物合成途徑中的重要參與者之一,調控著植物木脂素的合成,并參與植物對生物和非生物脅迫的抵御。該家族蛋白結構均含有一個典型的
DIR結構域,? 少數成員含有兩個串聯的DIR結構域。有些DIR蛋白還含有其他結構域, 比如C端融合的木菠蘿素類凝集素(Jacalin-related lectin, JRLs)結構域, 該結構域是JRL類凝集素蛋白的典型結構域,同抗病、抗蟲等植物防御體系有密切關系[13]。已有研究基于基因
組或轉錄組數據對小麥[3]、毛竹[14]、
水稻[15]和丹參[16]的
DIR基因家族成員進行鑒定分析,結果表明,不同物種的DIR家族在成員數量上、結構域數量以及類型上和進化模式上都存在著差異。
朝倉花椒是一種重要的經濟作物,本實驗室前期對朝倉花椒雌株結果期幼芽的轉錄 組進行測序,經拼接比對后,獲得了62150個表達序列標簽,以及68315個注釋基因[17]。在前期研究的基礎上,本研究通過注釋比對,從轉錄組數據庫中篩選得到28條DIR蛋白的序列。通過生物信息學分析可知,朝倉花椒的DIR蛋白家族均含有保守的DIR結構域,且多個氨基酸位點相對穩定,蛋白家族的蛋白質三級結構相對保守,且結構相似。然而,該家族編碼基因大小差異顯著,開放閱讀框最小為120 bp,最大為1209 bp,由此可知朝倉花椒中的
DIR蛋白兼具專一化與多樣化的特征。以朝倉花椒、蒺藜苜蓿、擬南芥和甜橙DIR家族共同構建的進化樹將其分成6個小組(I~VI),其中ZpDIR主要集中位于I與VI組,而2、3組主要包含的是MtDIR與CsDIR。有研究顯示,I組被歸為DIR-a亞家族[18],它包含了朝倉花椒、蒺藜苜蓿、擬南芥以及甜橙4個物種的DIR
基因,其中擬南芥DIR成員最多,為11個,而朝倉花椒、蒺藜苜蓿、以及甜橙的
DIR成員數量分別為7、4、4個。本研究通過對朝倉花椒
DIR基因家族進行鑒定和分析,對該家族蛋白的基本結構、生化性質以及進化關系等方面有了初步了解,為后期該基因功能的研究提供了理論基礎。
參考文獻:
[1]王玲. 杜仲Dirigent蛋白編碼基因
EuDIR3克隆及遺傳轉化[D].貴陽:貴州大學, 2019.
[2]BURLA V, KWON M, DAVIN LB, et al. Dirigent proteins and dirigent sites in lignifying tissues [J]. Phytochemistry (Oxford), 2001, 57(6): 883897.
[3]宋敏. 小麥JRL和DIR基因家族的鑒定與分析[D].南京:南京農業大學, 2013.
[4]田代華. 實用中藥詞典[M].北京:人民衛生出版社,2002: 869873.
[5]陳錫, 曾曉芳, 趙德剛. 朝倉花椒葉水浸提物對白菜種子萌發及幼苗生長的影響[J].種子, 2016, 35(2):3741.
[6]劉雪薇, 趙懿琛, 趙德剛. 朝倉花椒幾丁質酶類似基因的克隆及分析[J].基因組學與應用生物學, 2018, 37(4):15621569.
[7]李慧峰, 冉昆, 何平, 等.蘋果生長素響應因子(ARF)基因家族全基因組鑒定及表達分析[J].植物生理學報, 2015, 51(7): 10451054.
[8]GASTEIGER E, HOOGLAND C, GATTIKER A, et al. Protein identification and analysis tools on the ExPASy server[M].New Jersey:Human Press,2005:571607.
[9]COMBET C, BLANCHET C, GEOURJON C, et al. NPS@: network protein sequence analysis
[J].Trends in biochemical science, 2000, 25 (3): 147150.
[10]KELLY LA, MEZULIS S, Y ATES CM, et al. The phyre2 web portal for protein modelling, prediction and analysis[J].
protocals, 2015, 10 (6): 845858.
[11]BAILEY TL, ELKAN C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers
[J].Proceedings.International conference on intelligent system for molecular biology,1994, 2: 2836.
[12]ELLIS CM, NAGPAL P, YOUNG JC, et al. Auxin response factor1 and auxin response factor2 regulate senescence and floral organ abscission in Arabidopsis thaliana[J].Development, 2005, 132 (20): 45634574.
[13]向陽, 杜才富, 馬正強. Jacalin類凝集素研究進展[J].植物生理學報,2013, 49 (10): 10231029.
[14]陳家璐, 張智俊, 劉笑雨, 等. 毛竹
Dirigent基因家族的全基因組鑒定與分析[J].植物生理學報, 2019, 55(9):14061417.
[15]穰中文, 周清明. 水稻
dirigent基因家族生物信息學分析[J].湖南農業大學學報(自然科學版), 2013, 39(2):111120.
[16]馬金洋, 楊瑾冬, 李卿, 等. 丹參Dirigent基因家族的發現與生物信息學分析[J].基因組學與應用生物學, 2017, 36(4):15941610.
[17]趙懿琛, 劉雪薇, 李巖, 等. 朝倉花椒幼芽轉錄組測序數據組裝及基因功能注釋[J].基因組學與應用生物學, 2016, 35 (7): 18051819.
[18]RALPH S G, JANCSIK S, BOHLMANN J. Dirigent proteins in conifer defense II: Extended gene discovery, phylogeny, and constitutive and stress-induced gene expression in spruce (Picea spp.) [J]. Phytochemistry (Amsterdam), 2007, 68(14): 19751991.

