利用環(huán)境DNA-宏條形碼技術(shù)監(jiān)測(cè)蘇州地區(qū)小管福壽螺的入侵
陳曉 方靖怡 王萌 沈晴 孫振軍 王備新






摘要 :小管福壽螺Pomacea canaliculata是我國(guó)南方重要的外來入侵有害生物,目前已在江蘇蘇州的河道、湖塘等水體中廣泛發(fā)生,并呈現(xiàn)向北入侵趨勢(shì)。及時(shí)準(zhǔn)確地監(jiān)測(cè)小管福壽螺在水體中的入侵?jǐn)U散是有效防控其為害的關(guān)鍵。傳統(tǒng)觀察法受其生活史、發(fā)生狀況及環(huán)境等影響,難以在入侵早期及時(shí)監(jiān)測(cè)。新興的環(huán)境DNA-宏條形碼(environmental DNA metabarcoding)技術(shù)可實(shí)現(xiàn)對(duì)入侵生物快速、靈敏的監(jiān)測(cè)。本文分別采用環(huán)境DNA-宏條形碼技術(shù)和傳統(tǒng)觀察方法對(duì)蘇州地區(qū)河流、湖泊和運(yùn)河共38個(gè)樣點(diǎn)的小管福壽螺發(fā)生情況進(jìn)行了檢測(cè)。結(jié)果顯示,環(huán)境DNA-宏條形碼技術(shù)檢測(cè)到小管福壽螺的發(fā)生率(92.11%)遠(yuǎn)高于傳統(tǒng)觀察法(36.84%)。豐度閾值的設(shè)定和水體類型對(duì)環(huán)境DNA-宏條形碼技術(shù)的檢測(cè)結(jié)果有一定影響,本研究為今后運(yùn)用環(huán)境DNA-宏條形碼技術(shù)進(jìn)行福壽螺入侵監(jiān)測(cè)提供技術(shù)支持。
關(guān)鍵詞 :環(huán)境DNA(eDNA); 高通量測(cè)序; 小管福壽螺; 生物入侵
中圖分類號(hào):
S 40
文獻(xiàn)標(biāo)識(shí)碼: A
DOI: 10.16688/j.zwbh.2020389
Monitoring of the invasive species Pomacea canaliculata via environmental DNA metabarcoding in Suzhou city
CHEN Xiao1, FANG Jingyi1,2, WANG Meng1, SHEN Qing3, SUN Zhenjun3*, WANG Beixin1*
(1. College of Plant Protection, Nanjing Agricultural University, Nanjing 210095, China;
2. Wageningen University, Wageningen 6700 AK, The Netherlands; 3. Suzhou
Station of Plant Protection and Plant Quarantine, Suzhou 215008, China)
Abstract
Pomacea canaliculata is a dangerous invasive species in southern China. At present, P.canaliculata has widely occurred in rivers, lakes, and other water bodies in Suzhou, Jiangsu province, and shows a trend of northward invasion. Timely and accurate monitoring of its invasion and spreading in water bodies is the key to effectively prevent and control its harm. Owing to the effect of its life history, occurrence, and environment, traditional observation cannot monitor P.canaliculata in the early stage of invasion. The emerging environmental DNA metabarcoding technology shows rapid and sensitive monitoring of invasive organisms. In this paper, environmental DNA metabarcoding and traditional observation methods were used to detect the occurrence of P.canaliculata in 38 samples of rivers, lakes, and canals in Suzhou. The results showed that the detection rate of P.canaliculata by environmental DNA metabarcoding technique (92.11%) was much higher than that by traditional observation method (36.84%). The setting of abundance threshold and water body type have a certain impact on the detection results by environmental DNA metabarcoding technology. This study would provide technical support for the future use of this technology to monitor Pomacea.
Key words
environmental DNA; high-throughput sequencing; Pomacea canaliculata; biological invasion
外來物種入侵是重要的全球環(huán)境問題和生態(tài)安全問題[1]。入侵生物對(duì)農(nóng)、林、牧、漁業(yè)和交通航運(yùn)等產(chǎn)生直接或間接的破壞,造成重大的經(jīng)濟(jì)損失和生態(tài)安全問題。福壽螺Pomacea屬軟體動(dòng)物門Mollusca,腹足綱Gastropoda,瓶螺科Ampullariidae,是世界100種惡性外來入侵物種之一[2],具有適應(yīng)性強(qiáng)、生長(zhǎng)快、繁殖力高,食性雜且食量大等特點(diǎn),在局部區(qū)域?qū)λ尽④住⑸徟骸④蛯?shí)等農(nóng)作物造成嚴(yán)重危害。此外,福壽螺還是一些危害人體健康的寄生蟲的中間宿主,如卷棘口吸蟲Echinostoma revolutum和廣州管圓線蟲Angiostrongylus cantonensis等[3]。長(zhǎng)期以來福壽螺被認(rèn)為只有1種,即小管福壽螺Pomacea canaliculata,福壽螺通常也特指小管福壽螺,但現(xiàn)在通過分子生物學(xué)方法發(fā)現(xiàn)福壽螺屬包括多個(gè)種[4]。小管福壽螺自1981年引入廣東后,逐漸擴(kuò)散至浙江、江西、云南和四川等地,現(xiàn)已成為影響南方農(nóng)業(yè)生產(chǎn)的重要有害生物[5]。2003年中國(guó)國(guó)家環(huán)保總局將其列入首批16種外來入侵生物名單[6]。當(dāng)前,小管福壽螺作為外來物種已在江蘇蘇州定殖[7],在部分河道、湖塘發(fā)生量較大,并呈現(xiàn)逐漸向北(無錫、常州、鎮(zhèn)江、南京)擴(kuò)散的趨勢(shì)。入侵初期是防治入侵物種的重要時(shí)期,監(jiān)測(cè)預(yù)警是有效防控生物入侵的重要手段[1]。然而,小管福壽螺生活于水中,可潛藏在水底,非繁殖期有較強(qiáng)的隱蔽性[6],傳統(tǒng)的現(xiàn)場(chǎng)觀察和調(diào)查問卷法發(fā)現(xiàn)難度大,不利于早期預(yù)警。如何有效地開展早期監(jiān)測(cè)是當(dāng)前福壽螺防控中的重要工作內(nèi)容。
環(huán)境DNA(environmental DNA, eDNA)是指生物體經(jīng)由體表、尿液、糞便、黏液或細(xì)胞死亡裂解后等釋放到環(huán)境中的DNA片段[8]。隨著高通量測(cè)序技術(shù)的發(fā)展,DNA宏條形碼技術(shù)(DNA metabarcoding)可以從混合樣本中同時(shí)獲得更多的生物信息。環(huán)境DNA-宏條形碼技術(shù),即利用宏條形碼技術(shù)對(duì)環(huán)境樣本中分離的DNA進(jìn)行擴(kuò)增測(cè)序,可以同時(shí)鑒定混合樣本中多個(gè)分類單元[9](后文簡(jiǎn)稱為環(huán)境DNA技術(shù))。目前,環(huán)境DNA技術(shù)已成功應(yīng)用于入侵水生生物的監(jiān)測(cè),并展現(xiàn)出快速、靈敏等優(yōu)點(diǎn)。該技術(shù)可以在亞洲鯉魚入侵北美五大湖早期密度較低時(shí)檢測(cè)到其存在[1011]。Takahara等[12]發(fā)現(xiàn)環(huán)境DNA技術(shù)相比傳統(tǒng)觀察法能更準(zhǔn)確地反映入侵物種藍(lán)鰓太陽魚Lepomis macrochirus在日本的擴(kuò)散趨勢(shì)。此外,對(duì)云南梯田克氏原螯蝦Procambarus clarkia的環(huán)境DNA監(jiān)測(cè)結(jié)果也表明該技術(shù)的檢出率明顯高于傳統(tǒng)的誘捕法[13]。然而,當(dāng)前我國(guó)多數(shù)地區(qū)對(duì)福壽螺的發(fā)生監(jiān)測(cè)依然以走訪調(diào)查、現(xiàn)場(chǎng)觀察等為主[1416],尚未見利用環(huán)境DNA-宏條形碼技術(shù)進(jìn)行監(jiān)測(cè)的相關(guān)研究。入侵生物的早期預(yù)警需要在其豐度很低時(shí)監(jiān)測(cè)到其存在,因此低豐度序列的閾值篩選尤為重要。研究發(fā)現(xiàn),環(huán)境DNA在不同類型的水體中的富集也存在差異[17]。為此,還需明確篩選出的豐度閾值及各種環(huán)境因素對(duì)環(huán)境DNA技術(shù)監(jiān)測(cè)結(jié)果的影響。
本文旨在利用環(huán)境DNA技術(shù)監(jiān)測(cè)蘇州地區(qū)不同水體類型的38個(gè)樣點(diǎn)的小管福壽螺發(fā)生情況,探索環(huán)境DNA技術(shù)是否可以作為批量檢測(cè)入侵生物福壽螺的靈敏、高效的技術(shù),明確豐度閾值和水體類型對(duì)環(huán)境DNA技術(shù)檢測(cè)結(jié)果的影響,為今后運(yùn)用環(huán)境DNA-宏條形碼技術(shù)對(duì)福壽螺進(jìn)行監(jiān)測(cè)和早期預(yù)警提供技術(shù)參考。
1 材料與方法
1.1 研究區(qū)域和樣點(diǎn)設(shè)置
蘇州地區(qū)處于江蘇省南部,屬于長(zhǎng)江流域太湖水系,水系發(fā)達(dá),河網(wǎng)稠密,為福壽螺的繁殖和擴(kuò)散提供了良好條件[18]。本研究于2019年1月在蘇州市相城區(qū)、常熟市、張家港市及昆山市進(jìn)行水環(huán)境樣品及福壽螺生物樣品的采集,共設(shè)置了38個(gè)樣點(diǎn)(編號(hào)S01~S38),其中河流樣點(diǎn)24個(gè),湖泊樣點(diǎn)6個(gè)和運(yùn)河樣點(diǎn)8個(gè)(表1)。
1.2 環(huán)境DNA采集和福壽螺發(fā)生調(diào)查
采用過濾法收集水體中的環(huán)境DNA。每個(gè)樣點(diǎn)設(shè)3個(gè)采樣位置,各位置相隔5 m,每個(gè)位置在30~50 cm水深處用采樣瓶各采集500 mL水樣,合計(jì)1.5 L。將玻璃微纖維濾膜(GF/F,孔徑0.7 μm)放置于過濾裝置(Nalgene 300-4100過濾器)中,隨后將充分搖勻的水樣通過過濾裝置進(jìn)行過濾。水樣過濾后用鑷子將濾膜放入離心管中,并加入無水乙醇置于4℃移動(dòng)冰箱短期保存,帶回實(shí)驗(yàn)室后放在-20℃環(huán)境保存。每次過濾前用75%乙醇和超純水充分沖洗過濾裝置,以防止各樣點(diǎn)交叉污染,所有儀器和材料在使用前均滅菌處理。
采用現(xiàn)場(chǎng)觀察法記錄采樣點(diǎn)周邊100 m范圍內(nèi)福壽螺的發(fā)生情況,觀察到有空殼、卵塊、活體的水域均認(rèn)為有福壽螺存在,并隨機(jī)采集10只福壽螺活體帶回實(shí)驗(yàn)室。
1.3 DNA提取與擴(kuò)增
將38個(gè)樣點(diǎn)分3組進(jìn)行環(huán)境DNA提取與擴(kuò)增,每組包括12(或13個(gè))樣點(diǎn)收集的樣品以及1個(gè)陽性對(duì)照和1個(gè)陰性對(duì)照。陽性對(duì)照(SP)為10頭沖洗干凈的福壽螺在1.5 L無菌水內(nèi)培養(yǎng)72 h后用過濾法收集的環(huán)境DNA樣本,使用純凈水過濾后的樣本作為陰性對(duì)照(SN)。另外選取4頭活體福壽螺,分別提取其組織DNA后測(cè)序獲得物種COⅠ序列,構(gòu)建本地福壽螺序列數(shù)據(jù)庫。試驗(yàn)結(jié)束后10頭福壽螺均用75%乙醇?xì)⑺啦⒆鳛闃?biāo)本保存。
環(huán)境DNA擴(kuò)增前首先用無菌剪刀將濾膜剪碎成約3 mm的碎片,使用上海生工Ezup柱式動(dòng)物基因組DNA抽提試劑盒(Ezup Column Animal Genomic DNA Purification Kit)從濾膜中提取環(huán)境DNA。采用上游引物mlCOlintF:5′-GGWACWGGWTGAACWGTWTAYCCYCC-3′和下游引物jgHCO2198:5′-TANACYTCNGGRTGNCCRAARAAYCA-3′(用N替代原序列中的I)對(duì)樣品的環(huán)境DNA進(jìn)行擴(kuò)增[19]。在每對(duì)引物上游、下游的5′-末端分別添加8個(gè)核苷酸長(zhǎng)度的標(biāo)簽,用以區(qū)分各個(gè)測(cè)序組中的每個(gè)樣品。PCR反應(yīng)體系為50 μL。反應(yīng)條件為94℃ 5 min;95℃ 10 s,62℃ 30 s(每次循環(huán)該過程都比上1個(gè)循環(huán)降低1℃),72℃ 60 s,16個(gè)循環(huán);95℃ 10 s,46℃ 30 s,72℃ 30 s,25個(gè)循環(huán);72℃ 5 min。測(cè)序平臺(tái)為Illumina Hiseq 2 500。
福壽螺組織DNA提取試劑盒同上,利用上游引物L(fēng)CO1490:5′-GGTCAACAAATCATAAAGATATTGG-3′和下游引物HCO2198: 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′進(jìn)行擴(kuò)增[20]。反應(yīng)條件為94℃ 1 min;94℃ 30 s,45℃ 90 s,72℃ 1 min,6個(gè)循環(huán);94℃ 30 s,51℃ 90 s,72℃ 1 min,36個(gè)循環(huán);72℃ 5 min。測(cè)序平臺(tái)為ABI 3730XL。
1.4 數(shù)據(jù)處理與分析
利用SEED軟件(Version 2.1.05)對(duì)測(cè)序獲得的原始數(shù)據(jù)進(jìn)行處理與分析,將原始數(shù)據(jù)成對(duì)導(dǎo)入并進(jìn)行拼接組裝[21]。篩選出質(zhì)量Q≥30的序列,在去除過長(zhǎng)和過短序列后,按照標(biāo)簽將序列分組為各個(gè)樣點(diǎn)數(shù)據(jù)。去除引物與標(biāo)簽不匹配的錯(cuò)誤序列,刪去嵌合體。對(duì)所得優(yōu)質(zhì)序列按照大于97%的相似度進(jìn)行可操作分類單元(operational taxonomic units, OTUs)聚類。將獲得的OTUs序列與NCBI上的數(shù)據(jù)庫進(jìn)行BLAST比對(duì)。為防止NCBI中發(fā)布的序列錯(cuò)誤或比對(duì)出現(xiàn)錯(cuò)誤,我們同時(shí)將OTUs與基于福壽螺組織DNA COⅠ序列構(gòu)建的本地序列數(shù)據(jù)庫進(jìn)行比對(duì),完成OTUs的物種信息注釋。將經(jīng)過處理分析后的信息匯總,得到各樣點(diǎn)中監(jiān)測(cè)到的福壽螺OTUs的豐度表,篩選其中一致性(similarity)=100%,覆蓋度(coverage)=100%的結(jié)果為有效的可操作分類單元。
為了評(píng)估低豐度序列的準(zhǔn)確性,我們?cè)O(shè)置了序列豐度閾值和相對(duì)豐度閾值兩種篩選方式,研究不同閾值設(shè)置對(duì)檢測(cè)結(jié)果的影響[22]。序列豐度(n)即每個(gè)OTU中各個(gè)樣點(diǎn)測(cè)得的序列數(shù)。相對(duì)豐度(m)是指某樣點(diǎn)的目標(biāo)OTU序列數(shù)占該樣點(diǎn)檢測(cè)到的所有OTUs序列總數(shù)的比例。將得到的物種序列數(shù)進(jìn)行標(biāo)準(zhǔn)化處理,通過lg(n+1)進(jìn)行對(duì)數(shù)轉(zhuǎn)換。所有的分析及作圖通過R語言(Version 4.0.1)完成。利用R語言中的“pheatmap包”對(duì)處理后的小管福壽螺序列及每個(gè)樣點(diǎn)的總序列數(shù)據(jù)進(jìn)行對(duì)比統(tǒng)計(jì),制作豐度熱圖。為評(píng)估相對(duì)序列豐度是否合適作為篩選物種存在的方式,將標(biāo)準(zhǔn)化處理的數(shù)據(jù)利用R語言中的“l(fā)m包”進(jìn)行線性擬合。為探討兩種方法檢測(cè)到的福壽螺發(fā)生率是否與水體類型有關(guān),我們進(jìn)一步分析了河流、湖泊、運(yùn)河中的福壽螺檢出率,即環(huán)境DNA技術(shù)檢測(cè)到福壽螺序列或現(xiàn)場(chǎng)觀測(cè)到福壽螺的樣點(diǎn)數(shù)與檢測(cè)總樣點(diǎn)數(shù)的比值。
2 結(jié)果與分析
2.1 現(xiàn)場(chǎng)觀察法和環(huán)境DNA-宏條形碼技術(shù)檢測(cè)結(jié)果
現(xiàn)場(chǎng)觀察法在38個(gè)樣點(diǎn)中的14個(gè)樣點(diǎn)觀察到小管福壽螺發(fā)生痕跡,其中活體、卵塊和空殼都觀察到的有2個(gè)樣點(diǎn)(S19、S23),觀察到卵塊和空殼的有4個(gè)樣點(diǎn)(S20、S21、S35、S36),僅觀察到卵塊或空殼的樣點(diǎn)數(shù)分別有6個(gè)(S08、S16、S31、S34、S37、S38)和2個(gè)(S12、S15)。
利用環(huán)境DNA-宏條形碼技術(shù)在38個(gè)樣點(diǎn)和6組對(duì)照中共得到21 973 306條DNA序列,其中福壽螺的序列數(shù)共111 317條。在36個(gè)樣點(diǎn)中檢測(cè)到4 852條福壽螺序列,陽性對(duì)照組(SP)檢測(cè)到106 465條福壽螺序列,陰性對(duì)照組(SN)未檢測(cè)到福壽螺序列,說明試驗(yàn)操作無污染。通過USEARCH聚類得到兩個(gè)OTU,經(jīng)比對(duì)及注釋,其GenBank序列號(hào)分別為EF514972和EU528586,均為小管福壽螺。其中36個(gè)樣點(diǎn)及3個(gè)陽性對(duì)照中共檢測(cè)到103 141條序列的注釋結(jié)果為EF514972,遠(yuǎn)高于注釋結(jié)果為EU528586的8 176條序列(表2,圖1)。此外,從現(xiàn)場(chǎng)采集的福壽螺中獲得的COⅠ序列經(jīng)BLAST比對(duì)及注釋后結(jié)果同樣為小管福壽螺,驗(yàn)證了環(huán)境DNA技術(shù)檢測(cè)到的物種信息結(jié)果準(zhǔn)確。
2.2 不同序列豐度閾值對(duì)檢測(cè)結(jié)果的影響
不同序列豐度閾值下小管福壽螺COⅠ條碼序列檢出率的結(jié)果(表3)表明,在序列豐度閾值n≥3和相對(duì)豐度閾值m≥0.001%兩種閾值下檢測(cè)到小管福壽螺序列的樣點(diǎn)數(shù)一致。其中,環(huán)境DNA技術(shù)檢測(cè)到小管福壽螺COⅠ序列的樣點(diǎn)數(shù)為35個(gè),占總調(diào)查樣點(diǎn)的92.11%,在14個(gè)現(xiàn)場(chǎng)調(diào)查到福壽螺個(gè)體的樣點(diǎn)中有13個(gè)檢測(cè)到小管福壽螺COⅠ序列。但隨著序列豐度閾值和相對(duì)豐度閾值的提高,檢出率隨之下降。當(dāng)序列豐度閾值n≥15時(shí),小管福壽螺COⅠ序列的檢出率僅為57.89%,14個(gè)調(diào)查到福壽螺個(gè)體樣點(diǎn)中僅有7個(gè)樣點(diǎn)(50%)檢測(cè)到小管福壽螺序列;而序列相對(duì)豐度閾值m≥0.030%時(shí),小管福壽螺COⅠ序列的檢出率僅為47.37%,14個(gè)調(diào)查到福壽螺個(gè)體樣點(diǎn)中僅有5個(gè)樣點(diǎn)(35.71%)檢測(cè)到小管福壽螺COⅠ序列。
基于38個(gè)樣點(diǎn)及對(duì)照組共44個(gè)樣本的總序列數(shù)及小管福壽螺序列數(shù)的線性擬合結(jié)果顯示,總序列數(shù)與小管福壽螺序列數(shù)間不存在線性關(guān)系(P>0.05,圖2),這表明基于序列相對(duì)豐度閾值m檢測(cè)到的小管福壽螺發(fā)生率不能夠真實(shí)地反映福壽螺的實(shí)際發(fā)生情況。結(jié)合表3的結(jié)果,本文在后續(xù)分析中,以序列豐度閾值n作為環(huán)境DNA技術(shù)檢測(cè)小管福壽螺發(fā)生的指標(biāo)。
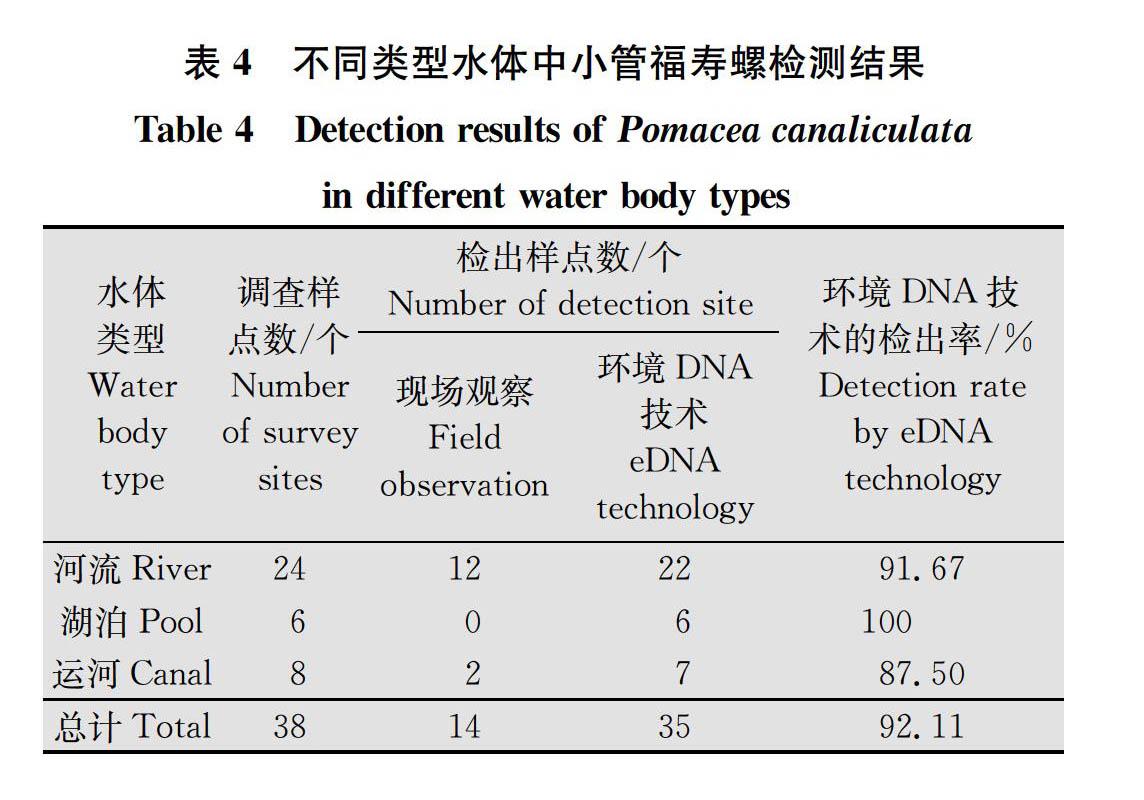
2.3 不同水體類型中小管福壽螺的檢測(cè)結(jié)果
當(dāng)序列豐度閾值n≥2與n≥3時(shí),從環(huán)境DNA中檢測(cè)到小管福壽螺的結(jié)果與現(xiàn)場(chǎng)觀察的結(jié)果一致(表3),且此時(shí)兩種方法檢測(cè)到小管福壽螺的樣點(diǎn)數(shù)均最多,現(xiàn)以序列豐度閾值n≥3作為小管福壽螺COⅠ條形碼檢出的閾值來計(jì)算其在不同水體類型中的檢出率(表4)。結(jié)果表明,湖泊中小管福壽螺檢出率最高,達(dá)到100%,河流和運(yùn)河檢出率分別為91.67%和87.50%。但是,現(xiàn)場(chǎng)觀察法并未在6個(gè)湖泊樣點(diǎn)中調(diào)查到小管福壽螺,而24個(gè)河流樣點(diǎn)中有12個(gè)樣點(diǎn)(50%)調(diào)查到小管福壽螺,8個(gè)運(yùn)河樣點(diǎn)中有2個(gè)樣點(diǎn)(25%)調(diào)查到小管福壽螺。綜上所述,38個(gè)調(diào)查樣點(diǎn)中,現(xiàn)場(chǎng)觀察法成功調(diào)查到小管福壽螺的共14個(gè)樣點(diǎn)(36.84%),遠(yuǎn)低于從環(huán)境DNA中檢測(cè)出小管福壽螺的35個(gè)樣點(diǎn)(92.11%)。
3 討論
目前我國(guó)報(bào)道有3種外來入侵福壽螺,即小管福壽螺、斑點(diǎn)福壽螺Pomacea maculata和Pomacea sp.,其中小管福壽螺與斑點(diǎn)福壽螺親緣關(guān)系近,形態(tài)與生活習(xí)性也相近,利用傳統(tǒng)分類方法很難區(qū)分二者[4]。基于DNA條形碼技術(shù)研究發(fā)現(xiàn),云南省、浙江省均已監(jiān)測(cè)到斑點(diǎn)福壽螺的入侵[4,23]。然而,此前的研究中,蘇州地區(qū)僅發(fā)現(xiàn)小管福壽螺,尚不確定是否有其他福壽螺入侵物種存在[24]。本文通過分子技術(shù)驗(yàn)證了蘇州地區(qū)現(xiàn)階段僅有小管福壽螺一種入侵物種存在。
針對(duì)入侵物種小管福壽螺的檢測(cè),環(huán)境DNA-宏條形碼技術(shù)相比傳統(tǒng)觀察法更加靈敏。在38個(gè)樣點(diǎn)中環(huán)境DNA技術(shù)在序列豐度閾值n≥1時(shí)檢測(cè)到小管福壽螺的樣點(diǎn)數(shù)有36個(gè),明顯高于現(xiàn)場(chǎng)觀察法(14個(gè))。當(dāng)序列豐度閾值設(shè)置為n≥3,環(huán)境DNA技術(shù)檢測(cè)到小管福壽螺的發(fā)生率(92.11%)遠(yuǎn)高于傳統(tǒng)觀察法(36.84%)。然而,利用環(huán)境DNA技術(shù)檢測(cè)也存在假陰性結(jié)果。現(xiàn)場(chǎng)觀察到小管福壽螺的14個(gè)樣點(diǎn)中S21樣點(diǎn)在現(xiàn)場(chǎng)觀察到福壽螺卵塊及空殼,但環(huán)境DNA技術(shù)并未檢測(cè)出,這可能與其在環(huán)境中的DNA含量少有關(guān),處于不同發(fā)育階段的生物其環(huán)境DNA釋放量存在差異[25],也有可能是生物信息分析過程中由于篩選標(biāo)準(zhǔn)高,序列被刪除而造成的假陰性結(jié)果[26]。
環(huán)境DNA技術(shù)中的生物信息學(xué)數(shù)據(jù)處理是影響結(jié)果的重要步驟。低豐度序列可能是人為因素或誤差產(chǎn)生的錯(cuò)誤序列,尤其是單體序列(singleton, n=1)[27]。設(shè)置豐度閾值是降低低豐度序列造成假陽性結(jié)果的一種常用方法[28]。但也不可否認(rèn)在檢測(cè)低豐度物種時(shí),低豐度序列可能反映了物種存在的重要信息[29]。在已有研究中,一般存在兩種篩選方法,一種是根據(jù)序列豐度閾值篩選[30],一種是根據(jù)序列相對(duì)豐度閾值[31]來去除低豐度序列的OTUs。本文研究了上述兩種豐度閾值對(duì)小管福壽螺檢測(cè)結(jié)果的影響,結(jié)果表明,從單個(gè)環(huán)境DNA樣本中檢測(cè)出小管福壽螺的序列數(shù)與總序列數(shù)沒有顯著相關(guān)性,而序列相對(duì)豐度閾值是基于總序列數(shù)計(jì)算得到的,說明基于序列相對(duì)豐度閾值m的結(jié)果并不能準(zhǔn)確反映小管福壽螺的入侵,針對(duì)低豐度生物的檢測(cè)時(shí)采取序列豐度閾值可能更合適。小管福壽螺序列數(shù)為n=1的樣點(diǎn)只有S22,在該樣點(diǎn)我們并未觀察到其物種存在痕跡,所以我們認(rèn)為該結(jié)果為假陽性結(jié)果,即與實(shí)際不相符。本研究中,序列豐度閾值為n≥3時(shí)利用環(huán)境DNA技術(shù)在35個(gè)樣點(diǎn)中檢測(cè)到小管福壽螺COⅠ條形碼。關(guān)于n值多少為最佳,針對(duì)不同物種閾值設(shè)置是否不同,仍需要進(jìn)一步研究。
環(huán)境DNA技術(shù)和現(xiàn)場(chǎng)觀察法在不同水體類型中的檢測(cè)結(jié)果也存在差異。在所有的湖泊樣點(diǎn)中,環(huán)境DNA技術(shù)都檢測(cè)到了小管福壽螺,這與我們前期在這些湖泊中發(fā)現(xiàn)有小管福壽螺分布的事實(shí)相符,而傳統(tǒng)觀察法在湖泊水體并未觀測(cè)到小管福壽螺。湖泊水體在監(jiān)測(cè)時(shí)受水深、生物習(xí)性等因素影響,而傳統(tǒng)觀察及捕撈多在沿岸區(qū)域進(jìn)行,底棲性的小管福壽螺在監(jiān)測(cè)時(shí),極有可能難以發(fā)現(xiàn)。在運(yùn)河水體中,利用環(huán)境DNA技術(shù)的檢出率(87.50%)也高于傳統(tǒng)觀察法(25%)。相比傳統(tǒng)觀察法只在河流水體中有較高的檢出率不同,環(huán)境DNA技術(shù)在3種水體類型中都有較高的檢出率,表明該方法受水體類型的影響較小。
本文的研究結(jié)果表明環(huán)境DNA技術(shù)對(duì)入侵水生生物的監(jiān)測(cè)有重要的應(yīng)用價(jià)值,該技術(shù)可以高效敏捷地監(jiān)測(cè)小管福壽螺的入侵分布。雖然該技術(shù)仍存在低豐度物種檢出率低,且不能定量的不足[32],但環(huán)境DNA技術(shù)具有可對(duì)大量的混合樣本快速檢測(cè),受時(shí)間和環(huán)境因素影響小,對(duì)生物和環(huán)境近乎無傷,操作簡(jiǎn)單便捷,省時(shí)省力靈敏度高等優(yōu)點(diǎn)。相比傳統(tǒng)觀察法,環(huán)境DNA-宏條形碼技術(shù)可在福壽螺入侵初期進(jìn)行監(jiān)測(cè),且受水體類型影響小。相信隨著環(huán)境DNA的獲取及數(shù)據(jù)分析方法的標(biāo)準(zhǔn)化及優(yōu)化,其必將成為未來入侵水生生物檢測(cè)的優(yōu)選方法。
參考文獻(xiàn)
[1] RICHARD N M, DANIEL S W, MARK L, et al. Biotic invasions: causes, epidemiology, global consequences, and control [J]. Ecological Applications, 2000, 10(3): 689710.
[2] LOWE S, BROWNE M, BOUDJELAS S, et al. 100 of the world’s worst invasive alien species: a selection from the global invasive species database[C]∥Invasive Species Specialist Group of the World Conservation Union. Auckland, New Zealand: ISSG Publications, 2000.
[3] 李小慧, 胡隱昌, 宋紅梅, 等. 中國(guó)福壽螺的入侵現(xiàn)狀及防治方法研究進(jìn)展[J]. 中國(guó)農(nóng)學(xué)通報(bào), 2009, 25(14): 229232.
[4] 楊倩倩, 劉蘇汶, 茹煒崠, 等. 基于DNA條形碼技術(shù)對(duì)浙江省外來入侵福壽螺進(jìn)行分子鑒定[J]. 生物多樣性, 2016, 24(3), 341350.
[5] 周宇, 袁雪穎, 楊子軒, 等. 福壽螺入侵中國(guó)的擴(kuò)散動(dòng)態(tài)及潛在分布[J]. 湖泊科學(xué), 2018, 30(5): 13791387.
[6] 中國(guó)環(huán)境保護(hù)總局. 中國(guó)第一批外來入侵物種名單[EB/OL]. (20030110) [20200704]. http:∥www.mee.gov.cn/gkml/zj/wj/200910/t20091022_172155.htm.
[7] 查國(guó)賢, 李俊, 沈晴, 等. 蘇州市農(nóng)業(yè)植物檢疫性有害生物發(fā)生現(xiàn)狀及防控對(duì)策[J]. 植物檢疫, 2018, 32(5): 7579.
[8] TABERLET P, COISSAC E, HAJIBABAEI M, et al. Environmental DNA [J]. Molecular Ecology, 2012, 21(8): 17891793.
[9] 陳煉, 吳琳, 劉燕, 等. 環(huán)境DNA metabarcoding及其在生態(tài)學(xué)研究中的應(yīng)用[J]. 生態(tài)學(xué)報(bào), 2016, 36(15): 45734582.
[10]JERDE C L, MAHON A R, CHADDERTON W L, et al. ‘Sight-unseen’ detection of rare aquatic species using environmental DNA[J]. Conservation Letters, 2011, 4(2): 150157.
[11]JERDE C L, CHADDERTO W L, MAHON A R, et al. Detection of Asian carp DNA as part of a Great Lakes basin-wide surveillance program [J]. Canadian Journal of Fisheries and Aquatic Sciences, 2013, 70(4): 522526.
[12]TAKAHARA T, MINAMOTO T, YAMANAKA H, et al. Estimation of fish biomass using environmental DNA [J/OL]. PLoS ONE, 2012, 7(4): e35868. DOI: 10.1371/journal. pone.0035868.
[13]馬竹欣. 利用環(huán)境DNA技術(shù)調(diào)查入侵種克氏原螯蝦在元陽梯田的分布[D]. 昆明: 云南大學(xué), 2016.
[14]劉雨芳, 李菲, 李玉峰, 等. 福壽螺在湖南的分布現(xiàn)狀、危害與擴(kuò)散風(fēng)險(xiǎn)預(yù)警[J]. 水生生物學(xué)報(bào), 2011, 35(6): 10671071.
[15]周兵, 謝春揚(yáng)迪, 閆小紅, 等. 江西省福壽螺的入侵危害現(xiàn)狀及稻田發(fā)生規(guī)律[J]. 生態(tài)與農(nóng)村環(huán)境學(xué)報(bào), 2015, 31(6): 902909.
[16]劉義滿, 戢小梅, 李長(zhǎng)林, 等. 湖北省福壽螺分布調(diào)查[J]. 長(zhǎng)江蔬菜, 2020(6): 5765.
[17]FEDIAJEVAITE J, PRIESTLEY V, ARNOLD R, et al. Meta-analysis shows that environmental DNA outperforms traditional surveys, but warrants better reporting standards [J]. Ecology and Evolution, 2021, 11(9): 48034815.
[18]蘇州市統(tǒng)計(jì)局. 蘇州統(tǒng)計(jì)年鑒—2019[M]. 北京: 中國(guó)統(tǒng)計(jì)出版社, 2019: 45.
[19]LERAY M, YANG J Y, MEYER C P, et al. A new versatile primer set targeting a short fragment of the mitochondrial COⅠ region for metabarcoding metazoan diversity: application for characterizing coral reef fish gut contents [J]. Frontiers in Zoology, 2013, 10(1): 34. DOI: 10.1186/1742-9994-10-34.
[20]FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates [J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294299.
[21]VETROVSKY T, BALDRIAN P, MORAIS D. SEED 2: a user-friendly platform for amplicon high-throughput sequencing data analyses [J]. Bioinformatics, 2018, 34(13): 22922294.
[22]ZHAN Aibin, XIONG Wei, HE Song, et al. Influence of artifact removal on rare species recovery in natural complex communities using high-throughput sequencing [J/OL]. PLoS ONE, 2014, 9(5): e96928. DOI: 10.1371/journal.pone.0096928.
[23]冉甄. 基于線粒體COⅠ基因?qū)υ颇鲜〔煌低鈦砣肭稚锔勐莸南到y(tǒng)發(fā)育學(xué)研究[D].昆明: 昆明醫(yī)科大學(xué), 2019.
[24]YANG Qianqian, LIU Suwen, HE Chao, et al. Invisible apple snail invasions: importance of continued vigilance and rigorous taxonomic assessments [J]. Pest Management Science, 2019, 75(5): 12771286.
[25]CHRISTOPHER R T, MICHAEL J S, JEN N, et al. Seasonality, DNA degradation and spatial heterogeneity as drivers of eDNA detection dynamics [J/OL]. Science of The Total Environment, 2021, 768: 144466. DOI: 10.1016/j.scitotenv.2020.144466.
[26]FURLAN E M, DAVIS J, DUNCAN R P. Identifying error and accurately interpreting eDNA metabarcoding results: A case study to detect vertebrates at arid zone waterholes [J]. Molecular Ecology Resources, 2020, 20(5): 12591276.
[27]KUNIN V, ENGELBREKTSON A, OCHMAN H, et al. Wrinkles in the rare biosphere: pyrosequencing errors can lead to artificial inflation of diversity estimates [J]. Environmental Microbiology, 2010, 12(1): 118123.
[28]GIGUET-COVEX C, PANSU J, ARNAUD F, et al. Long livestock farming history and human landscape shaping revealed by lake sediment DNA [J/OL]. Nature Communications, 2014, 5: 4211. DOI: 10.1038/ncomms4211.
[29]ZHAN Aibin, HULK M, SYLVESTER F, et al. High sensitivity of 454 pyrosequencing for detection of rare species in aquatic communities [J]. Methods in Ecology and Evolution, 2013, 4(6): 558565.
[30]CAREW M E, PETTIGROVE V J, METZELING L, et al. Environmental monitoring using next generation sequencing: rapid identification of macroinvertebrate bioindicator species [J/OL]. Frontiers in Zoology, 2013, 10(1): 45. DOI: 10.1186/1742-9994-10-45.
[31]ELBRECHT V, LEESE F. Can DNA-based ecosystem assessments quantify species abundance? Testing primer bias and biomass-sequence relationships with an innovative metabarcoding protocol [J/OL]. PLoS ONE, 2015, 10(7): e0130324. DOI: 10.1371/journal.pone.0130324.
[32]李杰, 楊婧, 黃原. 應(yīng)用DNA復(fù)合條形碼技術(shù)研究秦嶺水生動(dòng)物多樣性[J]. 生態(tài)學(xué)報(bào), 2016, 36(19): 61036112.
(責(zé)任編輯:楊明麗)

