山西蘋果褪綠葉斑病毒的分布及遺傳多樣性分析
續海紅 王燕飛 竇彥鑫 陳偉 楊凱
摘要 :蘋果褪綠葉斑病毒Apple chlorotic leaf spot virus (ACLSV) 是侵染蘋果的主要潛隱性病毒之一,在我國蘋果植株上發生普遍,嚴重威脅我國蘋果的品質與產量。本研究從山西省12個蘋果主產區隨機采集360份表現褪綠和斑駁等癥狀的蘋果葉片作為研究樣本,通過RT-PCR檢測,360份樣本中有209份樣本為ACLSV陽性,對209份陽性樣本的外殼蛋白(coat protein, CP)基因進行分離、測序、克隆,得到12個新的ACLSV分離物(分別命名為 Shanxi 1~Shanxi 12)。選擇17個來自不同國家的分離物與12個新的ACLSV分離物在核苷酸和氨基酸層面上進行序列一致性和系統發育分析。結果顯示,29個ACLSV分離物被劃分為2個不同進化群體。進一步對2個不同ACLSV群體進行選擇壓分析和中性檢驗,結果表明,組Ⅰ與組Ⅱ的ACLSV群體之間存在明顯的遺傳差異,其中負向選擇可能是ACLSV遺傳變異的原因之一。本研究較全面地分析了ACLSV的發生、危害,并對山西蘋果的ACLSV分離物進行了遺傳結構分析,為山西蘋果褪綠葉斑病毒病的防治提供了理論指導。
關鍵詞 :山西蘋果; 隱性病毒; 蘋果褪綠葉斑病毒; 外殼蛋白; 遺傳變異
中圖分類號:
S 432.41
文獻標識碼: A
DOI: 10.16688/j.zwbh.2020578
Analysis on distribution and genetic diversity of Apple leaf spot virus from Shanxi province
XU Haihong1, WANG Yanfei2, DOU Yanxin1, CHEN Wei2*, YANG Kai1,3
(1.Shanxi Key Laboratory of Germplasm Improvement and Utilization in Pomology, Institute of Pomology,
Shanxi Agricultural University, Taiyuan 030031, China; 2.School of Life Science, Shanxi Normal University,
Linfen 041000, China; 3. Institute of Cotton Research, Shanxi Agricultural University, Yuncheng 044000, China)
Abstract
Apple chlorotic leaf spot virus (ACLSV) is one of the main recessive viruses infecting apples.It occurs widely on apple plants in China, posing a serious threat to the quality and yield of apples in China.In this study, 360 samples of apple leaves with symptoms such as fading and mottling were randomly collected from 12 major apple production areas in Shanxi province.Based on RT-PCR test results, 209 out of 360 samples showed ACLSV-positive.The CP genes of 12 new ACLSV isolates, named as Shanxi 1—Shanxi 12, were isolated, sequenced and cloned from these positive samples.Phylogenetic analysis was performed based on the nucleotide and amino acid sequences of the CP genes of 29 ACLSV isolates (17 were obtained online).The results showed that these isolates were divided into two distinct evolutionary populations, with a possible relation to the region and host.The genetic diversity of different ACLSV populations was further studied.Selective pressure analysis and neutrality test showed that these isolates had significant genetic differences between group Ⅰ and group Ⅱ.Negative selection might play an important role in the genetic variation of ACLSV.The knowledge on the occurrence, damage, genetic structure and evolutionary mechanism of Shanxi ACLSV is useful for the design of sustainable management strategies for controlling ACLSV.
Key words
apple; recessive viruses; Apple chlorotic leaf spot virus; coat protein; genetic diversity
蘋果是世界四大水果之一,廣泛分布在許多國家和地區,如中國、歐洲、日本、澳大利亞和美國。中國作為最大的蘋果生產和消費國,天然的地理環境形成了很多蘋果優勢種植區,蘋果的種植面積和產量、人均占有量和出口量等均居世界前列[12],已成為農民增收,脫貧致富的重要支柱產業[34]。近些年隨著黃土高原優勢產區面積增加,渤海灣產區果園更新及西南冷涼高地新型果園的崛起,蘋果病毒病成為制約果園發展的首要病害,其中蘋果褪綠葉斑病毒病發生最為普遍,嚴重影響了蘋果的產量和質量以及果農的經濟效益[5]。目前蘋果病毒病缺乏有效的防治方法,因此蘋果病毒病的系統研究對蘋果產業的健康發展具有重要意義。
蘋果病毒病在世界范圍內廣泛傳播。目前已報道侵染蘋果的病毒有39種,中國已鑒定的蘋果病毒有17種[68]。蘋果病毒分為潛隱性病毒和非潛隱性病毒,其中蘋果莖溝病毒Apple stem grooving virus (ASGV)、蘋果褪綠葉斑病毒Apple chlorotic leaf spot virus (ACLSV)、蘋果莖痘病毒Apple stem pitting virus (ASPV)是蘋果上報道的最主要的3個潛隱性病毒[911]。ACLSV是侵染蘋果的主要潛隱性病毒之一,屬于乙型線形病毒科Betaflexiviridae,纖毛病毒屬Trichovirus[12]。ACLSV為正義單鏈RNA病毒,病毒粒子呈長線狀或絲狀,基因組大小約7 545~7 555 nt,5′端帶有帽狀結構, 3′端有一個Poly(A)尾,編碼3個開放式閱讀框(open reading frames, ORFs)[1315]。ORF1編碼蛋白大小為1 877~1 887 aa,分子量約為216 kDa。具有依賴RNA的RNA聚合酶(Pol)、類木瓜蛋白酶(P-Pro)、解旋酶(Hel)和甲基轉移酶(Mt)活性,在ACLSV復制過程中發揮重要功能[16]。 ORF2編碼運動蛋白(movement protein, MP),大小為446~460 aa,分子量約為50 kDa。參與ACLSV在細胞間的運動 [17]。ORF3編碼ACLSV的外殼蛋白(coat protein, CP)[18],大小為193 aa,分子量約為22 kDa。CP蛋白是重要的結構蛋白,序列相對保守, N端高度可變,是ACLSV株系劃分的重要依據[1920]。
本研究于2015年-2016年在山西省12個蘋果主產區隨機采集360份表現為褪綠和斑駁等癥狀的新鮮蘋果葉片。對ACLSV發生危害、分子變異等情況進行系統性調查和鑒定,了解ACLSV在山西蘋果主產區的遺傳與流行規律。此外,本研究借助生物學與生物信息學對分離獲得的山西ACLSV全長CP基因進行遺傳結構和分子變異分析,為山西ACLSV的防治提供參考。
1 材料與方法
1.1 植物樣品采集
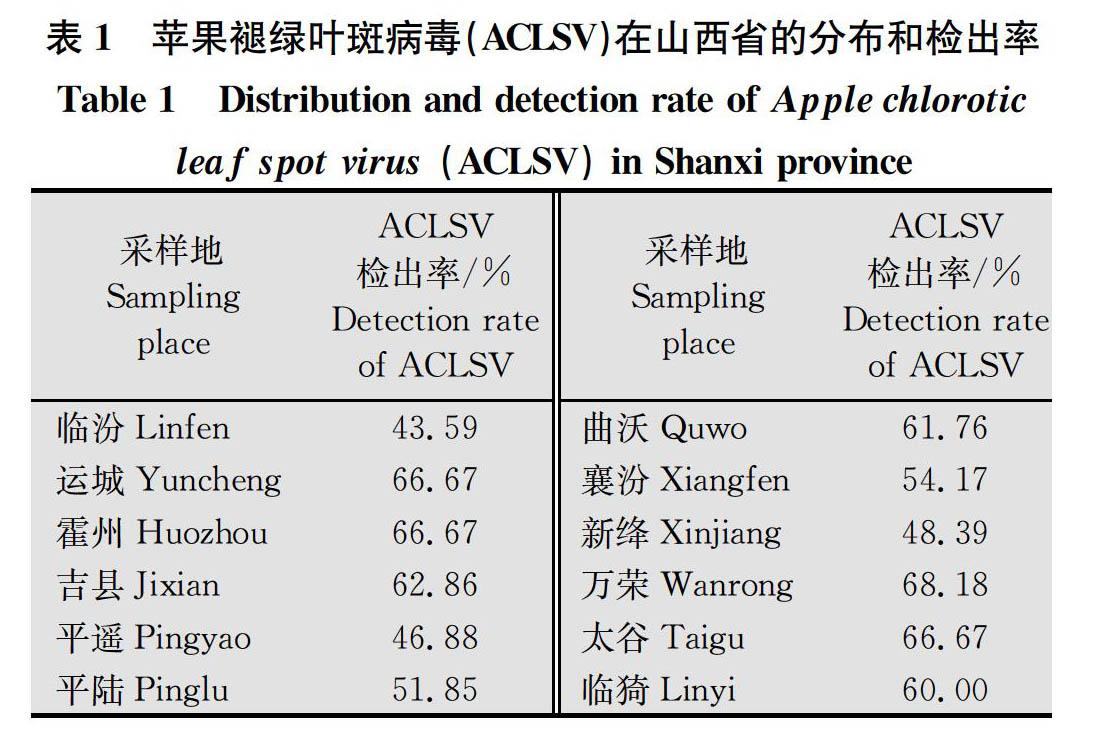
2015年和2016年的4月-6月,在山西省12個蘋果主產區(臨汾市堯都區、運城市區、霍州、吉縣、臨猗、平遙、平陸、曲沃、太谷、萬榮、襄汾、新絳)開展ACLSV病毒病調查,采集360份表現為褪綠或斑駁等癥狀的新鮮蘋果葉片,迅速放入冰盒,在24 h內送實驗室分析。
1.2 ACLSV引物設計
根據GenBank數據庫中的ACLSV(登錄號為 KC935956)序列中6727-6746位核苷酸設計引物ACLSV-CPF(5′-GAGARTTTCAGTTTGCTMGR-3′,M=A/C;R=A/G),7519-7543位核苷酸設計引物ACLSV-CPR(5′-AGTCTACAGGCTATTTATTATAAGT-3′),用于ACLSV檢測和全長CP基因克隆。目標片段長度為817 bp。
1.3 RNA提取和RT-PCR擴增與測序
使用植物總RNA提取試劑盒(Tiangen公司)提取蘋果葉片樣品的總RNA。使用Quant cDNA 第一鏈合成試劑盒(Tiangen公司)合成cDNA。以cDNA為模板進行PCR擴增,反應程序:94℃預變性3 min;94℃變性1 min,52℃退火1 min,72℃延伸2 min,35個循環; 72℃ 延伸10 min,4℃保存。反應體系為25 μL,其中,cDNA 2 μL,2×PCR Master Mix 12.5 μL,10 μmol/L正、反向引物各1 μL,ddH2O 8.5 μL。2%瓊脂糖凝膠電泳檢測RT-PCR擴增產物,使用瓊脂糖凝膠純化試劑盒(CW2302M,康維試劑)純化并回收陽性擴增條帶,ACLSV回收產物與PMD19-T載體16℃過夜連接,連接產物在水浴鍋中42℃熱激90 s轉化大腸桿菌 DH-5α,LB培養基(含有50 μg/mL Amp)上篩選陽性菌落,經PCR鑒定陽性的菌液送擎科生物技術有限公司測序。每個ACLSV陽性樣本至少進行3次重復測序。
1.4 山西ACLSV群體的遺傳多樣性分析
對本研究獲得的山西ACLSV的CP基因序列在NCBI上進行比對分析,獲得17條來自不同國家和寄主上的,與本研究序列相似的ACLSV CP基因序列。采用SDTv 1.2軟件[20]計算核苷酸序列和氨基酸序列的一致性。使用MEGA 7.0軟件[21]對本研究分離并克隆的山西ACLSV CP基因進行遺傳變異分析。基于山西ACLSV分離物CP基因核苷酸序列,通過最大似然法(construct/test maximum likelihood tree)中的general time reversible模型構建系統發育樹,自展值設定為1 000,低于50%的進化分支在本研究被自動舍棄。
1.5 山西ACLSV種群特征與遺傳學分析
借助DnaSP 5.10.01 軟件,基于CP基因的核苷酸序列,進行山西 ACLSV種群遺傳與進化分析,計算參數按照軟件設定參數進行[22]。
同時,計算Tajima’s D、Fu & Li’s D 和Fu & Li’s F 參數,對本研究獲得的山西ACLSV分離物進行中性檢驗[23]。使用DnaSP 5.0計算山西ACLSV分離物CP基因非同義突變頻率(Ka)和同義突變頻率(Ks)的比值,進行選擇壓分析,若Ka/Ks=1,表示這些群體處在中性或者近似中性選擇中;Ka/Ks<1,群體處在負向選擇中;如果Ka/Ks>1,群體處在正向選擇中。
1.6 山西ACLSV種群重組分析
基于CP基因的核苷酸序列,利用重組檢測軟件RDP 4.31自帶的7種檢測方法(MaxChi、RDP、SiScan、BootScan、Chimaera、GENECONV和3Seq)對ACLSV不同分離物重組和重組斷點進行自動識別,若至少4種不同方法檢測到同一重組事件,且P<10-6,則該事件被認定為陽性重組事件[2425]。
1.7 山西ACLSV種群差異檢驗和基因流分析
利用DnaSP 5.10.01 軟件,基于CP基因的核苷酸序列,分別計算反映山西ACLSV種群差異的5個檢驗值(Kst*、Ks*、Snn、 Z* 和Fst)[26]。通常在沒有遺傳分化的情況下,Kst*、Ks*、Snn、 Z*的值接近于0。通過1 000次檢驗,4個檢驗值(Kst*、Snn 、Ks*、Z*)均有統計學意義則能反映ACLSV種群遺傳差異情況[27]。Fst作為重要的基因分化系數,常被用來衡量種群間多樣性程度。一般來說,未分化至完全分化群體的Fst絕對值范圍為0~1。Fst的絕對值>0.33,表明基因流動不頻繁;Fst的絕對值<0.33,表明基因流動頻繁[28]。
2 結果與分析
2.1 山西ACLSV發生情況
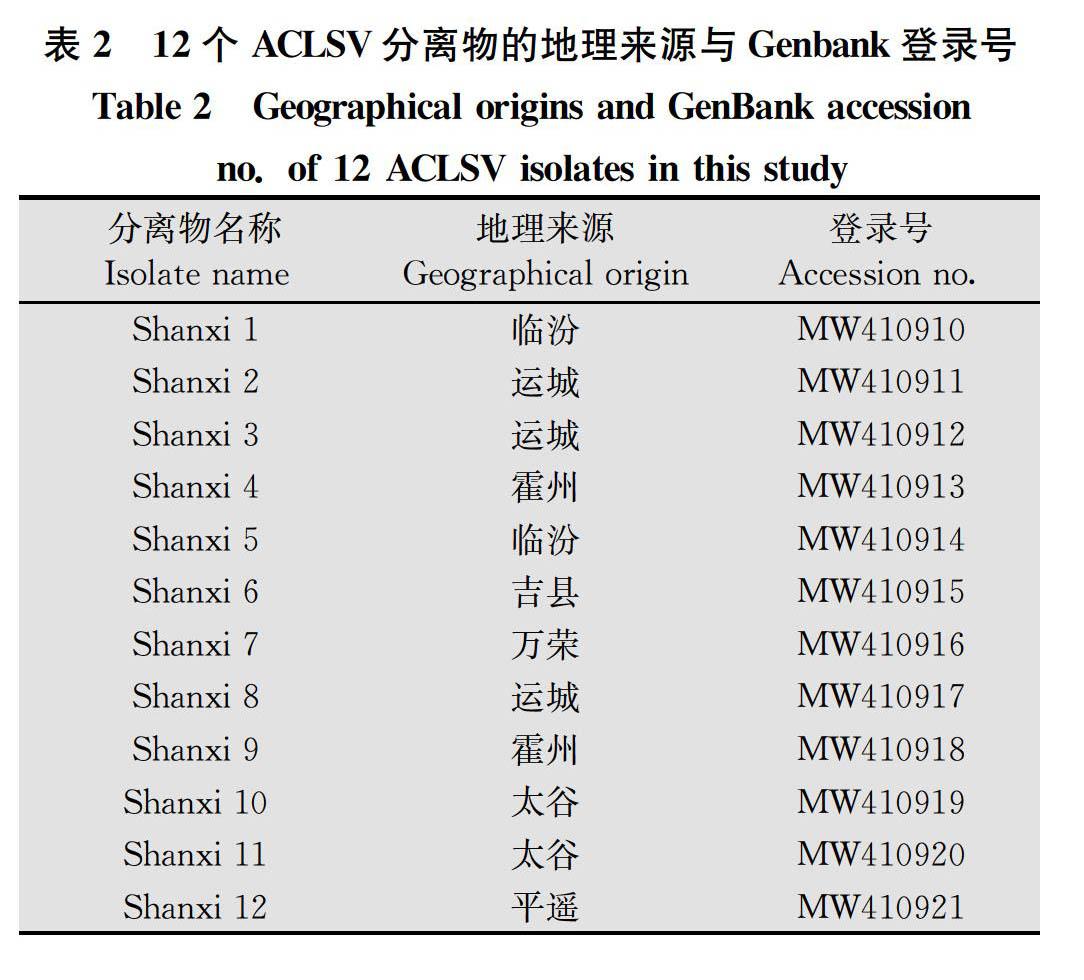
本研究在山西省12個蘋果主產區隨機采集了360份具有ACLSV特征的葉片樣本, 通過RT-PCR檢測發現其中有209份樣本被ACLSV浸染,各采樣點檢出率為43.59%~68.18%,平均檢出率為58.06%(表1)。萬榮地區檢出率最高,為68.18%,其余依次為運城、霍州、太谷、吉縣、曲沃、臨猗、襄汾、新絳、平遙和臨汾。臨汾的檢出率最低,為43.59%。對209份樣品的RT-PCR產物回收測序后,通過NCBI-BLAST分析(https:∥www.ncbi.nlm.nih.gov/),有197份樣品的RT-PCR產物核苷酸序列與已公布的ACLSV 的核苷酸和氨基酸序列一致性為100%,為已知的ACLSV分離物;而其余12份樣品RT-PCR產物核苷酸序列與已登錄的ACLSV分離物在核苷酸和氨基酸水平均有差異,為新的ACLSV分離物(Shanxi 1~Shanxi 12)(表2)。
2.2 山西 ACLSV 群體的遺傳多樣性分析
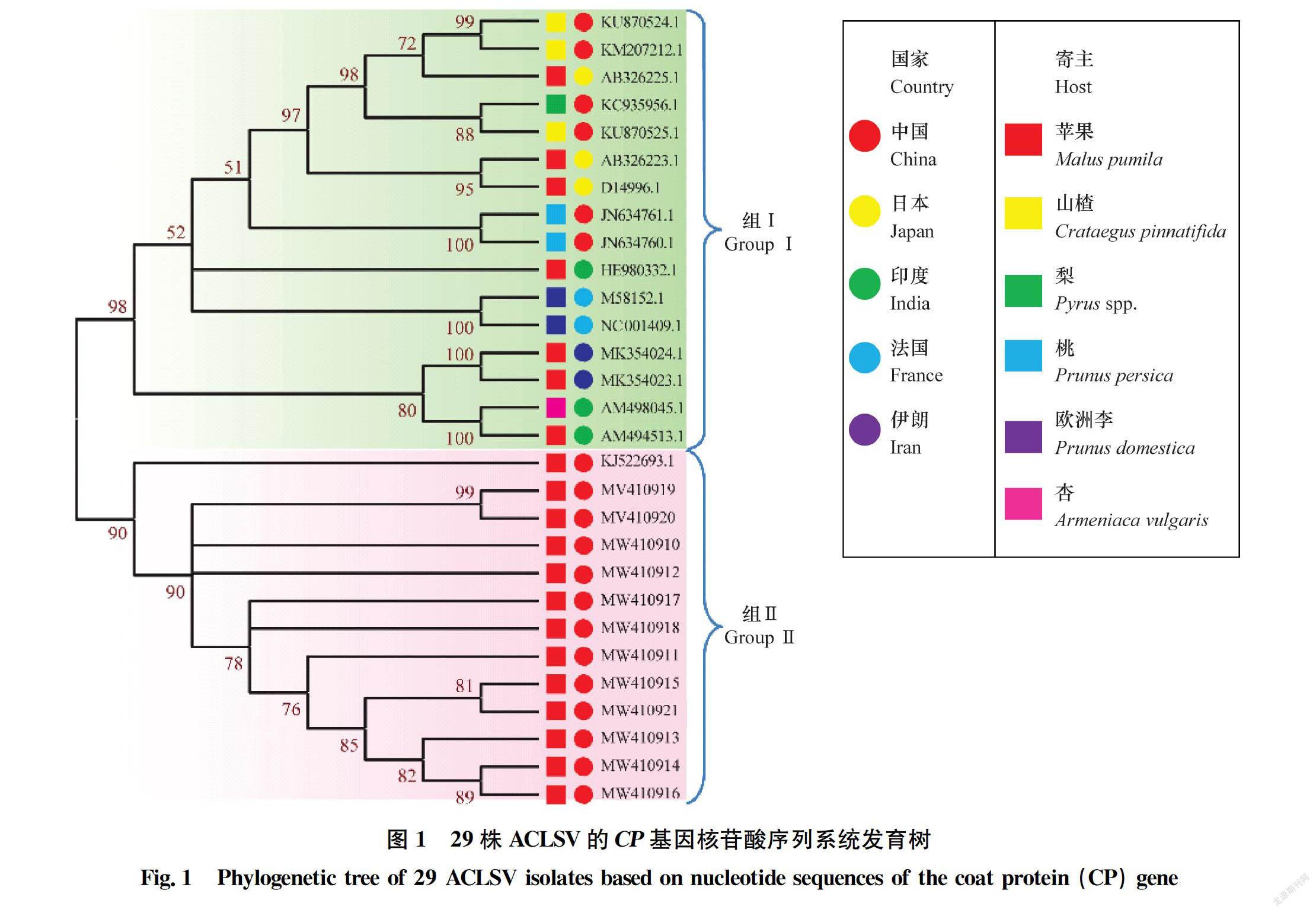
將來自NCBI數據庫的17個分離物與本研究分離的12個分離物,共計29個ACLSV分離物的CP基因進行了系統發育和遺傳變異分析。結果顯示,29個ACLSV分離物被分為2組(圖1)。其中,組Ⅰ包含16個分離物,這些分離物分別來自于5個國家的6個寄主。就國家而言,來自中國的分離物有6個,日本3個,印度3個,法國2個,伊朗2個。就寄主而言,分離自蘋果的有7個,山楂3個,桃子2個,李子2個,梨1個,杏1個。組Ⅱ包含13個ACLSV分離物,均是從中國的蘋果上分離獲得。包括本研究分離的12個山西ACLSV分離物與來自山東的分離物(KJ522693.1)。
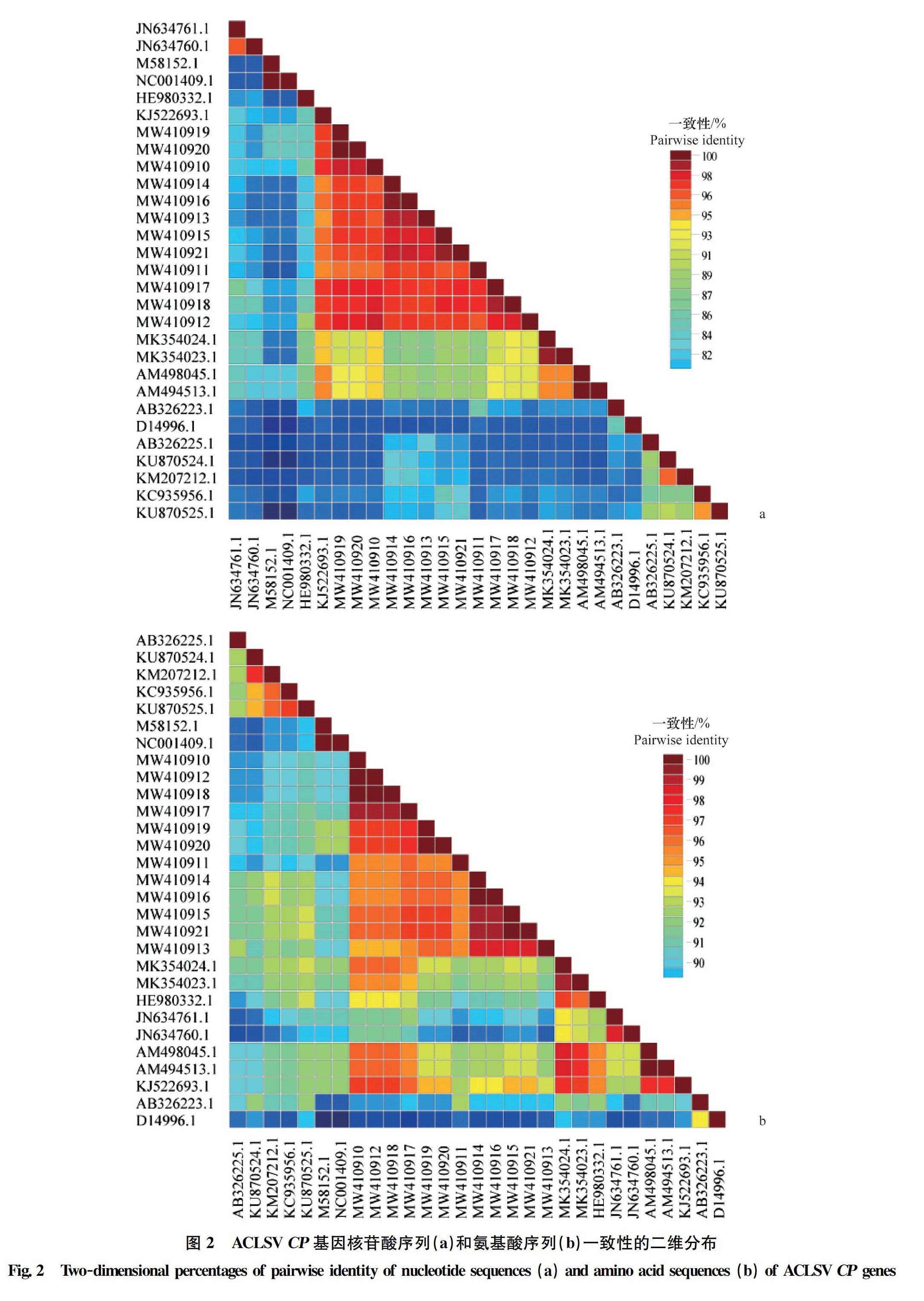
基于CP基因序列一致性分析表明,29個ACLSV分離物 CP基因核苷酸一致性為81.10%~100%,CP基因氨基酸一致性為88.60%~100%,這說明山西ACLSV中CP基因氨基酸在進化中具有較高保守性(圖2)。本研究獲得的山西分離物MW410910(Shanxi 1)與 KJ522693.1 的CP基因核苷酸一致性最高,為97.42%;MW410916(Shanxi 7)與D14996.1 序列的CP基因核苷酸一致性最低,為83.33%。其次,山西ACLSV分離物MW410910(Shanxi 1)、MW410912(Shanxi 3)和MW410918(Shanxi 9)與 KJ522693.1的氨基酸一致性最高,為97.93%;MW410917(Shanxi 8)與D14996.1序列的氨基酸一致性最低,為89.12%。
2.3 山西 ACLSV種群特征與變異情況
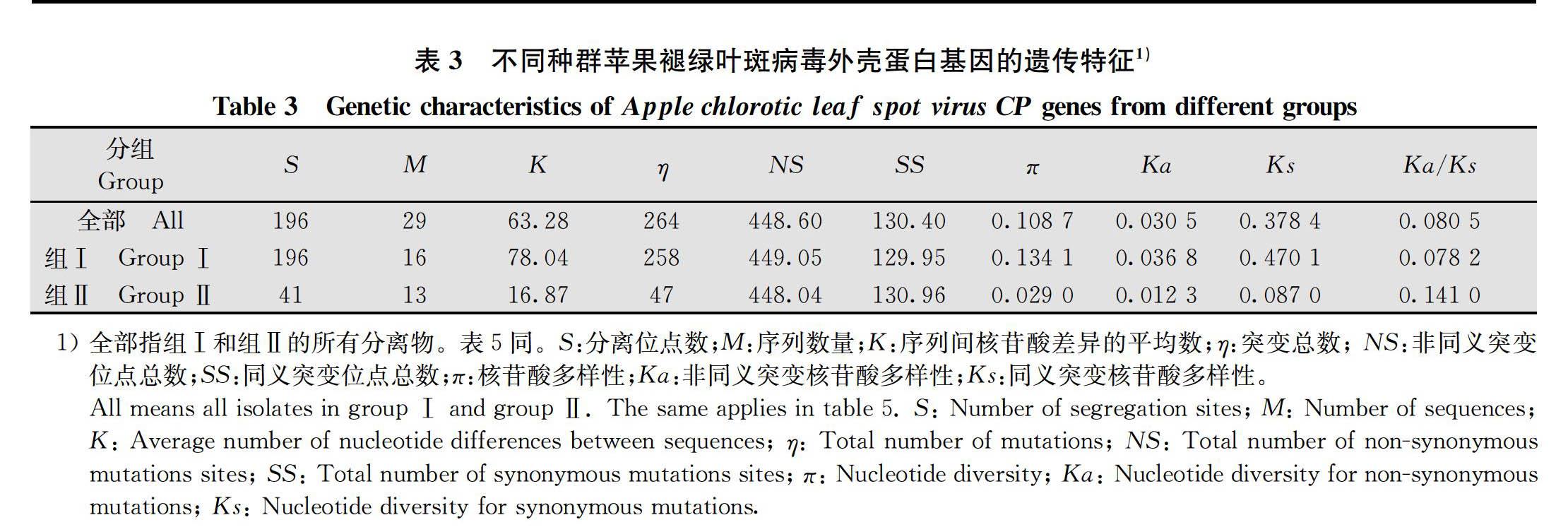
將來自于NCBI數據庫的17個分離物與本研究分離的12個分離物 共29個ACLSV 分離物按照系統發育關系分為2個組,并使用DnaSP 5.10.01軟件對ACLSV的種群特征進行分析。結果顯示,組Ⅰ的突變總數(η=258)、分離位點數(S=196)、序列間核苷酸差異的平均數(K=78.04)和核苷酸多樣性(π=0.134 1)都是最高的(表3)。隨后,對ACLSV CP基因的多態性進一步分析,結果顯示CP基因在1-50、201-250、326-375、401-450和501-550位點的多態性水平較高,對應Pi值分別為0.099 8、0.198 4、0.144 1、0.127 0、0.059和0.101 5(圖4a)。
2.4 山西ACLSV種群重組分析
重組是病毒遺傳變異的一個重要來源。為了研究重組在ACLSV種群進化中的作用,利用重組檢測軟件RDP 4.31對來自本研究的12個分離物和來自GenBank的 17個分離物的CP基因進行了重組事件檢測。檢測結果顯示,在29個ACLSV分離物中均未發現明顯的重組事件。
為了進一步分析ACLSV CP基因的變異和選擇壓,本研究計算了29個ACLSV分離物非同義突變位點和同義突變位點的比值(Ka/Ks)(表3)。結果顯示,ACLSV兩個系統發育分組的非同義突變頻率/同義突變頻率的比值(Ka/Ks)均小于1,表明ACLSV 的CP基因進化主要受負向選擇。進一步對2個進化枝的選擇壓差異進行分析,組Ⅰ和Ⅱ的Ka/Ks平均值分別為0.078 2和0.141 0(表3)。此外,組Ⅰ和組Ⅱ選擇壓的兩兩分析結果表明,組Ⅰ和組Ⅱ的Ka/Ks值分布存在差異(圖3b)。雖然ACLSV兩個組的Ka/Ks值均小于1,但是組Ⅱ的Ka/Ks值明顯高于組Ⅰ,表明組Ⅰ比組Ⅱ受到更強的負向選擇。
2.5 山西ACLSV的種群的差異檢驗和基因流分析
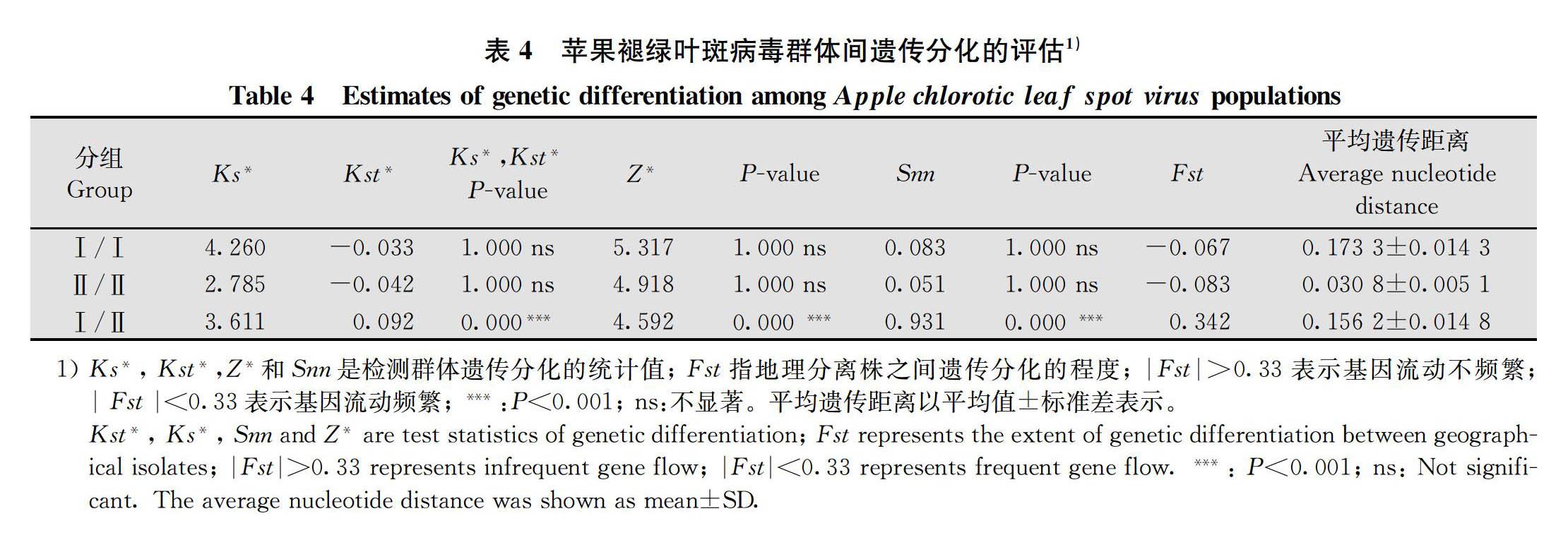
本研究評估了ACLSV種群組內與組間的遺傳分化程度。根據Ks*, Kst*, Z* 和 Snn的P值,我們發現組Ⅰ與組Ⅱ在遺傳分化上存在顯著差異,而組Ⅰ內和組Ⅱ內分離物之間沒有顯著差異。同時我們可以看到在組Ⅰ內和組Ⅱ內的|Fst|均小于0.33,組Ⅰ與組Ⅱ組間的|Fst|大于0.33。說明ACLSV分離物在組內的基因流動非常頻繁,組間的基因流動很少。進一步計算了組內與組間的遺傳距離,結果顯示,組Ⅰ內和組Ⅱ內分離物間的遺傳距離分別為(0.173 3±0.014 3)和(0.030 8±0.005 1),而組Ⅰ與組Ⅱ間的遺傳距離為(0.156 2±0.014 8)(表4)。
2.6 山西ACLSV中性檢驗和群體動態
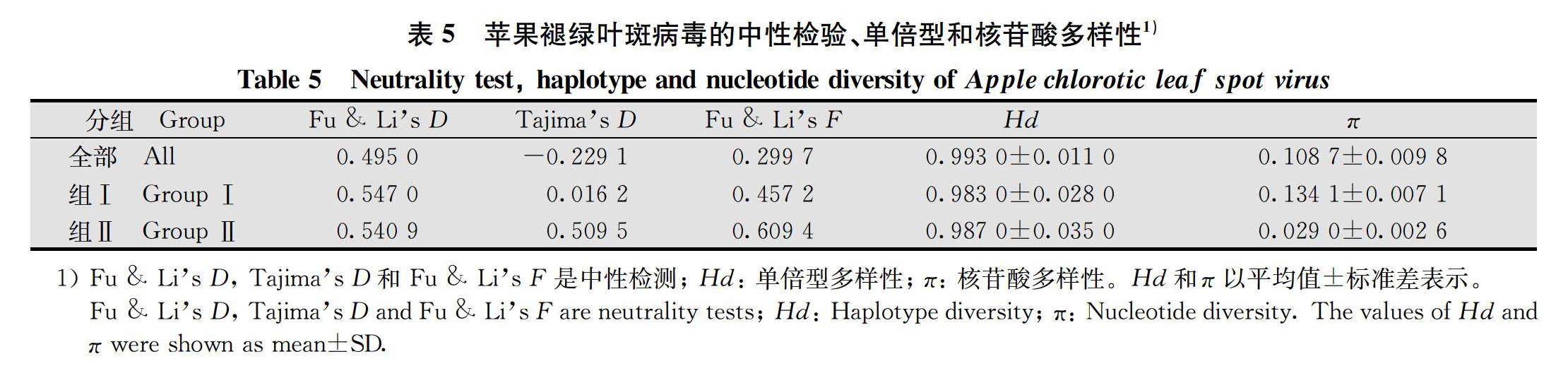
本研究基于Fu & Li’s D、Tajima’s D、Fu & Li’s F 統計檢驗值評估了29個ACLSV分離物CP基因的核苷酸多樣性模式(表5)。研究結果顯示,ACLSV組Ⅰ和組Ⅱ的Tajima’s D、Fu & Li’s D、Fu & Li’s F均為正值,但這些值均沒有顯著差異,這可能是由于種群瓶頸結構或/和平衡選擇造成的。此外,單倍型多樣性(Hd=0.987 0)高和核苷酸多樣性(π=0.029 0)低均在組Ⅱ中出現。綜上結果表明,山西ACLSV種群多樣性可能是由于平衡選擇造成的。
3 結論與討論
蘋果褪綠葉斑病毒是侵染蘋果的主要潛隱性單鏈RNA病毒,嚴重威脅蘋果產業的健康發展。本研究發現,采樣的山西9個產區ACLSV的發病率超過50%,給果農造成巨大的經濟損失。此外, ACLSV的寄主范圍廣,除可以侵染蘋果外,還可侵染梨、桃、李、杏、櫻桃等多種果樹,對果樹的生長、產量和質量均造成嚴重的影響[29]。ACLSV在不同宿主和環境中會發生突變以適應不同寄主,而高突變率加重了ACLSV危害[30]。本研究通過對山西及GenBank中登記的ACLSV分離物CP基因核苷酸序列、氨基酸序列、種群多樣性和種群結構進行分析,基本明確了山西12個蘋果產區的ACLSV存在變異,種群多樣性較高,是危害山西蘋果優產、高產的重要因素,有效預防ACLSV已經成為山西蘋果產區亟待解決的問題。
山西蘋果ACLSV種群是較為穩定的類群。ACLSV分離物系統發育分析和核酸序列一致性分析顯示,來自中國山西蘋果的分離物單獨分為一組;其他的分離物(如中國山楂、中國桃子、日本蘋果和印度蘋果等)分為一組。這表明ACLSV分離株的遺傳多樣性主要是由地理和寄主因素共同決定的,該結果與2015年李科等報道的山東蘋果分離物的聚類與地理或寄主并無明顯關系的結果相反[18],這可能是由于本研究僅是基于CP基因分析,ACLSV遺傳多樣性是否與地理或寄主因素相關還需進一步驗證。同時我們發現中國山西蘋果的ACLSV種群與其他種群之間的遺傳多樣性存在明顯差異,其種群內的突變數和核苷酸多樣性都是最小,說明山西蘋果的ACLSV種群相比其他已知的ACLSV分離物多樣性較低。結合前人研究結果ACLSV在自然環境存在較大的由環境因子、地理位置、寄主等引起的負向選擇壓力[31]。可能是這種負選擇壓力影響了山西蘋果ACLSV種群的CP基因變異,同時,不同ACLSV種群CP基因的Ka/Ks值表現出高度多樣性,這些都證明ACLSV種群可能經歷了不同的壓力。本研究中來自山西蘋果的ACLSV種群受到負選擇壓遠小于組Ⅰ,說明該種群與其他ACLSV種群相比正趨于穩定。
地域和寄主的負向選擇壓力可能是山西蘋果ACLSV種群變異的主要因素。有研究報道ACLSV種群之間存在顯著的遺傳差異和頻繁的基因交流[6],而本研究發現,ACLSV分離株在組內的基因交流非常頻繁,而在組Ⅰ與組Ⅱ之間很少出現基因交流。同時發現,組Ⅱ內的遺傳距離小于組Ⅰ與組Ⅱ間的遺傳距離,但有趣的是組Ⅰ內的遺傳距離大于組Ⅰ與組Ⅱ間的遺傳距離。這可能是由于組Ⅰ的分離物來自不同國家和寄主,而組Ⅱ的分離物均來自中國蘋果。不同的宿主、環境和載體都可能對ACLSV產生選擇壓力,在強大的選擇壓力下ACLSV能夠非常快速地進化,從而提高其遺傳多樣性。通過對山西ACLSV群體進行中性檢驗發現山西蘋果ACLSV種群可能發生了平衡選擇或種群收縮[32]。 以上結果進一步證實中國山西蘋果的ACLSV種群與其他種群之間存在明顯的遺傳差異,同時山西蘋果ACLSV種群是非常保守的。ACLSV作為單鏈RNA病毒,其RNA聚合酶缺乏DNA聚合酶的校對能力[33]。
參考文獻
[1] 劉鳳之, 王海波, 胡成志. 我國主要果樹產業現狀及“十四五”發展對策[J].中國果樹, 2021(1): 15.
[2] 鄭國富. 我國蘋果出口貿易發展的特征、問題與升級戰略[J].中國果樹, 2021(6): 8992.
[3] 王金政, 毛志泉, 叢佩華, 等. 新中國果樹科學研究70年——蘋果[J]. 果樹學報, 2019, 36(10): 12551263.
[4] 王樹桐, 王亞南, 曹克強. 近年我國重要蘋果病害發生概況及研究進展[J]. 植物保護, 2018, 44(5): 1325.
[5] 姚潤東, 史文森, 孫吳潤澤, 等. 中國西南主要蘋果產區潛隱性病毒分子鑒定[J]. 四川大學學報(自然科學版), 2019, 56(2): 357362.
[6] CHEN Wei, JING Xiaoya, SUN Liuqing, et al. Genome cloning and genetic diversity of Apple chlorotic leaf spot virus[J]. Indian Journal of Biochemistry & Biophysics, 2019, 56: 514519.
[7] 李保華, 王彩霞, 董向麗. 我國蘋果主要病害研究進展與病害防治中的問題[J]. 植物保護, 2013, 39(5): 4654.
[8] ABTAHI F, SHAMS-BAKHSH M, SAFAIE N, et al. Incidence and genetic diversity of apple chlorotic leaf spot virus in Iran [J]. Journal of Plant Pathology, 2019, 101(3): 513519.
[9] 秦子禹, 孫建設, 王娜, 等. 蘋果莖痘病毒TaqMan探針實時熒光定量RT-PCR檢測方法的建立[J]. 園藝學報, 2015, 42(7): 14001408.
[10]MA Xiaofang, HONG Ni, MOFFETT P, et al. Genetic diversity and evolution of Apple stem pitting virus isolates from pear in China [J]. Canadian Journal of Plant Pathology, 2016, 38(2): 218230.
[11]陳雅寒, 孫平平, 馬強, 等. 東北冷寒產區蘋果褪綠葉斑病毒檢測及其分子多樣性分析[J]. 園藝學報, 2019, 46(12): 23972405.
[12]BRAKTA A, THAKUR P D, HANDA A. First report of apple top working disease caused by viruses (Apple stem grooving virus, Apple chlorotic leaf spot virus, and Apple stem pitting virus) in apple in India [J]. Plant Disease, 2013, 97(7): 1001.
[13]CHEN Shanyi, ZHOU Ying, YE Ting, et al. Genetic variation analysis of apple chlorotic leaf spot virus coat protein reveals a new phylogenetic type and two recombinants in China[J]. Archives of Virology, 2014, 159(6): 14311438.
[14]NICKEL O, SILVA F N, FAJARDO T V M, et al. Characterization and genetic variability of coat protein genes of Apple chlorotic leaf spot virus isolates from southern Brazil[J]. Tropical Plant Pathology, 2017, 43(2): 109116.
[15]WANG M, DAI H. First report of apple chlorotic leaf spot virus in hawthorn in China[J]. Plant Disease, 2015, 99(1): 164.
[16]URBANOVICH O. Molecular variability of Apple chlorotic leaf spot virus isolated in Belarus [J]. Environmental and Experimental Biology, 2016, 14(3): 121126.
[17]KESHAVARZ T, HAJNAJARI H. Status of the Apple chlorotic leaf spot virus infection in native and imported apple tree cultivars in the national collection of Kamalshahr horticulture [J]. Journal of Plant Protection, 2019, 33(4): 387396.
[18]李科, 時洪偉, 荊陳沉, 等. ACLSV山東蘋果分離物基因重組及CP序列多樣性分析[J]. 中國農業科學, 2015, 48(14): 28572867.
[19]RANA T, CHANDEL V, KUMAR Y, et al. Molecular variability analyses of Apple chlorotic leaf spot virus capsid protein[J]. Journal of Biosciences, 2010, 35(4): 605615.
[20]HE Zhen, YASAKA R, LI Wenfeng, et al. Genetic structure of populations of sugarcane streak mosaic virus in China: comparison with the populations in India[J]. Virus Research, 2016, 211: 103116.
[21]張虎, 景曉雅, 孫柳清, 等. 玉米矮花葉病毒分離物基因組克隆及多樣性分析[J]. 中國農業科技導報, 2019. 21(9): 3643.
[22]HOSSEINI H, MEHRVAR M, ZAKIAGHL M, et al. Comparative genetic diversity of potato virus Y populations based on coat protein gene [J]. Acta Virologica, 2017, 61(2): 161174.
[23]XIE Xiansheng, CHEN Wei, FU Qiang, et al. Molecular variability and distribution of Sugarcane Mosaic Virus in Shanxi, China [J/OL]. PLoS ONE, 2016, 11(3): e0151549. DOI: 10.1371/journal.pone.0151549.
[24]DALMON A, DESBIEZ C, COULON M, et al. Evidence for positive selection and recombination hotspots in Deformed wing virus (DWV) [J/OL]. Scientific Reports, 2017, 7: 41045. DOI: 10.1038/srep41045.
[25]WANG Yanan, WU Beilei, BORTH W B, et al. Molecular characterization and distribution of two strains of Dasheen mosaic virus on taro in Hawaii [J]. Plant Disease, 2017, 101(12): 19801989.
[26]SAMIEI A, MEHRVAR M, ZAKIAGHL M, et al. Distribution and phylogenetic analysis of the 3′UTR and coat protein gene of Iranian Beet black scorch virus[J]. Journal of Plant Diseases and Protection, 2019, 126(6): 535542.
[27]MURPHY J F, MORAWO T. Comparative evaluation of disease induced by three strains of Tobacco etch virus in Capsicum annuum L.[J]. Plant Disease, 2016, 101(1): 217223.
[28]LI Xiangdong, ZHU Tiansheng, YIN Xiao, et al. The genetic structure of Turnip mosaic virus population reveals the rapid expansion of a new emergent lineage in China [J/OL]. Virology Journal, 2017, 14(1): 165. DOI: 10.1186/s12985-017-0832-3
[29]秦子禹, 孫建設, 王娜, 等. 一種高效的蘋果褪綠葉斑病毒TaqMan探針實時熒光定量RT-PCR檢測方法[J]. 植物保護學報, 2015, 42(4): 7479.
[30]SONG Yansu, HONG Ni, WANG Liping, et al. Molecular and serological diversity in Apple chlorotic leaf spot virus from sand pear (Pyrus pyrifolia) in China [J]. European Journal of Plant Pathology, 2011, 130(2): 183196.
[31]GAO Fangluan, LIN Wuzhen, SHEN Jianguo, et al. Genetic diversity and molecular evolution of Arabis mosaic virus based on the CP gene sequence [J]. Archives of Virology, 2016, 161(4): 10471051.
[32]TORRICO A K, CELLI M G, CAFRUNE E E, et al. Genetic variability and recombination analysis of the coat protein gene of Strawberry mild yellow edge virus [J]. Australasian Plant Pathology, 2016, 45(4): 401409.
[33]XING Fei, ROBE B L, ZHANG Zhijia, et al. Genomic analysis, sequence diversity, and occurrence of Apple necrotic mosaic virus, a novel ilarvirus associated with mosaic disease of apple trees in China [J]. Plant Disease, 2018, 102(9): 18411847.
(責任編輯:楊明麗)

