蠶豆VfGASA1基因的異源過表達延遲擬南芥開花
高營 林云 袁星星 薛晨晨 陳新 朱月林


摘要: GASA(Gibberellic acid-stimulated in Arabidopsis)蛋白是一類受赤霉素調控的小分子蛋白質,參與植物生長發育的多條途徑。本研究在蠶豆(Vicia faba)中同源克隆了VfGASA1基因,該基因的開放閱讀框全長為363 bp,編碼1個包含120個氨基酸殘基的蛋白質,VfGASA1在蠶豆葉片和嫩莢中高表達,亞細胞定位結果顯示,VfGASA1是一個定位于質外體的分泌蛋白。異源過表達VfGASA1導致轉基因擬南芥開花延遲、蓮座葉數量增多,外施赤霉素能夠恢復這一現象。實時熒光定量結果顯示,轉基因擬南芥植株中FT基因顯著下調,而DELLA基因中的GAI基因顯著上調,說明VfGASA1可能是通過間接抑制DELLA蛋白的表達從而調控植物開花。本研究結果為蠶豆的開花調控育種提供了理論依據。
關鍵詞: 蠶豆;VfGASA1;擬南芥;開花時間;赤霉素
中圖分類號: S643.6 文獻標識碼: A 文章編號: 1000-4440(2021)01-0044-09
Heterologous overexpression of Vicia faba VfGASA1 gene delays flowering in transgenic Arabidopsis
GAO Ying1, 2, LIN Yun2, YUAN Xing-xing2, XUE Chen-chen2, CHEN Xin2, ZHU Yue-lin1
(1.College of Horticulture, Nanjing Agricultural University, Nanjing 210095, China;2.Institute of Industrial Crops, Jiangsu Academy of Agricultural Sciences, Nanjing 210014, China)
Abstract: Gibberellic acid-stimulated in Arabidopsis (GASA) protein is a kind of protein with small molecular weight regulated by gibberellin and participates in various pathways of plant growth and development. In this study, the GASA gene VfGASA1 in faba bean (Vicia faba) was got by homologous cloning. The open reading frame(ORF) of VfGASA1 was 363 bp in length and encoded a protein containing 120 amino acid residues. VfGASA1 was highly expressed in the leaves and capsules of faba beans, and subcellular localization showed that VfGASA1 was a secretory protein located in the apoplast. Heterologous overexpression of VfGASA1 in Arabidopsis could delay flowering and increase the number of rosette leaves, while the phenomenon could be recovered by the application of exogenous gibberellin. Results of real-time PCR (RT-PCR) showed that the expressional level of ‘florigen gene (FT) was significantly down-regulated in transgenic Arabidopsis while GAI (one of DELLA genes) was significantly up-regulated. Therefore, VfGASA1 might regulate the flowering of plants by indirect inhibiting the expression of DELLA genes. This study provides theoretic basis for breeding programs targeted on the flowering control of faba bean.
Key words: Vicia faba;VfGASA1;Arabidopsis;flowering time;gibberellin
開花是指被子植物由營養生長向生殖生長的轉變,是植物生命史的重要相變,也是農作物的重要農藝性狀之一;開花時間受到不同信號的影響,在擬南芥中有光周期、自主途徑、春化途徑及赤霉素(GA)信號途徑等[1-4]。一些關鍵的調控因子負責整合這些信號途徑,包括FT、SOC1以及LFY的表達[5-7],這些調控因子激活LFY、AP1和FUL在花序分生組織側部的表達,誘導花原基的產生[8]。其中,FT是開花調控的整合因子和決定基因,各信號途徑最終通過影響其表達水平而誘導開花。FT基因的表達量受到CO蛋白的正向調控[9],CO蛋白在光照條件下積累,在黑暗條件下降解,引起FT的節律表達和蛋白質積累,從而影響開花[10]。
赤霉素是重要的植物激素之一,參與種子萌發、節間伸長、葉片發育、開花時間調控、花器官建成以及花粉成熟等生理過程[11-15]。DELLA蛋白是赤霉素信號途徑中的核心抑制因子,能夠結合眾多下游蛋白質,阻礙GA信號的傳遞[16]。擬南芥共有5個DELLA基因(GAI、RGA、RGL1、RGL2和RGL3),這些基因編碼的蛋白質均可以抑制開花[17]。研究發現,DELLA蛋白可以結合CO蛋白,阻止CO蛋白對下游FT基因的調控,從而抑制植物開花[18];DELLA蛋白中的RGA蛋白可以結合FLC蛋白并促進FLC蛋白對SOC1和FT基因的抑制作用[19]。當植物感受到GA信號后,GA的受體蛋白GID1通過結構的改變與DELLA蛋白互作,使其泛素化并降解,受DELLA蛋白綁定的轉錄因子得以釋放并行使功能,從而使得下游基因響應表達[20-21]。
植物體內有大量的基因受到GA的調控,但這些基因是如何行使功能的,目前尚不明確。GASA (Gibberellic acid-stimulated in Arabidopsis) 是一類受赤霉素調節的基因,最早在番茄GA合成缺陷突變體gib1中被鑒定[22],目前已在多種單子葉植物(如水稻、玉米)[23-24]和雙子葉植物(如擬南芥、草莓)[25-26]中被克隆。研究發現大多數GASA基因表達量都受到赤霉素的影響,這些基因可以調控植物生長發育,包括種子萌發、細胞伸長、細胞分裂、開花時間、脅迫響應和激素信號交流等[27-28]。GASA蛋白屬于小分子多肽,一般長80~120個氨基酸,具有3個結構域:N端的信號肽、中間的親水區域和C端高度保守的GASA結構域[29]。在模式植物擬南芥中共有15個GASA家族基因(AtGASA1~AtGASA15)[27-28],其中AtGASA4、AtGASA6、AtGASA7、AtGASA8和AtGASA13受GA誘導表達量上調,而AtGASA1、AtGASA5、AtGASA9 和AtGASA11受GA誘導表達量下調[30]。AtGASA4能夠促進擬南芥開花[31],而AtGASA5卻抑制擬南芥開花[32],說明GASA基因家族在植物生長發育中出現了功能分化,但有關GASA蛋白的研究還不明確。
蠶豆是重要的冬季食用豆作物,在中國有廣泛的種植[33],在南方地區,蠶豆一般在冬季播種,次年春季收獲鮮莢,促進蠶豆田間適量提早開花有助于蠶豆提前上市。前人研究發現,苗期春化處理可以導致蠶豆葉片中大量bHLH、ERF以及WRKY基因家族轉錄因子的基因表達量發生顯著變化[34],此外還有91個參與不同路徑的蛋白質在春化處理后有顯著積累[35],但是關于進一步的機理研究以及其他途徑對蠶豆開花時間影響的研究未見報道。由于蠶豆的基因組巨大,測序和組裝的難度極大,其可用基因組信息較少[36],因此有關蠶豆基因的克隆和功能研究較少。
本研究以通蠶鮮6號為研究材料,同源克隆VfGASA1基因,對VfGASA1蛋白的同源性和亞細胞定位進行研究,并在擬南芥中過表達VfGASA1基因,以期研究該基因對蠶豆開花及其他性狀的影響,豐富蠶豆開花機制研究的理論基礎。
1 材料與方法
1.1 材料及其 RNA 的提取
本試驗采用的蠶豆(Vicia faba)品種為通蠶鮮6號,由江蘇省農業科學院提供。按照根、莖、葉片、花、嫩莢、籽粒等組織取樣后放入液氮中速凍,存于-80 ℃冰箱備用。用RNA提取試劑[天根生化科技(北京)有限公司]提取總RNA。
1.2 基因克隆
蒺藜苜蓿(Medicago truncatula)是豆科中與蠶豆親緣關系較近的物種[37],根據美國國家生物信息中心(NCBI)網站(www.ncbi.nlm.nih.gov)提供的蒺藜苜蓿MtGASA13(LOC11422708)的序列信息,設計引物VfGASA-F1和VfGASA-R1(表1)進行PCR擴增,擴增程序:94 ℃預變性2 min;98 ℃變性15 s,55 ℃退火30 s,68 ℃延伸1 min,終延伸5 min,循環數為33。PCR產物通過瓊脂糖凝膠電泳檢測,并利用DNA回收試劑盒[擎科生物科技(南京)有限公司]純化回收,測序獲得VfGASA1序列。本研究所用引物根據需求按照序列信息設計,采用的軟件為Primer 5。
1.3 同源比對
通過在線BLAST (https://blast.ncbi.nlm.nih.gov/Blast.cgi)序列比對,將同源性較高、不同植物品種的GASA家族基因進行系統進化分析,使用DNAMAN軟件進行多序列分析,系統進化樹用 MEGA 10.0 本地軟件鄰接法構建;利用InterPro網站(www.ebi.ac.uk/interpro)對蛋白質結構進行預測。
1.4 載體構建
根據pCAMBIA1305.1-GFP序列設計VfGASA1的接頭引物VfGASA1-F2和VfGASA1-R2并擴增,Xba Ⅰ酶切產生的線性化載體和PCR產物通過重組酶(南京諾唯贊生物科技股份有限公司)進行重組連接,構建
35S∶∶VfGASA1-GFP融合載體。利用DH5α大腸桿菌感受態轉化連接產物,通過測序獲得正確的單克隆菌株,菌株擴繁后利用質粒提取試劑盒[天根生化科技(北京)有限公司]提取質粒,并將其轉入農桿菌感受態中。
1.5 亞細胞定位
將農桿菌在28 ℃ 條件下培養至OD600為0.5~0.8,收集菌體并用浸染液重懸至OD600為0.1左右,選擇生長28 d左右健康本氏煙草(Nicotiana benthamiana)的倒三至倒五葉,利用注射器進行浸染,浸染后48 h內在激光共聚焦顯微鏡下觀察熒光。觀察到熒光后利用30%蔗糖水溶液處理葉片30 min,觀察煙草葉肉細胞質壁分離后的熒光情況。
1.6 擬南芥浸染
向250 ml LB液體培養基中加入農桿菌菌液,28 ℃、220 r/min培養過夜。至菌液OD600達到1.0左右,室溫離心收集菌體,配制植物浸染液,用等體積的浸染液重懸菌體。將長勢良好的擬南芥花苞浸入農桿菌浸染液中,4 min后拿出并用保鮮膜包裹,避光倒伏放置24 h,然后按常規方法培育植株至結實,收獲T0代種子。
1.7 RT-PCR分析
利用TaKaRa實時定量PCR試劑盒SYBR Green Ⅱ熒光染料法進行基因的表達量分析。設計特異性引物(表1),反應體系為:SYBR Green Premix Ex Taq Ⅱ 10.0 μl,Primer F/R 各0.8 μl,滅菌水6.4 μl,DNA模板2.0 μl,總體系為20.0 μl。進行Real Time PCR兩步法擴增反應,每份樣品設3個生物學重復。試驗結果利用相對定量2-△△Ct方法計算表達量。
1.8 數據統計和分析
數據采用Excel軟件繪圖,用SPSS 23.0統計軟件對平均數進行單因素方差分析(ANOVA)比較。
2 結果與分析
2.1 蠶豆GASA1基因的克隆與同源比對
根據與蠶豆親緣關系較近的蒺藜苜蓿中的MtGASA13(基因識別號:LOC11422708)序列設計引物(表1),從蠶豆葉片cDNA中擴增出全長約 630 bp的單一條帶,根據測序結果預測該基因編碼序列(CDS)全長363 bp,編碼1個包含120個氨基酸殘基的蛋白質,將該基因命名為VfGASA1。進化樹分析結果(圖1A)顯示,與VfGASA1同源性最高的蛋白質是來自苜蓿的MtGASA13(登錄號:XP_003625068.1),相似性為88.3%,其次是鷹嘴豆CaGASA1蛋白(登錄號:XP_004493436.1),相似性為85.8%,這也和豆科植物與苜蓿、鷹嘴豆有較近的親緣關系一致[38]。在模式植物擬南芥(Arabidopsis thaliana)中,與蠶豆VfGASA1蛋白同源性最高的蛋白質是AtGASA15(登錄號:NP_001319515.1),相似性約為69.0%。根據InterPro網站(www.ebi.ac.uk/interpro)的預測,VfGASA1蛋白的一級結構包含3個結構域,前24個氨基酸殘基是一段信號肽,第61至第120個氨基酸是GASA結構域,中間是一段不保守區域,與其他物種中同源蛋白相比,VfGASA1蛋白在GASA結構域內高度保守,包含12個半胱氨酸殘基,而在信號肽區域的差異較大(圖1B)。
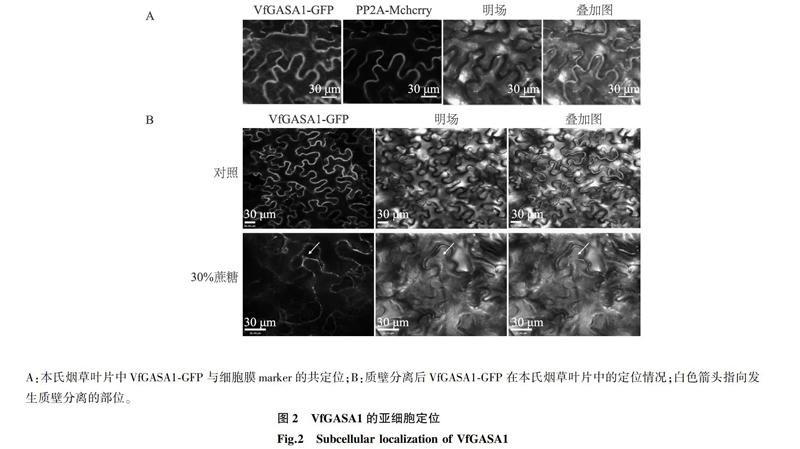
2.2 VfGASA1亞細胞定位
根據TMHMM網站(http://www.cbs.dtu.dk/services/TMHMM/)的預測,VfGASA1蛋白是一個分泌蛋白,可能定位于質外體,這與大部分GASA蛋白的定位有差別,為檢測VfGASA1蛋白的亞細胞定位情況,將重組質粒35S∶∶VfGASA1-GFP以及PP2A-mCherry共同轉入本氏煙草葉片中進行瞬時表達,結果發現綠色熒光蛋白(GFP)信號與膜marker的熒光重疊,均出現在細胞輪廓上(圖2A),繼續用30%蔗糖溶液對煙草葉片進行質壁分離處理,發現在質壁分離后的細胞膜和細胞壁上均能觀察到GFP熒光(圖2B),綜合以上結果推測VfGASA1是一個定位在質外體的分泌蛋白。
2.3 VfGASA1基因的表達模式
GASA家族基因參與植物側根、葉片、莖稈、花器官和種子的生長發育,因此VfGASA1的表達模式會決定其最終功能。我們通過RT-PCR對蠶豆不同部位VfGASA1的表達量進行測定,結果顯示,VfGASA1在嫩莢中表達量最高,在葉中也有很高的表達量,在花器官中有一定的表達量,但是在莖和籽粒中表達量很低,而在根中幾乎檢測不到表達(圖3),說明VfGASA1是一個在嫩莢和葉片中高表達的基因。
2.4 VfGASA1在擬南芥中抑制開花的機制
VfGASA1蛋白與擬南芥體內AtGASA15編碼蛋白高度同源,此前未有關于AtGASA15功能的研究,為進一步研究VfGASA1在植物體內的作用,我們購買了擬南芥突變體gasa15,并在Col-0型(野生型)擬南芥中過表達了VfGASA1,在T2代篩選得到陽性轉基因株系L16和L24。定量結果顯示,在這2個陽性株系體內的VfGASA1有較高的表達量(圖4A),過表達株系在長日照條件下(光照16 h/黑暗8 h),開花時間明顯延遲(圖4B、圖4C),播種后第23 d,野生型以及gasa15突變體擬南芥均已開花,而L16、L24仍未抽薹(圖4B),L16、L24株系在開花時蓮座葉數量也較野生型及gasa15突變體擬南芥顯著增加(圖4D),說明外源VfGASA1基因的過表達可以延遲擬南芥的開花,gasa15突變體開花時間與野生型擬南芥比沒有差異,這可能是由其他GASA基因的功能冗余導致的。
為明確VfGASA1是否參與開花信號途徑,我們對長日照條件下擬南芥開花途徑中的關鍵基因進行了連續定量檢測,結果發現過表達植株AtFT的表達模式與野生型一致,但是轉基因植株中AtFT的表達量顯著降低,尤其在光照16 h后(圖4E)。AtCO的表達模式及表達量在野生型與轉基因擬南芥之間沒有明顯差異(圖4F),此外,開花途徑負調控基因FLC的相對表達量在L24植株中極顯著上調,而正向調控基因SOC1的相對表達量顯著下調(圖4G),這些結果表明在過表達植株中開花途徑被抑制。GA途徑主要通過解除DELLA對CO的綁定,促進FT的表達從而誘導開花,因此我們對比了野生型和轉基因株系DELLA基因的表達量,5個DELLA基因中,GAI在過表達株系L24中極顯著上調(圖4H)。
通過對開花基因以及GAI的定量分析,我們推測VfGASA1參與了赤霉素調控開花的過程,為驗證這一猜想,我們對轉基因擬南芥植株外施GA3處理,結果發現赤霉素處理后過表達VfGASA1株系L24與野生型的開花時間沒有明顯差異(圖5),并且相對于未處理的植株,開花時間明顯提前,外源赤霉素可以恢復VfGASA1對開花的延遲作用,說明VfGASA1缺失參與了赤霉素對開花時間的影響,并且起到負調控的作用,可能是通過促進DELLA基因中GAI基因的表達,從而抑制開花。
3 討論
中國是世界第一大蠶豆消費國和生產國[39],在蠶豆生產中晚花會導致蠶豆受到高溫脅迫,從而使產量降低[40]以及異交率大幅提升[41]。近年來,蠶豆晚花給蠶豆的產量提升以及育種工作帶來了極大的困擾,因此蠶豆晚花是亟待解決的問題。目前生產上常用苗期春化處理的方式促進蠶豆早花,但是這種方式需要大量前期投入,而且對某些春化不敏感品種沒有效果,因此尋找其他辦法,例如外源赤霉素處理等來促進早花對于蠶豆生產非常重要。
赤霉素信號途徑通過解除DELLA蛋白對CO蛋白的抑制,提升CO蛋白對FT的轉錄激活能力,提高FT的表達,從而促進植物開花[18-19]。但是關于赤霉素下游基因如何行使功能的研究較少,GASA家族基因是一類赤霉素下游響應基因[27-28],前人的研究發現部分GASA成員可以參與赤霉素對植物開花的調控,例如AtGASA4能夠促進擬南芥提早開花[31],AtGASA5能夠抑制擬南芥開花[32],此外GASA家族成員還能夠調控株型建成以及種子大小等[27-28],顯示出該家族成員在分子育種中的潛力。
蠶豆龐大的基因組是蠶豆基因克隆和分子育種的巨大障礙[36],前期關于蠶豆的研究較少,大部分集中于抗病和抗列當寄生基因的QTL定位[42],以及產量相關性狀的QTL定位[43],關于蠶豆開花時間的研究僅有春化的相關報道[34-35]。本研究通過同源比對的方式從蠶豆cDNA中克隆出1個GASA家族基因并命名為VfGASA1,發現VfGASA1是一個定位在質外體的分泌蛋白,受到赤霉素的調控,在擬南芥中異源過表達VfGASA1能夠顯著延遲擬南芥的開花時間,熒光定量的結果顯示在轉基因植株中擬南芥DELLA基因中GAI基因的相對表達量顯著上調,說明VfGASA1可能是通過影響DELLA基因的表達,從而參與赤霉素對開花時間的調節,考慮到VfGASA1定位在質外體,因此VfGASA1不會直接調控DELLA基因的表達,應該存在某種間接途徑影響DELLA基因的表達,這需要后續的深入研究。
參考文獻:
[1] SONG Y H, SHIM J S, KINMONTH-SCHULTZ H A, et al. Photoperiodic flowering: time measurement mechanisms in leaves[J]. Annual Review of Plant Biology, 2015, 66: 441-464.
[2] TURCK F, FORNARA F, COUPLAND G. Regulation and identity of florigen: FLOWERING LOCUS T moves center stage[J]. Annual Review of Plant Biology, 2008, 59: 573-594.
[3] KING R W, EVANS L T. Gibberellins and flowering of grasses and cereals: prizing open the lid of the “florigen” black box[J]. Annual Review of Plant Biology, 2003. 54(1): 307-328.
[4] KIM D H, DOYLE M R, SUNG S, et al. Vernalization: winter and the timing of flowering in plants[J]. Annual Review of Cell and Developmental Biology, 2009, 25(1): 277-299.
[5] PIN P A, NILSSON O. The multifaceted roles of FLOWERING LOCUS T in plant development[J]. Plant Cell and Environment, 2012, 35(10): 1742-1755.
[6] LEE J, LEE I. Regulation and function of SOC1, a flowering pathway integrator[J]. Journal of Experimental Botany, 2010, 61(9): 2247-2254.
[7] MOYROUD E, KUSTERS E, MONNIAUX M, et al. LEAFY blossoms[J]. Trends in Plant Science, 2010, 15(6): 346-352.
[8] WELLMER F, RIECHMANN J L. Gene networks controlling the initiation of flower development[J]. Trends in Genetics, 2010, 26(12): 519-527.
[9] SAMACH A, ONOUCH H, GOLD S E, et al. Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis[J]. Science, 2000, 288(5471): 1613-1616.
[10]TIWARI S B, SHEN Y, CHANG H C, et al. The flowering time regulator CONSTANS is recruited to the FLOWERING LOCUS T promoter via a unique cis-element[J]. New Phytologist, 2010, 187(1): 57-66.
[11]DAVIERE J M, ACHARD P. Gibberellin signaling in plants[J]. Development, 2013, 140(6): 1147-1151.
[12]LIU Z, BOACHON B, LUGAN R, et al. A conserved cytochrome P450 evolved in seed plants regulates flower maturation[J]. Molecular Plant, 2015, 8(12): 1751-1765.
[13]OGAWA M, HANADA A, YAMAUCHI Y, et al. Gibberellin biosynthesis and response during Arabidopsis seed germination[J]. The Plant Cell, 2003, 15(7): 1591-1604.
[14]SRIKANTH A, MARKUS S. Regulation of flowering time: all roads lead to Rome[J]. Cellular and Molecular Life Sciences, 2011, 68(12): 2013-2037.
[15]WANG Y, ZHAO J, LU W, et al. Gibberellin in plant height control: old player, new story[J]. Plant Cell Reports, 2017, 36(3): 391-398.
[16]MURASE K, HIRANO Y, SUN T, et al. Gibberellin-induced DELLA recognition by the gibberellin receptor GID1[J]. Nature, 2008, 456(7221): 459-463.
[17]GALVAO V C, HORRER D, KUTTNER F, et al. Spatial control of flowering by DELLA proteins in Arabidopsis thaliana[J]. Development, 2012, 139(21): 4072-4082.
[18]XU F, LI T, XU P B, et al. DELLA proteins physically interact with CONSTANS to regulate flowering under long days in Arabidopsis[J]. FEBS Letters, 2016, 590(4): 541-549.
[19]Li M, AN F, LI W, et al. DELLA proteins interact with FLC to repress flowering transition[J]. Journal of Integrative Plant Biology, 2016, 58(7): 642-655.
[20]GRIFFITHS J, MURASE K, RIEU I, et al. Genetic characterization and functional analysis of the GID1 gibberellin receptors in Arabidopsis[J]. The Plant Cell Online, 2006, 18(12): 3399-3414.
[21]WILLIGE B C, GHOSH S, NILL C, et al. The DELLA domain of GA INSENSITIVE mediates the interaction with the GA INSENSITIVE DWARF1A gibberellin receptor of Arabidopsis[J]. The Plant Cell Online, 2007, 19(4): 1209-1220.
[22]SHI L, GAST R T, GOPALRAJ M, et al. Characterization of a shoot-specific, GA3- and ABA-regulated gene from tomato[J]. The Plant Journal: for Cell and Molecular Biology, 1992, 2(2): 153-159.
[23]LEE S C, KIM S, HAN H K, et al. A gibberellin-stimulated transcript, OsGASR1, controls seedling growth and alpha-amylase expression in rice[J]. Journal of Plant Physiology, 2017, 214: 116-122.
[24]ZIMMERMANN R, SAKAI H, HOCHHOLDINGER F. The Gibberellic Acid Stimulated-Like gene family in maize and its role in lateral root development[J]. Plant Physiology, 2010, 152(1): 356-365.
[25]SUN S, WANG H, YU H, et al. GASA14 regulates leaf expansion and abiotic stress resistance by modulating reactive oxygen species accumulation[J]. Journal of Experimental Botany, 2013, 64(6): 1637-1647.
[26]DE LA FUENTE JOSE I, IRAIDA A, CRISTINA C, et al. The strawberry gene FaGAST affects plant growth through inhibition of cell elongation[J]. Journal of Experimental Botany, 2006, 57(10): 2401-2412.
[27]ZHANG S, WANG X. One new kind of phytohormonal signaling integrator: up-and-coming GASA family genes[J]. Plant Signaling & Behavior, 2017, 12(2): 6453-6459.
[28]NAHIRNAK V, NATALIA INES A, HORACIO ESTEBAN H, et al. Snakin/GASA proteins: involvement in hormone crosstalk and redox homeostasis[J]. Plant Signaling & Behavior, 2012, 7(8): 1004-1008.
[29]AUBERT D, CHEVILLARD M C, DORNE A M, et al. Expression patterns of GASA genes in Arabidopsis thaliana: the GASA4 gene is up-regulated by gibberellins in meristematic regions[J]. Plant Molecular Biology, 1998, 36(6): 871-883.
[30]ZHANG S C, WANG X J. Expression pattern of GASA, downstream genes of DELLA, in Arabidopsis[J]. Chinese Science Bulletin, 2008, 53(24): 3839-3846.
[31]ROXRUD I, LID S E, FLETCHER J C, et al. GASA4, one of the 14-member Arabidopsis GASA family of small polypeptides, regulates flowering and seed development[J]. Plant & Cell Physiology, 2007, 48(3): 471-483.
[32]ZHANG S, YANG C, PENG J, et al. GASA5, a regulator of flowering time and stem growth in Arabidopsis thaliana[J]. Plant Molecular Biology, 2009, 69(6): 745-759.
[33]O'SULLIVAN D M , DEEPTI A. Advances in faba bean genetics and genomics[J]. Frontiers in Genetics, 2016(7): 150-160.
[34]GAO B, BIAN X C, YANG F, et al. Comprehensive transcriptome analysis of faba bean in response to vernalization[J]. Planta, 2019, 251(1): 22-41.
[35]CAO Y Y, BIAN X C, CHEN M X, et al. iTRAQ-based quantitative proteomic analysis in vernalization-treated faba bean (Vicia faba L.)[J]. PLoS One, 2017, 12(11): 10-28.
[36]COOPER J W, WILSON M H, DERKS M F L, et al. Enhancing faba bean (Vicia faba L.) genome resources[J]. Journal of Experimental Botany, 2017, 68(8): 1941-1953.
[37]CRUZ-IZQUIERDO S, AVILA C M, SATOVIC Z, et al. Comparative genomics to bridge Vicia faba with model and closely-related legume species: stability of QTLs for flowering and yield-related traits[J]. Theoretical and Applied Genetics, 2012, 125(8): 1767-1782.
[38]YANG K, TIAN Z, CHEN C, et al. Genome sequencing of adzuki bean (Vigna angularis) provides insight into high starch and low fat accumulation and domestication[J]. Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(43): 13213-13218.
[39]LIU Q, DONNER E, YIN Y, et al. The physicochemical properties and in vitro digestibility of selected cereals, tubers and legumes grown in China[J]. Food Chemistry, 2006, 99(3): 470-477.
[40]NAYAK G K, ROBERTS S P M, GARRATT M, et al. Interactive effect of floral abundance and semi-natural habitats on pollinators in field beans (Vicia faba)[J]. Agriculture Ecosystems & Environment, 2015, 199(27): 58-66.
[41]BISHOP J, JONES H E, OSULLIVAN D M, et al. Elevated temperature drives a shift from selfing to outcrossing in the insect-pollinated legume, faba bean (Vicia faba) [J]. Journal of Experimental Botany, 2017, 68(8): 2055-2063.
[42]KAUR S, KIMBER R B E, COGAN N O I, et al. SNP discovery and high-density genetic mapping in faba bean (Vicia faba L.) permits identification of QTLs for ascochyta blight resistance[J]. Plant Science, 2014, 217/218(41): 47-55.
[43]CRUZ-LZQUIERDO S, AVILA C M, SATOVIC Z, et al. Comparative genomics to bridge Vicia faba with model and closely-related legume species: stability of QTLs for flowering and yield-related traits[J]. Theoretical and Applied Genetics, 2012, 125(8): 1767-1782.
(責任編輯:陳海霞)
收稿日期:2020-04-29
基金項目:國家食用豆產業技術體系生物防治與綜合防控崗位科學家項目(CARS-08-G15);江蘇特糧特經產業技術體系集成創新中心項目[JATS(2019)399]
作者簡介:高 營(1994-),女,河南南陽人,碩士研究生,從事豆類作物分子育種研究。(E-mail)3099618289@qq.com
通訊作者:朱月林,(E-mail)ylzhu@njau.edu.cn

