Janus激酶2選擇性抑制劑HipHop藥效團模型與3D QSAR模型的建立
梁 妍,陳小兵,黃麗蕓,袁章林,董開心,劉燕華,鐘山裕,李豫園,許 軍*
(1.江西中醫藥大學藥學院,南昌 330004;2.贛南衛生健康職業學院,贛州 341000)
Janus激酶2(JAK2)是酪氨酸激酶(JAK)家族的4個激酶亞型之一,是一種非受體型酪氨酸蛋白激酶,廣泛分布于人體的各種組織細胞中[1]。JAK2可介導促紅細胞生成素(EPO)、粒細胞-巨噬細胞集落刺激因子(GMCSF)、白細胞介素-3(IL-3)及生長因子在內的多種細胞因子信號傳導,在人體正常造血功能中起到非常重要的作用[2-3]。研究表明,JAK-信號轉導及轉錄激活蛋白(JAK-STAT)信號通路能快速將膜外信號轉導進入細胞核內,通過調控下游相關基因的表達促進細胞的增殖生存、抑制凋亡,并在實體瘤、淋巴瘤腫瘤及炎癥性疾病中發揮重要作用[4-5],而JAK2-STAT信號通路的持續激活與人類多種腫瘤尤其是造血系統惡性腫瘤的形成有關,如慢性髓系白血病、多發性骨髓瘤和急性髓性白血病等。JAK2抑制劑通過影響JAK2-STAT通路而降低一些炎癥反應細胞因子和化學因子從而影響其產物的調控達到臨床效果[6]。JAK2選擇性抑制劑可針對JAK2激酶特異區域選擇性結合到激酶催化區,從而產生抑制效應。目前,JAK2抑制劑主要應用于骨髓增生性疾病與實體瘤的治療中,但其在臨床使用中出現了耐藥性、毒性和不良反應等問題,因此尋找新型、療效更強、不良反應更小的JAK2選擇性抑制劑可為臨床應用提供更多、更好的選擇。
藥效團模型是一種基于配體的藥物設計工具,通過已有化合物的三維結構信息構建藥效團模型是藥物發現的一種重要手段[7-8]。基于分子共同特征藥效團(HipHop)模型用于發現一系列配體小分子所共有的化學特征,并基于這些共同特性結構的比對疊合自動生成藥效團模型,其不僅可篩選具有潛在活性的化合物,而且可以使用共有的特征藥效團搜索化合物數據庫來尋找可能的先導分子。
定量構效關系(QSAR)模型試圖發現能夠同生物大分子靶標在形狀和電荷(靜電勢)上互補的小分子。QSAR模型是基于小分子的立體和靜電場構建的回歸模型,可用于預測未知配體小分子的活性及觀察受體-配體間有利和不利的相互作用。與2D QSAR相比,3D QSAR更能間接反映配體小分子和蛋白大分子之間的非鍵相互作用特征,具有更加豐富的物理化學內涵,因此得到了迅速發展和廣泛應用。
利用定性的HipHop藥效團模型技術和定量的QSAR模型方法構建JAK2選擇性抑制劑模型,旨在發現獲得可以篩選能力強且可以指導先導物改造的最優模型,為JAK2選擇性抑制劑的研究提供參考。
1 HipHop藥效團模型的建立
1.1材料與方法 通過Discovery Studido 4.5軟件中HipHop算法構建JAK2選擇性抑制劑藥效團模型。搜集大量已報道文獻中的活性JAK2抑制劑小分子,從中篩選出對JAK2激酶具有選擇性和共同特性結構且活性較強的9個化合物作為訓練集,27個化合物作為測試集[9-11]。測試集化合物包括活性和非活性小分子,20個非活性小分子來源于NCBI數據庫。活性小分子(化合物1~27)結構見圖1,其半數抑制濃度(IC50)值分別為0.6、0.8、0.9、1.4、1.4、0.7、1.1、1.3、1.8、3.0、5.6、1.5、8.0、1.0、1.3、1.8、1.0、31.9、3.2、3.2、15.5、7.1、9.7、2.0、5.0、2.0、9.0 nmol·L-1。

圖1 活性JAK2選擇性抑制劑(1~27)的分子結構式
1.2化合物前處理 在Chem Draw軟件中將上述27個化合物的結構畫出,保存格式為MDL Molfile。首先利用Discovery Studido 4.5軟件的Prepare Ligands 模塊和 Full Minimization 模塊對化合物進行處理,產生三維結構、加立場CHARMm和能量最小化等。然后在表格瀏覽器中將訓練集分子的 Principal 和 MaxOmitFeat屬性定義,Principal 屬性定義了分子的活性水平,MaxOmitFeat 屬性定義了每個分子中允許不與藥效團模型匹配的特征元素的個數。如果所選取的分子有截然不同的藥效特征類型和尺寸,那么 Principal 和 MaxOmitFeat 屬性設置的不同對最終構建的藥效團模型影響較大。最后在Feature Mapping模塊中選擇要匹配的特征元素,包括氫鍵受體、氫鍵供體、疏水中心、正電離子中心、負電離子基團和芳環基團[12]。
1.3模型建立 在 Common Feature Pharmacophore Generation模塊中,展開Conformation Generation參數組,在下拉列表中選擇BEST,將Maximum Conformation(構象上限數)設為200,意為對每個小分子產生最多200個構象用以表征小分子的構象空間;將Energy Threshold(能量閾值)設置為10,意為只有同最低能構象的能量差值在能量閾值10 kcal·mol-1之內的構象被保留[13];其他參數則為默認值。點擊Run運行作業,運行時間大概需要5 min。作業完成后打開Report 鏈接,從Summary 一欄可知,此次運算一共產生了10個藥效團,每個藥效團的詳細信息見表1。排名第1的藥效團評分高于其他,因此選擇第1個藥效團模型進行后續工作。
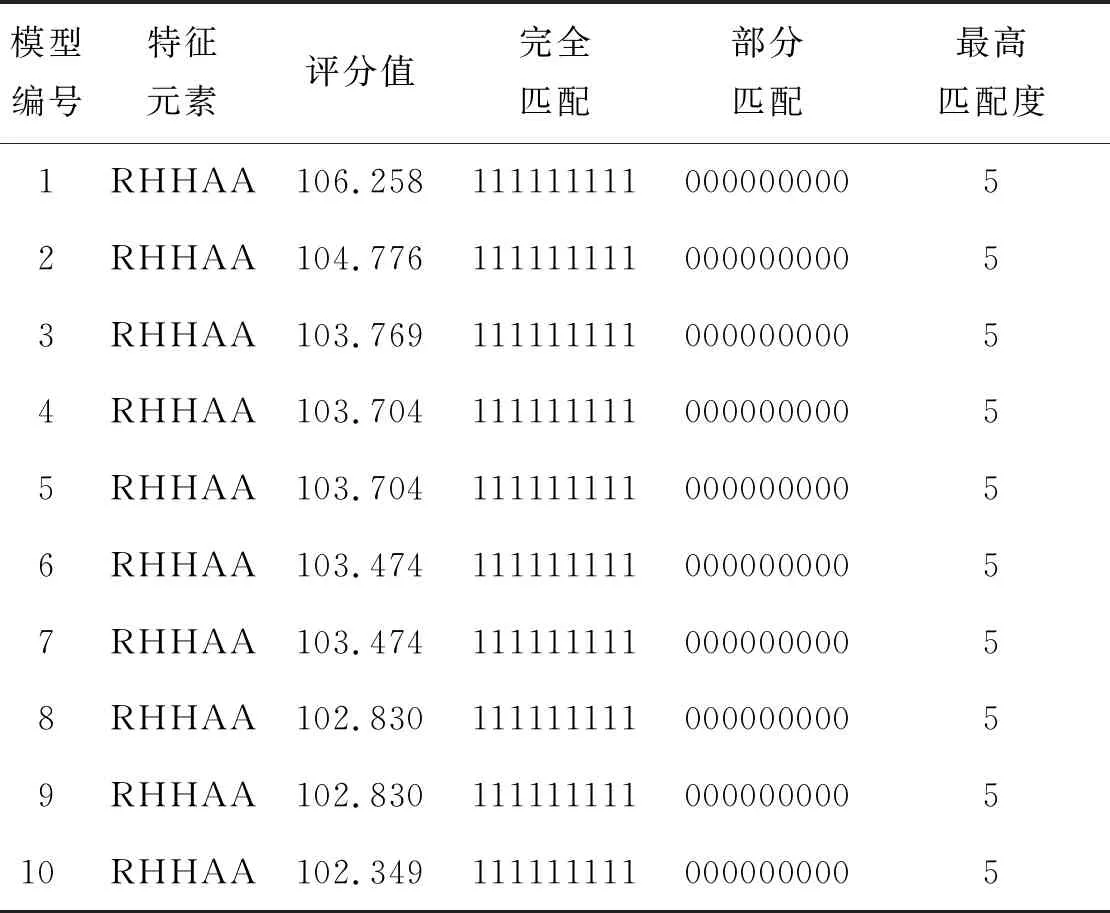
表1 JAK2選擇性抑制劑HipHop模型結果的輸出參數
最優藥效團模型由5個特征元素構成,分別為1個芳環中心(黃色)、2個疏水特征(藍色)、2個氫鍵受體特征(綠色),最優藥效團模型空間排列方式見圖2A。根據評分值可知,化合物1與藥效團的疊合效果最好,疊合效果見圖2B。

圖2 HipHop產生的最優藥效團模型(A)及其疊合結果(B,化合物1)
1.4測試集驗證 展開Conformation Generation參數組,點擊Conformation Generation右邊選項,選擇BEST模式,構象上限數設為200,能量閾值設為10。運行后,得到具有JAK2選擇性抑制作用的18個小分子全部命中,而非活性分子命中1個,說明該模型具有較高的預測力和篩選能力。但是評分值代表該化合物結構與藥效團的匹配程度,并不等同于IC50值或半數抑制常數(Ki)值[14]。因此HipHop為定性藥效團模型,在一定范圍內可預測化合物是否具有活性,指導該藥物的研發,但不能給出準確的活性數值。該模型具有一定的局限性,建立3D QSAR模型可以在其基礎上預測該類化合物的活性,并對其化合物結構的改造提供指導。
2 3D QSAR模型的建立
2.1材料與方法 通過Discovery Studido 4.5軟件中3D QSAR模塊構建JAK2選擇性抑制劑的3D QSAR模型。訓練集與測試集的活性小分子結構與HipHop中的相反,3D QSAR訓練集中去除了2個活性較弱的小分子。在構建模型時,化合物活性數之間的差值一般在1~2之間效果最佳,因此活性數據一般轉換為lgIC50。本方法利用能量格點作為描述符構建了一個偏最小二乘(PLS)模型,該能量格點由2種用于測量靜電勢和立體效應的探針計算得到。
2.2化合物前處理 在Small Molecules模塊中的Minimize Ligands選中所有訓練集化合物,批量對化合物進行疊合能量優化,疊合效果決定了最終模型的可信度。見圖3。由圖3可知,訓練集分子結構疊合效果較好,有利于模型的建立。

圖3 訓練集分子優化疊合效果圖
2.3模型建立 打開Creat QSAR Model模塊,將16個訓練集小分子設置到參數表中的Input Ligands選項;將9個測試集小分子設置到參數表中的Input Test Ligands。Activity Property為arNCTRlogRBA,在Model Name欄中輸入 Grid Based Model,其余參數保留默認設置。點擊Run運行該作業,該作業大概需要運行1 min。待作業完成后,在Report文件的Results部分,點擊View Results查看運行結果。以訓練集分子的實驗活性值為橫坐標(x1)、預測值為縱坐標(y1),訓練集的多元線性回歸模型(MLR)線性回歸方程為y1=0.986x1+0.006。R12值越接近1,表示預測結果越準確。本實驗模型R12=0.986,即建立的該模型所預測的化合物lgIC50值與其真實值十分接近,說明該模型成功建立。
2.4QSAR模型的驗證 為了驗證模型的預測能力,對所設置的測試集進行外部驗證。在Report窗口中,點擊View Test Results鏈接查看該模型對于測試集化合物活性值預測的可信度,以測試集分子的實驗活性值為橫坐標(x2)、預測值為縱坐標(y2),測試集MLR的線性回歸方程為y2=0.047x2+0.465,R22=0.273,表明該模型具有一定的可信度。
2.53D QSAR的3D格點 3D QSAR 模型除了預測能力,其相應的3D格點圖也具有一定的作用。格點包含表征靜電勢和表征范德華作用兩部分內容。在Report中,點擊View Training Set Aligned to ModelEP,視圖窗口中顯示了訓練集分子匹配至3D QSAR模型的靜電場系數等,見圖4。紅色區域表示該區域取代基的負電性越強越有利于提高化合物的活性,如帶負電荷的原子,羧酸、亞磺酸中的羥基氧等;藍色區域表示該區域取代基的正電性越強越有利于提高化合物的活性,如帶正電荷的原子,伯、仲、叔脂肪胺中的氮原子等。
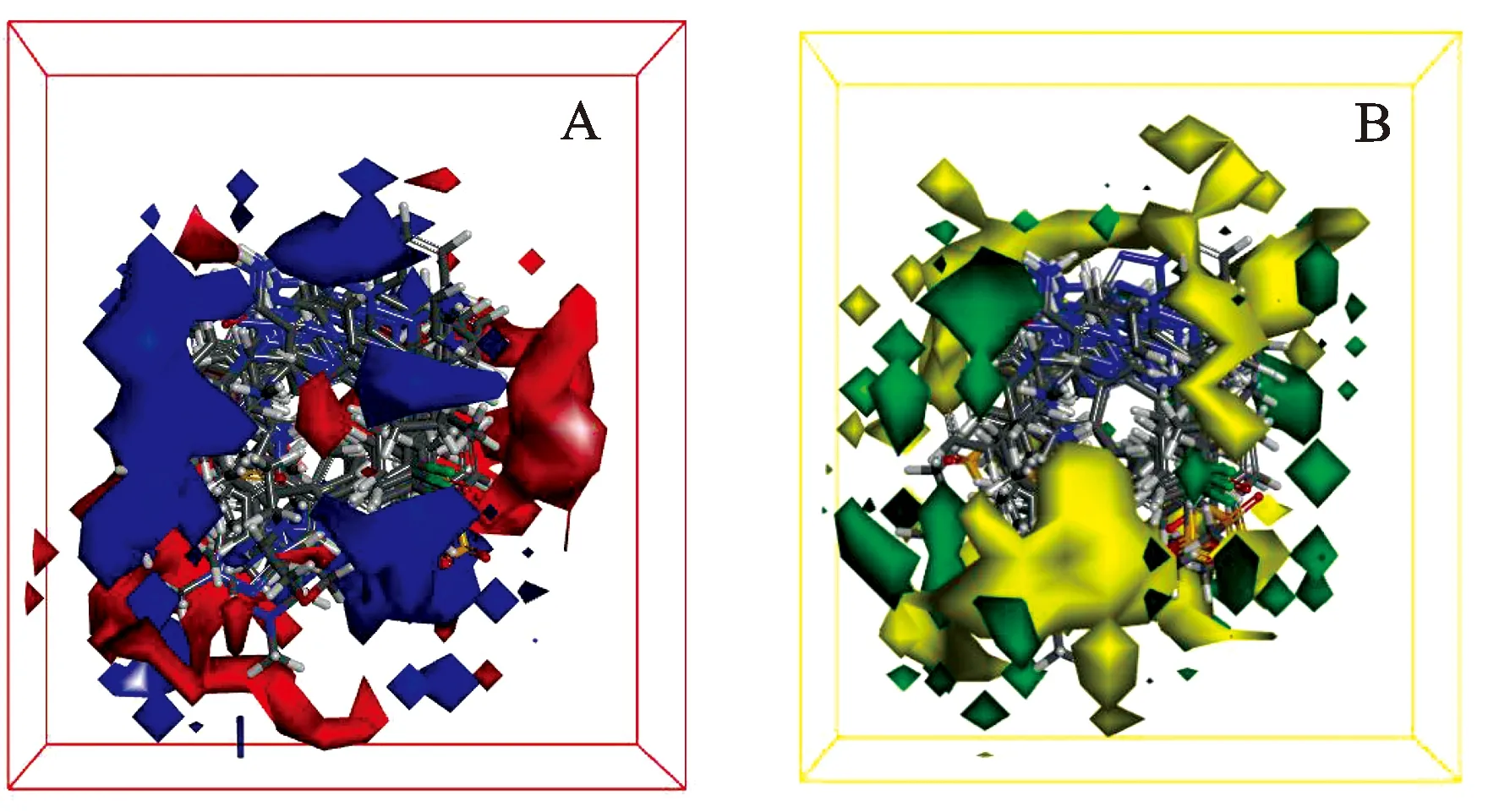
圖4 3D QSAR模型的靜電場系數等高圖(A)與立體場系數等高圖(B)
在Report中,點擊 View Training Set Aligned to ModelVDW,視圖窗口中顯示了訓練集分子匹配至3D QSAR模型的立體場系數等高圖。黃色區域表示該區域取代基體積的增大不利于提高化合物的活性;綠色區域表示該區域取代基體積的增大有利于提高化合物的活性。例如化合物1、2、4、12等,隨著氨基位引入的取代基體積的增大,化合物活性不斷提高。因此對該類化合物進行結構優化設計時,應優先引入體積較大的取代基團,以期提高化合物的生物活性。
3 結論
將從已報道文獻中篩選出具有JAK2選擇性抑制作用強和共同特性結構的化合物分別從定性及定量方面建立模型。根據驗證結果可知,HipHop藥效團模型可預測化合物是否具有活性,且篩選能力較強,但不能預測化合物活性的強弱,存在一定的限制性,而3D QSAR模型可以補足此缺點,能夠預測該類化合物的活性值且對其結構的修飾指明方向。因此,2個模型的結合有利于JAK2選擇性抑制的發現,為該類藥物的研究提供指導。

