基于KASP標記的葡萄品種鑒定
王富強,張建,溫常龍,樊秀彩,張穎,孫磊,劉崇懷,姜建福
基于KASP標記的葡萄品種鑒定
王富強1,張建2,溫常龍2,樊秀彩1,張穎1,孫磊1,劉崇懷1,姜建福1
1中國農業科學院鄭州果樹研究所,鄭州 450009;2北京市農林科學院北京蔬菜研究中心,北京 100097
【】開發一組能夠區分中國主栽葡萄品種的競爭性等位基因特異性PCR(KASP)分子標記,為中國葡萄品種保護、品種登記和市場維權等提供技術支撐。基于前人篩選的60個葡萄高多態性SNP位點,轉化為KASP標記。分別使用23份代表性品種和76份主要栽培品種對轉化成功的KASP標記進行初篩、復篩、驗證,獲得一組高質量的KASP標記,并用于構建76份葡萄品種的DNA指紋圖譜。60個SNP位點有3個不具備基因組特異性,6個無法設計KASP-PCR引物,最終51個SNP成功轉化為KASP標記,轉化率達到89.47%。利用LGC-SNPline平臺對23份代表性品種進行基因分型,51個KASP標記全部成功分型。基于次要等位基因頻率(MAF)大于0.25、多態性信息含量(PIC)大于0.35、缺失率小于0.2、雜合率小于0.6等參數,初步篩選出27個優質KASP標記。使用構建指紋圖譜的76份品種復篩出22個高質量KASP標記。22個KASP標記的缺失率均小于0.12,PIC均大于0.3,雜合率在0.4—0.6的標記占77.27%,MAF大于0.3的標記占95.45%。此外,利用這22個標記對同一品種不同樹體提取的23份代表性品種的DNA擴增檢測,前后兩次檢測分型結果穩定一致,表明這22個標記具有較好的重復性和穩定性。進一步將基于22個標記獲得的76份葡萄品種分型結果轉化為二元編碼數據,得到76份葡萄品種的指紋圖譜。經鄰接聚類分析和群體結果分析,可將76份葡萄品種分為3類,并能正確區分二倍體和多倍體。僅用10個標記(VIT_15_18567587、VIT_6_4258638、VIT_8_3320936、VIT_12_22228357、VIT_11_19390306、VIT_16_17950801、VIT_18_11138668、VIT_12_739916、VIT_16_13454358、VIT_16_21202286)就能區分70份品種,其中有54份品種達到品種鑒定的標準(差異位點數≥2)。從60個葡萄SNP中成功轉化51個KASP標記,并篩選出22個高質量KASP標記,構建了76份葡萄品種的SNP指紋圖譜,首次驗證了KASP技術在我國葡萄品種鑒定中的可行性。
葡萄;KASP標記;品種鑒定;指紋圖譜;遺傳多樣性分析
0 引言
【研究意義】葡萄為葡萄科(Vitaceae)葡萄屬(L.)多年生藤本植物[1],全世界至少有23 000個品種(http://www.vivc.de/),其適應性強,適栽范圍廣。不同地區間葡萄品種交流頻繁,而部分引種或地方品種又沒有確切的科學名稱,難免發生同物異名或同名異物現象[2]。另外,當前葡萄苗木市場管理暫不完善,隨意更改品種名稱、炒作品種等不良現象時有發生,極大損害了育種家的利益,不利于葡萄產業的健康、可持續發展。因此,尋找一種經濟、高效、準確的品種鑒別方法,對于提高我國葡萄種質資源管理效率和品種保護能力均具有重要意義。【前人研究進展】DNA分子標記具有周期短、受環境影響小、可實現高通量檢測等優點,為葡萄品種鑒定提供了新的手段。其中SSR(simple sequence repeat)和SNP(single nucleotide polymorphism)分子標記已被國際植物新品種權保護聯盟(UPOV)BMT分子測試指南[3]和國內《植物品種鑒定DNA指紋方法總則》(NY/T 2594-2016)[4]定為優先推薦的兩種標記方法。目前,國內已研發出一套用于中國葡萄品種鑒定的30個SSR標記體系[5],并利用其中8個引物區分國內290份葡萄品種[6]。但因SSR標記數量和檢測通量有限、檢測成本偏高、數據讀取費時耗力等因素[7],限制了更大范圍的葡萄品種鑒定工作。SNP分子標記作為最新一代標記,具有數量多、具二態性、穩定遺傳等優點[8],加上各種SNP高通量檢測平臺的出現,可以很好彌補SSR標記的技術缺陷[9],已在生物學、農業、醫學和生物進化等領域得到了廣泛應用[10]。其中,高通量SNP檢測技術主要有基因芯片和LGC公司推出的KASP(kompetitive allele specific PCR)技術,適用于不同檢測需求[9]。基因芯片技術主要適用于少樣本多位點檢測,如我國自主研發的Wheat 660K[11]、Rice SNP50[12]、Maize 50K[13]基因芯片,單次位點檢測量達上萬個,不足之處是當前使用成本偏高,開發難度較大;KASP技術則適于多樣本、少位點的檢測,具有高效、靈活、準確、低成本的特點[14],國內外研究者已利用KASP技術分別構建小麥[15-16]、玉米[17]、水稻[10]、棉花[18]、甘藍[19]、黃瓜[20]等作物的核心標記體系,為相應作物的品種鑒定和品種保護等提供了便利。而SNP標記在葡萄品種鑒定中,主要使用成本較高的基因芯片和測序技術,如Cabezas等[21]已構建了針對歐亞種葡萄的SNP鑒定體系,并確定了48個SNP為鑒定品種的標準位點;LAUCOU等[22]基于18K SNPs葡萄基因芯片從945份材料中篩選出14個高多態性SNP標記用于歐洲地區栽培品種鑒定。【本研究切入點】目前國際上篩選出的SNP標記僅在二倍體歐亞種中得到應用,而我國主栽的葡萄多為多倍體品種,因此有必要開發適用范圍更廣的SNP標記。雖然李貝貝等[23]和LIANG等[24]利用不同重測序技術獲得上萬個SNP位點,分別對304份和472份葡萄種質進行了遺傳多樣性分析,但沒有篩選出用最少的SNP位點組合來鑒別葡萄品種的具體結果。在實際的葡萄品種鑒定工作中,研究者希望用最少的位點區分更多的樣本,以達到高效、準確、廉價的鑒定目的,比較而言,KASP技術更適合SNP分子標記開發。【擬解決的關鍵問題】本研究基于前人開發的葡萄SNP標記,以國內主栽葡萄品種、新育成品種為試材,首次嘗試利用KASP技術篩選出一套能夠鑒別中國主栽葡萄品種的核心KASP標記,并構建葡萄品種SNP指紋圖譜,為我國葡萄新品種保護、市場維權等提供技術支持。
1 材料和方法
1.1 試驗材料
試驗材料于2020年6月采自中國農業科學院鄭州果樹研究所國家果樹種質鄭州葡萄圃。
KASP標記初篩:使用23份葡萄代表性品種,其具備不同倍性、不同種性、不同用途等特點,如二倍體歐亞種釀酒品種‘赤霞珠’‘黑比諾’,二倍體歐美雜種鮮食品種‘陽光玫瑰’‘金手指’,三倍體歐美雜種鮮食品種‘夏黑’,四倍體歐美雜種鮮食品種‘巨峰’‘巨玫瑰’等(表1)。KASP標記復篩:使用76份葡萄品種,其中13份國內主栽品種、63份近年新育成品種(表2)。

表1 KASP標記初篩使用的23份葡萄代表品種信息
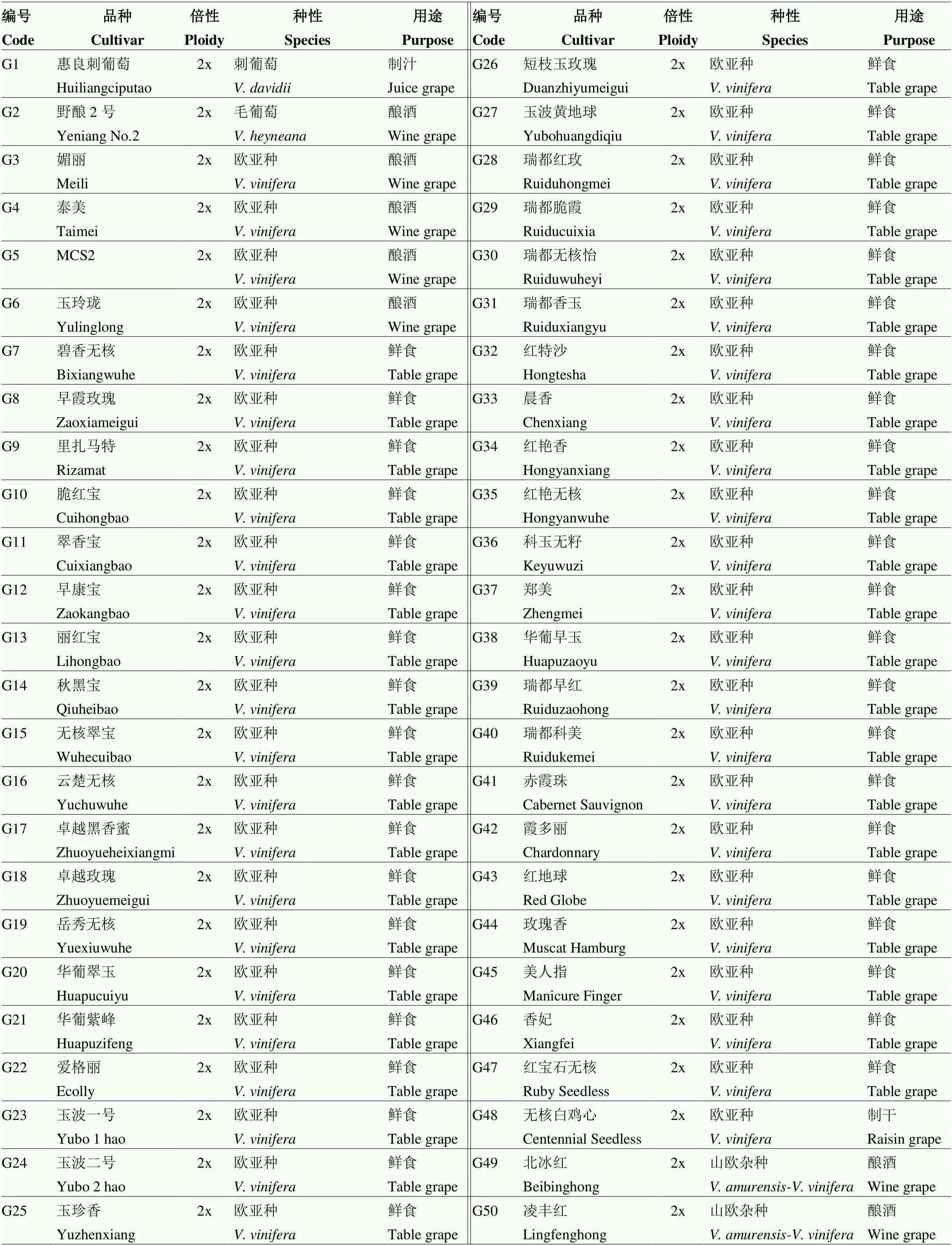
表2 KASP標記復篩使用的76份葡萄品種信息
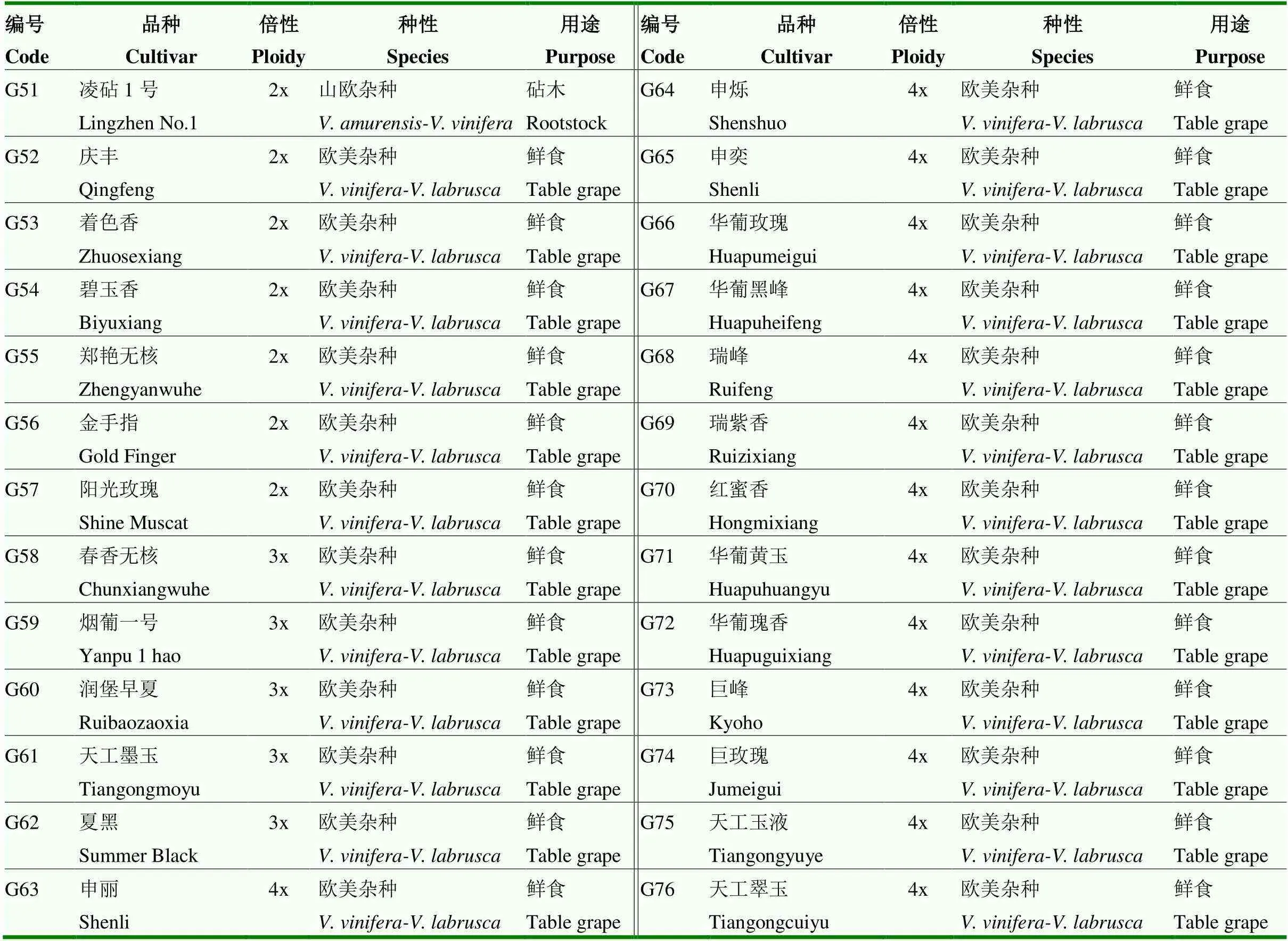
續表2 Continued table 2
1.2 DNA提取與檢測
使用愛森生物科技有限公司的植物基因組DNA提取試劑盒,提取葡萄葉片DNA,采用NanoDrop 1000 spectrophotometer(Themo Scientific)紫外分光光度計檢測DNA濃度和純度(OD260/OD280=1.7—2.0),并用超純水將濃度稀釋至50 ng·μL-1,按編號裝入96孔PCR板中,用于高通量擴增檢測[8]。
1.3 KASP標記轉化和引物合成
根據前人研究結果,共選用60個SNP位點作為KASP標記的篩選庫,分別來自CABEZAS等[21]的46個和LAUCOU等[22]的14個SNP位點。為保證篩選的SNP位點具有基因特異性,從Ensemblplants網站下載SNP位點前后各100 bp的葡萄DNA序列(https://plants.ensembl.org/Vitis_vinifera/Gene),首先在NCBI中進行比對(https://blast.ncbi.nlm.nih.gov/ Blast.cgi?PROGRAM=blastn&PAGE_TYPE=BlastSearch&LINK_LOC=blasthome;Standard databases為RefSeq Representative genomens;Organism為wine grape (taxid: 29760)),去除非特異性的位點。然后,將SNP位點信息前后各100 bp的特異性DNA序列在建有LGC公司KASP檢測技術平臺的北京市農林科學院蔬菜改良中心設計并合成引物。其中引物設計要求兩條正向引物的變性溫度在62.5℃左右,溫差不超過1℃,GC含量在50%—55%;反向引物的變性溫度在66℃,GC含量不超過60%。
1.4 PCR擴增與標記檢測
PCR反應體系(10.14 μL)為KASP Master mix 5 μL、KASP Primer mix 0.14 μL和模板DNA(50 ng·μL-1)5 μL。PCR反應條件:第一輪94℃ 15 min;94℃ 20 s,61—55℃ 60 s,10個Touch Down循環(每個循環降低0.6℃);第二輪94℃ 20 s,55℃ 60 s,26個循環。擴增產物使用熒光微孔板檢測儀檢測,然后采用LGC公司開發的SNP viewer 2.0軟件讀取檢測數據[20]。
1.5 數據統計與分析
根據SNP分型結果數據計算相關遺傳多樣性參數,包括MAF(次要等位基因頻率)、PIC(多態信息含量)[20,25]、Heterozygosity(雜合度)、Missing rate(缺失率)等,均在Excel 2019中使用公式進行計算:
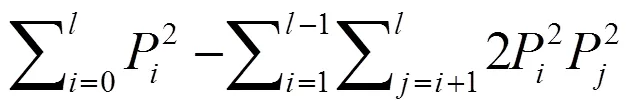
式中,P和P是SNP的兩個等位基因在所有被測品種中的發生頻率,是樣本數量[20]。
依據SNP標記具有的二態性特點,將分型數據轉化為二元編碼數據,野生型(與葡萄基因組一致)表示為(1、0),突變體表示為(0、1),雜合基因型表示為(1、1),缺失位點記為(999、999)[8],然后利用PowerMarker V3.25軟件的鄰接算法(Neighbor- joining)計算76個葡萄品種的遺傳距離[26],并用Figtree v1.4.4構建聚類圖。
使用STRUCTURE 2.3.4進行貝葉斯聚類[27],并確定最佳的類群分組。設定等位變異頻率特征數(遺傳群體數)K=1—15,Burn-in周期為100 000,MCMC的重復次數為100 000次,采用混合模型和相關等位基因頻率,對不同的K值進行15次重復運行,然后將后綴為“_f”的結果文件壓縮,上傳到“STRUCTURE HARVESTER”網站(http://taylor0.biology.ucla.edu/ struct_harvest/),根據Evanno等[28]的方法計算得到?K和似然值的對數函數lnp(D),分別針對基因庫數(K)建模,確定最佳K值。
采用北京市農林科院蔬菜改良中心提供的Perl腳本計算出最優組合標記[20]。
2 結果
2.1 KASP標記轉化結果
對60個SNP位點進行特異性分析,與葡萄參考基因組(Standard databases:RefSeq Representative genomens;Organism:wine grape (taxid: 29760))序列比對后,發現3個非特異性標記,最終57個SNP位點成功設計出51個KASP標記,轉化率為89.47%(附表1)。
2.2 KASP標記的初篩
利用KASP技術對23份葡萄品種進行基因分型,51個KASP標記全部成功分型。其中,MAF值大于0.25的標記有31個,PIC值大于0.35的標記有35個,缺失率小于0.2的標記有44個,雜合率小于0.6的標記有50個,同時滿足以上4個條件的標記有27個,并用于下一步的復篩(圖1)。
2.3 KASP標記的復篩及DNA指紋圖譜構建
使用初篩出的27個KASP標記對76份葡萄品種進行基因分型。結果表明,VIT_3_5724485、VIT_4_680574、VIT_11_5406647、VIT_15_18031506、VIT_18_3829207等5個標記分型結果不具備差異性,并舍棄;其余22個KASP標記在76份樣本中均分型成功(圖2)。其中,22個標記的缺失率均小于0.12,雜合率在0.4—0.6的占77%,PIC值在0.4—0.5的占95%,MAF值大于0.3的占95%(圖3)。此外,使用篩出的22個KASP標記對同一品種不同樹體提取的23份代表性品種的DNA擴增檢測,前后兩次檢測的分型結果一致,表明篩選的22個KASP標記具有較好的重復性和穩定性,可以初步作為一組核心標記用于葡萄的品種鑒定。將這22個標記獲得的76份葡萄品種分型結果轉化為二元編碼數據,得到76份中國主要栽培葡萄品種的SNP指紋圖譜(圖4)。
2.4 76個葡萄品種的聚類分析和群體結構分析
基于22個KASP標記的分型結果,利用PowerMarker V3.25和Figtree v1.4.4軟件繪制76份葡萄品種的聚類圖(圖5-a)。76份葡萄品種可以分為3大類:第Ⅰ大類由19份多倍體歐美雜交品種單獨聚為一大類(圖5-a中標紅色的分枝),其中‘春香無核’‘潤堡早夏’‘天工墨玉’同為‘夏黑’的芽變品種,和‘夏黑’聚在一起;親本之一同為‘紅富士’的‘天工翠玉’‘天工玉液’2份品種也在該大類中,但因另一親本不同,沒有緊密聚在一起;‘巨玫瑰’‘華葡黃玉’‘華葡瑰香’同為‘巨峰’和‘沈陽玫瑰’的雜交后代,和‘巨峰’聚在一起;其他含有‘巨峰’親緣的多倍體品種也與‘巨峰’聚在一起。第Ⅱ大類由39份二倍體品種組成,包含35份歐亞種、3份歐美雜種、1份毛葡萄(圖5-a中標綠色的分枝),其中‘早康寶’和‘麗紅寶’、‘翠香寶’和‘秋黑寶’、‘鄭艷無核’和‘慶豐’、‘云楚無核’和‘岳秀無核’、‘瑞都紅玫’‘瑞都脆霞’‘瑞都香玉’‘瑞都早紅’5組分別出自同一雜交組合后代群體的品種,各自聚類在一起;具有‘玫瑰香’親緣關系的‘脆紅寶’‘晨香’‘卓越玫瑰’‘華葡翠玉’‘華葡早玉’5份品種與‘玫瑰香’聚類在一起;同樣,具有‘紅地球’親緣關系的‘玉波一號’‘玉波黃地球’‘云楚無核’‘岳秀無核’‘華葡翠玉’‘華葡紫峰’‘紅艷無核’7份品種也與‘紅地球’聚類在一起;‘紅特沙’與親本之一‘里扎馬特’緊密聚在一起。第Ⅲ大類主要由11份歐亞種、3份歐美雜種、3份山歐雜種、1份刺葡萄,共18份品種組成,同為二倍體品種(圖5-a中標藍色的分枝),其中‘赤霞珠’‘北冰紅’‘愛格麗’‘媚麗’‘泰美’‘玉玲瓏’6份釀酒葡萄品種聚在該大類中;‘北冰紅’‘凌豐紅’2份出自同一育種單位的山歐雜交品種,和‘惠良刺葡萄’緊密聚類在一起;‘玉波二號’‘短枝玉玫瑰’同為‘達米娜’和‘紫地球’的雜交后代,緊密聚在一起;由‘金手指’和‘紫地球’雜交獲得的‘玉珍香’也與‘金手指’聚在該類群中。
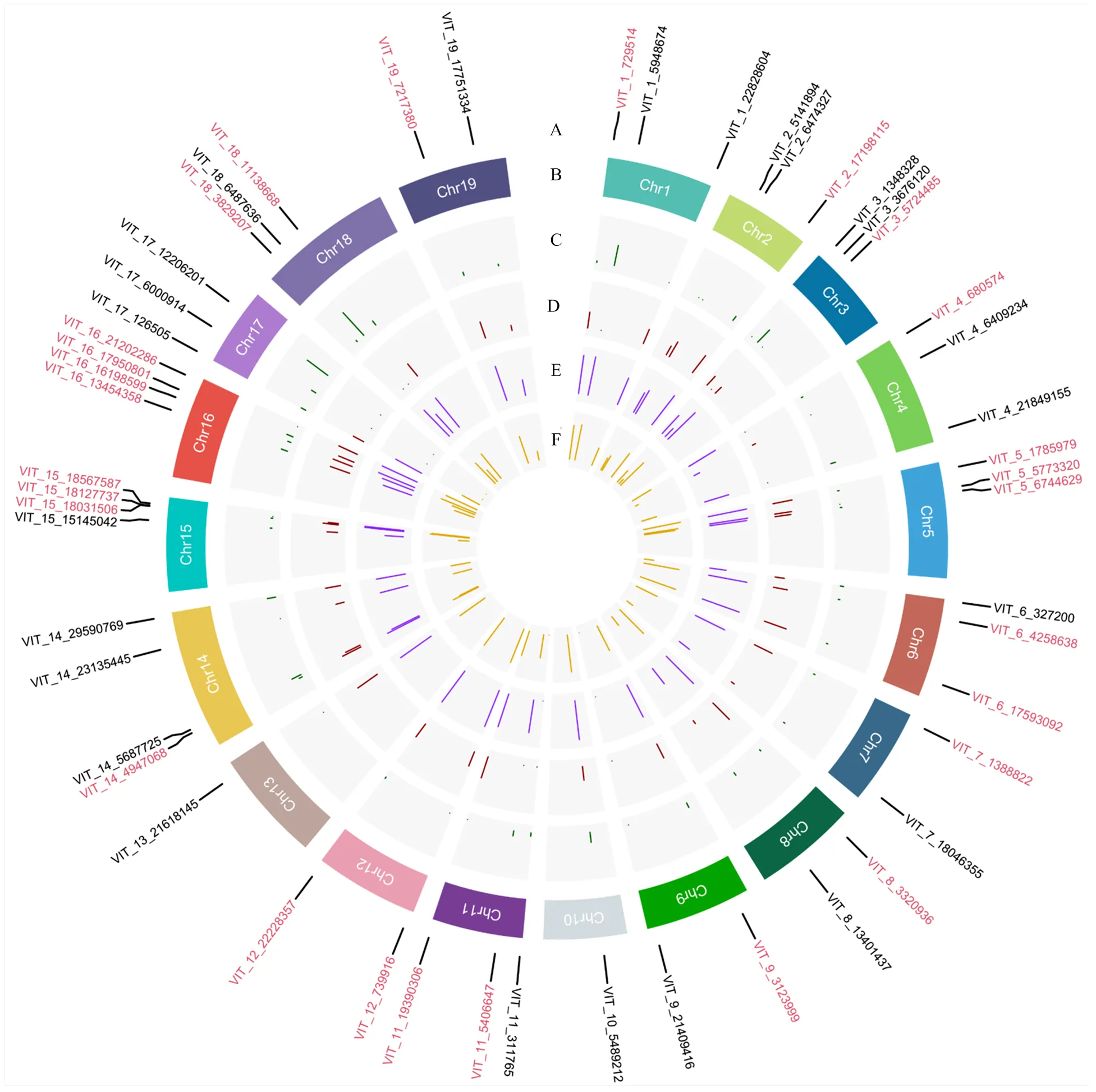
A:51個KASP標記的名稱(紅色字體為初篩出的27個高質量KASP標記);B:葡萄染色體號;C:缺失率;D:雜合率;E:多態性信息含量;F:次要等位基因頻率
用STRUCTURE 2.3.4進行種群遺傳結構相關性分析,根據EVANNO等[28]的方法,用最大似然值?K來確定K值。本研究中當K=3時,?K取得最大值,推斷76份葡萄品種分為3個類群(圖5-b、圖5-c),Ⅰ、Ⅱ、Ⅲ類分別占總群體數的39.47%、38.16%、22.37%,其中Ⅰ類群包含18個歐美雜種品種,占總歐美雜種群體數(25份)的72.00%,Ⅱ和Ⅲ群體中共包含36份歐亞種品種,占總歐亞種群數(46份)的78.26%,與聚類分析結果具有一定的一致性。
2.5 KASP標記在品種鑒定中的應用
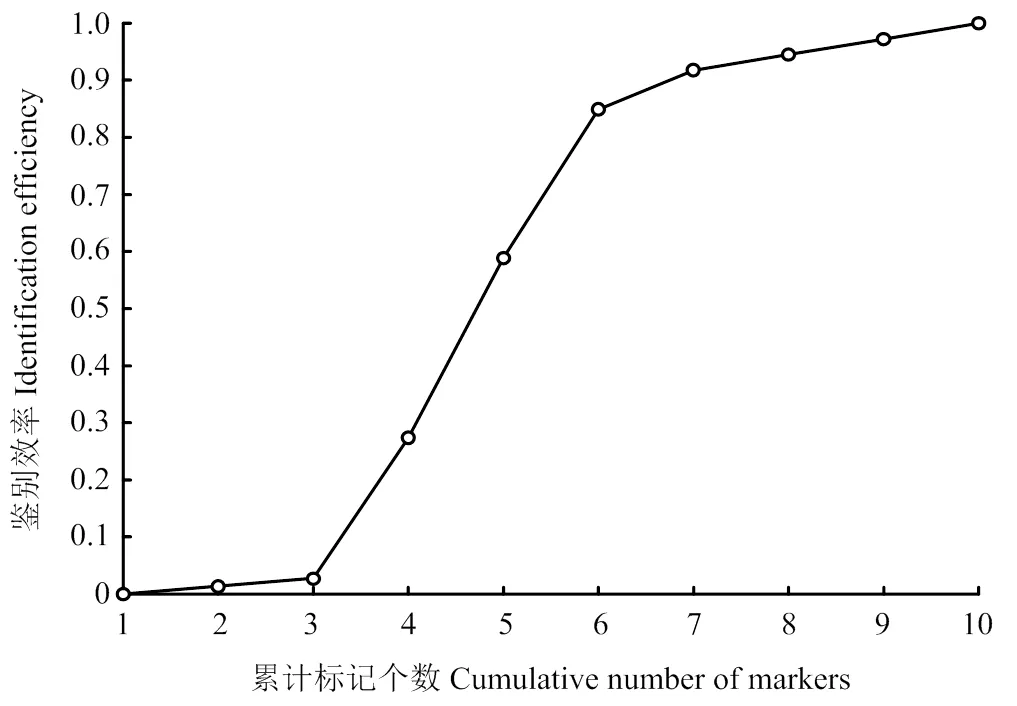
對76份葡萄品種的指紋數據比對分析,發現3組基因型一致的品種分別為:‘夏黑’和‘潤堡早夏’(‘夏黑’的芽變品種),‘天工翠玉’和‘天工墨玉’(同一家育種單位選育的品種),‘里扎馬特’和‘紅特沙’(親本之一為‘里扎馬特’)。采用北京市農林科學院蔬菜改良中心提供的Perl腳本計算出一組僅用10個標記就能區分70份品種的最優組合標記(圖6),有54份葡萄品種達到品種鑒定的標準(差異位點數≥2)[29]。其中,中國主栽的‘紅地球’‘玫瑰香’‘美人指’‘香妃’‘紅寶石無核’‘碧香無核’‘金手指’‘陽光玫瑰’‘赤霞珠’9份葡萄品種含有至少2個KASP標記的基因型可區別于其他品種。新育成的品種中,‘脆紅寶’‘翠香寶’‘早康寶’‘麗紅寶’‘秋黑寶’‘無核翠寶’6份“寶”系列品種,‘瑞都無核怡’‘瑞都科美’‘瑞都脆霞’‘瑞都香玉’‘瑞都早紅’‘瑞都紅玫’6份“瑞都”系列品種,‘玉波一號’‘玉波二號’‘玉珍香’‘短枝玉玫瑰’‘玉波黃地球’5份“玉”系列品種,‘華葡紫峰’‘華葡翠玉’‘華葡早玉’‘華葡黃玉’‘華葡玫瑰’5份“華葡”系列品種,同一親本后代的‘云楚無核’‘岳秀無核’及‘慶豐’‘鄭艷無核’、單親后代的‘MCS2’‘泰美’、山歐雜種的‘北冰紅’‘凌豐紅’‘凌砧1號’、刺葡萄種的‘惠良刺葡萄’以及多倍體‘天工玉液’‘煙葡一號’等品種間均有不少于2個KASP標記的差異。
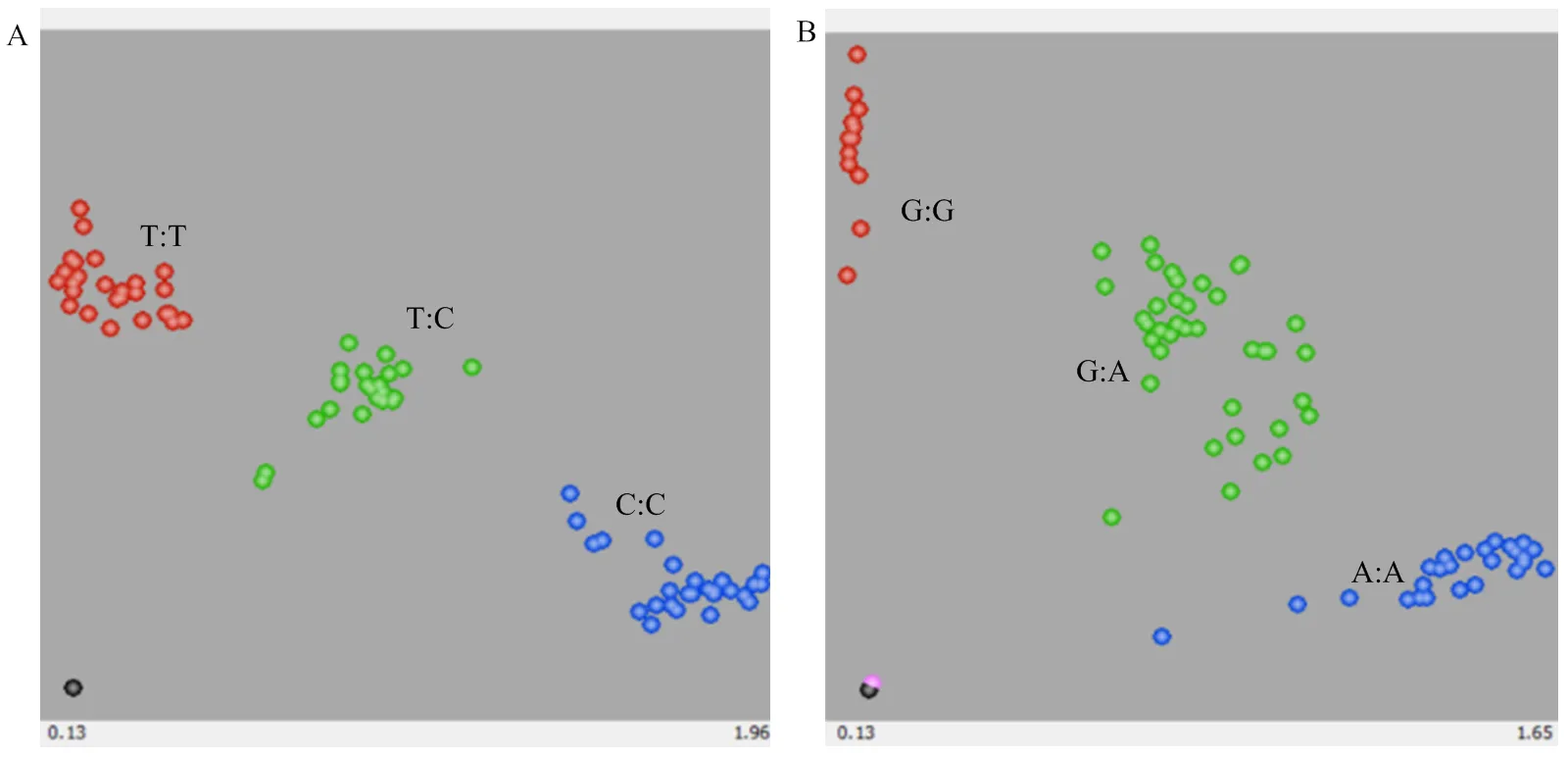
圖2 基于76份葡萄品種,VIT_6_4258638(A)和VIT_16_21202286(B)KASP標記熒光檢測結果
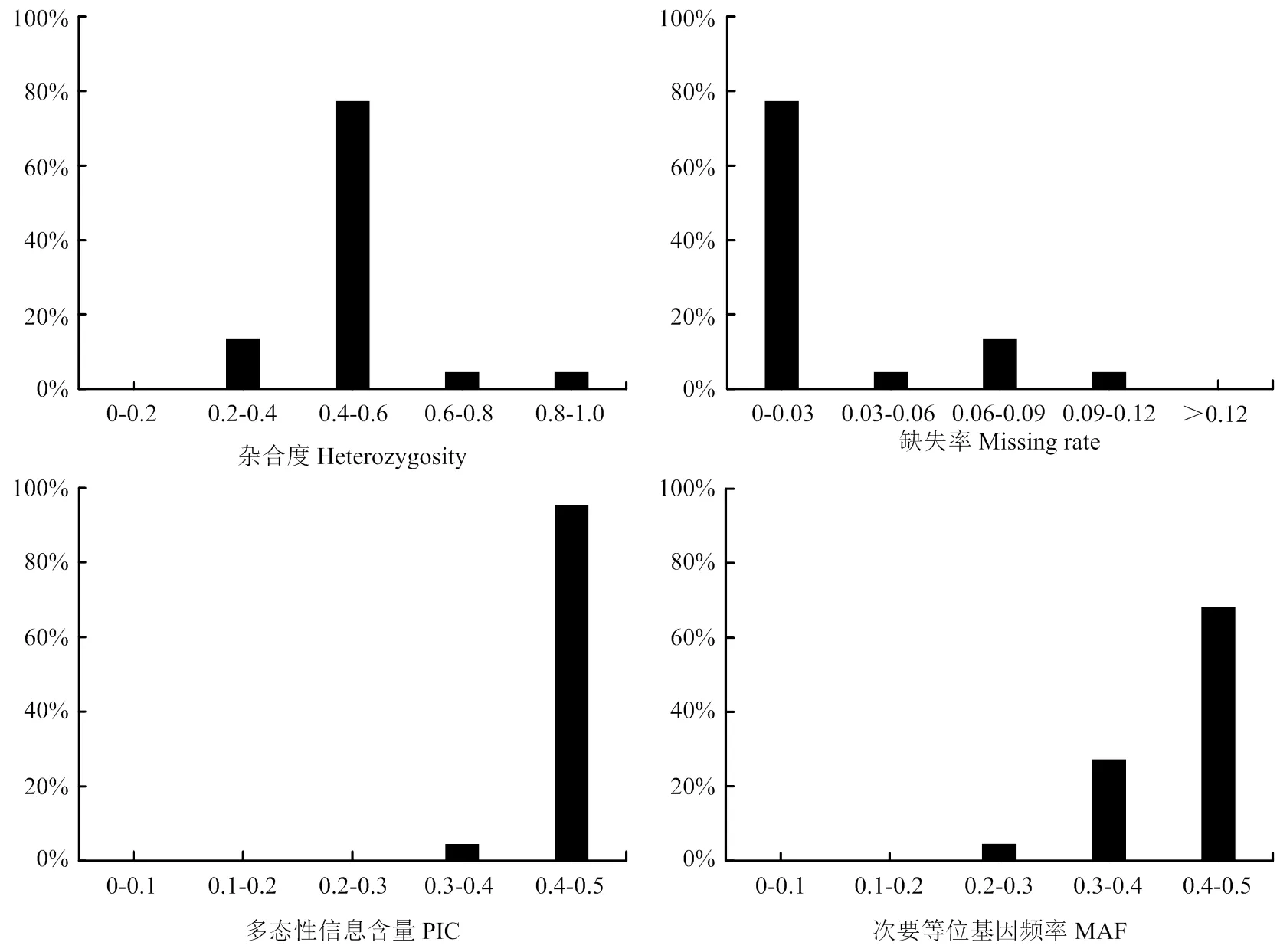
圖3 22個KASP標記基于76份葡萄品種獲得的缺失率、雜合度、多態性信息含量、次要等位基因頻率值在不同范圍內所占百分比
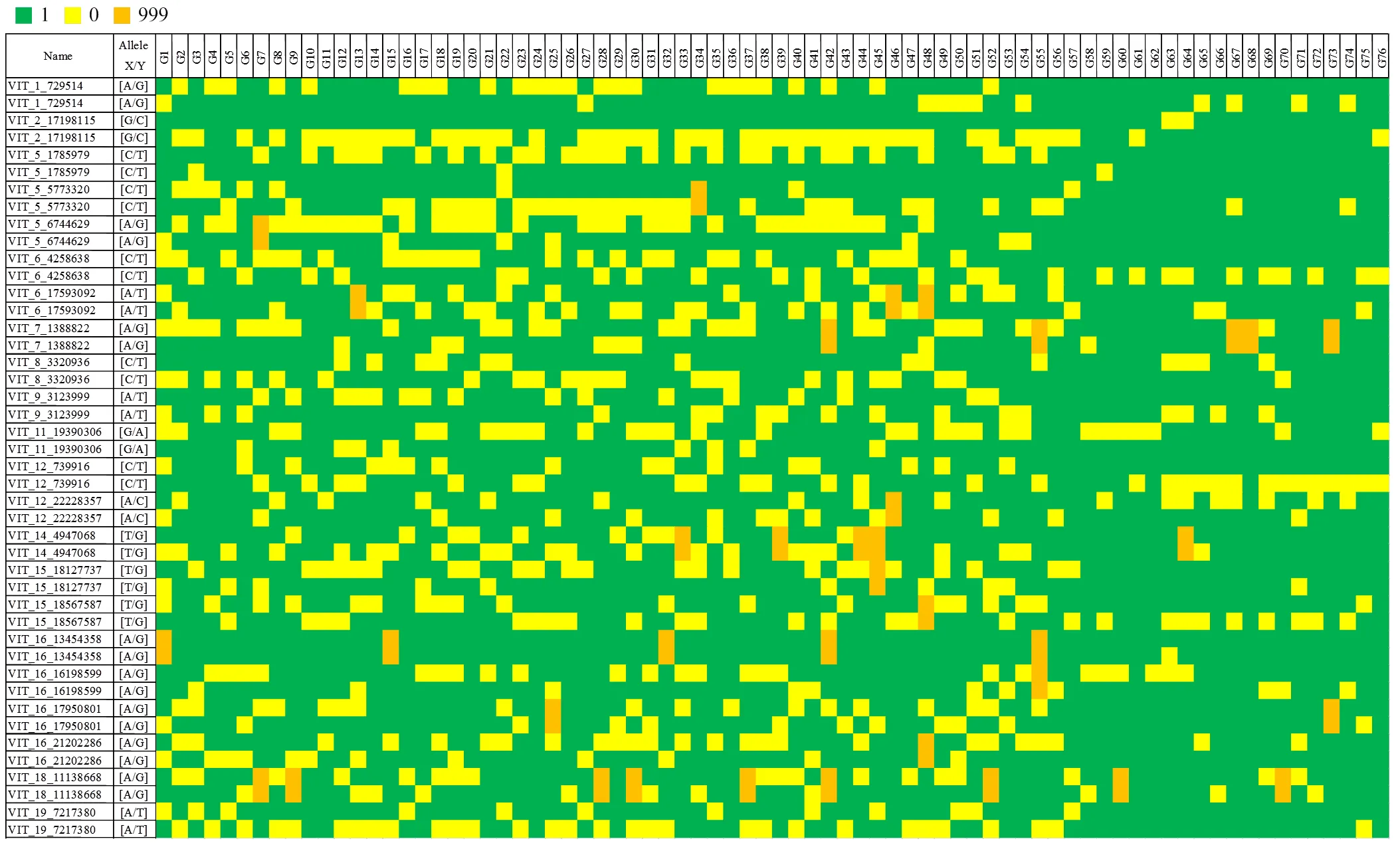
第1列:KASP標記名稱;第2列:KASP標記對應的突變堿基類型;其余列:依次為76個主要栽培葡萄品種的基因型
3 討論
中國作為東亞種群的集中分布區,包含至少38個種[1],保存各類葡萄種質資源達3 000多份[30],且中國新育成的葡萄品種數量還以每年至少20份的速度增長[31]。但由于人們頻繁的引種交流和長期的葡萄傳播過程,同物異名或同名異物的現象時有發生[2],如云南‘水晶’、貴州‘水晶’、關口葡萄實為尼加拉的同物異名品種,鹽井‘黑珍珠’和茨中‘教堂’葡萄實為‘巴柯’的同物異名品種[32]。此外,葡萄無性繁殖能力強,成活率高,育種家的成果一旦被竊取,極易被擴繁、易名、炒作,不僅侵害育種家的利益,而且加大了葡萄苗木市場的管理難度。
構建DNA指紋圖譜技術(DNA-Fingerprinting)對于快速準確的品種鑒定和純度鑒定、維護育種家的利益等具有重大意義[8]。當前,研究者已在水稻[29]、玉米[17]、棉花[33]、甘藍[34]等多種作物中進行了以SSR和SNP核心標記為主的指紋圖譜構建。在葡萄作物上,李貝貝等[6]利用8對引物構建國內最大群體量的SSR指紋圖譜。但SSR標記在實際應用中還存在數量和檢測通量有限、位點存在一定突變率、數據準確讀取費時耗力、無法有效區分芽變品種等不足[15]。與SSR相比,SNP是基于單核苷酸的突變,突變頻率更低,遺傳穩定性更高,批量檢測時平均每個孔位的使用成本比SSR的低1/10—1/20;同時,SNP也是構建DNA指紋數據庫的推薦標記之一,但目前國內還沒有利用SNP標記構建葡萄指紋圖譜的研究[35]。而構建SNP指紋圖譜首先要選擇合適的SNP分型技術。目前,至少有20種SNP分型技術,其中SEMAGN等[14]以玉米為材料,從轉化率、準確性、成本、靈活性等角度對比分析了KASP和Illumina GoldenGate兩種技術,認為KASP基因分型技術在少位點檢測大樣本群體時具有更大的優勢。趙勇等[36]基于53份大豆材料,使用4個SNP比較GBS、CAPS、KASP三種分型方法,也認為KASP技術更具有靈活實用性。劉麗華等[9]同樣認為KASP技術可實現高通量的少量位點檢測大量群體的目的,更適合作物品種鑒定工作。馬麗等[17]、李志遠等[8]、劉寶平等[37]和LI等[38]基于KASP技術分別篩選出22個、59個、48個和50個核心KASP標記,用于玉米、甘藍、番茄和白菜等作物的品種鑒定中。
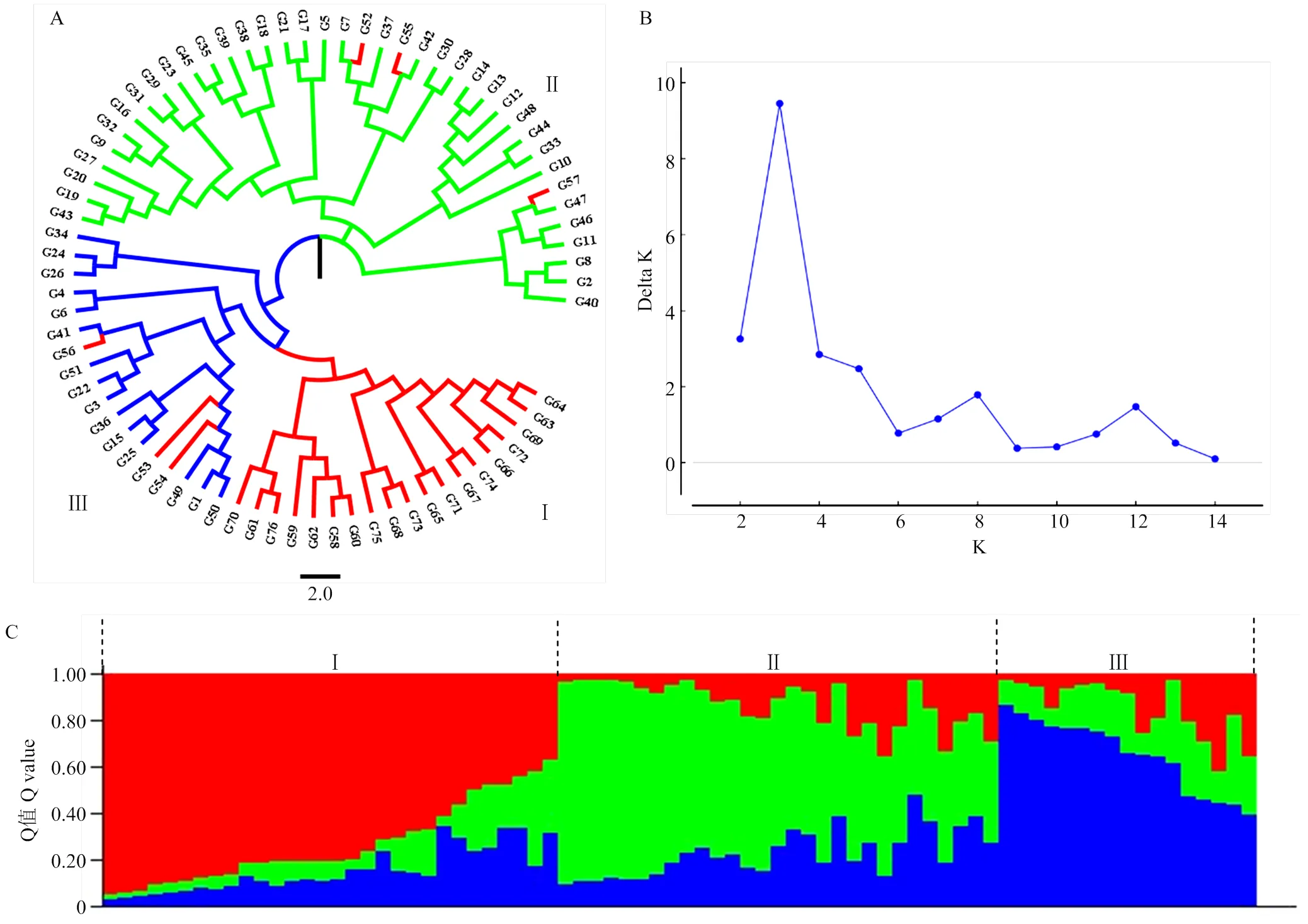
A:鄰接聚類分析;B:K值與?K趨勢變化圖;C:K=3時群體遺傳結構分析圖
1: VIT_15_18567587; 2: VIT_6_4258638; 3: VIT_8_3320936; 4: VIT_12_ 22228357; 5: VIT_11_19390306; 6: VIT_16_17950801; 7: VIT_18_ 11138668; 8: VIT_12_739916; 9: VIT_16_13454358; 10: VIT_16_21202286
本研究基于KASP技術,利用前人篩選的SNP位點,首次嘗試構建我國76份主要栽培葡萄品種的SNP指紋圖譜。基于KASP分型技術的檢測原理,選擇的SNP位點需要在群體中穩定遺傳、具備高多態性。同時,為保證分型結果準確,篩選的標記應具有基因組特異性。本研究使用的SNP位點分別來自CABEZAS等[21]對1 324份葡萄材料有效分型驗證的46個SNPs和LAUCOU等[22]基于18K SNPs葡萄基因芯片對945份材料篩出的14個SNPs,保證了選擇的位點具有足夠的區分能力。
為保證構建的76份葡萄指紋圖譜的真實性,本研究基于檢測獲得的基因型數據進行聚類分析和群體分析,兩種分析結果一致。聚類分析結果表明所有的多倍體歐美雜種起源于二倍體,但又區別于二倍體,在聚類圖中單獨聚為一類。其他二倍體品種分為兩大枝,符合對應的親緣關系,與王富強等[5]基于30對核心SSR引物對52份葡萄品種聚類分析結果具有一致性,首次驗證了KASP分子標記可用于研究多倍體和二倍體葡萄之間的遺傳進化關系,使用KASP標記構建葡萄指紋圖譜具有可行性。
在品種鑒定分析中,因‘春香無核’‘潤堡早夏’‘天工墨玉’同為‘夏黑’的芽變品種,以及親本之一為‘里扎馬特’的‘紅特沙’,遺傳相似度極高,使用22個KASP標記無法有效區分。只有‘無核白雞心’的芽變品種‘科玉無籽’與‘無核白雞心’含有6個純合基因型的差異,說明芽變品種的鑒定依然是當前品種鑒定的難點,需要用更多的標記進行區分。而按照SSR分子標記的品種有效區分標準,本研究獲得的10個標記(VIT_15_18567587、VIT_6_4258638、VIT_8_3320936、VIT_12_22228357、VIT_11_19390306、VIT_16_17950801、VIT_18_11138668、VIT_12_739916、VIT_16_13454358、VIT_16_21202286)可完全區分54份品種,包含了我國主栽的‘紅地球’‘紅寶石無核’‘金手指’‘陽光玫瑰’等品種,以及親緣關系較近的“寶”系列、“瑞都”系列、“玉”系列、“華葡”系列品種,這與使用SSR標記區分相關品種具有一致性[5],表明使用KASP標記對葡萄進行品種鑒定的實用性,且比SSR標記具有更快更準、更易判讀等優勢。
4 結論
從60個葡萄SNP位點,成功轉化51個KASP標記,并篩選出22個高質量KASP標記,構建了76份中國主要栽培葡萄品種的SNP指紋圖譜。經鄰接聚類分析和群體結果分析,可將76份葡萄品種分為3類,并能正確區分開二倍體和多倍體。僅用10個標記就能區分70份品種,其中有54份品種達到品種鑒定的標準(差異位點數≥2),首次驗證了KASP技術在我國葡萄品種鑒定中的可行性。
[1] 孔慶山. 中國葡萄志. 北京: 中國農業科學技術出版社, 2004: 16.
KONG Q S. Chinese Ampelography. Beijing: Chinese Agricultural Science and Technology Press, 2004: 16. (in Chinese)
[2] LI B B, JIANG J F, FAN X C, ZHANG Y, SUN H S, ZHANG G H, LIU C H. Molecular characterization of chinese grape landraces (L.) using microsatellite DNA markers. Hortscience, 2017, 52(4): 533-540.
[3] BUTTON P. The international union for the protection of new varieties of plants (UPOV) recommendations on variety denominations. Proceedings of the Fifth International Symposium on the Taxonomy of Cultivated Plants, 2008, 799: 191-200.
[4] 植物品種鑒定-DNA指紋方法-總則. 中華人民共和國農業行業標準. NY/T 2594—2016.
General guideline for identification of plant varieties using DNA markers. Agricultural Industry Standard of the People’s Republic of China, NY/T 2594—2016. (in Chinese)
[5] 王富強, 李貝貝, 樊秀彩, 張穎, 劉崇懷, 姜建福. 葡萄品種SSR分子鑒定體系的建立及應用. 果樹學報, 2020, 37(9): 1281-1293.
WANG F Q, LI B B, FAN X C, ZHANG Y, LIU C H, JIANG J F. Establishment and application of SSR molecular identification system in grapevine.Journal of Fruit Science, 2020, 37(9): 1281-1293. (in Chinese)
[6] 李貝貝, 姜建福, 張穎, 樊秀彩, 孫海生, 張國海, 劉崇懷. 葡萄品種DNA指紋數據庫的構建及遺傳多樣性分析. 植物遺傳資源學報, 2018, 19(2): 338-350.
LI B B, LIU C H, JIANG J F, ZHANG Y, FAN X C, SUN H S, ZHANG G H, LIU C H. DNA fingerprinting and genetic diversity analysis of grape (L.) cultivars based on SSR markers. Journal of Plant Genetic Resources, 2018, 19(2): 338-350. (in Chinese)
[7] RASHEED A, HAO Y F, XIA X C, KHAN A, XU Y B, VARSHNEYR K, HE Z H. Crop breeding chips and genotyping platforms: Progress, challenges, and perspectives. Molecular Plant, 2017, 10(8): 1047-1064.
[8] 李志遠, 于海龍, 方智遠, 楊麗梅, 劉玉梅, 莊木, 呂紅豪, 張揚勇. 甘藍SNP標記開發及主要品種的DNA指紋圖譜構建. 中國農業科學, 2018, 51(14): 2771-2787.
LI Z Y, YU H L, FANG Z Y, YANG L M, LIU Y M, ZHUANG M, LV H H, ZHANG Y Y. Development of SNP markers in cabbage and construction of DNA fingerprinting of main varieties. Scientia Agricultura Sinica, 2018, 51(14): 2771-2787. (in Chinese)
[9] 劉麗華, 龐斌雙, 劉陽娜, 李宏博, 王娜, 王拯, 趙昌平. 基于SNP標記的小麥高通量身份鑒定模式. 麥類作物學報, 2018, 38(5): 529-534.
LIU L H, PANG B S, LIU Y N, LI H B, WANG N, WANG Z, ZHAO C P. High-throughput identification mode for wheat varieties based on SNP markers. Journal of Triticeae Crops, 2018, 38(5): 529-534. (in Chinese)
[10] YANG G L, CHEN S P, CHEN L K, SUN K, HUANG C H, ZHOU D H, HUANG Y T, WANG J F, LIU Y Z, WANG H, CHEN Z Q, GUO T. Development of a core SNP arrays based on the KASP method for molecular breeding of rice. Rice, 2019, 12(1): 21.
[11] SUN C W, DONG Z D, ZHAO L, REN Y, ZHANG N, CHEN F. The wheat 660K SNP array demonstrates great potential for marker- assisted selection in polyploid wheat. Plant Biotechnology Journal, 2020, 18(6): 1354-1360.
[12] CHEN H D, XIE W B, HE H, YU H H, CHEN W, LI J, YU R B, YAO Y, ZHANG W H, HE Y Q, TANG X Y, ZHOU F S, DENG X W, Zhang Q F. A high-density SNP genotyping array for rice biology and molecular breeding. Molecular Plant, 2014, 7(3): 541-553.
[13] XU C, REN Y H, JIAN Y Q, GUO Z F, ZHANG Y, XIE C X, FU J J, WANG H W, WANG G Y, XU Y B, LI P, ZOU C. Development of a maize 55 K SNP array with improved genome coverage for molecular breeding. Molecular Breeding, 2017, 37(3): 1-12.
[14] SEMAGN K, BABU R, HEARNE S, OLSEN M. Single nucleotide polymorphism genotyping using kompetitive allele specific PCR (KASP): Overview of the technology and its application in crop improvement.Molecular Breeding, 2014, 33(1): 1-14.
[15] RASHEED A, WEN W E, GAO F M, ZHAI S N, JIN H, LIU J D, GUO Q, ZHANG Y J, DREISIGACKER S, XIA X C, HE Z H. Development and validation of KASP assays for genes underpinning key economic traits in bread wheat. Theoretical and Applied Genetics, 2016, 129(10): 1843-1860.
[16] GREWAL S, HUBBART-EDWARDS S, YANG C Y, DEVI U, BAKER L, HEATH J, ASHLING S, SCHOLEFIELD D, HOWELLS C, YARDE J, ISAAC P, KING I P, KING J. Rapid identification of homozygosity and site of wild relative introgressions in wheat through chromosome-specific KASP genotyping assays. Plant Biotechnology Journal, 2020, 18(3): 743-755.
[17] 馬麗, 鐘敬, 應繼鋒, 沈志成, 劉法新, 張立陽, 鄭秀婷, 張先文, 阮祥經, 林海艷, 傅軍, 劉歡. 基于KASP技術開發的玉米核心SNP標記及其應用. ZL201910623802.0. 2019-09-20.
MA L, ZHONG J, YING J F, SHEN Z C, LIU F X, ZHANG L Y, ZHENG X T, ZHANG X W, YUAN X J, LIN H Y, FU J, LIU H. Corn core SNP markers based on KASP technology and their application. ZL201910623802.0. 2019-09-20. (in Chinese)
[18] 匡猛, 王延琴, 周大云, 馬磊, 方丹, 徐雙嬌, 楊偉華, 魏守軍, 馬峙英. 基于單拷貝SNP標記的棉花雜交種純度高通量檢測技術. 棉花學報, 2016, 28(3): 227-233.
KUANG M, WANG Y Q, ZHOU D Y, MA L, FANG D, XU S J, YANG W H, WEI S J, MA Z Y. High-throughput genotyping assay technology for cotton hybrid purity based on single-copy SNP markers. Cotton Science, 2016, 28(3): 227-233. (in Chinese)
[19] LI Z Y, YU H L, LI X, ZHANG B, REN W J, LIU X P, FANG Z Y, YANG L M, ZHUANG M, LV H H, ZHANG Y Y. Kompetitive allele-specific PCR (KASP) genotyping and heterotic group classification of 244 inbred lines in cabbage (L. var.). Euphytica: International Journal of Plant Breeding, 2020, 216: 1086-1099.
[20] ZHANG J, YANG J J, ZHANG L K, LUO J, ZHAO H, ZHANG J N, WEN C L. A new SNP genotyping technology target SNP-seq and its application in genetic analysis of cucumber varieties. Scientific Reports, 2020, 10(1): 275-305.
[21] CABEZAS J A, IBá?EZ J, LIJAVETZKY D, VéLEZ D, BRAVO G, RODRíGUEZ V, CARRE?O I, JERMAKOW A M, CARRE?O J, RUIZ-GARCíA L, THOMAS M R, MARTINEZ-ZAPATER J M. A 48 SNP set for grapevine cultivar identification. BMC Plant Biology, 2011, 11(1): 153.
[22] LAUCOU V, LAUNAY A, BACILIERI R, LACOMBE T, ADAM- BLONDON AF, BERARD A, CHAUVEAU A, DE-ANDRES M T, HAUSMANN L, IBANEZ J, LE PASLIER M C, MAGHRADZE D, MARTINEZ-ZAPATER J M, MAUL E, PONNAIAH M, TOPFER R, PEROS J P, BOURSIQUOT J M. Extended diversity analysis of cultivated grapevinewith 10K genome-wide SNPs. PloS ONE, 2018, 13(2): e0192540.
[23] 李貝貝, 張恒, 姜建福, 張穎, 樊秀彩, 房經貴, 劉崇懷. 基于SLAF-seq技術的葡萄種質遺傳多樣性分析. 園藝學報, 2019, 46(11): 2109-2118.
LI B B, ZHANG H, JIANG J F, ZHANG Y, FAN X C, FANG J G, LIU C H. Analysis of genetic diversity of grape germplasms using SLAF-seq technology. Acta Horticulturae Sinica, 2019, 46(11): 2109-2118. (in Chinese)
[24] LIANG Z C, DUAN S C, SHENG J, ZHU S S, NI X M, SHAO J H, LIU C H, NICK P, DU F, FAN P G, MAO R Z, ZHU Y F, DENG W P, YANG M, HUANG H C, LIU Y X, DING Y Q, LIU X J, JIANG J F, ZHU Y Y, LI S H, HE X H, CHEN W, DONG Y. Whole-genome resequencing of 472accessions for grapevine diversity and demographic history analyses. Nature Communications, 2019, 10(1): 511-519
[25] BOTSTEIN D, WHITE R L, SKOLNICK M, DAVIS R W. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314-331.
[26] 趙久然, 李春輝, 宋偉, 王元東, 張如養, 王繼東, 王鳳格, 田紅麗, 王蕊. 基于SNP芯片揭示中國玉米育種種質的遺傳多樣性與群體遺傳結構. 中國農業科學, 2018, 51(4): 626-634.
ZHAO JR, LI C H, SONG W, WANG Y D, ZHANG R Y, WANG J D, WANG F G, TIAN H L, WANG R. Genetic diversity and population structure of important chinese maize breeding germplasm revealed by SNP-chips. Scientia Agricultura Sinica, 2018, 51(4): 626-634. (in Chinese)
[27] FALUSH D, STEPHENS M, PRITCHARD J K. Inference of population structure using multilocus genotype data: linked loci and correlated allele frequencies. Genetics, 2003, 164(4): 1567-1587.
[28] EVANNO G, REGNUAT S, GOUSET J. Detecting the number of clusters of individuals using the software structure: A simulation study. Molecular Ecology, 2005, 14(8): 2611-2620.
[29] 水稻品種鑒定-DNA指紋方法. 中華人民共和國農業行業標準. NY/T 1433—2007.
Identification of rice (L.) varieties using microsatellite markers. Agricultural Industry Standard of the People’s Republic of China, NY/T 1433—2007. (in Chinese)
[30] 段長青, 劉崇懷, 劉鳳之, 王忠躍, 劉延琳, 徐麗明. 新中國果樹科學研究70年—葡萄. 果樹學報, 2019, 36(10): 1292-1301.
DUAN C Q, LIU C H, LIU F Z, WANG Z Y, LIU Y L, XU L M. Fruit scientific research in new China in the past 70 years: grape. Journal of Fruit Science, 2019, 36(10): 1292-1301. (in Chinese)
[31] 孟聚星, 姜建福, 張國海, 孫海生, 樊秀彩, 張穎, 吳久赟, 劉崇懷. 我國育成的葡萄新品種系譜分析. 果樹學報, 2017, 34(4): 393-409.
MENG J X, JIANG J F, ZHANG G H, SUN H S, FAN X C, ZHANG Y, WU J Y, LIU C H. Pedigree analysis of grape cultivars released in China. Journal of Fruit Science, 2017, 34(4): 393-409. (in Chinese)
[32] 李貝貝, 張穎, 樊秀彩, 李民, 劉崇懷, 姜建福. ‘巴柯’和‘尼加拉’葡萄疑似同物異名品種的SSR及形態學分析. 果樹學報, 2019, 36(4): 393-400.
LI B B, ZHANG Y, FAN X C, LI M, LIU C H, JIANG J F. Analysis of suspected synonyms of ‘Baco Noir’ and ‘Niagara’ by SSR markers and morphology. Journal of Fruit Science, 2019, 36(4): 393-400. (in Chinese)
[33] 匡猛, 楊偉華, 許紅霞, 王延琴, 周大云, 馮新愛. 中國棉花主栽品種DNA指紋圖譜構建及SSR標記遺傳多樣性分析. 中國農業科學, 2011, 44(1): 20-27.
KUANG M, YANG W H, XU H X, WANG Y Q, ZHOU D Y, FENG X A. Construction of DNA Fingerprinting and analysis of genetic diversity with SSR markers for cotton major cultivars in China. Scientia Agricultura Sinica, 2011, 44(1): 20-27. (in Chinese)
[34] 結球甘藍品種鑒定技術規程-SSR分子標記法. 中華人民共和國農業行業標準. NY/T 2473—2013.
Protocol for identification of cabbage varieties SSR marker method. Agricultural Industry Standard of the People’s Republic of China, NY/T 2473—2013. (in Chinese)
[35] 王富強, 樊秀彩, 張穎, 劉崇懷, 姜建福. SNP分子標記在作物品種鑒定中的應用和展望. 植物遺傳資源學報, 2020, 21(5): 1308-1320.
WANG F Q, FAN X C, ZHANG Y, LIU C H, JIANG J F. Application and prospect of SNP molecular markers in crop variety identification. Journal of Plant Genetic Resources, 2020, 21(5): 1308-1320. (in Chinese)
[36] 趙勇, 劉曉東, 趙洪錕, 袁翠平, 齊廣勛, 王玉民, 董英山. 大豆SNP分型方法的比較. 分子植物育種, 2017, 15(9): 3540-3546.
ZHAO Y, LIU X D, ZHAO H K, YUAN C P, QI G X, WANG Y M, DONG Y S. Comparison of methods for SNP genotyping in soybean. Molecular Plant Breeding, 2017, 15(9): 3540-3546. (in Chinese)
[37] 劉寶平, 王均帥, 焦珍珍, 邢海星, 張東峰, 馬睿, 趙小翠, 尹增奇, 吳坤生. 一套適用于番茄DNA指紋庫構建的KASP引物組合及其應用. ZL201910916095.4. 2019-12-24.
LIU B P, WANG J S, JIAO Z Z, XING H X, ZHANG D F, MA R, ZHAO X C, YIN Z Q, WU K S. A set of KASP markers combinations suitable for the construction of tomato DNA fingerprint library and their application. ZL201910916095.4. 2019-12-24. (in Chinese)
[38] LI P R, SU T B, WANG H P, ZHAO X Y, WANG W H, YU Y J, ZHANG D S, WEN C L, YU S C, ZHANG F L. Development of a core set of KASP markers for assaying genetic diversity insubsp.Makino. Plant Breeding, 2019, 138: 309-324.
Identification of Grape Cultivars Based on KASP Markers
WANG FuQiang1, ZHANG Jian2, WEN ChangLong2, FAN XiuCai1, ZHANG Ying1, SUN Lei1, LIU ChongHuai1, JIANG JianFu1
1Zhengzhou Fruit Research Institute, Chinese Academy of Agricultural Sciences, Zhengzhou 450009;2Beijing Vegetable Research Center, Beijing Academy of Agricultural and Forestry Sciences, Beijing 100097
【】This study was aimed to develop a set of kompetitive allele specific PCR (KASP) markers that can be used to distinguish the main cultivated grape cultivars in China, so as to provide the technical support for domestic grape cultivar protection, cultivar registration and market right protection. 【】60 highly polymorphic SNP loci from previous studies in grapes were selected to design KASP markers. 23 representative grape cultivars and 76 main cultivated grape cultivars in China were used for preliminary screening, re-screening, and verification of the successfully transformed KASP markers. A set of high-quality KASP markers was further selected to establish the DNA fingerprint database for the 76 grape cultivars. 【】Among the 60 SNP loci, 3 were not genome-specific, 6 could not be used to design KASP-PCR markers, and 51 could be successfully transformed to KASP markers with a conversion rate of 89.47%. Next, 23 representative grape cultivars were successfully genotyped by using the 51 KASP markers on LGC-SNP line platform. Then, 27 KASP markers of high quality were selected successfully based on the minor allele frequency (MAF) greater than 0.25, polymorphism information content (PIC) greater than 0.35, deletion rate less than 0.2, and heterozygosity rate less than 0.6. Finally, 22 KASP markers were successfully re-screened in the 76 main cultivated grape cultivars. The missing rate of the 22 markers was less than 0.12, the PIC value of them was greater than 0.30, the heterozygosity rate of 0.40-0.60 was 77.27%, and the MAF of greater than 0.30 was 95.45%. In addition, the consistent genotype data were acquired with different trees from each of the 23 representative cultivars by using the 22KASP markers, indicating that these 22 KASP markers had good reproducibility and stability for grape cultivar identification. The SNP genotyping results of 76 grape cultivars based on 22 markers were converted to binary coding data, and the fingerprints of 76 grape cultivars were obtained. Based on neighbor-joining cluster analysis, the 76 grape cultivars could be divided into 3 populations. Diploid and polyploidy cultivars were also correctly identified. The 10 markers (VIT_15_18567587, VIT_6_4258638, VIT_8_3320936, VIT_12_22228357, VIT_11_19390306, VIT_16_17950801, VIT_18_11138668, VIT_12_739916, VIT_16_ 13454358, VIT_16_21202286) were enough to be able to distinguish 70 of the 76 cultivars, of which 54 cultivars met the criteria for variety identification (the number of different loci ≥2). 【】From 60 grape SNPs, 51 KASP markers were successfully transformed, and 22 high-quality KASP markers were selected successfully for developing the fingerprints of the 76 main grape cultivars in China. It was the first time to verify the feasibility of KASP genotyping in the identification of grape cultivars in China.
grape; KASP marker; cultivar identification; fingerprint; genetic diversity analysis

10.3864/j.issn.0578-1752.2021.13.012
2020-08-26;
2021-01-23
國家重點研發計劃(2019YFD1001401)、國家現代農業產業技術體系(CARS-29-yc-1)、中國農業科學院科技創新工程(CAAS-ASTIP- 2019-ZFRI)
王富強,E-mail:82101185031@caas.cn。通信作者姜建福,E-mail:jiangjianfu@caas.cn
(責任編輯 趙伶俐)

