東海溫州水域4類常見耐藥基因的流行情況研究*
金晶磊 周前進① 邵鑫斌 王瑤華 陳 炯① 閆茂倉 苗 亮 唐 標 張嚴峻
東海溫州水域4類常見耐藥基因的流行情況研究*
金晶磊1, 2周前進1, 2①邵鑫斌3王瑤華3陳 炯1, 2①閆茂倉3苗 亮2唐 標4張嚴峻5
(1. 寧波大學 農產品質量安全危害因子與風險防控國家重點實驗室 寧波 315211; 2. 寧波大學海洋學院 寧波 315832; 3. 浙江省海洋水產養殖研究所 浙江省近岸水域生物資源開發與保護重點實驗室 溫州 325005; 4. 浙江省農業科學院 農產品質量標準研究所 杭州 310021; 5. 浙江省疾病預防控制中心 杭州 310051)
磺胺類、四環素、喹諾酮類和多粘菌素抗生素是醫學與養殖業中的常用抗生素, 其中多粘菌素在畜牧養殖業中常作為促長劑添加。調查分析了這4類抗生素的常見耐藥基因在東海溫州近海水域的分布情況。利用抗性平板篩選溫州近海水域對磺胺類、四環素類及喹諾酮類抗生素耐藥的可培養細菌分離株, 對耐藥分離株通過PCR方法進行目標耐藥基因檢測; 對目標耐藥基因陽性的耐藥分離株進行多粘菌素耐藥基因檢測以及采用微量二倍稀釋法測定對上述4類抗生素的耐藥性。結果表明, 利用抗性平板篩選獲得可培養耐藥分離株1 605株, 從中篩選到目標耐藥基因陽性的51株, 檢出率為3.18%, 包括含磺胺類耐藥基因35株、含四環素耐藥基因36株、含喹諾酮耐藥基因17株、含多粘菌素耐藥基因2株; 51株耐藥菌歸于7個菌屬, 其中氣單胞菌屬(sp) 43株, 占比84.31%, 枸櫞酸桿菌屬(sp)和希瓦氏菌屬(sp)各2株, 埃希氏菌屬(sp.)、類芽孢桿菌屬(sp.)、sp.及sp.各1株; 上述2株非常見分離株不統計在內, 常見的49株均對四環素耐藥, 41株對多粘菌素耐藥, 對磺胺類和喹諾酮類耐藥的分離株均為34株; 同時, 發現23株對4類抗生素均產生耐藥, 16株對多達3種抗生素耐藥, 多重耐藥現象普遍存在。同時, 鑒定出陽性的條件致病菌維氏氣單胞菌和豚鼠氣單胞菌各1株。綜上, 溫州市近海水域中含有常見耐藥基因的耐藥分離株檢出率較低, 但同一耐藥基因的細菌分布多樣, 對磺胺類、四環素類、喹諾酮類及多粘菌素類等4類抗生素表現出較強的耐藥性, 多重耐藥現象明顯。研究結果將為評估溫州近海水域的細菌耐藥性風險, 規范抗生素的使用提供科學依據。
東海溫州水域; 耐藥基因; 多重耐藥;
由于抗生素的常年使用, 未代謝的抗生素在環境中積累和轉化, 對環境中的微生物進行持續不斷的選擇, 造成耐藥基因與耐藥菌的流行, 對公共衛生與生態環境造成了巨大的威脅(Arias, 2009; Kennedy, 2013; Willyard, 2017)。海洋作為地球上耐藥基因與耐藥菌的主要儲存庫之一, 不但自身擁有豐富的耐藥基因與耐藥菌, 其他來源的耐藥菌與耐藥基因還可能通過雨水沖刷、潮汐、污水排放等方式進入海洋環境(Chen, 2013; Hatosy, 2015)。近海水域作為人類活動的主要區域之一, 來自于畜牧與水產養殖、臨床醫療、工業生產活動的抗生素殘留、耐藥菌、耐藥基因等污染物可通過一系列方式傳入其中, 增加近海水域中耐藥基因與耐藥菌的種類與豐度, 對海洋生態環境造成危害。此外, 耐藥基因還可能通過菌種之間的遺傳物質傳遞從而傳入海洋致病菌中, 增加了海洋耐藥病原菌病害發生與流行的可能性(Normark, 2002)。
溫州海域面積約11 000 km2, 海岸線長355 km, 擁有436個島嶼, 是我國重要的海水養殖區, 做好溫州近海水域耐藥基因的監控與評估, 對溫州海水養殖環境及公共衛生健康均有重要的意義。針對溫州地區陸地環境中耐藥基因的流行情況, 有學者進行過一定的調查分析。嚴春等(2016)從醫院重癥監護室中分離到攜帶有、、等超廣譜β-內酰胺酶基因的肺炎克雷伯菌; Liao等(2020)從病患體內分離的大腸桿菌中檢測到多粘菌素耐藥基因; Lu等(2017)則通過熒光定量PCR方法從豬糞中檢測到磺胺類、四環素類、喹諾酮類抗生素的耐藥基因及多種β-內酰胺酶基因。同時, 也有個別學者對溫州海洋水體環境和海產品中耐藥基因的分布情況做過調查。Chen等(2017)調查發現溫州海水養殖水域有著較高的磺胺類抗生素殘留, 利用熒光定量PCR方法檢測到較高豐度基因污染; 吳越等(2020)對溫州水產品上分離的哈維氏弧菌進行耐藥分析, 從中檢測到了磺胺類抗生素耐藥基因和, 四環素耐藥基因、及, 喹諾酮類耐藥基因等, 且多數菌株出現多重耐藥現象。本研究通過抗性平板法于溫州近海水域海水中分別篩選了磺胺類、四環素類、喹諾酮類抗生素抗性的可培養耐藥菌分離株, 通過PCR方法檢測耐藥菌對應的耐藥基因攜帶情況, 在含有目標耐藥基因的耐藥菌分離株中進一步測試多粘菌素耐藥基因的污染情況, 綜合分析四類抗生素耐藥基因在溫州近海水域的分布; 利用16S rDNA測序, 鑒定耐藥菌分離株的種屬特性, 分析耐藥基因在不同種屬細菌中的分布; 同時, 通過微量二倍稀釋法測定耐藥菌對受試抗生素的最小抑菌濃度(Minimal Inhibit Concentration, MIC), 分析其多重耐藥現象, 研究將為評估溫州近海水域的細菌耐藥性風險, 規范抗生素的使用提供科學依據。
1 材料與方法
1.1 樣品采集
于2019—2020年冬、春兩季, 沿東海溫州近海水域, 選擇25個采樣點進行海水樣品的采集(表1)。每個采樣地點以采水器采集表層海水樣品(采樣深度: 水面下15 cm) 1 500 mL置于2.5 L的無菌聚乙烯扁方桶中。采集的海水樣品在16—20 °C下運輸并貯存, 用于后續的實驗室分析。
表1 溫州近海水域海水樣品的采樣點

Tab.1 Location of seawater sampling sites off Wenzhou City
1.2 耐藥分離株培養
每份樣品, 分別取500 mL海水樣品, 經過0.22 μm的無菌混合纖維素濾膜(生工生物工程(上海)股份有限公司, 中國)過濾以收集細菌。在無菌條件下將濾膜剪碎, 放置于10 mL的無菌磷酸緩沖鹽溶液(Phosphate Buffer Saline, PBS)中, 經渦旋振蕩器充分震蕩后, 吸取50 μL分別涂布于含有不同種類抗生素(磺胺類: 4 μg/mL甲氧芐啶; 四環素類: 16 μg/mL四環素; 喹諾酮類: 1 μg/mL環丙沙星)的2216E瓊脂(青島海博生物技術有限公司, 中國)平板上, 并置于28 °C下培養16 h。根據菌落的形態、顏色、大小等形態學特征于每個平板上差異挑選一定數量的單菌落分別接種至2216E液體培養基(青島海博生物技術有限公司, 中國)中過夜培養。由于某些細菌生長較為緩慢, 此后每隔12 h進行菌株的補篩。耐藥分離株使用終濃度25%的甘油保存于–80 °C用于后續實驗。
1.3 目標耐藥基因與16S rDNA的分子鑒定
針對上述3類抗生素, 我們分別選擇了在海洋環境中較為普遍的耐藥基因, 對收集到的耐藥分離株進行耐藥基因的鑒定(表2), 然后對耐藥基因陽性的耐藥分離株進行其他兩類抗生素耐藥基因的鑒定。在前期實驗中, 無法利用抗性平板從多粘菌素耐藥的分離株鑒定到耐藥基因, 而文獻報道攜帶基因的多重耐藥質粒的水平傳播是引起基因流行的主要原因(Sun, 2018), 因此, 我們對磺胺類、四環素類和喹諾酮類耐藥基因陽性的耐藥分離株進行基因的測定, 以期能夠獲得多粘菌素類抗生素耐藥基因的流行情況。耐藥基因陽性的耐藥分離株通過16S rDNA序列測定進一步確定其種屬, 引物信息見表2。鑒定過程的操作簡述如下: 將步驟1.2中凍存的耐藥分離株復蘇后, 使用煮沸法粗提細菌的基因組DNA。利用PCR方法, 按照文獻提供的方法克隆耐藥分離株的對應耐藥基因片段(Lane, 1991; Aminov2001; Ng, 2001; Park, 2006; Hoa, 2008; Rutgersson, 2014; Rebelo, 2018; Borowiak, 2020)。PCR產物經過1.5%的瓊脂糖凝膠電泳檢測后送至上海生工生物工程有限公司測序, 測序結果上傳至NCBI的BLAST檢索系統進行同源性對比分析。對含有目標耐藥基因的耐藥分離株, 按照文獻方法擴增16S rDNA基因序列, PCR產物經上述方法鑒定后送至上海生工生物工程有限公司進行測序, 結果同樣上傳至NCBI的BLAST檢索系統進行同源性對比分析。
表2 本研究所用引物

Tab.2 Primers used in this study
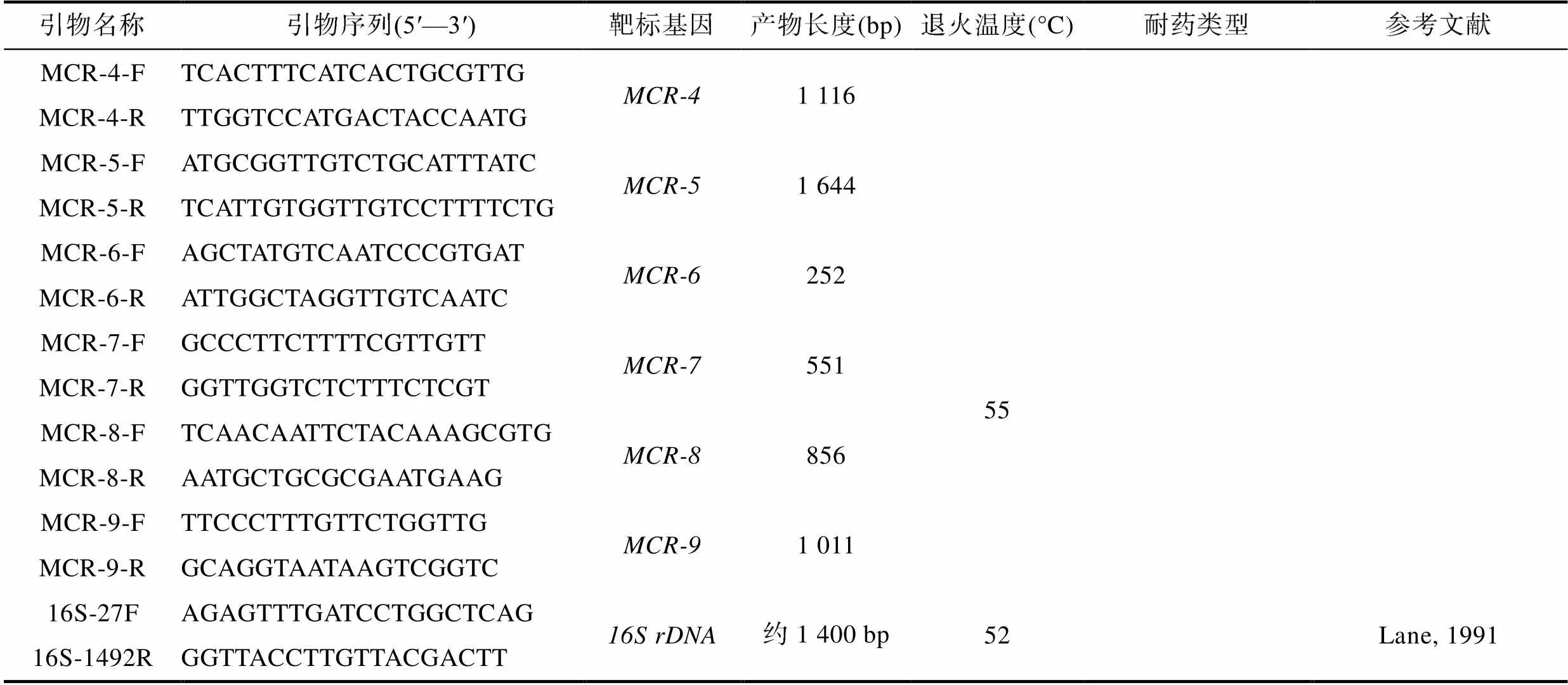
續表
1.4 藥物敏感性實驗
對含有目標耐藥基因的耐藥分離株進行藥物敏感性實驗。藥物敏感性實驗采用微量二倍稀釋法, 參照美國臨床和實驗室標準協會(Clinical and Laboratory Standards Institute, CLSI)的標準判斷耐藥分離株的藥物敏感性, 以大腸桿菌() ATCC25922作為質控菌株。在選擇目標耐藥基因所對應的抗生素時, 磺胺類抗生素選用甲氧芐啶, 四環素類抗生素選用四環素, 喹諾酮類抗生素選用環丙沙星, 多粘菌素類抗生素選用多粘菌素E。同時, 在96孔板上設置無藥無菌的陰性對照與無藥有菌的陽性對照。于每個孔中加入上述稀釋至1×104cfu/mL的菌液5 μL, 置于28 °C培養箱中靜置16—24 h后, 取出96孔板置于酶標儀上測定600的吸光度值(optical density, OD)。在陽性對照生長良好, 陰性對照未有無污染時, 以OD值判斷能夠抑制細菌生長的最低藥物濃度作為該菌株的最小抑菌濃度。同時根據文獻對于多重耐藥菌(multidrug-resistant organism, MDRO)的定義, 對含有目標耐藥基因的耐藥分離株進行多重耐藥分析, 統計其多重耐藥率(Magiorakos, 2012)。
2 實驗結果
2.1 耐藥分離株的分離鑒定
如表3所示, 從25個采樣點的海水樣品中共分離純化獲得耐藥分離株1 605株, 通過分子鑒定, 共計51株耐藥分離株檢測出目標耐藥基因, 占總耐藥分離株數量的3.18%。51株耐藥分離株中, 從平陽縣鄰近水域分離29株, 占該地區耐藥分離株總數的19.33%。其中23株檢出, 檢出率為15.33%; 21株檢出, 檢出率為14.00%; 1株檢出, 檢出率為0.67%; 4株檢出a, 6株檢出, 檢出率為6.67%。蒼南縣鄰近水域分離20株, 占該地區耐藥分離株總數的5.33%。其中10株檢出, 檢出率為2.6%; 15株檢出, 檢出率為4.00%; 1株檢出, 檢出率為0.27%; 7株檢出(), 檢出率為1.87%。樂清市鄰近水域分離2株, 占該地區耐藥分離株總數的0.28%, 2株均檢測出, 檢出率為0.28%。
表3 不同來源樣品耐藥分離株的分離鑒定結果
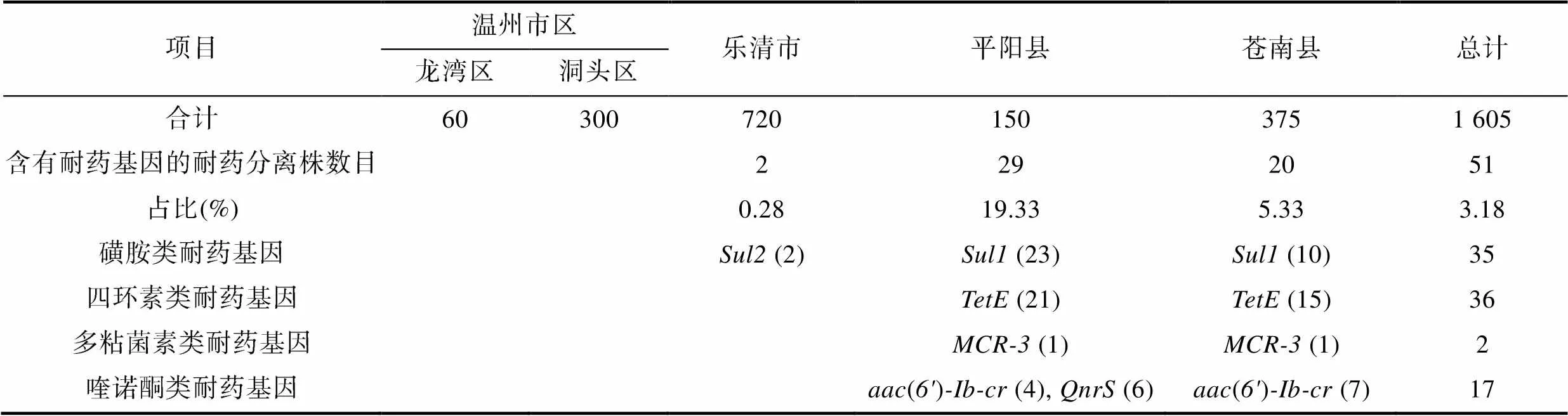
Tab.3 Identification of antibiotic resistant bacterial isolates from 25 sampling sites
2.2 目標耐藥基因及目標耐藥基因陽性耐藥分離株的分子鑒定
利用平板法, 51株含有目標耐藥基因的耐藥分離株中, 磺胺類耐藥基因檢測到和, 其中檢出33株, 占比68.63%,檢出2株, 占比3.92%; 四環素類耐藥基因檢測到, 共計36株, 占比70.56%; 多粘菌素類耐藥基因檢測到, 共2株, 占比3.92%; 喹諾酮類耐藥基因檢測出和, 其中檢出10株, 占比19.61%,檢出6株, 占比11.76%。具體如表4所示。
表4 51株含有目標耐藥基因的耐藥分離株的耐藥基因鑒定結果
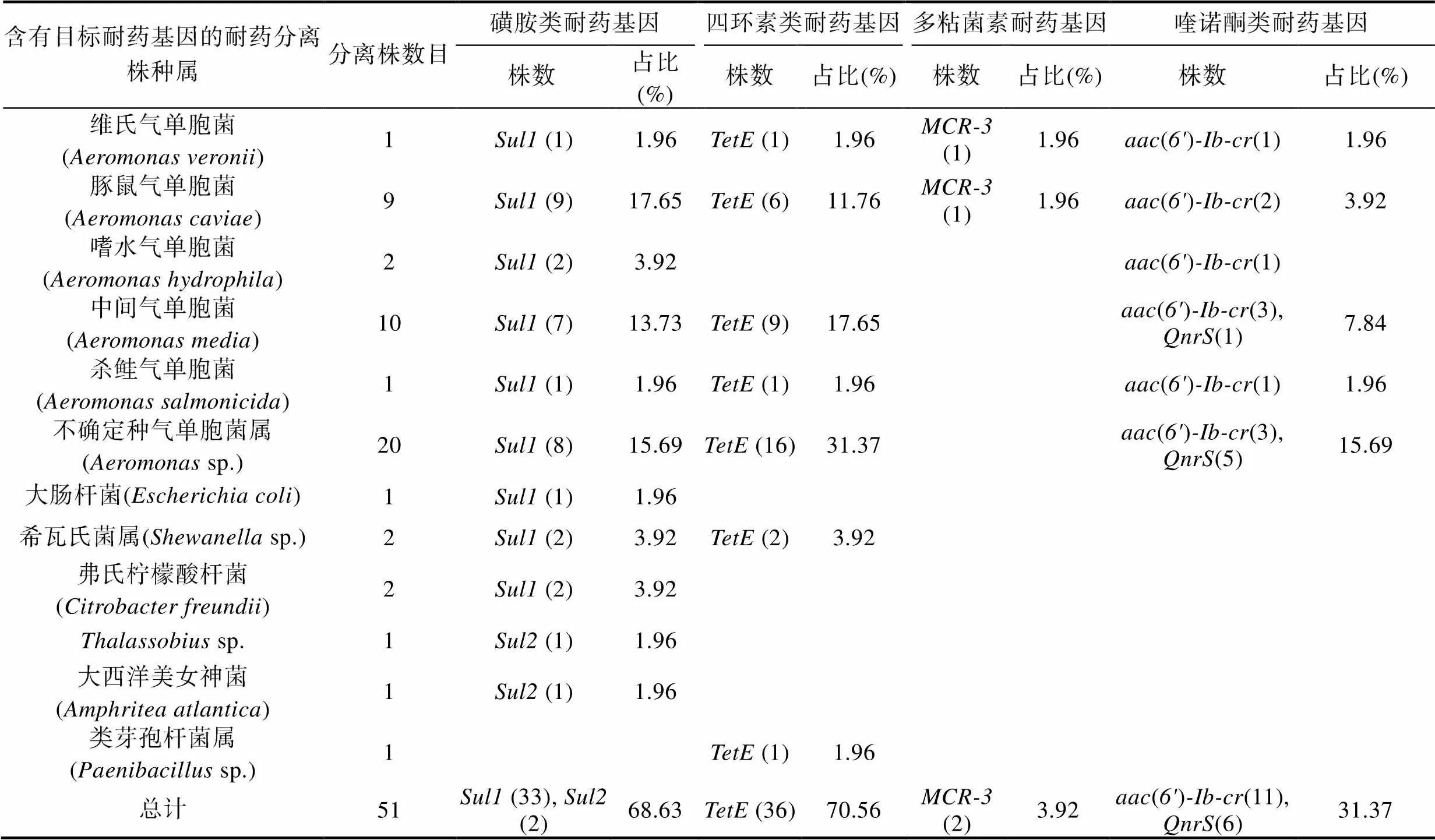
Tab.4 Molecular identification of resistant genes from 51 antibiotic resistant isolates
51株含有目標耐藥基因的耐藥分離株, 通過16S rDNA序列分析鑒定為7個不同屬, 分別為氣單胞菌屬(sp.)、埃希氏菌屬(sp.)、希瓦氏菌屬(sp.)、枸櫞酸桿菌屬(sp.)、sp.、sp.、類芽孢桿菌屬(sp.)。其中, 氣單胞菌屬共計43株, 占比84.32%, 包括維氏氣單胞菌1株, 占比1.96%, 豚鼠氣單胞菌9株, 占比17.65%, 嗜水氣單胞菌2株, 占比3.92%, 中間氣單胞菌10株, 占比19.61%, 殺鮭氣單胞菌1株, 占比1.96%, 不確定種的氣單胞菌屬細菌20株, 占比39.22%; 希瓦氏菌屬2株, 占比3.92%; 埃希氏菌屬1株, 為大腸桿菌, 占比1.96%; 枸櫞酸桿菌屬2株, 為弗氏檸檬酸桿菌, 占比3.92%;sp.和類芽孢桿菌屬各有1株, 占比為3.92%;sp.屬的大西洋美女神菌1株, 占比1.96%。耐藥基因主要分布于氣單胞菌屬細菌中(圖1, 表4)。此外, 對51株耐藥分離株進一步分析發現2株中間氣單胞菌, 1株殺鮭氣單胞菌和2株其他不確定種的氣單胞菌中同時檢出與, 1株維氏氣單胞菌和1株豚鼠氣單胞菌中同時檢出、與(圖1, 表4)。具體如表4所示。從區域上歸類, 可以發現上述51株耐藥分離株中, 蒼南縣鄰近水域20株, 平陽縣鄰近水域29株, 樂清市鄰近水域2株, 溫州其他縣區鄰近水域未檢出目標耐藥基因陽性的耐藥分離株(表5)。

圖1 51株耐藥分離株耐藥基因的分布
注: 橫坐標代表不同種類的耐藥基因, 縱坐標代表含有該耐藥基因的耐藥分離株數, 柱狀圖中不同顏色的色塊表示不同種屬的耐藥分離株, 高度代表耐藥分離株數目
2.3 藥物敏感性測定結果
盡管在使用相應濃度的抗生素測試時,sp.分離株和大西洋美女神菌仍舊表現出耐藥性, 但是由于在CLSI的標準中沒有這2株分離株的耐藥標準, 故相關數據計算時, 未列入其中。49株含有目標耐藥基因的耐藥分離株對4種抗生素的藥物敏感性試驗結果如圖2和表6所示: 僅對1種抗生素耐藥的分離株有2株, 占比4.08%, 對2種抗生素耐藥的分離株有8株, 占比16.33%, 對3種抗生素耐藥的分離株有16株, 占比32.65%, 對4種抗生素耐藥的分離株有23株, 占比46.94%。其中, 蒼南縣鄰近水域有10株, 占本地區分離總數的50.00%, 平陽縣鄰近水域有13株, 占本地區分離株總數的48.82% (表6)。49株耐藥分離株均對四環素耐藥, 41株耐藥分離株對多粘菌素E耐藥, 占比83.67%; 34株耐藥分離株對環丙沙星耐藥, 占比69.39%; 34株耐藥分離株對甲氧芐啶耐藥, 占比69.39%。多數條件致病菌(如豚鼠氣單胞菌、維氏氣單胞菌、中間氣單胞菌、殺鮭氣單胞菌、嗜水氣單胞菌、大腸桿菌等)對3類以上抗生素具有較高的MIC值(環丙沙星MIC>1 μg/mL; 多粘菌素E的MIC>8 μg/mL;四環素MIC>16 μg/mL; 甲氧芐啶MIC>64 μg/mL) (圖3), 不同來源的耐藥分離株在四種抗生素的MIC分布上未見顯著差別(圖4)。
3 討論
耐藥基因和耐藥菌能夠經由多種途徑進入近海水體, 擴散傳播, 給海洋生態環境和公共衛生帶來巨大威脅(Normark, 2002; Chen, 2013)。本文初步分析了東海溫州近海水域可培養細菌中磺胺類、四環素類、喹諾酮類和多粘菌素類耐藥基因的流行情況。研究通過抗性平板法篩選了耐藥分離株1605株, 獲得含目標耐藥基因的51株, 分屬于氣單胞菌屬等7個屬, 分別攜帶有耐藥基因、、、、()以及等, 其中39株存在多重耐藥現象; 此外, 研究還分離獲得了攜帶多粘菌素耐藥基因的條件致病菌維氏氣單胞菌和豚鼠氣單胞菌各1株, 表明多粘菌素的耐藥基因在海洋水體中有流行。
上述51株含目標耐藥基因的耐藥菌集中發現于平陽縣和蒼南縣鄰近水域(分別是29、20株), 樂清市鄰近水域有少量發現(2株), 表明盡管溫州地區近海水域整體耐藥基因流行水平低(含有目標耐藥基因的耐藥菌占比僅為3.18%), 但相對于溫州其他海域, 平陽縣與蒼南縣鄰近海域耐藥基因流行情況要更為突出。據溫州市統計局的資料, 平陽縣海水養殖規模很小, 畜牧業不發達, 其近海水域與西灣風景區相鄰, 景區較高的人流量與景區內部小而分散的社區醫院可能是其耐藥基因與耐藥菌的重要來源; 蒼南縣是溫州海水與畜牧養殖的主要縣市之一, 本研究蒼南鄰近水域的采樣點位于河口附近, 上游的養殖排放物容易在此沉積, 一定規模的養殖和特殊的地理環境可能是蒼南縣采樣點耐藥基因流行的重要原因。
表5 含有目標耐藥基因的耐藥分離株的16S rDNA鑒定結果
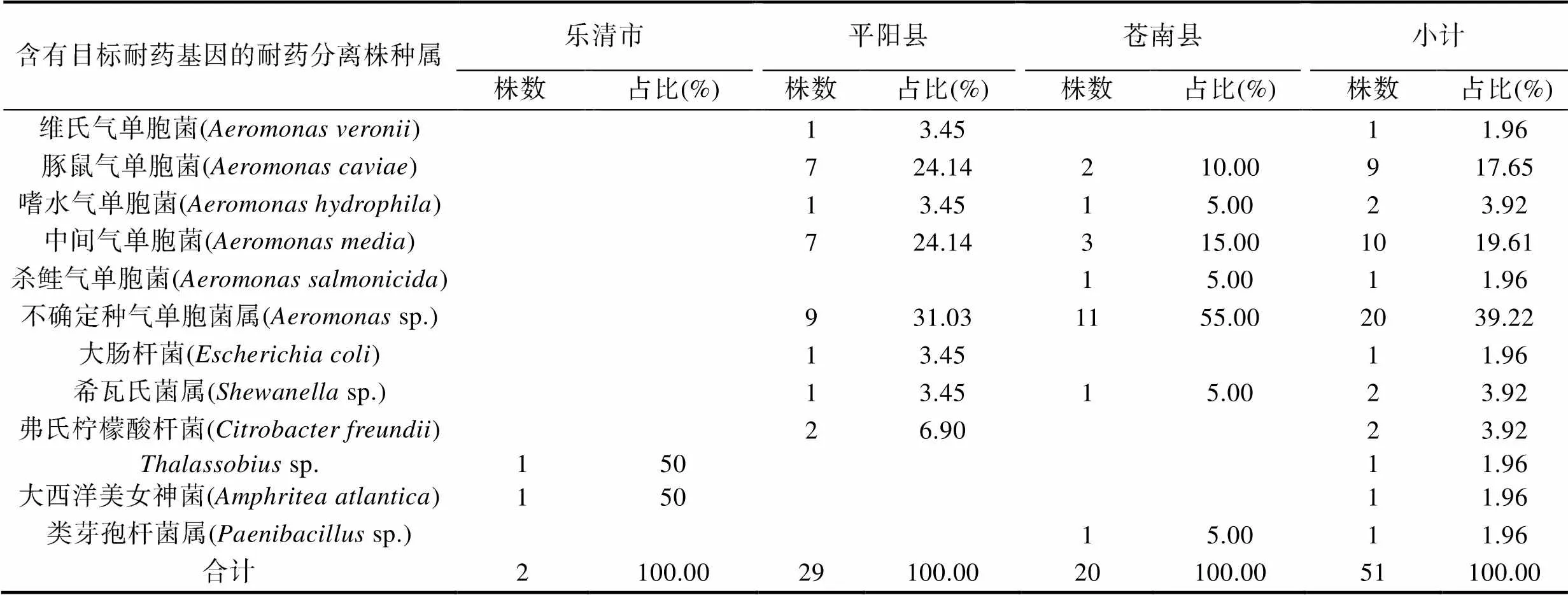
Tab.5 16S rDNA sequencing of 51 antibiotic resistant isolates
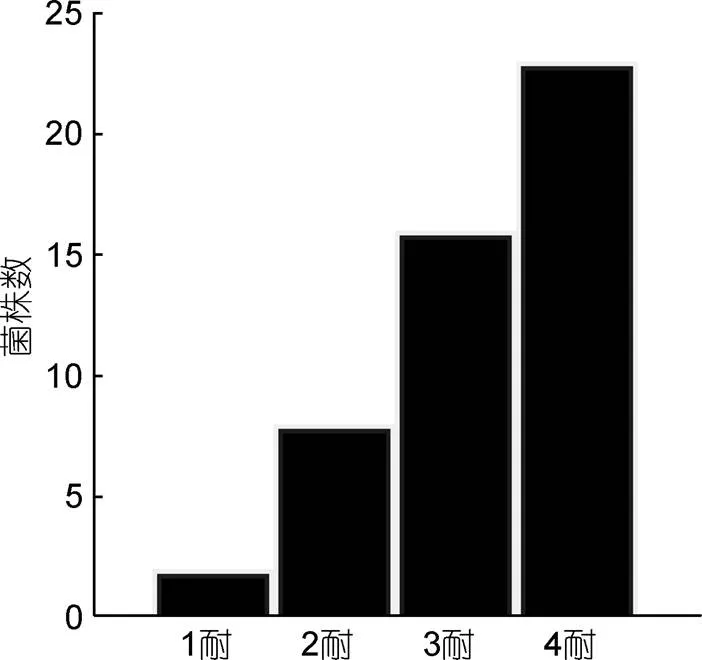
圖2 含有目標基因的耐藥分離株的多重耐藥分析結果
注: 1耐: 對一種抗生素有耐藥性; 2耐: 對兩種抗生素有耐藥性; 3耐: 對三種抗生素有耐藥性; 4耐: 對四種抗生素有耐藥性
已有研究發現, 海水環境中存在多種四環素類耐藥基因(包括、、、、、等), 耐藥基因的分布差異與地理環境及細菌耐藥性的進化有關(Kim, 2004; Balassiano, 2007; Nikolakopoulou, 2008)。國內海水樣品與水產品源細菌中檢出的四環素耐藥基因類型多為、等(趙敏等, 2014; 王鑰等, 2015), 國外發現從食品中分離的氣單胞菌和養殖海水與底泥中能大量檢出(Balassiano, 2007; Tamminen, 2011)。本研究從其他1605株耐藥分離株中檢測出含有的耐藥分離株36株, 耐藥基因檢出率上低于國內已有報道(王鑰等, 2015; 吳越等, 2020)。耐藥基因、和是介導磺胺類抗生素耐藥的主要原因之一(Hoa, 2008)其中,與是國內養殖海水與水產品中主要流行的磺胺類耐藥基因,的流行與耐藥基因在細菌間的轉移有關(馬辰婕等, 2017; 汪濤等, 2017; 吳越等, 2020)。本研究中檢測到的磺胺類耐藥基因以為主(平陽縣23株, 蒼南縣10株), 有少量的(樂清市2株), 未檢測到基因, 在耐藥基因類型上與已有報道一致, 但是耐藥基因的檢出率低于已有報道(黃志堅等, 2012; 吳越等, 2020)。上述結果表明溫州近海水域中磺胺類與四環素類耐藥基因流行情況并不嚴重, 但集中出現在平陽和蒼南兩個近海區域。喹諾酮類抗生素的耐藥情況在不同種屬與不同來源的細菌中差異較大。本研究從平陽縣臨近水域檢測到含有喹諾酮類耐藥基因的耐藥分離株11株, 蒼南縣臨近水域7株, 低于已有報道, 可能與不同的藥物使用策略有關(Zhao, 2012; 張舒婷等, 2019; 趙姝等, 2019)。喹諾酮類耐藥基因與均能夠介導較低水平的喹諾酮類藥物耐藥, 但是攜帶會提高細菌染色體自發突變的頻率, 從而導致高水平的喹諾酮類耐藥細菌的產生(Martínez, 1998), 而()同時具有氨基糖苷類與喹諾酮類耐藥活性, 增加了細菌在抗菌藥物選擇下的適應能力, 說明在藥物使用策略上要減少喹諾酮類藥物的使用并且避免與氨基糖苷類藥物的聯用, 防止高水平喹諾酮類藥物耐藥菌的出現和()基因的流行。另外, 多重耐藥質粒和攜帶多種耐藥基因的整合子是導致細菌多重耐藥的主要原因(馬辰婕等, 2017; Sun, 2018)。本研究于平陽和蒼南鄰近水域篩選出至少含有3類耐藥基因的多重耐藥分離株39株(占比達79.59%), 暗示該區域的多種耐藥基因可能集中在整合子或質粒等載體上傳播, 提升細菌對多種抗生素的耐藥能力。
表6 51株含有目標耐藥基因的耐藥分離株對4種抗生素藥物的MIC值
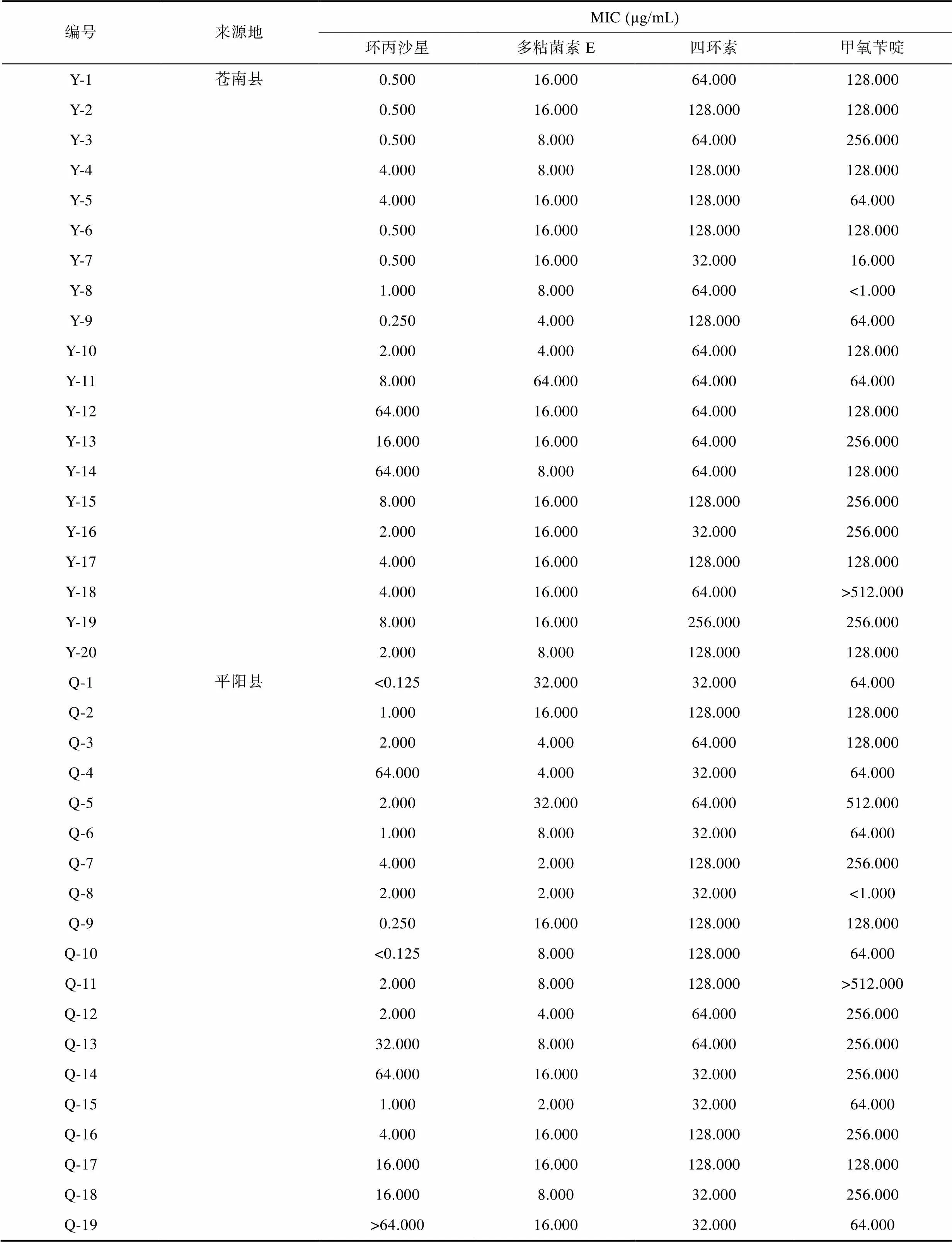
Tab.6 MIC value of 51 antibiotic resistant isolates containing target antibiotic resistance genes
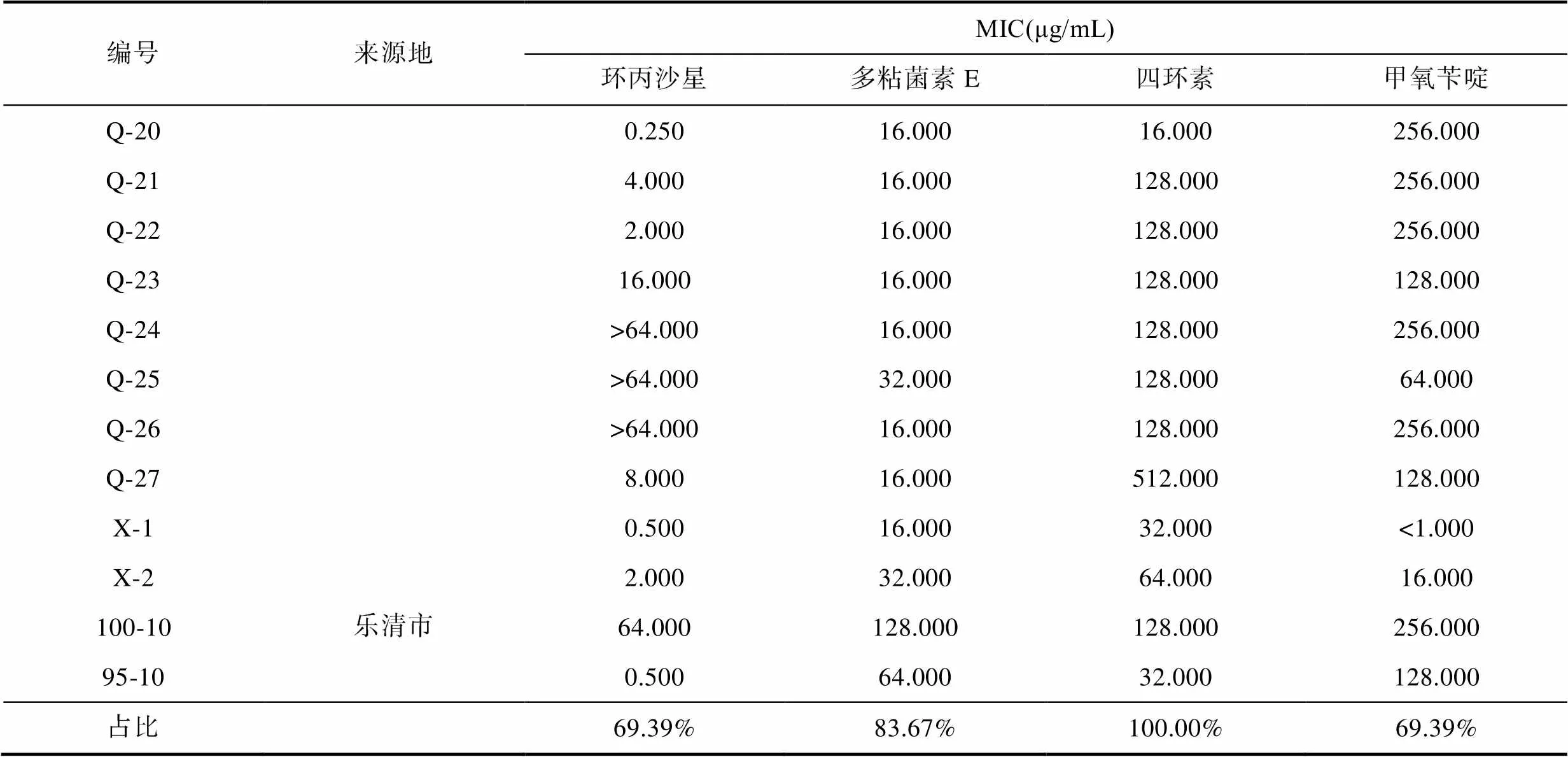
續表
注: 統計時, 排除了1株與1株大西洋美女神菌()
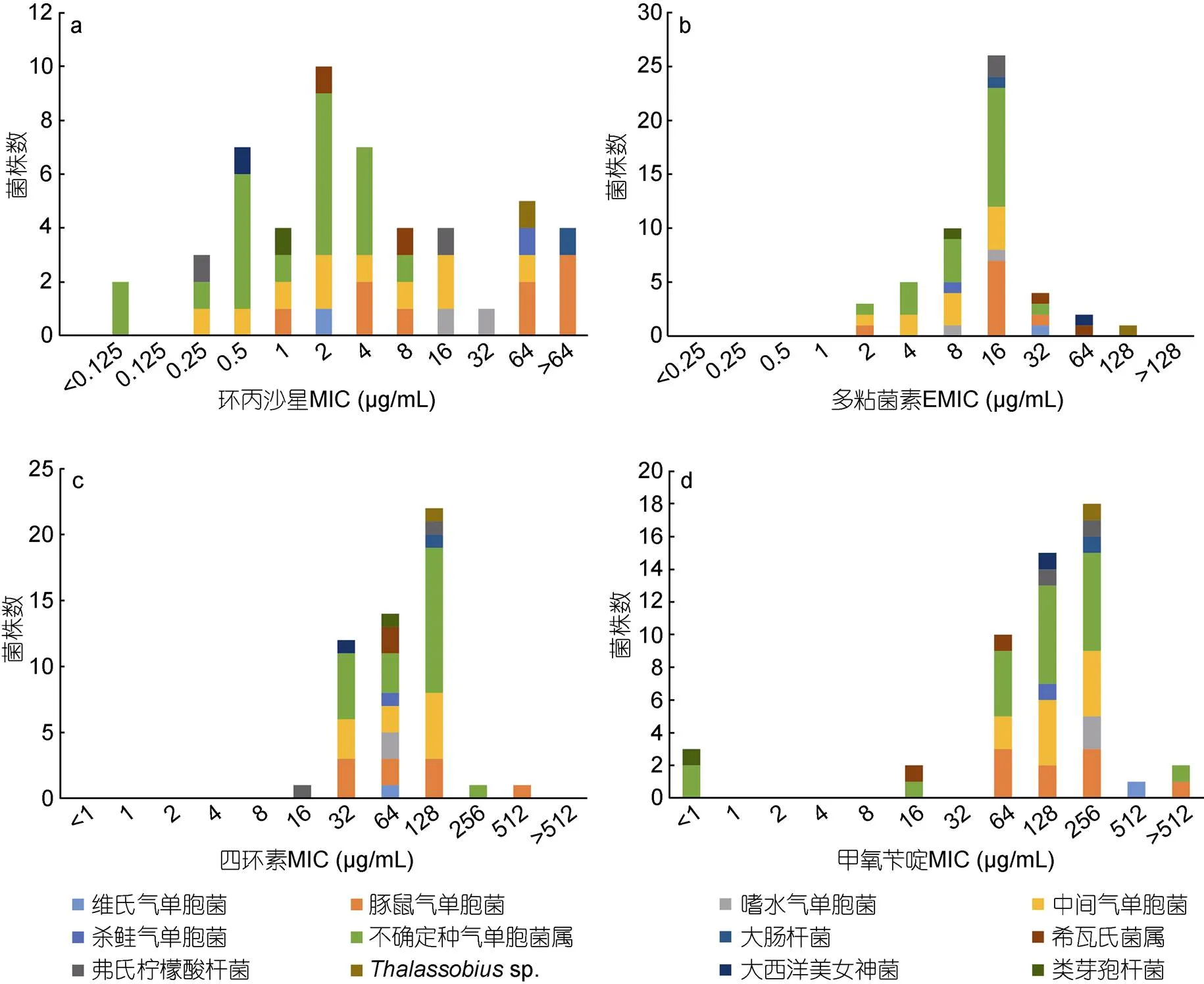
圖3 51株耐藥分離株的四種抗生素的MIC分布
注: 51株不同種屬的耐藥分離株對: 環丙沙星(a)、多粘菌素E(b)、四環素(c)、甲氧芐啶(d)的MIC分布。橫坐標代表耐藥分離株對該種抗生素的MIC值, 縱坐標代表耐藥分離株的數目, 柱狀圖中不同顏色的色塊表示不同種屬的耐藥分離株, 其高度代表耐藥分離株數目
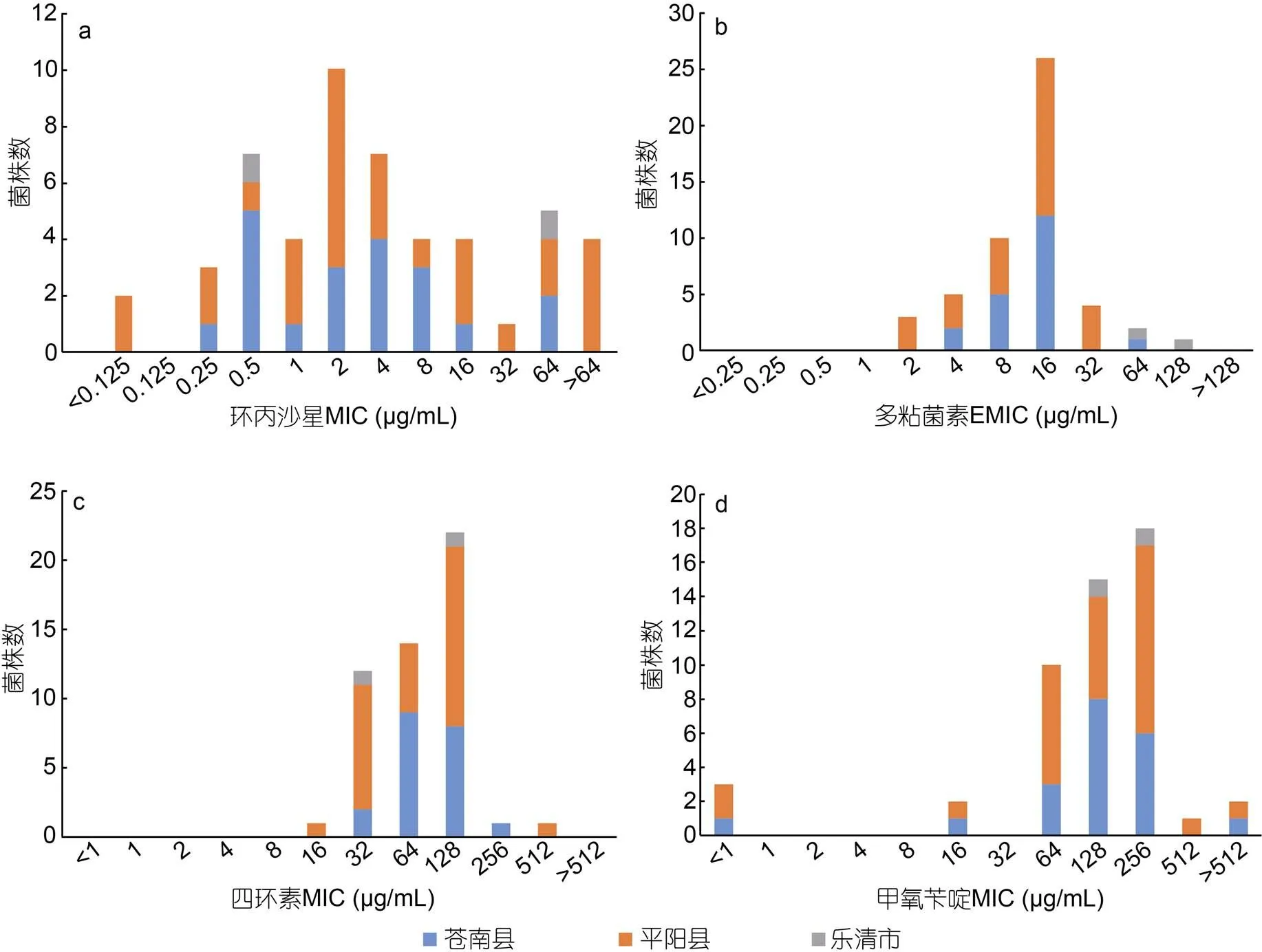
圖4 不同采樣地點的耐藥分離株的四種抗生素MIC分布差異分析
注: 來自蒼南縣、平陽縣及樂清市的耐藥分離株對: 環丙沙星(a)、多粘菌素E(b)、四環素(b)、甲氧芐啶(d)的MIC分布差異。橫坐標代表耐藥分離株對該種抗生素的MIC值, 縱坐標代表耐藥分離株的數目, 柱狀圖中不同顏色的色塊表示不同來源的耐藥分離株, 其高度代表耐藥分離株數目
水產養殖中通常很少使用多粘菌素類藥物, 部分資料表明水產中通常只有弧菌具有較高的多粘菌素耐藥性(劉開放等, 2017; 張西萌等, 2020)。質粒介導的可移動的多粘菌素耐藥基因至等是目前認為介導多粘菌素耐藥的主要因素之一, 使得多粘菌素的使用受到極大限制(Sun, 2018; Borowiak, 2020)。海洋水體中系列基因的報道相對較少,與在氣單胞菌中有檢測到(Eichhorn, 2018; Ma, 2018)。本研究在平陽鄰近水域檢出含有的維氏氣單胞菌, 在蒼南鄰近水域檢出含有的豚鼠氣單胞菌, 盡管與國外水產動物分離的氣單胞菌結論一致(Eichhorn, 2018), 整體檢出率也較低, 但的來源是值得關注的問題。有研究認為水體環境是的主要傳播途徑, 氣單胞菌屬是其重要的儲存庫(Eichhorn, 2018)。因此, 本研究在溫州近海水域的兩種氣單胞菌中檢出, 可能是外源多粘菌素殘留導致的定向選擇結果。
我國近海海域或者水產養殖動物中分離的耐藥菌一般以弧菌屬與氣單胞菌屬為主(黃志堅等, 2012; Ye, 2013; Dong, 2019)。本研究發現溫州近海水域分離的耐藥菌以氣單胞菌為主, 占比達到84.31%, 未發現弧菌屬細菌, 表明氣單胞菌屬細菌可能具有高效的耐藥基因轉移方式, 成為該地區耐藥基因傳播的重要途徑。在氣單胞菌屬細菌中, 海洋水體和水產養殖中常以維氏氣單胞菌和嗜水氣單胞菌為主, 畜禽養殖中多為豚鼠氣單胞菌(黃志堅等, 2012; 鄧玉婷等, 2019; 劉小芳等, 2021)。本研究鑒定的23株條件致病性氣單胞菌屬細菌中, 包括中間氣單胞菌10株、豚鼠氣單胞菌9株、嗜水氣單胞菌2株、殺鮭氣單胞菌和維氏氣單胞菌各1株。其中, 豚鼠氣單胞菌檢出的數量較多, 與先前的報道有所差異(黃志堅等, 2012; 鄧玉婷等, 2019), 可能是外源性細菌輸入的結果。氣單胞菌普遍存在于近海水域和水生動物體內, 可以作為溫州近海水域耐藥基因流行情況監測的指標微生物, 為該地區抗生素的合理使用提供科學參考。
4 結論
本研究調查分析了2019—2020年期間東海溫州近海水域磺胺類、四環素類、喹諾酮類以及多粘菌素類耐藥基因和耐藥菌的分布情況。對篩選獲得的51株耐藥基因陽性的耐藥分離株分析發現, 對四類抗生素產生耐藥的比例較高, 其中對四環素類抗生素的耐藥比例最高, 達100%。同時, 盡管含耐藥基因的耐藥分離株檢出率較低, 但同一耐藥基因的細菌種類豐富, 但又以氣單胞菌屬細菌為主, 占比84.31%。多重耐藥現象嚴重, 含3種及以上耐藥基因的耐藥菌占比76.47%。另外, 獲得了多粘菌素耐藥基因陽性的條件致病菌維氏氣單胞菌和豚鼠氣單胞菌各1株。上述系列研究結果將為評估溫州近海水域的細菌耐藥性風險, 規范抗生素的使用提供科學依據。
馬辰婕, 吳小梅, 林 茂等, 2017. 水產養殖環境耐藥細菌中復合1型整合子的流行特征. 微生物學通報, 44(9): 2089—2095
王 玥, 蘇 潔, 明紅霞等, 2015. 浴場海水中大腸桿菌耐藥性及其耐藥基因研究. 海洋學報, 37(12): 123—128
鄧玉婷, 譚愛萍, 張瑞泉等, 2019. 廣東主要水產養殖地區氣單胞菌耐藥狀況的調查分析. 南方農業學報, 50(11): 2375—2383
劉小芳, 任 燕, 張德鋒等, 2021. 魚源氣單胞菌的毒力基因檢測、分型及致病力. 水產學報, 45(3): 462—471
劉開放, 步 君, 章 翔等, 2017. 華南地區海水養殖水域中哈維氏弧菌的抗藥性分析. 海南大學學報自然科學版, 35(2): 131—139
嚴 春, 張 穎, 何 衛等, 2016. NICU患兒肺炎克雷伯菌耐藥基因攜帶和同源性分析. 中國衛生檢驗雜志, 26(18): 2637—2640, 2645
汪 濤, 楊再福, 陳勇航等, 2017. 磺胺類抗性基因的產生及演變研究進展. 環境污染與防治, 39(11): 1251—1255
吳 越, 鄭伊諾, 陸榮茂等, 2020. 溫州地區水產品中哈維氏弧菌的耐藥分析. 水產科學, 39(6): 837—843
張西萌, 付溥博, 魏海燕等, 2020. 北京進出口水產品中259株霍亂弧菌分離株的耐藥性研究. 食品安全質量檢測學報, 11(14): 4901—4906
張舒婷, 陳賽賽, 2019. 青島海水浴場多重耐藥菌的分布特征研究. 生物技術通報, 35(4): 57—63
趙 姝, 李 健, 馬立才等, 2019. 海水養殖動物源弧菌喹諾酮類藥物耐藥表型與基因型分析. 海洋漁業, 41(4): 463—471
趙 敏, 汪開毓, 王 均等, 2014. 斑點叉尾鮰源維氏氣單胞菌對四環素類抗生素的耐藥性及耐藥基因的檢測. 水生生物學報, 38(2): 386—392
黃志堅, 陳旭凌, 路曉峰等, 2012. 水產養殖生物和養殖環境細菌鑒定及抗生素抗性基因檢測. 中山大學學報(自然科學版), 51(6): 92—96
Aminov R I, Garrigues-Jeanjean N, Mackie R I, 2001. Molecular ecology of tetracycline resistance: development and validation of primers for detection of tetracycline resistance genes encoding ribosomal protection proteins. Applied and Environmental Microbiology, 67(1): 22—32
Arias C A, Murray B E, 2009. Antibiotic-resistant bugs in the 21st century—a clinical super-challenge. The New England Journal of Medicine, 360(5): 439—443
Balassiano I T, De Freire Bastos M D C, Madureira D J, 2007. The involvement of tetA and tetE tetracycline resistance genes in plasmid and chromosomal resistance ofin Brazilian strains. Memorias Do Instituto Oswaldo Cruz, 102(7): 861—866
Borowiak M, Baumann B, Fischer J, 2020. Development of a Novel mcr-6 to mcr-9 Multiplex PCR and Assessment of mcr-1 to mcr-9 Occurrence in Colistin-ResistantIsolates from Environment, Feed, Animals and Food (2011—2018) in Germany. Frontiers in Microbiology, 11: 80
Chen B W, Yang Y, Liang X M, 2013. Metagenomic profiles of antibiotic resistance genes (ARGs) between human impacted estuary and deep ocean sediments. Environmental Science & Technology, 47(22): 12753—12760
Chen C Q, Zheng L, Zhou J L, 2017. Persistence and risk of antibiotic residues and antibiotic resistance genes in major mariculture sites in Southeast China. Science of the Total Environment, 580: 1175—1184
Dong P Y, Cui Q J, Fang T T, 2019. Occurrence of antibiotic resistance genes and bacterial pathogens in water and sediment in urban recreational water. Journal of Environmental Sciences, 77: 65—74
Eichhorn I, Feudi C, Wang Y, 2018. Dentification of novel variants of the colistin resistance gene mcr-3 inspp. from the national resistance monitoring programme GERM-Vet and from diagnostic submissions. Journal of Antimicrobial Chemotherapy, 73(5): 1217—1221
Hatosy S M, Martiny A C, 2015. The ocean as a global reservoir of antibiotic resistance genes. Applied and Environmental Microbiology, 81(21): 7593—7599
Hoa P T P, Nonaka L, Viet P H, 2008. Detection of the sul1, sul2, and sul3 genes in sulfonamide-resistant bacteria from wastewater and shrimp ponds of north Vietnam. Science of the Total Environment, 405(1/3): 377—384
Kennedy D, 2013. Time to deal with antibiotics. Science, 342(6160): 777
Kim S R, Nonaka L, Suzuki S, 2004. Occurrence of tetracycline resistance genes(M) and(S) in bacteria from marine aquaculture sites. FEMS Microbiology Letters, 237(1): 147—156
Lane D J, 1991. 16S/23S rRNA sequencing. In: Stackebrandt E, Goodfellow M eds. Nucleic Acid Techniques in Bacterial Systematics. New York, USA: Wiley, 115—147
Liao W, Lin J, Jia H, 2020. Resistance and heteroresistance to colistin inisolates from Wenzhou, China. Infection and Drug Resistance, 13: 3551—3561
Lu X M, Li W F, Li C B, 2017. Characterization and quantification of antibiotic resistance genes in manure of piglets and adult pigs fed on different diets. Environmental Pollution, 229: 102—110
Ma S Z, Sun C T, Hulth A, 2018. Mobile colistin resistance gene-5 in porcine. Journal of Antimicrobial Chemotherapy, 73(7): 1777—1780
Magiorakos A P, Srinivasan A, Carey R B, 2012. Multidrug-resistant, extensively drug-resistant and pandrug-resistant bacteria: an international expert proposal for interim standard definitions for acquired resistance. Clinical Microbiology and Infection, 18(3): 268—281
Martínez-Martínez L, Pascual A, Jacoby G A, 1998. Quinolone resistance from a transferable plasmid. The Lancet, 351(9105): 797—799
Ng L K, Martin I, Alfa M, 2001. Multiplex PCR for the detection of tetracycline resistant genes. Molecular and Cellular Probes, 15(4): 209—215
Nikolakopoulou T L, Giannoutsou E P, Karabatsou A A, 2008. Prevalence of tetracycline resistance genes in Greek seawater habitats. The Journal of Microbiology, 46(6): 633—640
Normark B H, Normark S, 2002. Evolution and spread of antibiotic resistance. Journal of Internal Medicine, 252(2): 91—106
Park C H, Robicsek A, Jacoby G A, 2006. Prevalence in the United States of()encoding a ciprofloxacin-modifying enzyme. Antimicrobial Agents and Chemotherapy, 50(11): 3953—3955
Rebelo A R, Bortolaia V, Kjeldgaard J S, 2018. Multiplex PCR for detection of plasmid-mediated colistin resistance determinants,-1,-2,-3,-4 and-5 for surveillance purposes. Eurosurveillance, 23(6): 17-00672
Rutgersson C, Fick J, Marathe N, 2014. Fluoroquinolones andgenes in sediment, water, soil, and human fecal flora in an environment polluted by manufacturing discharges. Environmental Science & Technology, 48(14): 7825—7832
Sun J, Zhang H M, Liu Y H, 2018. Towards understanding MCR-like colistin resistance. Trends in Microbiology, 26(9): 794—808
Tamminen M, Karkman A, L?hmus A, 2011. Tetracycline resistance genes persist at aquaculture farms in the absence of selection pressure. Environmental Science & Technology, 45(2): 386—391
Willyard C, 2017. The drug-resistant bacteria that pose the greatest health threats. Nature, 543(7643): 15
Ye L, Lu Z, Li X H, 2013. Antibiotic-resistant bacteria associated with retail aquaculture products from Guangzhou, China. Journal of Food Protection, 76(2): 295—301
Zhao J Y, Dang H Y, 2012. Coastal seawater bacteria harbor a large reservoir of plasmid-mediated quinolone resistance determinants in Jiaozhou Bay, China. Microbial Ecology, 64(1): 187—199
INVESTIGATION ON RESISTANCE GENES AGAINST FOUR TYPES OF COMMON ANTIBIOTICS IN WENZHOU WATERS OF THE EAST CHINA SEA
JIN Jing-Lei1, 2, ZHOU Qian-Jin1, 2, SHAO Xin-Bin3, WANG Yao-Hua3, CHEN Jiong1, 2, YAN Mao-Cang3, MIAO Liang2, TANG Biao4, ZHANG Yan-Jun5
(1. State Key Laboratory for Managing Biotic and Chemical Threats to the Quality and Safety of Agro-products, Ningbo University, Ningbo 315211, China; 2. School of Marine Science, Ningbo University, Ningbo 315832, China; 3. Zhejiang Key Laboratory of Exploitation and Preservation of Coastal Bio-resource, Zhejiang Mariculture Research Institute, Wenzhou 325005, China; 4. Institute of Quality and Standard for Agro-Products, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China; 5. Zhejiang Provincial Center for Disease Control and Prevention, Hangzhou 310051, China)
Sulfonamide, tetracyclines, quinolone, and polymyxin are antibiotics commonly used in medicine and animal breeding, of which polymyxin is often used as addictive to promote animal growth. The prevalence of the common resistance genes against the four types of antibiotics in coastal water of Wenzhou, Zhejiang, East China was investigated. Firstly, culturable bacterial strains resistant to sulfonamides, tetracyclines, and quinolones were isolated from the coastal waters of Wenzhou using the resistance plate method, in which the common resistance genes against sulfonamides, tetracyclines, and quinolones were identified using polymerase chain reaction. Secondly, polymyxin resistance-associatedgenes were detected from the bacterial isolates carrying the resistance genes against sulfonamides, tetracyclines, and quinolones. Finally, the antimicrobial resistance of all the isolates carrying the four types of the resistance gene were determined using microdilution broth method. The results show that 1 605 culturable bacterial isolates were resistant to sulfonamides, tetracyclines, and quinolones, of which 51 were proven to carry different resistance genes against sulfonamides (35 bacterial isolates), tetracyclines (36 bacterial isolates), quinolones (17 bacterial isolates), and polymyxin (2 bacterial isolates), with a detection rate of 3.18%. These bacterial isolates could be classified into 7 genera, i.e.,sp. (43 isolates, 84.31%),sp.,sp.,sp.,sp.,sp., andsp. Except forsp. andsp., the remaining 49 isolates were all resistant to sulfonamides, of which 41 were resistant to tetracyclines, 34 to quinolones and polymyxin, 23 to all the four antibiotics, and 16 to three antibiotics, indicating a ubiquitous multi-drug resistance in the coastal waters in Wenzhou. In addition, 2 bacterial isolates carrying resistance genewere identified, i.e.,and. Therefore, a small amount of antibiotic resistant isolates carrying common resistant genes against sulfonamides, tetracyclines, quinolones, and polymyxin were identified in the coastal waters, which characterized a diverse multi-drug resistance and strong resistance tolerance. These findings provide a reference for evaluating the risk of bacterial resistance and standardizing the use of antibiotics in Wenzhou coastal waters.
Wenzhou waters of the East China Sea; resistance genes; multi-drug resistance;
X172; Q789; S931.3
10.11693/hyhz20210200040
* 浙江省重點研發計劃, 2021C02059號; 農產品質量安全危害因子與風險防控國家重點實驗室自主設計課題, 2010DS700124-ZZ2008號; 農產品質量安全危害因子與風險防控國家重點實驗室2020開放基金項目, KF20200105號; 浙江省近岸水域生物資源開發與保護重點實驗室開放基金, J2018004號。金晶磊, E-mail: jinjinglei321@163.com
陳 炯, 研究員, 博士生導師, E-mail: jchen1975@163.com, chenjiong@nbu.edu.cn; 周前進, 副教授, 碩士生導師, E-mail: mumu2325@163.com
2021-02-06,
2021-03-31

