高產堿性纖維素酶細菌的篩選鑒定及其酶學特性與發酵條件研究
馬振剛 熊亮 張真 羅堿

摘要:【目的】從自然環境中分離篩選高產纖維素酶的菌株,開展堿性纖維素酶的酶學特性分析,為該菌株及所產纖維素酶的綜合開發利用打下基礎。【方法】采用羧甲基纖維素鈉(CMCNa)平板篩選法篩選纖維素酶高產菌株,利用生理生化分析結合分子生物學法對菌株進行鑒定,并通過3,5-二硝基水楊酸(DNS)法研究其活性特征與發酵條件。【結果】在長期覆蓋枯樹葉的土壤中分離獲得1株高產堿性纖維素酶的菌株,經鑒定該菌株為蠟樣芽孢桿菌(Bacillus cereus),名稱為B. cereus strain CQNUX 3-1。酶活性分析顯示該菌株胞外分泌液具有內切葡聚糖酶、外切葡聚糖酶及β-葡萄糖苷酶的活性,其酶活力分別達107.7、33.1和155.6 U/mL。酶學特征分析表明3種酶組分均具有較好的耐堿和一定的耐高溫能力。其中,內切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶的最佳反應溫度分別為70、60和40 ℃;最佳反應pH分別為8.0、9.0和9.0;Fe3+能增加3種酶的酶活力,而β-葡萄糖苷酶具有較好的EDTA、尿素和Cu2+耐受性。發酵條件對菌株產酶的分析結果表明,該菌株發酵溫度在37 ℃較適宜;發酵第4 d時的酶活力達最大值;該菌株能在堿性發酵環境下生長并產酶,在初始pH為7.0時發酵酶活力最高。【結論】篩選獲得的纖維素酶高產菌株B. cereus strain CQNUX 3-1所生產的纖維素酶具有較高的反應溫度適用性和較強的堿耐受性,菌株發酵產酶溫度適中,且有較寬的發酵pH適用范圍,可作為堿性纖維素酶生產資源菌株,具有應用于纖維素酶制劑制備與生產、纖維素資源綜合利用等領域的潛力。
關鍵詞: 堿性纖維素酶產生菌;篩選鑒定;酶學特性;發酵條件;蠟樣芽孢桿菌
中圖分類號: S154.39? ? ? ? ? ? ? ? ? ? ? ? ? ? 文獻標志碼: A 文章編號:2095-1191(2021)03-0722-10
Screening and identification of a strain with high yield of alkaline cellulase and its enzyme characterizations and
fermentation conditions
MA Zhen-gang, XIONG Liang, ZHANG Zhen, LUO Jian
(College of Life Sciences, Chongqing Normal University, Chongqing? 401331, China)
Abstract:【Objective】Strains with high yield of cellulase were isolated from the natural environment,and the enzymatic characteristics of screened alkaline cellulase were analyzed which could lay a foundation for the comprehensive development and utilization of the cellulase produced by this strain. 【Method】The carboxymethyl cellulose sodium(CMCNa)plate screening method was used to isolate and screen high yield cellulose-producing strains and then the isolated strains were identified by physiological and biochemical analysis and molecular biology method. The characteristics of enzyme activity and fermentation conditions were studied by 3,5-dinitrosalicylic acid(DNS) method. 【Result】A high alkaline cellulase producing strain was isolated from the soil samples which were long-term covered by rotting leaves and this strain was identified and named as Bacillus cereus strain CQNUX 3-1. The results of enzyme activity tests showed that strain CQNUX 3-1 exhibited the activity of endoglucanase,exoglucanase and β-glucosidase and were 107.7,33.1 and 155.6 U/mL,respectively. Analysis of enzymatic characterization showed that the three enzymes had better alkaline resistance and certain high temperature performance. The optimum reaction temperature of endoglucanase,exoglucanase and β-glucosidase were 70,60 and 40 ℃,respectively. The best reaction pH were 8.0,9.0 and 9.0 respectively. Fe3+ could? increase the enzyme activity of three enzymes,while β-glucosidase showed better tolerance to EDTA,urea and Cu2+. The effect of fermentation conditions on the enzyme production showed that the optimum fermentation temperature of the strain was 37 ℃ and the enzyme activity could reach to the maximum after four days fermentation. Finally,the bacteria could grow and produce enzymes under alkaline fermentation environment. Enzyme activity was the best when the initial pH was 7.0. 【Conclusion】A strain with high alkaline cellulase producing is selected. The cellulase produced by B. cereus strain CQNUX 3-1 has high reaction temperature applicability and strong alkali resistance. The strain fermentation enzyme has moderate temperature and has a wide range of fermentation pH. This strain can be used as a alkaline cellulase production resource strain, with the potential for the preparation and production of cellulase preparations and the comprehensive utilization of cellulose resources.
Key words: alkaline cellulase producing strain; screening and identification; enzyme characterization; fermentation conditions; Bacillus cereus
Foundation item: National Natural Science Foundation of China(31770160); Science and Technology Research Project of Chongqing Municipal Education Commission(KJQN201800524)
0 引言
【研究意義】纖維素是自然界中分布最廣、儲量最多的碳水化合物,占植物界碳含量的50%以上,為地球上最豐富的生物質資源和天然可再生能源物質(高鳳菊和李春香,2005;Ragauskas et al.,2006)。但由于植物纖維素具有不溶于水的結晶狀剛性結構,且通常被木質素層覆蓋或包圍,因此其開發利用面臨較大困難(高蕾蕾和李迎秋,2017),約有80%纖維素未得到充分利用,對纖維素廢棄物的大量丟棄或焚燒處理還帶來嚴重的環境污染問題(師璐等,2017)。目前,降解天然纖維素的方法主要包括酸水解、熱化學處理及酶水解法等(劉鎖珠等,2017)。其中,酶水解法因具有快速、高效和環保等優點而被廣泛應用于環境保護、食品工業、動物生產和中藥成分提取等領域(趙君,2017)。尤其是在食品行業領域內,纖維素酶在果蔬加工、白酒釀造、醬油釀造、食醋釀造、啤酒加工、飲料加工和茶葉加工等工藝過程中均具有重要作用(高蕾蕾和李迎秋,2017)。纖維素酶是由多種酶組成的多酶系統,包括纖維二糖水解酶、內切葡聚糖酶(CMCase)和α-葡萄糖苷酶(da Silva Delabona et al.,2012;Shanmugapriya et al.,2012)。但酶水解法在應用過程中易受底物、反應條件和酶活力等因素的影響,致使酶在諸多生產環境下無法有效水解纖維素而導致纖維素轉化率和降解物回收效率降低(Mojovi? et al.,2006;Eijsink et al.,2008)。因此,開發獲得高活力且對pH、溫度、離子及添加物等反應條件具有較好耐受性的纖維素酶對提高酶水解法工作效率具有重要意義。【前人研究進展】產纖維素的真菌包括曲霉屬、鐮刀菌屬、青霉菌屬和木霉屬,是獲得水解植物纖維素所需的酶復合物的來源(da Silva Delabona et al.,2012)。其中,里氏木霉在工業乙醇生產業、食品業、飼料生產行業、紡織品和紙漿紙張生產領域已有推廣應用(Fischer et al.,2020);但絲狀真菌(包括曲霉屬、青霉屬和木霉屬等)因生長較慢、菌體產色、纖維素酶產物抑制及低酶活力等(Lo et al.,2005),在一定程度上限制了其在纖維素酶生產和纖維質降解等領域的推廣與應用(閆訓友等,2004;文少白等,2010)。細菌因具有生長快、增殖周期短、代謝能力強等特點已成為獲得微生物源纖維素酶的理想來源之一(Rajendran et al.,2019)。至今,越來越多產纖維素酶的細菌菌株被篩選獲得,包括芽孢桿菌屬(Bacillus)(Hassan and Sohail,2020)、梭菌屬(Clostridium)(Gaur and Tiwari,2015)等均已用于生產纖維素酶。新的纖維素產生菌株,如藤黃色桿菌(Luteibacter)(李正風等,2020)和纖維單胞菌(Cellulomonas)(Shi et al.,2020)等也陸續被篩選得到。作為菌株資源的篩選來源,土壤(Reddy et al.,2018)、沙漠 (Shi et al.,2020;Thamer and Pravej,2020)及垃圾堆(Ma et al.,2015)、腸道和糞便(Peristiwati et al.,2018;Rajendran et al.,2019)、秸稈堆肥(李正風等,2020;Mohammadipour et al.,2020)等越來越受到研究人員的關注。其中,土壤作為資源微生物篩選的寶庫已成為各類微生物資源的重要篩選來源。Alvarado等(2020)利用新方法從土壤中分離出對革蘭氏陰性和革蘭氏陽性測試菌具有拮抗活性的放線菌;Rushabh等(2020)從番茄根際土壤中分離得到1株能產生吲哚-3-醋酸的細菌,并通過薄層色譜和高效液相對產物性質進行分析;Casta?eda-Cisneros等(2020)從山谷農業土壤中分離出鏈霉菌,并對其產生的內切葡聚糖酶(CMCase)和木聚糖酶(Xyl)的產量進行評價;Koul等(2021)從桑樹組織和根際土壤中分離得到2株蛋白酶產生菌。另外,具有降解農藥莠去津(Ye et al.,2016)、產生生物活性代謝物(Awad et al.,2018)、降解淀粉(Gudeta,2018)等能力的微生物也從土壤樣品中被篩選獲得。【本研究切入點】由于不同行業中纖維素酶的反應條件迥異,生產條件也較苛刻,迫切需要在苛刻或極端條件下能有效降解的纖維素酶。已報道纖維素酶產生菌的發酵條件與酶反應條件較溫和,而針對高產量的極端條件纖維素降解菌的研究報道極為少見。【擬解決的關鍵問題】通過平板篩選法,利用細菌鑒定結合分子生物學鑒別系統鑒定分離獲得高產堿性纖維素酶菌株,并對該菌株的纖維素酶類型、產酶發酵條件和粗酶的最適反應條件進行全面分析,為該菌株及所產纖維素酶的綜合開發利用打下基礎。
1 材料與方法
1. 1 試驗材料
1. 1. 1 樣品來源 土壤樣品采集于重慶師范大學(大學城校區)樹林下長期堆積腐爛枯葉的區域,分三點取樣,采樣深度10 cm。
1. 1. 2 培養基 (1)羧甲基纖維素篩選培養基:羧甲基纖維素鈉5.0 g,K2HPO4 1.0 g,MgSO4·7H2O 0.5 g,蛋白胨1.0 g,瓊脂20.0 g,NaCl 0.5 g,NaH2PO4 1.0 g,剛果紅0.2 g,蒸餾水1000 mL。(2)初步發酵培養基:CMCNa 5.0 g,蛋白胨2.5 g,酵母膏0.5 g,MgSO4 0.3 g,KH2PO4 2.0 g,NaCl 1.0 g,(NH4)2SO4 1.0 g,CaCl2·2H2O 0.3 g,FeSO4·7H2O 0.005 g,MnSO4 0.0016 g,ZnCl2 0.0017 g,CoCl2 0.0017 g,蒸餾水1000 mL,自然pH。(3)LB固體培養基:胰蛋白胨10.0 g,酵母提取物5.0 g,NaCl 10.0 g,瓊脂20.0 g,蒸餾水1000 mL。(4)β-葡萄糖苷酶篩選培養基:含0.1%七葉苷和0.05%檸檬酸高鐵銨的LB的培養基。
1. 1. 3 試劑 (1)常用試劑:七葉苷和檸檬酸高鐵銨購于Sigma公司,剛果紅、濾紙和CMCNa購自生工生物工程(上海)股份有限公司。(2)DNS試劑:稱45.5 g酒石酸鉀鈉加入125 mL熱水中,攪拌溶解后加入1.575 g的3,5-二硝基水楊酸和65.5 mL的2 mol/L NaOH,再加入1.25 g重蒸酚和1.25 g亞硫酸鈉,攪拌溶解,冷卻,用蒸餾水定容至250 mL,貯存在棕色瓶中備用。
1. 2 試驗方法
1. 2. 1 纖維素酶產生菌初步篩選 取5.0 g樣品加入到495 mL蒸餾水中,37 ℃搖床振蕩15 min后靜置30 min,取100 μL懸濁液加入到900 μL滅菌水中,混勻,稀釋至10-1;各吸取50 μL 10-1濃度的土樣懸濁液,將稀釋液涂布在羧甲基纖維素瓊脂篩選培養基上,放置于37 ℃恒溫箱中培養24 h后,觀察菌落生長情況及透明水解圈大小。
1. 2. 2 篩選平板復篩 觀察在篩選培養基上周圍有明顯透明水解圈的菌落,挑取透明圈直徑與菌落直徑比較大的菌株進行多次重復劃線純化,置于37 ℃恒溫箱中培養24 h直到獲得純菌株。將純化后的菌株接種至液體LB培養基中,于37 ℃、150 r/min條件下振蕩培養至渾濁,4 ℃保存備用。
1. 2. 3 菌株形態學觀察與生理生化分析 取分離菌株劃線接種在固體LB培養基上以獲取單菌落,觀察菌落特征,包括顏色、光澤、表面狀況、形狀、大小和質地等特征(張紀忠,1990)。生理生化特征分析主要參照《伯杰氏系統細菌學手冊》和《微生物學實驗手冊》(周德慶,1986;Sneath et al.,1986)。
1. 2. 4 基于16S rDNA序列的系統進化分析 以分離菌株總DNA為模板,采用細菌16S rDNA序列的通用引物8F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-ACGGTTACCTTGTTACGACTT-3')進行16S rDNA序列PCR擴增。PCR擴增程序:95 ℃預變性5 min;94 ℃ 40 s,55 ℃ 30 s,72 ℃ 90 s,進行32個循環;72 ℃延伸10 min;4 ℃ 保存(陳麗燕等,2011)。PCR產物送至生工生物工程(上海)股份有限公司進行序列測定。測序獲得的序列通過NCBI在線BLAST程序進行同源檢索并下載同源序列,使用ClustalX進行多重序列比對,并通過MEGA 7.0中的鄰接法(Neighbor-joining,NJ)構建系統發育進化樹。
1. 2. 5 纖維素酶粗酶液制備 將分離菌株按20%的比例接種到含250 mL發酵培養基的錐形瓶中,于30 ℃、170 r/min條件下振蕩培養1~6 d。每天取出1瓶發酵液,10000 r/min離心3 min,收集上清液即為細胞胞外酶液。
1. 2. 6 3,5-二硝基水楊酸(DNS)法測定酶活力
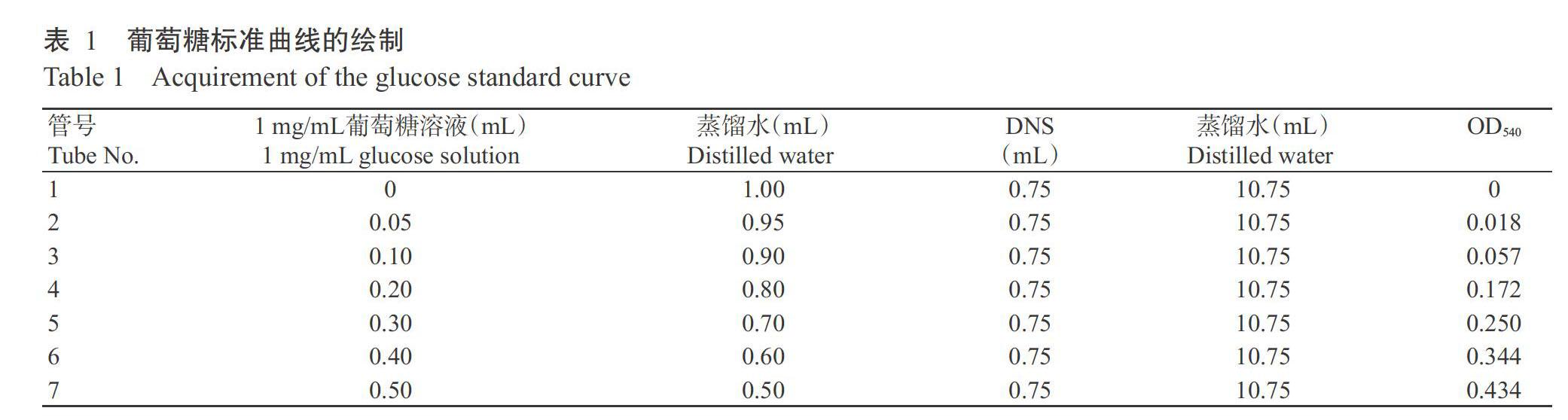
(1)葡萄糖標準曲線繪制:準確稱取104.8 mg恒重葡萄糖,用蒸餾水溶解并定容于100 mL容量瓶中,配制成1 mg/mL標準葡萄糖溶液,作為標準葡萄糖母液備用。按照表1完成各試劑的添加及反應,冷卻后分別在540 nm下比色,記錄吸光值(OD540),繪制葡萄糖標準曲線。
(2)菌株酶活力測定:分別以羧甲基纖維素鈉、濾紙和七葉苷為底物測定纖維素酶各組分(內切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶)的酶活力(Kirstahler et al.,2015)。0.5 mL菌體發酵上清液加入1.0 mL底物反應液,再加入0.5 mL pH 7.0的磷酸鹽緩沖液,在35 ℃水浴下反應30 min;加入1.0 mL DNS顯色反應液,沸水浴10 min,終止反應。冷卻后補足至5.0 mL,540 nm波長下測定OD540。酶活力定義:在35 ℃下,1.0 mL酶液每分鐘水解底物生成1 μg葡萄糖的酶量,稱為1個酶活力單位,以U/mL表示。
1. 2. 7 反應條件對菌株酶活力的影響 取發酵培養2 d后離心收集的上清液作為粗酶液,保持反應pH為7.0,測定不同反應溫度(10、 20、30、40、50、60、70、80和90 ℃)對纖維素酶活力的影響。收集發酵4 d后的粗酶液,在40 ℃反應條件下測定不同反應pH(5.0、6.0、7.0、8.0、9.0、10.0和11.0)對酶活力的影響。取發酵培養2 d后離心收集的上清液作為粗酶液,在溫度40 ℃和pH 7.0條件下,分別配制含有不同離子或化合物(K+、Ca2+、Fe3+、Mg2+、Ba2+、Cu2+、Na+、Zn2+、EDTA和尿素)的反應緩沖液,測定不同添加物對纖維素酶活力的影響。纖維素酶各組分的酶活力測定按前述方法進行,每組試驗均設3次獨立重復。
1. 2. 8 菌株發酵條件優化 將分離菌株按2%的接種量接種于發酵培養基中進行產酶條件優化。在180 r/min轉速的振蕩培養條件下,分別測定培養基初始pH(4.0、5.0、6.0、7.0、8.0、9.0、10.0、11.0和12.0)、培養溫度(20、25、30、35、40和45 ℃)及培養時間(1、2、3、4、5、6和7 d)對菌株產纖維酶活力的影響。
1. 3 統計分析
每個試驗條件下計算獲得3組平行試驗數據,使用SPSS 22.0對不同試驗條件下所獲取的數據進行T檢驗,使用GraphPad Prism 5進行圖表制作。
2 結果與分析
2. 1 纖維素酶產生菌的篩選與分離結果
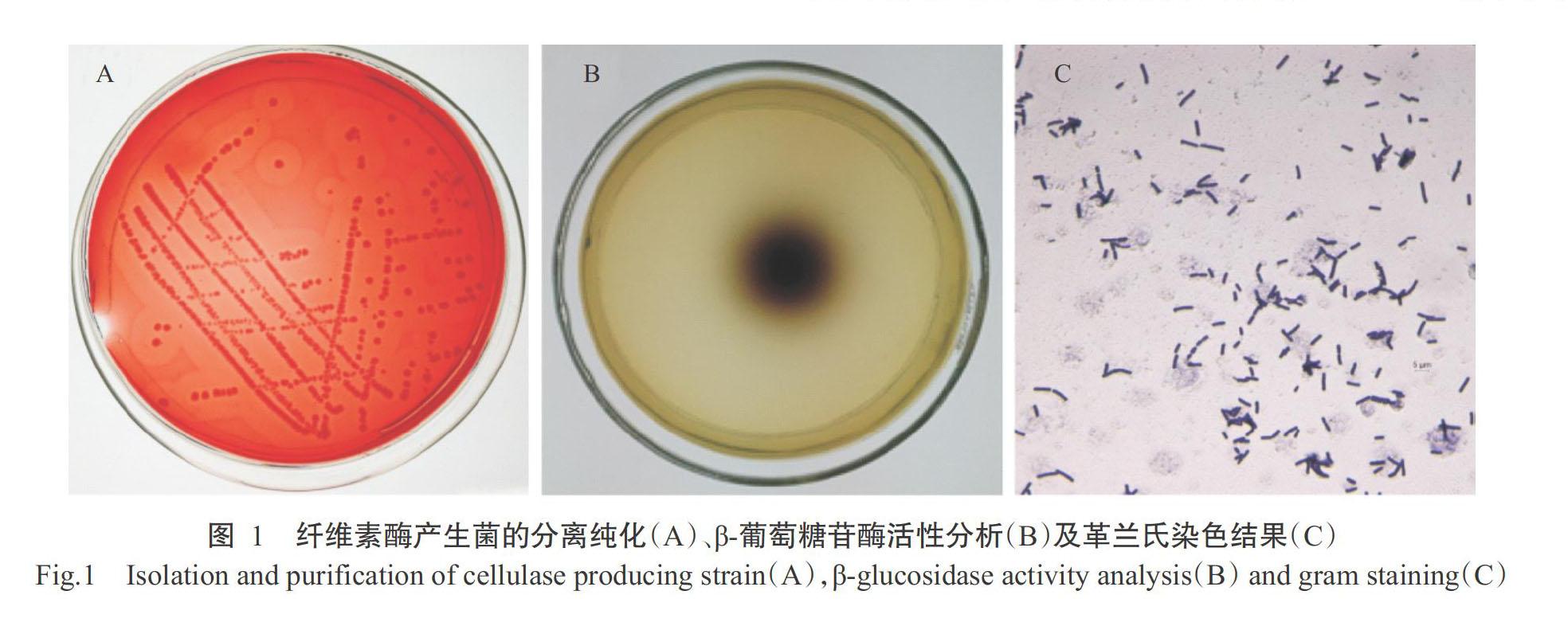
收集到的土壤樣品按前述方法進行稀釋,均勻涂布于以CMCNa為唯一碳源的篩選培養基上,37 ℃培養72 h后,挑選透明圈直徑(H)和菌落直徑(C)比值最大的分離菌株進行反復劃線純化,如圖1-A所示。結果表明,該分離菌株的H/C為5.08。在β-葡萄糖苷酶篩選培養基上菌落周圍能產生黑圈(圖1-B),表明其具有產生β-葡萄糖苷酶的能力。
2. 2 分離菌株的鑒定結果
2. 2. 1 菌落形態及生理生化鑒定 對純化獲得的細菌單菌落進行形態學觀察,其菌落為白色,偏大,扁平,圓形,邊緣不規則,不分泌色素(圖1-A)。顯微鏡觀察發現,其菌體呈長桿狀,革蘭氏陽性菌,無莢膜(圖1-C)。隨后,對分離菌株進行葡萄糖發酵、硝酸還原等生理生化鑒定,結果(表2)表明,該菌株為革蘭氏陽性菌,菌體為長桿狀,能利用葡萄糖、蔗糖、果糖,可水解淀粉,七葉苷、接觸酶、尿素分解和硝酸還原試驗等呈陽性,甲基紅(M.R)反應和V-P試驗呈陰性。各項生理生化指標特征與芽孢桿菌屬的代表種一致(周德慶,1986)。
2. 2. 2 基于16S rDNA序列的系統發育分析 提取分離菌株的基因組作為模板(圖2-A),采用細菌16S rDNA序列的通用擴增引物進行PCR擴增,擴增產物單一,且其分子量約1.6 kb,結果如圖2-B所示。
對PCR產物進行測序,獲得長度為1516 bp的DNA序列(GenBank登錄號MW314599)。多重序列比對分析結果表明,分離菌株的16S rDNA序列與B. cereus strain 55-4的16S rDNA具有最高的序列同源性。從GenBank中下載獲得系統發育分析所需的同源基因序列,使用ClustalX進行多重序列對比后,用MEGA 7.0構建系統發育進化樹,約束條件為Bootstrap方法1000個重復檢測。結果(圖2-C)表明,分離菌株與B. cereus的不同分離菌株聚為一枝,且與B. cereus strain 55-4進化距離最近。
綜上所述,分離菌株的菌落形態、生理生化特征與芽孢桿菌屬一致,16S rDNA序列與B. cereus strain 55-4的同源性達99%,在系統發育進化樹中與B. cereus菌株能聚為一枝,表明其進化關系較近。因此,確定分離菌株為蠟樣芽胞桿菌(B. cereus),其名稱為B. cereus strain CQNUX 3-1。
2. 3 纖維素酶各組分的酶活力分析結果
2. 3. 1 葡萄糖標準曲線 按前述方法,利用分光光度法獲得以葡萄糖濃度為橫坐標、OD540為縱坐標所得的標準曲線回歸方程為:y=0.8996x-0.0051,R2=0.9957。
2. 3. 2 不同底物條件下酶活力的測定 根據前述方法取發酵第4 d的粗酶液,底物分別為羧甲基纖維素鈉、濾紙和七葉苷,分別測定內切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶的酶活力。結果表明,粗酶液對3種底物均具有分解作用,其中降解七葉苷的β-葡萄糖苷酶活力高達155.6 U/mL,降解CMCNa的內切葡聚糖酶活力為107.7 U/mL,降解濾紙的外切葡聚糖酶活力為33.1 U/mL。
2. 4 反應條件對CQNUX 3-1菌株酶活力的影響
2. 4. 1 反應溫度對纖維素酶各組分酶活力的影響
取發酵培養2 d后離心收集的上清液作為粗酶液,保持反應pH為7.0,測定不同反應溫度對纖維素酶活力的影響,結果(圖3-A)表明,溫度對纖維素酶各組分酶活力的影響有所不同。其中,β-葡萄糖苷酶最適反應溫度為40 ℃,隨著反應溫度的增加,其酶活力明顯下降,但直至溫度達80 ℃時仍能保留69%左右的酶活力,約為79.3 U/mL;而外切葡聚糖酶和內切葡聚糖酶受反應溫度影響小,其最適溫度分別為60和70 ℃。
2. 4. 2 反應pH對纖維素酶各組分酶活力的影響
收集發酵4 d后的粗酶液,在40 ℃的反應條件下測定不同反應pH對酶活力的影響,結果(圖3-B)表明,β-葡萄糖苷酶酶活力最高,其最適反應pH為9.0;內切葡聚糖酶最適反應pH為8.0,且在pH達10.0時,仍保留92%的酶活力;外切葡聚糖酶活力受pH影響較低,在pH 7.0~9.0時酶活力相對較高。
2. 4. 3 離子和添加物對纖維素酶各組分酶活力的影響 取發酵培養2 d后離心收集的上清液作為粗酶液,在溫度為40 ℃和pH為7.0的條件下,分別配制含有各種離子的反應緩沖液,測定不同離子及添加物對纖維素酶活力的影響,結果(圖3-C)表明,Ca2+、Fe3+和Cu2+離子可顯著促進內切纖維素酶的活力,K+、Ca2+、Fe3+、Mg2+和Ba2+可增加β-葡萄糖苷酶的活力,僅Fe3+能促進外切葡聚糖酶的活力。可見,Fe3+在CQNUX 3-1菌株纖維素酶的應用過程中可作為極佳的酶活力促進離子。此外,Cu2+對外切葡聚糖酶有顯著的抑制作用,另外2種酶則具有良好的Cu2+耐受能力;β-葡萄糖苷酶對EDTA和尿素也具有較好的耐受性。
2. 5 發酵生產條件的優化
2. 5. 1 發酵時間對酶發酵的影響 制備發酵培養基,于37 ℃、150 r/min的恒溫搖床上對CQNUX 3-1菌株分別發酵培養2、3、4、5和6 d,離心獲得培養上清液作為粗酶液。在pH 7.0、40 ℃條件下反應,測定發酵時間對產纖維素酶各組分酶活力的影響,結果(圖4-A)表明,內切葡聚糖酶和β-葡萄糖苷酶在發酵第4 d酶活力最高,縮短或延長發酵時間均使得酶活力明顯降低;而外切葡聚糖酶在發酵第2~3 d時酶活力較高,隨著發酵時間的延長,酶活力有較小程度的降低。因此,該菌株發酵的最佳時間應控制在4 d。
2. 5. 2 初始pH對酶發酵的影響 使用具有不同初始pH的發酵培養基,于37 ℃、150 r/min的恒溫搖床上進行發酵,分析初始pH對酶發酵的影響,結果(圖4-B)表明,β-葡萄糖苷酶在發酵初始pH為7.0時的酶活力最高,且在pH 7.0~9.0時酶活力較穩定;而當pH達12.0時酶發酵受到明顯影響。外切葡聚糖酶在pH為5.0的條件下酶活力最高,pH 9.0~10.0時的酶活力為最高酶活力的77%,當pH達12.0時酶發酵受到明顯抑制。內切葡聚糖酶的活力在發酵pH為5.0時最高,其酶活力受發酵初始pH的影響較小。
2. 5. 3 發酵溫度對酶發酵的影響 分別在不同溫度下對菌株進行發酵培養4 d,粗酶液在pH 7.0、40 ℃的條件下反應,測定不同發酵溫度對纖維素酶各組分酶活力的影響,結果(圖4-C)表明,β-葡萄糖苷酶和外切葡聚糖酶的最佳發酵溫度為37 ℃,在42 ℃時仍能分別獲得90%和66%的酶活力;而內切葡聚糖酶的最適發酵溫度為30 ℃,其在37 ℃發酵可保留57%的酶活力。溫度低于25 ℃將不利于纖維素的發酵生產。CQNUX 3-1菌株的最佳發酵條件符合工業上通過液體發酵獲得纖維素的發酵要求(閆訓友等,2004)。
3 討論
纖維素作為最常見的有機聚合物,被認為是通過生物轉化生產不同產品的最重要自然可再生資源(Rajendran et al.,2019),但目前約有80%的纖維素尚未得到充分利用而被廢棄,進而引起環境污染等問題(Ragauskas et al.,2006;Illavarasi,2014)。微生物的酶解已被廣泛用于降解天然纖維素(Robson and Chambliss,1989),尋求高產纖維素酶生產微生物菌株成為高效降解纖維素的有效方法(Kersten and Cullen,2007)。本研究利用CMCNa平板篩選法,從長期覆蓋腐爛枯葉的土壤中篩選獲得1株高產堿性纖維素酶的菌株,經鑒定為蠟樣芽孢桿菌,命名為B. cereus strain CQNUX 3-1。目前,產纖維素酶微生物的報道包括放線菌、細菌和真菌等,其中放線菌因其產酶量低而研究較少;真菌中的絲狀真菌因產酶活力較強而備受關注;而細菌也因為產量不高、產酶組分單一、分泌性差等原因很少在工業中被應用,但細菌因其較快的發酵速度、較低的發酵營養條件要求等也具有較大的應用潛力。尤其是污物纖維素類分解和飼料發酵等環節均在氧含量較低或缺氧的條件下進行,此時厭氧及兼性厭氧型產纖維素酶細菌更具應用價值。出芽孢的細菌因能形成芽孢而對酸、堿和高溫環境耐受性較強,具有更高的工業生產應用潛力(吳敏峰等,2006)。因此,具有分泌能力且高產纖維素酶的出芽孢型細菌資源篩選對有效降解纖維素具有重要意義。目前,蠟樣芽胞桿菌(Bacillus cereus)已被報道能分泌產生蛋白酶(陳營等,2001)、膠原酶(李曄等,2016)和脂肪酶(韓雪和童攀,2017)等,是胞外酶生產的重要菌種資源。但蠟樣芽胞桿菌產纖維素酶的研究較少,已報道的蠟樣芽胞桿菌菌株產酶的活力并不高,主要集中在4.95~66.22 U/mL(禤金彩等,2014;汪彬等,2016)。相對而言,CQNUX 3-1菌株具有較佳的纖維素酶生產能力,致使其在纖維素的生物轉化過程中具有更佳的應用潛力。
對菌株產生的纖維素酶組分分析結果表明,CQNUX 3-1菌株具有較高的內切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶3種酶活力,分別達107.7、33.1和155.6 U/mL。近幾年,已報道來自自然界中的纖維素酶生產菌種達200多種(文少白等,2010),其中細菌種類繁多,包括纖維桿菌(Cellulomonas)、纖維粘菌(Cytophaga)及芽孢桿菌(Bacillus)等(劉娣,2008;陳麗燕等,2011),其內切葡聚糖酶(或CMCase)活力為17.08~358.276 U/mL(何楠等,2017;羅奉奉等,2017);而自然菌種經誘變選育后其CMCase活力可達453.2 U/mL(郭建強等,2017)。因此,CQNUX 3-1菌株可作為高產纖維素酶菌種誘變選育的重要候選資源。對纖維素酶系各組分的酶學性質進行分析,結果表明內切葡聚糖酶最適反應溫度為70 ℃,最適反應pH為8.0;外切葡聚糖酶最適反應溫度為60 ℃,在pH 7.0~9.0時酶活力最高;β-葡萄糖苷酶最適反應溫度為40 ℃,最適pH為9.0。目前,已報道的微生物源纖維素酶的最適反應溫度主要在45~65 ℃(高蕾蕾和李迎秋,2017),表明CQNUX 3-1菌株的纖維素酶具有較好的酶促反應溫度適用性,具有作為優良耐熱性堿性纖維素酶酶制劑而被應用的前景。一般認為,纖維素酶的最適反應pH在4.0~5.5(張鳳梅,2016),而CQNUX 3-1菌株產生的纖維素酶各組分在堿性條件下均表現出較高的酶活力,暗示其在堿性工業生產過程中具有較好的應用性(Long et al.,2020)。此外,Cu2+只對外切葡聚糖酶有顯著抑制作用,對其他2種酶的酶活力則具有良好的Cu2+耐受能力,較其他已報道的菌株具有明顯優勢(趙玉蓉等,2005;Zhang et al.,2008)。同時發現,β-葡萄糖苷酶對EDTA和尿素有較好的耐受性,表明該纖維素酶在洗滌工業和食品加工業廢水處理過程中有較好的應用前景。對菌株發酵條件的優化結果表明,CQNUX 3-1菌株各組分酶的最適發酵溫度為30~37 ℃,β-葡萄糖苷酶的最適發酵pH為7.0,內切葡聚糖酶和外切葡聚糖酶的最適發酵pH為5.0,且其在pH 7.0~10.0的堿性條件仍具有較高的發酵酶活力。內切葡聚糖酶和β-葡萄糖苷酶的最適發酵時間為4 d,而外切葡聚糖酶在發酵第2~3 d時的酶活力即達最高值。由此可見,CQNUX 3-1菌株具有較好的發酵耐酸堿性,且在較堿性發酵環境中仍具有較好的產酶能力。CQNUX 3-1菌株發酵產酶溫度適中,具有較寬的發酵pH條件適應性,使該菌株能更好地適應工業發酵生產條件,提示其具備應用于農業廢棄物生物轉化、環境纖維質污染物降解和纖維素資源綜合利用等領域的潛力,且可為之提供重要的菌株資源(Sadhu and Maiti,2013)。但本研究對該菌株所生產的純酶分離與酶活力分析尚待推進,后續研究工作將重點開展CQNUX 3-1菌株所產纖維素酶的分離純化,并對純酶的比活力、酶活特性及底物降解產物進行分析,為該菌在工業生產中的應用打下更加全面、堅實的基礎。
4 結論
篩選獲得的纖維素酶高產菌株B. cereus strain CQNUX 3-1所生產的纖維素酶具有較高的反應溫度適用性和較強的堿耐受性,菌株發酵產酶溫度適中,且有較寬的發酵pH適用范圍,可作為堿性纖維素酶生產資源菌株,具有應用于纖維素酶制劑制備與生產、纖維素資源綜合利用等領域的潛力。
參考文獻:
陳麗燕,張光祥,黃春萍,熊艷,李敏,常麗梅,張曉喻. 2011. 兩株高產纖維素酶細菌的篩選、鑒定及酶學特性[J]. 微生物學通報,38(4):531-538. doi:10.13344/j.microbiol.china.2011.04.017. [Chen L Y,Zhang G X,Huang C P,Xiong Y,Li M,Chang L M,Zhang X Y. 2011. Isolation,identification and enzymatic characteristics of cellulose-producing strains with high cellulase activity[J]. Micro-biology China,38(4):531-538.]
陳營,桂遠明,胡光源. 2001. 幾種離子及有機物對蠟樣芽胞桿菌蛋白酶活力的影響[J]. 中國微生態學雜志,13(3):146-148. doi:10.13381/j.cnki.cjm.2001.03.010. [Chen Y,Gui Y M,Hu G Y. 2001. Effects of some ions and organic compounds on protease activities of Bacillus cereus[J]. Chinese Journal of Microecology,13(3):146-148.]
高鳳菊,李春香. 2005. 真菌與細菌纖維素酶研究進展[J]. 唐山師范學院學報,27(2):7-10. doi:10.3969/j.issn.1009-9115.2005.02.003. [Gao F J,Li C X. 2005. Studies on cellulase from Eubacterium and Bacterium[J]. Journal of Tangshan Normal University,27(2):7-10.]
高蕾蕾,李迎秋. 2017. 纖維素酶及其在食品行業中的應用[J]. 食品工業,38(2):271-274. [Gao L L,Li Y Q. 2017. Ce-llulase and its application in food industry[J]. The Food Industry,38(2):271-274.]
郭建強,柴秀娟,龐學兵,裴娟,王愛英. 2017. 芽胞桿菌高產纖維素酶菌株的誘變選育與培養基優化[J]. 核農學報,31(2):263-270. doi:10.11869/j.issn.100-8551.2017.02. 0263. [Guo J Q,Chai X J,Pang X B,Pei J,Wang A Y. 2017. Mutation breeding and medium optimization of Bacillus sp. with high cellulase yielding[J]. Journal of Nuclear Agricultural Sciences,31(2):263-270.]
韓雪,童攀. 2017. 一株產脂肪酶蠟狀芽孢桿菌的分離鑒定[J]. 江漢大學學報(自然科學版),45(1):68-71. doi:10. 16389/j.cnki.cn42-1737/n.2017.01.011. [Han X,Tong P. 2017. Isolation of a lipase-producing strain of Bacillus cereus[J]. Journal of Jianghan University(Natural Science Edition),45(1):68-71.]
何楠,令利軍,馮蕾,蘇敏,姚彥敏,宋雪梅. 2017. 1株產纖維素酶細菌的篩選、鑒定及生長特性[J]. 微生物學雜志,37(1):43-49. doi:10.3969/j.issn.1005-7021.2017.01.007. [He N,Ling L J,Feng L,Su M,Yao Y M,Song X M. 2017. Isolation,identification and growth characteristics of a cellulase-producing bacterium[J]. Journal of Microbiology,37(1):43-49.]
李曄,張西軒,曹廣秀,張真,阮海華. 2016. 產膠原酶的蠟樣芽胞桿菌發酵條件優化及酶的分離純化[J]. 微生物學通報,43(7):1419-1428. doi:10.13344/j.microbiol.china.150519. [Li Y,Zhang X X,Cao G X,Zhang Z,Ruan H H. 2016. Fermentation optimization and purification conditions for collagenase from Bacillus cereus[J]. Microbiology China,43(7):1419-1428.]
李正風,朱杰,唐麗,董高峰,吳濤,廖頭根,張偉,夏玉珍,王奕權,李巖. 2020. 煙草秸稈中產纖維素酶細菌篩選、鑒定及酶活測定[J]. 西南農業學報,33(3):645-650. doi:10.16213/j.cnki.scjas.2020.3.027. [Li Z F,Zhu J,Tang L,Dong G F,Wu T,Liao T G,Zhang W,Xia Y Z,Wang Y Q,Li Y. 2020. Isolation,identification and cellulase enzyme activity determination of cellulase-producing bacteria from tobacco straw[J]. Southwest China Journal of Agri-cultural Sciences,33(3):645-650.]
劉娣. 2008. 秸稈纖維素高效降解真菌的篩選、鑒定及其纖維素酶基因克隆[D]. 北京:中國農業科學院. [Liu D. 2008. Screening,identification and cloning of cellulase gene of straw cellulose degrading fungi[D]. Beijing:Chinese Aca-demy of Agricultural Sciences.]
劉鎖珠,李龍,付冠華,王利紅,強巴央宗,李家奎. 2017. 藏豬源高產纖維素酶菌株的篩選及鑒定[J]. 西北農林科技大學學報(自然科學版),45(3):43-50. doi:10.13207/j.cnki.jnwafu.2017.03.007. [Liu S Z,Li L,Fu G H,Wang L H,Qiangba Y Z,Li J K. 2017. Screening and identification of high-yield cellulase degrading bacteria strain from Tibetan pigs[J]. Journal of Northwest A & F University(Natural Science Edition),45(3):43-50.]
羅奉奉,張昌偉,莫亞玲,岑忠用. 2017. 桑園土壤中高產纖維素酶菌株的篩選與鑒定[J]. 福建農業學報,32(4):425-430. doi:10.19303/j.issn.1008-0384.2017.04.014. [Luo F F,Zhang C W,Mo Y L,Cen Z Y. 2017. Screening and identification of high cellulase-producing bacteriafrom soil at mulberry fields[J]. Fujian Journal of Agricultural Scien-ces,32(4):425-430.]
師璐,李陽,李國龍,馬昕,顧金剛,李順意. 2017. 高產纖維素酶枯草芽胞桿菌S-16的篩選及其發酵工藝優化[J]. 生物資源,39(2):118-124. doi:10.14188/j.ajsh.2017.02.007. [Shi L,Li Y,Li G L,Ma X,Gu J G,Li S Y. 2017. Screening of high yield cellulase Bacillus subtilis S-16 and optimization of its fermentation process[J]. Biotic Resources,39(2):118-124.]
汪彬,郭照輝,杜東霞. 2016. 纖維素降解菌的分離、鑒定及其產酶特性研究[J]. 湖南農業科學,(9):7-9. doi:10.16498/j.cnki.hnnykx.2016.09.003. [Wang B,Guo Z H,Du D X. 2016. Research on isolation,identification and enzyme production characterization of cellulose degrading bacteria[J]. Hunan Agricultural Science,(9):7-9.]
文少白,李勤奮,侯憲文,李光義,鄧曉. 2010. 微生物降解纖維素的研究概況[J]. 中國農學通報,26(1):231-236. [Wen S B,Li Q F,Hou X W,Li G Y,Deng X. 2010. Recent advances in microbial degradation of cellulose[J]. Chinese Agricultural Science Bulletin,26(1):231-236.]
吳敏峰,耿秀蓉,祝小,潘康成. 2006. 產纖維素酶芽孢桿菌的分離鑒定[J]. 飼料工業,27(20):21-24. doi:10.3969/j.issn.1001-991X.2006.20.007. [Wu M F,Geng X R,Zhu X,Pan K C. 2006. Isolation and identification producing cellulase of Bacillus sp.[J]. Feed Industry,27(20):21-24.]
禤金彩,廖龍,龍寒,何秀苗. 2014. 一株產纖維素酶蠟樣芽孢桿菌的分離鑒定及酶學性質初步研究[J]. 南方農業學報,45(6):984-988. doi:10.3969/j:issn.2095-1191.2014.6. 984. [Xuan J C,Liao L,Long H,He X M. 2014. Isolation and identification of Bacillus cereus producing cellulase and enzymatic properties analysis[J]. Journal of Sou-thern Agriculture,45(6):984-988.]
閆訓友,史振霞,張惟廣,劉志敏. 2004. 纖維素酶在食品工業中的應用進展[J]. 食品工業科技,25(10):140-142. doi:10.3969/j.issn.1002-0306.2004.10.052. [Yan X Y,Shi Z X,Zhang W G,Liu Z M. 2004. Progresses in cellulase and its applications in food industry[J]. Science and Technology of Food Industry,25(10):140-142.]
張鳳梅. 2016. 微生物纖維素酶的研究概況及應用前景[J]. 青海畜牧獸醫雜志,46(3):48-50. doi:10.3969/j.issn. 1003-7950.2016.03.021. [Zhang F M. 2016. Research over-view and application prospect of microbial cellulase[J]. Chinese Qighai Journal of Animal and Veterinary Sciences,46(3):48-50.]
張紀忠. 1990. 微生物分類學[M]. 上海:復旦大學出版社. [Zhang J Z. 1990. Microbiological taxonomy[M]. Shanghai:Fudan University Press.]
趙君. 2017. 纖維素酶的研究與展望[J]. 中國飼料添加劑,(4):9-10. [Zhao J. 2017. Research and prospect of cellulase[J]. Chinese Feed Additive,(4):9-10.]
趙玉蓉,金宏,陳清華,王紅權,沈維軍,朱立濤. 2005. 金屬離子對纖維素酶及木聚糖酶活性影響的研究[J]. 飼料博覽,(1):1-3. doi:10.3969/j.issn.1001-0084.2005.01.001. [Zhao Y R,Jin H,Chen Q H,Wang H Q,Shen W J,Zhu L T. 2005. Effects of metal ions on the activity of feed enzymes[J]. Feed Review,(1):1-3.]
周德慶. 1986. 微生物學實驗手冊[M]. 上海:上海科學技術出版社. [Zhou D Q. 1986. Microbiology laboratory ma-nual[M]. Shanghai:Shanghai Science and Technology Press.]
Alvarado M,Clemente-Casares P,Moreno D A,De Groot P W. 2020. Micromundo upside down:Targeted searching for antibiotics-producing bacteria from soil with reverse antibiosis approaches[J]. Frontiers in Microbiology,11:577550. doi:10.3389/fmicb.2020.577550.
Awad N E,Kassem H A,Hamed M A,El-Feky A M,Elnaggar M A A,Mahmoud K,Ali M A. 2018. Isolation and characterization of the bioactive metabolites from the soil derived fungus Trichoderma viride[J]. Mycology,9(1):1-11. doi:10.1080/21501203.2017.1423126.
Casta?eda-Cisneros Y E,Mercado Y,Anducho-Reyes M A,?lvarez Cervantes J,Ponce-Lira B,Evangelista-Martínez Z,Téllez A. 2020. Isolation and selection of streptomyces species from semi-arid agricultural soils and their potential as producers of xylanases and cellulases[J]. Cu-rrent Microbiology,77(2):3460-3472. doi:10.1007/s00284- 020-02160-7.
da Silva Delabona P,Pirota R D B,Codima C A,Tremacoldi C R,Rodrigues A,Farinas C S. 2012. Using amazon fo-rest fungi and agricultural residues as a strategy to produce cellulolytic enzymes[J]. Biomass and Bioenergy,37:243-250. doi:10.1016/j.biombioe.2011.12.006.
Eijsink V G,Vaaje-Kolstad G,V?rum K M,Horn S J. 2008. Towards new enzymes for biofuels:Lessons from chiti-nase research[J]. Trends in Biotechnology,26(5):228-235. doi:10.1016/j.tibtech.2008.02.004.
Fischer A J,Maiyuran S,Yaver D S. 2020. Industrial relevance of trichoderma reesei as an enzyme producer[M]//Mach-Aigner A R,Martzy R. Methods in Molecular Bio-logy,New York:Humana Press:23-43.
Gaur R,Tiwari S. 2015. Isolation,production,purification and characterization of an organic-solvent-thermostable alkalophilic cellulase from Bacillus vallismortis RG-07[J]. BMC Biotechnology,15(1):19. doi:10.1186/s12896-015-0129-9.
Gudeta D. 2018. Isolation and characterization of starch degrading rhizobacteria from soil of Jimma University Main Campus,Ethiopia[J]. African Journal of Microbiology Research,12(32):788-795. doi:10.5897/ajmr2018.8873.
Hassan M,Sohail M. 2020. Characterization of cellulases from thermophilic bacilli and their application for the saccharification of sugarcane bagasse[J]. Pakistan Journal of Botany,52(5):1785-1791. doi:10.30848/PJB2020-5(39).
Illavarasi S. 2014. Isolation and identification of cellulase producing bacteria from cow dung[J]. Scrutiny International Research Journal of Microbiology and Bio Techniques,1(1):6-12.
Kersten P,Cullen D. 2007. Extracellular oxidative systems of the lignin-degrading basidiomycete phanerochaete chrysosporium[J]. Fungal Genetics and Biology,44(2):77-87. doi:10.1016/j.fgb.2006.07.007.
Kirstahler P,Gunther M,Grumaz C,Lindemann E,Rupp S,Zibek S,Sohn K. 2015. Draft genome sequence of amantichitinum ursilacus igb-41,a new chitin-degrading bacterium[J]. Microbiology Resource Announcements,3(6):e01309-15. doi:10.1128/genomeA.01309-15.
Koul D,Chander D,Manhas R S,Chaubey A. 2021. Isolation and characterization of serratiopeptidase producing bacteria from mulberry phyllosphere[J]. Current Microbiology,78:351-357. doi:10.1007/s00284-020-02280-0.
Lo C M,Zhang Q,Lee P,Ju L K,2005. Cellulase production by Trichoderma reesei using sawdust hydrolysate[J]. Applied Biochemistry and Biotechnology, 122:561-573.
Long C,Ro?sch C,de Vries S D,Schols H,Venema K. 2020. Cellulase and alkaline treatment improve intestinal microbial degradation of recalcitrant fibers of rapeseed meal in pigs[J]. Journal of Agricultural and Food Chemistry,68(39):11011-11025. doi:10.1021/acs.jafc.0c03618.
Ma Z,Tang J,Lan X,Wen H,Chen Q,Li T,Xu J,Li C,Zhou Z. 2015. Isolation of a thermostable alkaline cellulase-producing bacterium strain from a garbage dump[J]. International Journal of Agriculture and Biology,17(3):625-630. doi:10.17957/IJAB/17.3.13.485.
Mohammadipour Z,Enayatizamir N,Ghezelbash G,Moezzi A. 2020. Bacterial diversity and chemical properties of wheat straw-based compost leachate and screening of cellulase producing bacteria[J]. Waste and Biomass Valorization,12:1293-1302. doi:10.1007/s12649-020-01119-w.
Mojovi? L,Nikoli? S,Rakin M,Vukasinovi? M. 2006. Production of bioethanol from corn meal hydrolyzates[J]. Fuel,85(12-13):1750-1755. doi:10.1016/j.fuel.2006. 01.018.
Peristiwati,Natamihardja Y S,Herlini H,2018. Isolation and identification of cellulolytic bacteria from termites gut (Cryptotermes sp.)[J]. Journal of Physics:Conference Series,1013(1):012173. doi:10.1088/1742-6596/1013/1/012173.
Ragauskas A J,Williams C K,Davison B H,Britovsek G J P,Cairney J,Eckert C A,Frederick W J,Hallett J P,Leak D J,Liotta C L. 2006. The path forward for biofuels and biomaterials[J]. Science,311(5760):484-489. doi:10.1126/ science.1114736.
Rajendran S,Croos A M B,Kapilan R. 2019. Isolation of a cellulase producing Bacillus cereus from cow dung[J]. Journal of the National Science Foundation of Sri Lanka,47(2):261-267. doi:10.4038/jnsfsr.v47i2.9168.
Reddy K V,Vijayalashmi T,Ranjit P,Raju M N. 2018. Characterization of some efficient cellulase producing bacteria isolated from pulp and paper mill effluent contaminated soil[J]. Brazilian Archives of Biology and Technology,60:e17160226. doi:10.1590/1678-4324-2017160226.
Robson L M,Chambliss G H. 1989. Cellulases of bacterial ori-gin[J]. Enzyme and Microbial Technology,11(10):626-644. doi:10.1016/0141-0229(89)90001-X.
Rushabh S,Kajal C,Prittesh P,Amaresan N,Krishnamurthy R. 2020. Isolation,characterization,and optimization of indole acetic acid-producing Providencia species(7MM11)and their effect on tomato(Lycopersicon esculentum) seedlings[J]. Biocatalysis and Agricultural Biotechnology,28:101732. doi:10.1016/j.bcab.2020.101732.
Sadhu S,Maiti T K. 2013. Cellulase production by bacteria:A review[J]. British Microbiology Research Journal,3(3):235-258. doi:10.9734/BMRJ/2013/2367.
Shanmugapriya K,Saravana P,Krishnapriya M M,Mythili A,Joseph S. 2012. Isolation,screening and partial purification of cellulase from cellulase producing bacteria[J]. International Journal of Advanced Biotechnology and Research,3(1):509-514.
Shi Y L,Sun Y,Ruan Z Y,Su J,Yu L Y,Zhang Y Q. 2020. Cellulomonas telluris sp. nov.,an endoglucanase-produ-cing actinobacterium isolated from badain jaran desert sand[J]. International Journal of Systematic and Evolutionary Microbiology,70(1):631-635. doi:10.1099/ijsem.0.0038 06.
Sneath P H A,Mair N S,Sharpe E,Holt J G. 1986. Bergeys manual of systematic bacteriology(volume 2)[M]. Baltimore:Williams and Wilkins Company.
Thamer H A,Pravej A. 2020. Isolation and molecular identifications of cellulase producing bacteria from desert soil[J]. Research Journal of Biotechnology,15(3):72-78.
Ye J,Zhang J,Gao J,Li H,Liang D,Liu R. 2016. Isolation and characterization of atrazine-degrading strain Shewanella sp. YJY4 from cornfield soil[J]. Letters in Applied Microbiology,63(1):45-52. doi:10.1111/lam.12584.
Zhang C,Dai L,Yu H,Bo Z,Jin F. 2008. Purification and characterization of piceid-β-glucosidase from Aspergillus oryzae[J]. Process Biochemistry,42(1):83-88. doi:10. 1016/j.procbio.2006.07.019.
(責任編輯 王 暉)

