利用多時序激光點云數據提取棉花表型參數方法
陽旭 胡松濤 王應華 楊萬能 翟瑞芳


摘要: 當前,能夠實現作物表型參數高效、準確的測量和作物生育期表型參數的動態量化研究是表型研究和育種中亟待解決的問題之一。本研究以棉花為研究對象,采用三維激光掃描LiDAR技術獲取棉花植株的多時序點云數據,針對棉花植株主干的幾何特性,利用隨機抽樣一致算法(RANSAC)結合直線模型完成主干提取,并對剩余的點云進行區域增長聚類,實現各葉片的分割;在此基礎上,完成植株體積、株高、葉長、葉寬等性狀參數的估計。針對多時序棉花激光點云數據,采用匈牙利算法完成相鄰時序作物點云數據的對齊、葉片器官對應關系的建立。同時,對各植株表型參數動態變化過程進行了量化。本研究針對3株棉花的4個生長點的點云數據,分別完成了主干提取、葉片分割,以及表型參數測量和動態量化。試驗結果表明,本研究所采用的主干提取及葉片分割方法能夠實現棉花的枝干和葉片分割。提取的株高、葉長、葉寬等表型參數與人工測量值的決定系數均趨近于1.0;同時,本研究實現了棉花表型參數的動態量化過程,為三維表型技術的實現提供了一種有效的方法。
關鍵詞: 棉花表型參數;LiDAR;主干提取;葉片分割;點云數據對齊;三維表型
中圖分類號: TP391.4????????? 文獻標志碼: A?????????? 文章編號: 202102-SA003
引用格式:陽旭, 胡松濤, 王應華, 楊萬能, 翟瑞芳. 利用多時序激光點云數據提取棉花表型參數方法[J]. 智慧農業(中英文), 2021, 3 (1): 51-62.
Citation:YANG Xu, HU Songtao, WANG Yinghua, YANG Wanneng, ZHAI Ruifang. Cotton phenotypic trait extraction using multi-temporal laser point clouds[J]. Smart Agriculture, 2021, 3 (1): 51-62. (
1? 引? 言
近年來,隨著農作物表型組學和表型技術的發展,三維表型技術由于能夠獲取比二維表型技術多一個維度的信息,逐漸引起了高通量表型研究人員的關注和興趣。其中,激光雷達(Light Detection and Ranging,LiDAR)技術作為一種通過發射激光脈沖快速獲取目標空間數據的有效手段,既能無損地獲取目標對象的三維信息,也克服了傳統的計算機視覺技術重建作物的算法復雜度高和耗時長等不利因素,為三維空間中表型參數的自動測量提供了一種新思路。農作物是一種典型的形態結構隨著時間變化而不斷發生變化的對象,對不同生育期的作物進行持續的點云數據采集,既能夠獲取作物某一生長點的表型參數,也可以實現作物表型參數的動態變化過程,為育種學家提供更具參考價值的表型數據。
現階段,作物的三維點云信息主要通過2種途徑獲取。一是利用基于圖像的計算機視覺方法重建作物目標,如葡萄[1]、番茄[2]、大豆[3]、玉米[4]以及植物根系[5]等。基于圖像的計算機視覺方法一般通過相機標定、特征提取、特征匹配以及光束法平差等關鍵步驟實現,對獲取的圖像有約束性要求,如保證相鄰視點影像間較高的重疊度,且生成的點云數據質量和圖像數量關系較大。當作物本身缺乏足夠特征時,需引入結構光等獲得較豐富的紋理特征,以期獲得較高精度的點云數據。Nguyen等[6]搭建結構光系統獲取植物多視影像,獲得多視點云,并完成點云間的配準,構建了植株的完整三維模型;在此基礎上,完成葉片數量、株高、葉片大小和節間距等參數的測量。Ni等[7]利用三臺相機獲取120幅藍莓簇圖像數據,重建藍莓點云及模型。針對獲取的二維圖像數據,利用Mask-RCNN實現單粒藍莓的分割及成熟度檢測,并將分割結果反投回三維空間,在三維空間中完成各藍莓表型參數的提取。二是利用LiDAR技術直接獲取目標三維信息。該類技術首先獲取作物對象不同視點的三維點云數據,再利用最臨近點迭代法(Iterative Closest Point,ICP)完成多視點的點云配準,生成目標完整的三維模型。利用LiDAR技術開展三維表型研究可概括為2個方面:點云數據處理方法和表型參數提取方法研究。從作物尺度進行劃分,LiDAR技術可分別應用在單株作物和大田作物中,對應地獲取不同尺度的表型參數。針對大田作物,提取的表型參數主要包括平均冠層高度、投影面積、植株密度、生物量等信息。Jiang等[8]對田間獲取的激光點云數據研發了一套和藍莓收獲相關的表型參數獲的三維表型技術,包括大小、尺度等。Jin等[9]提出了一種均值歸一化向量生長算法(Median Normalized Vector Growth,MNVG)實現田間玉米的莖葉分割,完成了葉面角、莖高、株高等表型參數的測量。Sun等[10]針對重建的田間成熟期棉花植株,利用密度聚類方法實現了棉鈴球的分割和計數,為棉花估產提供數據支持。劉守陽等[11]通過構建的數字化植物表型平臺(Digital Plant Phenotyping Platform,D3P)獲取田間小麥的三維冠層場景,記錄了綠色葉面積指數、平均傾角和散射光截獲率等信息,在此基礎上模擬虛擬LiDAR實驗,生成了三維點云數據,展示了LiDAR數據在田間冠層光截獲和冠層結構方面的潛力。針對單株作物,主要采用近距離或手持式激光掃描對象,從葉、莖、甚至根等器官角度完成各表型參數的提取。其中,點云配準和點云分割分別是獲取三維點云模型和提取表型參數的關鍵步驟。馬福峰等[12]提出了一種基于植株點云鄰域幾何特征約束的配準方法,選取關鍵點并估計其中每個點的支撐鄰域估算出鄰域幾何特征,利用幾何特征實現點云的初始配準。Chaudhury和Barron[13]將不同視點的點云投影至二維平面上,通過定位節點位置來建立點云間的初始對應關系,該算法實現的前提是點云數據中存在直線特征。點云分割是獲取表型參數的前提。適用于作物器官的點云分割方法主要3種:①基于三維區域增長的方法,通過利用區域內部點云的相似屬性,制定合適的區域增長規則來完成,主要應用在植株葉片的分割中,如油菜葉片[14]、綠蘿和龜背竹[15]等;②基于模型擬合的點云分割方法,利用植株中的直線、圓柱體、平面等典型幾何模型來完成,在作物主莖的分割中應用較多,如向日葵[16]和玉米主莖[17]等;③基于機器學習的方法,代表性的算法包括支持向量機(Support Vector Machine,SVM)和均值漂移(Mean Shift)等各種聚類算法。Wahabzada等[18]在獲取植株模型的基礎上,使用無監督聚類的方法,完成了葡萄、小麥和大麥的葉片和莖稈的分割。喻垚慎等[19]融合了點云數據的多維特征,利用人工標記數據為樣本,結合SVM分類器實現了樹木枝葉分離,屬于有監督分類方法。
按照一定的時間間隔多次采集作物對象的點云數據即可得到作物的多時序點云數據,這些數據中包含了作物對象的時空形態變化信息。針對多時序作物點云數據的研究可為作物表型參數的動態變化提供強有力的手段和方法,同時,也可為構建作物的生長模型提供數據基礎。Su等[20]使用激光掃描儀獲取了玉米6個生長階段的點云數據,計算了玉米株高、葉面積和投影葉面積3個表型參數,并研究表型參數的動態變化趨勢。Chaudhury等[21]構建了基于激光掃描儀的近景視覺系統,對野生擬南芥和大麥植株完成不同視點的點云完成配準,通過構建表面不規則網實現植株基于網格的面積計算和體積計算,并繪制了植株從觀測第1天到第22天的面積和體積連續變化曲線。Sun等[22]使用激光掃描系統對大田棉花完成了全生育期的表型參數提取和動態變化分析,探討了表型參數和產量之間的關系。An等[23]對擬南芥進行了連續10天的二維影像數據采集,分別從二維影像和三維模型的角度對比葉片表型參數測量結果的差異性,結果表明與傳統的二維影像相比,三維模型能夠獲得精度更高的表型參數。以上研究表明,雖然三維表型技術已經取得了一定的進展,但針對不同作物植株,數據處理方法各異,因此,其潛力還有待進一步挖掘。
棉花是中國四大經濟作物之一,是紡織工業的主要原材料,最大程度地提高棉花產量是栽培和育種的首要目標,對中國的農業經濟發展意義重大,而各生育期的表型參數可為該目標的實現提供數據基礎。為進一步挖掘三維表型技術在單株棉花植株上的潛力,本研究以單株棉花為研究對象,利用激光掃描技術獲取多時序點云數據,研究主干和葉片的分割算法,實現株高、葉長、葉寬、葉面積,以及棉花植株體積等表型參數的計算。在此基礎上,實現棉花植株表型參數的動態量化過程,從而為三維空間的高通量表型組學及構建作物生長模型提供方法支撐和數據依據。
2? 材料與方法
2.1 點云數據獲取
本研究所使用ZGScan手持式激光掃描儀(武漢中觀自動化科技有限公司)獲取3株棉花共12個時間點的點云數據。在掃描過程中使用的輔助掃描工具包括激光掃描標記點、支架以及掃描數據輸入和存儲的惠普筆記本電腦。使用戴爾計算機進行后期點云數據分析和處理。該計算配置為:Windows 64位操作系統,16 GB運行內存,處理器為主頻3.4 GHz的Inter(R) Core i7-3770 CPU。圖1分別為移植后的棉花植株和掃描設備。棉花植株于2018年8月10日在實驗室中進行培養,待長至約16 cm后移至栽培盆中種植。
株高是作物形態學重要參數之一,葉片是作物進行光合作用和呼吸作用的主要載體。棉花生長前期,葉和莖稈的生長最活躍。莖稈的生長,葉片數和葉面積的增長為后期的營養生長和生殖生長積累大量的有機物,是影響棉花高產的重要因素之一。鑒于莖、葉對作物生長發育的重要性,本研究主要探討LiDAR技術在棉花株高、葉長、葉面積,以及體積等重要表型參數測量方面的潛力,針對3株棉花的4個時間點進行點云數據采集,具體為種植之后的第46天、第49天、第58天和第63天,共采集12組棉花點云數據。隨著棉花植株的生長,其形態結構發生了變化,主要表現為植株漸高,部分新葉萌出,部分葉片面積增大,以及少數老葉脫落,葉傾角漸大,節間距有少許增大等趨勢。使用Geomagic Studio 2013軟件對采集的原始棉花點云數據進行預處理,包括刪除栽培盆、支架等噪聲數據。完成棉花植株點云數據預處理后,棉花植株的點云數據分別如圖2所示。
2.2 棉花器官分割方法
后續研究需要通過作物點云數據提取包括株高、葉長、葉寬、以及體積等一系列表型參數,并對其動態變化進行量化。因此,需要對棉花植株點云數據進行器官分割,即將完整的棉花植株點云數據分割成若干互不相交的點云子集。
2.2.1 主干提取
由圖2可以看出,棉花植株的主干趨于筆直。因此,根據棉花植株的形態結構以及點云數據特點,采用基于模型的檢測方法——隨機采樣一致性(Random Sample Consensus,RANSAC)算法結合直線模型提取主干部分。RANSAC算法是一種通過隨機采樣觀測數據估計模型參數的方法。首先,在原始數據集中隨機指定一組數據集合,設定一個適合該數據的參數模型以及置信度參數。然后,通過迭代選擇原始數據的隨機子集來不斷優化模型的參數。達到迭代終止條件后,選取局內點數量最多的模型即為所求結果。對于多分枝株型的作物,可以采取多次使用RANSAC的方式,直到所有的類直線型枝干都被提取出為止。
2.2.2 葉片分割
在使用RANSAC方法對植株進行主干分割后,得到包括多個葉片的彼此分離的點云數據,只需要將葉片點云進行聚類即可。因此,對提取主干后的剩余點云數據使用歐式聚類分割算法即可得到獨立的葉片。歐式聚類分割算法的步驟如下。
(1)為輸入的點云數據集P創建K-D樹;
(2)構建一個聚類C的空列表以及需要檢驗的點的隊列Q;
(3)在當前點云數據集P中隨機選取點P_i,執行下面的步驟。
①將P_i添加到當前隊列Q;
②對于每一個屬于Q中的點P_i:
在半徑r < dth的范圍內搜索與點P_i相鄰的點的集合P_k^i;
③對于每一個屬于P_k^i集合中的點,檢查當前點是否已經被處理過,如果沒有則將其添加到隊列Q中;
④當隊列Q中的所有點全部處理后,將隊列Q中的點添加到聚類C中,同時將隊列Q重置為空;
(4)當P中的所有點都被處理完并且屬于聚類C中的一部分時結束算法。
2.3 相鄰生育期作物點云配準及葉片對應關系建立
為研究同一株作物生長變化及相鄰時期表型參數動態變化,需要將相鄰時期的作物點云數據進行配準[24]。主要包括兩個步驟:(1)點云數據粗配準;(2)建立作物葉片器官間的一一對應關系。
2.3.1 相鄰生育期點云粗配準
點云配準根據精度可分為粗配準和精配準。粗配準是源點云與目標點云在完全未知任何初始相對位置情況下的配準。該操作的主要目的是在初始條件未知的情況下,快速估算一個大致的點云配準矩陣。較典型的是基于全局搜索策略的代表算法采樣一致性初始配準(SAmple Consensus Initial Alignment,SAC-IA)算法。該算法在源點云與目標點云之間,隨機選取幾何特征一致的點組成點對而非所有存在對應關系的組合,通過計算對應點對的變換關系得到最優解[25]。
首先,計算點云的快速點特征直方圖(Fast Point Feature Histograms,FPFH)。通過參數化查詢點與鄰域點之間的空間差異,形成描述查詢點的K鄰域幾何屬性的多維直方圖。
計算每個點的FPFH后,使用采樣一致性初始配準算法完成點云粗配準,具體如下。
(1)從待配準點云S中選取n個采樣點,為保證所采樣的點具有不同的FPFH特征,采樣點兩兩之間的距離應滿足大于預先給定最小距離閾值d;
(2)在目標點云R中查找與點云S中采樣點具有相似FPFH特征的一個或多個點,從這些相似點中隨機選取一個點作為點云S在目標點云R中的一一對應點;
(3)計算對應點之間的變換矩陣,通過求解對應點變換后的“距離誤差和”函數來判斷當前配準變換的性能。此處的距離誤差和函數多使用Huber誤差函數表示,記為H(l_i),見公式(1)。
H(l_i )={■(1/2 l_i^2,??????????????????????????????????????????????? ?????????????????????????? ??????????????????? ??????????????????????????????????????????????? ??????????????????????????????????? ??????????? ??????????????????????????????????????????????? ??????????????????????????????????????????? ??? ??????????????????????????????????????????????? ??????????????????????????????????????????????? ??? ??????????????????????????????????????????? ??????????????????????????????????????????????? ??????????? ??????????????????????????????????? ??????????????????????????????????????????????? ??????????????????? ??????????????????????????? ??????????????????????????????????????????????? ??????????????????????????? ??????????????????? ??????????????????????????????????????????????? ??????????????????????????????????? ??????????? ??????????????????????????????????????????????? ??? ??????????????????????????????????????? ??? ??????????????????????????????????????????????? ??????????????????????????????????????????????? ??? ??????????????????????????????????????????? ??????????????????????????????????????????????? ??????????? ??????????????????????????????????? ??????????????????????????????????????????????? ??????????????????? ??????????????????????????? ??????????????????????????????????????????????? ??????????????????????????? ??????????????????? ??????????????????????????????????????????????? ??????????????????????????????????? ??????????? ??????????????????????????????????????????????? ??????????????????????????????????????????? ??? ??????????????????????????????????????????????? |(|l_i | )|
2.3.2 相鄰生育期棉花葉片對應
為量化作物表型參數的動態變化過程,在作物點云數據粗對齊的基礎上,需要建立植株葉片間的對應關系。
對于同一株作物T_i和T_(i+1)時期的兩組點云,作物T_i時期葉片數為m,T_(i+1)時期葉片數為n。隨著植物的生長,可能會發生新葉長出或老葉脫落等現象,因此,葉數m和n可能不同,下面將分別討論葉片數m和n相同和不同的兩種數據處理情況。
(1)當m=n時。計算T_i和T_(i+1)時期植株每個葉片的中心點,然后分別計算T_i時期每個葉片中心點到T_(i+1)時期每個葉片中心點的三維歐氏距離,將T_i和T_(i+1)時期葉片中心點的三維歐式距離構建一個m × n的鄰接矩陣M,M(i,j) 表示T_i時期第i個葉片中心到T_(i+1)時期第j個葉片中心的三維歐式距離。設有2組葉片分別為L1、L2、L3和L1'、L2'、L3',圖3描述了這兩組葉片各中心點的歐式距離。矩陣的行標和列標分別代表T_i和T_(i+1)時期的各葉片編號。T_i和T_(i+1)時期植株的點云數據處于同一個坐標系下,使用匈牙利算法(Hungarian Algorithm)來對矩陣M求解,使得相鄰時期葉片中心點的三維歐式距離之和最小,此時即為相鄰時期植株葉片間的最佳匹配,如表1中加粗距離值所示。可得出結論,L1和L2'為同一組葉片,L2和L1'為同一組葉片,而L3則和L3'形成配對關系。
(2)當m < n時。對于同一株作物T_i和T_(i+1)兩個時期植株有效葉片數目不同的情況,將T_i時期的葉片數目補充為n個,同時將T_i時期n-m個葉片中心到T_(i+1)時期每個葉片中心的距離都記為0,并構建一個n × n的矩陣,然后使用匈牙利算法來對該矩陣進行求解,使得T_i和T_(i+1)兩時期葉片中心三維歐式距離之和最小,此時得到T_i時期m個葉片與T_(i+1)時期m個葉片的對應關系。T_(i+1)時期剩余n-m個葉片在T_i時期無葉片與其對應,則記為新長出的葉片。
各生育期的棉花葉片間一一對應關系建立后,即可完成株高、葉長、葉寬等一系列表型參數的計算,并可以構建各表型參數的時序變化圖。
3 ?試驗和結果分析
3.1 棉花主干和葉片分割結果
RANSAC實現作物主干提取的理想的結果是葉柄與主干分離,主干部分的噪聲少并且主干以外的部分葉片數與人工計數結果相同。在使用RANSAC算法實現作物主干提取過程中,不同的參數對結果產生不同的影響。點到模型的距離閾值過小,則提取到的主干不全,距離閾值過大則不能正確地提取主干部分。如果迭代次數過少,則不能得到最優的模型局內點,迭代次數過大會增加算法運行時間。因此,合適的參數選擇至關重要。在迭代次數固定的情況下,分別討論了閾值為1.0、6.0和18.0 mm的主干提取結果,如圖3(a)所示。經過多次試驗發現當迭代次數為2000次,距離閾值設置在5.0~10.0 mm時,如表2所示,可以得到較為理想的棉花植株主干提取結果。
對于多分枝株型的作物,可以采取多次使用RANSAC的方式,直到所有的類直線型枝干都被提取出為止。使用RANSAC算法進行分割的評價標準根據目的而定。在本試驗中,目視評價指標是主干應能反映出作物的株高;量化RANSAC算法提取主干精度的評價指標是主干實際長度與提取主干長度的比值,比值越接近1,精度相對越高。
歐式聚類分割算法的理想結果是分割后的聚類的葉片數量與人工計數結果相同且分割結果中葉片完整。算法實現棉花葉片的分割過程中,需要設置三個參數,近鄰搜索半徑、最小聚類點數和最大聚類點數。最小聚類點數和最大聚類點數用于排除噪聲以及避免輸出近鄰搜索半徑設置不佳時的錯誤結果,根據葉子的點云分布情況,一般最大聚類點設置大于點云數量的一半、最小聚類點設置不超過點云數量的10%;近鄰搜索半徑和點與點之間的距離均值有關,如果近鄰搜索半徑過大,則點云無法分割,如果過小則會導致分割結果錯誤。經過一系列的試驗驗證,各參數如下設置時,可以取得較好的分類結果:最小聚類點數為800個,最大聚類點數為155,000個,近鄰搜索半徑為120 mm。如圖3(b)所示,最小聚類點數為100個時,主干提取后留下的非葉片點云噪聲被單獨分割出來,對最終結果產生影響;距離閾值為80 mm時,由于葉片區域平均點間距大于葉柄區域,閾值小于葉片區域平均點云間距時,葉片被分割為大量細碎的點,因此在最小聚類點數的限制下只有較為密集的葉柄部分和葉片中的密集區域會被輸出。故當提高最小聚類點數到800個后,非葉片點云噪聲被排除;而提高距離閾值到120 mm后,葉片與葉柄便可以構成一個整體。
在當前點云數據集中,種子點Pi可隨機選取,由于聚類針對的是去除主干后的點云數據,各個葉片已有一定距離間隔,因此,只需要選擇合適的距離閾值和迭代次數,聚類后的葉片點集數量均保持不變。
3.2 棉花葉片對應關系建立
利用2.3節介紹的方法分別實現不同生育期作物點云數據及葉片間的一一對應關系。圖4是1號棉花第46天和第49天葉片中心三維歐式距離構建的矩陣。經過使用匈牙利算法計算后得到最佳匹配,陰影框內數值表示最佳匹配時對應葉片中心的三維歐式距離,棉花植株T_(i+1)時期9號葉片在T_i時期無葉片與其對應,為新長出的葉片。
多時序的棉花植株間各葉片的對應關系建立結果如圖5所示,不同時序點云中的同一葉片采用同一顏色渲染。可以注意到,頂部的新生葉片未能在前期數據中找到對應葉片,而植株底部的紅色葉片隨著植株的長大成為老葉脫落。
3.3 表型參數計算及動態變化
3.3.1 株高和體積
通過在提取主干過程中得到的作物主干部分來測量棉花作物的株高。分別獲取主干的最低點和最高點的三維坐標,兩者之間的歐氏距離即為植株高度。將通過點云計算的作物表型參數與人工使用卷尺測量的表型參數進行對比,從而評價通過作物點云數據提取作物表型參數的準確性。精度評估通過絕對誤差、相對誤差及線性回歸決定系數R2(Coefficient of Determination)來實現。其中絕對誤差可描述為作物點云數據計算的表型參數值與人工測量參數值的差值;相對誤差可描述為作物表型參數的絕對誤差與人工測量的參數值之比乘以100%,如公式(2)所示。
E_R=|Y-L|/L×100% (2)其中,E_R為相對誤差;Y為點云數據計算的表型參數值;L為人工測量參數值。
R2用于度量作物點云數據計算的表型參數與人工測量參數的相關性,公式(3)所示。
R^2=(∑_(i=1)^n?〖(Y_i 〖-?Y)〗^2-∑_(i=1)^n?〖(Y_i-Y ?_i )^2 〗〗)/(∑_(i=1)^n?〖(Y_i 〖-?Y)〗^2 〗) (3)其中,n為作物植株葉片數;Yi為人工測量的表型參數值;?Y為人工測量表型參數值的平均值;Y ?_i為點云計算表型參數值。
包含給定三維空間中點集的最小凸多面體被定義為這些點的凸包,凸包的生成和三維空間中點云的分布緊密相關。因此,最小包圍凸包常用來表示三維空間中作物植株的體積。棉花體積計算過程:輸入原始植株點云數據,通過MATLAB獲得棉花植株的凸包,即可自動計算凸包的體積,如圖6(a)所示。
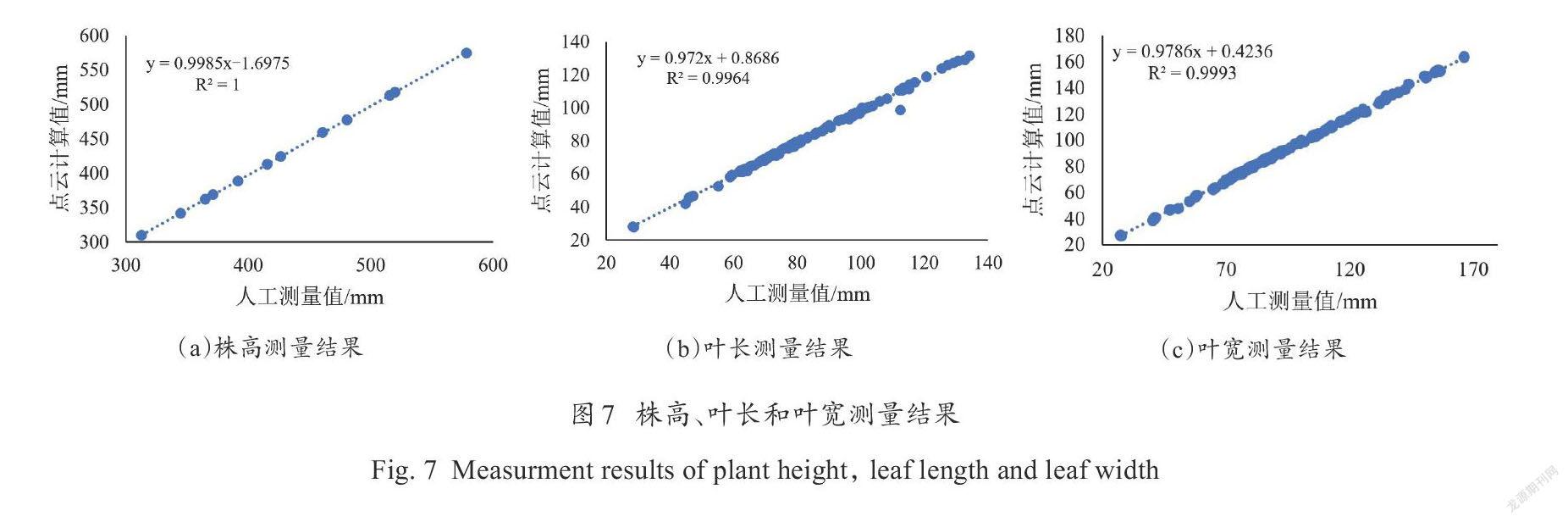
圖7為對株高、葉長、葉寬等參數進行精度評定,通過對12株棉花所有計算出的株高(如表2所示),葉長和葉寬與人工量測數據進行比較(圖7)。可以看出,這三者與人工測量值具備了高度的相關性,株高、葉長和葉寬的決定系數分別為1.0000、0.9964、0.9993,均趨近于1.0。該結果表明通過三維激光掃描技術可獲取作物的高精度表型參數。主要由于:1)所使用的近景激光掃描系統掃描精度較高,可達0.03 mm;2)與基于影像的二維表型量測方法相比,三維激光點云數據多了一個維度的信息,因而在表型參數測量上更具優勢。
3.3.2 葉長、葉寬和葉面積估算
棉花葉片存在一定弧度的彎曲。因此,直接利用葉片兩端點的歐式距離值來表描述葉長并不適用。本研究采用一種逐漸逼近的方法來測量葉片長度[26]。假設葉片兩端點分別為A(X_A,Y_A,Z_A)和B(X_B,Y_B,Z_B),AB兩端點間的距離為L_AB,取AB中點C(圖6(b)),分別計算點A、C和點B、C之間的距離l_AC和l_BC。令l_AB=l_AC + l_BC,設定l_AB和L_AB間長度偏差的閾值d,比較l_AB和L_AB;若|l_AB-L_AB | < d,令:
lAB = L_AB + d?? (4)
則l_AB為葉長;否則分別對AC和BC繼續處理。用B替換AC段中端點標號C,AC段更新為AB段。該過程一直迭代執行,直到距離差值滿足閾值條件為止。葉寬也采用逐漸逼近法完成計算。
對棉花植株點云數據進行葉片器官分割得到每個獨立的葉片點云數據,使用Geomagic Studio 2013軟件對葉片點云數據進行封裝,獲得葉片的網格數據,之后計算各網格的面積之和,即為葉片的面積。在網格化過程中,需要對網格數量和距離值進行約束,如果網格數量越多,則目標表面越精細,計算出的面積值與真實值越接近。本研究設置的網格數量為2,500,000個,距離值為0,因此,能夠保證得到足夠多的網格數量,也即較高精度的面積估值。封裝后的葉片如圖6(c)和6(d)所示。
3.3.3 表型參數動態變化估計
棉花植株點云數據獲取過程中進行了多次的作物點云數據采集,因此可以將同一作物不同生育期提取的表型參數按照先后順序進行對比分析,從而實現作物相鄰時期表型參數變化過程,并將其結果可視化,為育種和遺傳學相關研究提供數據基礎。
3株棉花的4個時間序列的株高、體積的變化趨勢如圖8(a)和8(b)所示,葉長、葉寬、葉面積的動態變換趨勢分別如圖8(c)~8(k)所示。從圖中可以看出,該觀測期內,株高的變化趨勢較為平緩,呈穩步上升態勢。體積的增長趨勢和株高較相似,主要原因在于體積的計算和株高相關性較強。老葉面積變化不大,新葉面積增加趨勢明顯,如1號棉花中頂部的三片葉子(圖5(a))中分別渲染為紫色、深藍色、藍綠色。葉面積和葉長的變化趨勢較為一致,同樣因為葉面積與葉長有較強的相關性。
本研究中,從1株棉花植株的點云數據采集開始到輸出各表型參數結果值,累計耗時約30 min,其中數據采集約15~20 min,主干提取和葉片聚類在30 s內完成,體積計算約11 s,單葉的葉面積估算約耗時30 s。此過程中,數據采集較為耗時,該過程與采集對象的復雜度有關,對象越復雜,則耗時越長。
4? 結論與討論
為挖掘三維激光掃描技術在作物表型組學中的應用,本研究利用激光掃描技術獲取了棉花的多時序三維激光點云數據,實現了三維空間內株高、葉長、葉寬、葉面積及體積等表型參數獲取和動態量化。主要取得的結果如下。
(1)利用RANSAC算法結合直線模型提取了3株棉花共12組點云的主干提取,并對剩余點云使用歐式聚類算法完成每一株棉花的葉片分割。完成了棉花植株的葉長、葉寬、葉面積、株高和體積等參數的計算,對比試驗結果表明,提取的株高、葉長、葉寬與人工測量值均具有高度的相關性,決定系數R2趨近于1.0,克服了傳統測量方法主觀性強、具有一定的破壞性等缺點,為后續產量等研究奠定基礎。
(2)獲取同一株棉花作物不同生育期的多時序點云數據,研究了作物點云的粗配準和葉片器官的一一對應方法,實現了對應關系的可視化。并根據器官間的對應關系實現了棉花表型參數變化的動態量化過程。通過研究莖稈、葉片不同時期的動態變化,可為剖析棉花個體的器官生長發育過程提供精確的信息,也可為構建作物生長的動態模型提供可靠的數據基礎。
本研究的不足之處在于,由于側重于方法研究,采集數據時,暫未覆蓋棉花植株的完整生育期,無法開展棉花植株的全生育期的各表型參數的獲取和動態變化研究。因此,在后續工作中,將主要利用全生育期的棉花點云數據,實現全生育期表型參數的動態變化可視化和深度數據挖掘。同時,本研究方法也可推廣至包含直線型的作物對象如玉米、油菜等的表型研究中。
參考文獻:
[1]??? BRIGLIA N, WILLIAMS K, WU D, et al. Image-based assessment of drought response in grapevines[J]. Frontiers in Plant Science, 2020, 11: 595.
[2]??? ROSE J C, PAULUS S, KUHLMANN H. Accuracy analysis of a multi-view stereo approach for phenotyping of tomato plants at the organ level[J]. Sensors, 2015, 15: 9651-9665.
[3]??? GUAN H, LIU M, MA X, et al. Three-dimensional reconstruction of soybean canopies using multisource imaging for phenotyping analysis[J]. Remote Sensing, 2018, 10(8): ID 1206.
[4]??? WANG Y, WEN W, WU S, et al. Maize plant phenotyping: Comparing 3D laser scanning, multi-view stereo reconstruction, and 3D digitizing estimates[J]. Remote Sensing, 2018, 11(1): ID 63.
[5]??? MASUDA T. 3D shape reconstruction of plant roots in a cylindrical tank from multiview images[C]// 2019 IEEE/CVF International Conference on Computer Vision Workshop (ICCVW). Piscataway, New York, USA: IEEE, 2019: 2149-2157.
[6]??? NGUYEN T, SLAUGHTER D, MAX N, et al. Structured light-based 3D reconstruction system for plants[J]. Sensors, 2015, 15(8). 18587-18612.
[7]??? NI X, LI C, JIANG H, et al. Three-dimensional photogrammetry with deep learning instance segmentation to extract berry fruit harvestability traits[J]. ISPRS Journal of Photogrammetry and Remote Sensing, 2021, 171: 297-309.
[8]??? JIANG Y, LI C, TAKEDA F, et al. 3D point cloud data to quantitatively characterize size and shape of shrub crops[J]. Horticulture Research, 2019, 6(1): 43-59.
[9]??? JIN S, SU Y, WU F, et al. Stem-leaf segmentation and phenotypic trait extraction of individual maize using terrestrial LiDAR data[J]. IEEE Transactions on Geoscience and Remote Sensing, 2018, 57(3): 1336-1346.
[10]? SUN S, LI C, CHEE P W, et al. Three-dimensional photogrammetric mapping of cotton bolls in situ based on point cloud segmentation and clustering[J]. Journal of Photogrammetry and Remote Sensing, 2020, 160: 195-207.
[11]? 劉守陽, 金時超, 郭慶華, 等. 基于數字化植物表型平臺(D3P)的田間小麥冠層光截獲算法開發[J]. 智慧農業(中英文), 2020, 2(1): 87-98.LIU S, JIN S, GUO Q, et al. An algorithm for estimating field wheat canopy light interception based on digital plant phenotyping platform[J]. Smart Agriculture, 2020, 2(1): 87-98.
[12]? 馬福峰, 耿楠, 張志毅. 基于鄰域幾何特征約束的植株三維形態配準方法研究[J]. 計算機應用與軟件, 2016, 33(9): 184-189.MA F, GENG N, ZHANG Z. On 3D plant morphology registration method based on geometrical feature constraint of neighborhood[J]. Computer Applications and Software, 2016, 33(9): 184-189.
[13]? CHAUDHURY A B M, BARRON J L. Junction-based correspondence estimation of plant point cloud data using subgraph matching[J]. IEEE Geoscience & Remote Sensing Letters, 2016, 13(8): 1119-1123.
[14]? YANG L, ZHAI R, YANG X, et al. Segmentation of plant organs point clouds through super voxel-based region growing methodology[J]. Computer Engineering and Applications, 2019, 55(16): 197-203..
[15]? LI D, CAO Y, TANG X, et al. Leaf segmentation on dense plant point clouds with facet region growing[J]. Sensors, 2018, 18(11): ID 3625.
[16]? GELARD W, DEVY M, HERBULOT A, et al. Model-based segmentation of 3D point clouds for phenotyping sunflower plants[C]// 12th International Joint Conference on Computer Vision, Imaging and Computer Graphics Theory and Applications Porto. Portugal, 2017: ID hal-02733980.
[17]? 梁秀英, 周風燃, 陳歡, 等. 基于運動恢復結構的玉米植株三維重建與性狀提取[J]. 農業機械學報, 2020, 51(6): 209-219.LIANG X, ZHOU F, CHEN H, et al. Three-dimensional maize plants reconstruction and traits extraction based on structure from motion[J].Transactions of the CSAM, 2020, 51(6): 209-219.
[18]? WAHABZADA M, PAULUS S, KERSTING K, et al. Automated interpretation of 3D laserscanned point clouds for plant organ segmentation[J]. BMC Bioinformatics, 2015, 16(1): ID 248.
[19]? 喻垚慎, 云挺, 楊緒兵. 基于激光點云數據的植物器官多維特征分割方法[J]. 數據采集與處理, 2015, 30(5): 1054-1061.YU Y, YUN T, YANG X. Multi dimension feature segmentation method of foliage organs based on laser point cloud data[J]. 2015, 30(5): 1054-1061.
[20]? SU Y, WU F, AO Z, et al. Evaluating maize phenotype dynamics under drought stress using terrestrial LiDAR[J]. Plant Methods, 2019, 15(1): ID 11.
[21]? AYAN CHAUDHURY, CHRISTOPHER WARD, ALI TALASAZ. Machine vision system for 3D plant phenotyping[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2019, 16(6): 2009-2022.
[22]? SUN S, LI C, PATERSON A H, et al. In-field high throughput phenotyping and cotton plant growth analysis using LiDAR[J]. Frontiers in Plant Science, 2018, 9: 1-17.
[23]? AN N, WELCH S, MARKELZ R, et al. Quantifying time-series of leaf morphology using 2D and 3D photogrammetry methods for high-throughput plant phenotyping[J]. Computers & Electronics in Agriculture, 2017, 135: 222-232.
[24]? 陽旭. 基于多時相點云數據的作物表型參數獲取及動態量化方法研究[D]. 湖北: 華中農業大學, 2019.YANG X. Research on crop phenotypic parameters acquisition and dynamic quantification method based on multi-temporal point cloud data[D]. Hubei: Huazhong Agricultural University, 2019.
[25]? RUSU R B, HOLZBACH A, BLODOW N, et al. Fast geometric point labeling using conditional random fields[C]// 2009 IEEE/RSJ International Conference on Intelligent Robots and Systems. Piscataway, New York, USA: IEEE, 2009: 7-12.
[26]? 史蒲娟, 翟瑞芳, 常婷婷, 等. 基于單目視覺和激光掃描技術的油菜植株模型重建及株型參數測量[J]. 華中農業大學學報, 2017, 36(3): 63-68.
Cotton Phenotypic Trait Extraction Using Multi-Temporal Laser Point Clouds
YANG Xu1,2, HU Songtao1, WANG Yinghua4, YANG Wanneng3,4, ZHAI Ruifang1*
(1.College of Informatics, Huazhong Agricultural University, Wuhan 430070, China; 2.Shenzhen Fortune Trend Technology Co., Ltd., Wuhan 430070, China; 3.National Key Laboratory of Crop Genetic Improvement, Huazhong Agricultural University, Wuhan 430070, China; 4.College of Plant Science & Technology, Huazhong Agricultural University, Wuhan 430070, China)
Abstract: To cope with the challenges posed by the rapid growth of world population and global environmental changes, scholars should employ genetic and phenotypic analyses to breed crop varieties with improved responses to limited resource environments and soil conditions to increase crop yield and quality. Therefore, the efficient, accurate, and non-destructive measurement of crop phenotypic traits, and the dynamic quantification of phenotypic traits are urgently needed for crop phenotypic research, and breeding as well as for modern agricultural development. In this study, cotton plants were taken as research objects, and the multi-temporal point cloud data of cotton plants were collected by using three-dimensional laser scanning technology. The multi-temporal point clouds of three cotton plants at four time points were collected. First, RANSAC algorithm was implemented for main stem extraction on the original point cloud data of cotton plants, then region growing based clustering was carried out for leaf segmentation. Plant height was estimated by calculating the end points of the segmented main stem. Leaf length and width measurements were conducted on the segmented leaf parts. In addition, the volume was also estimated through the convex hull of the original point cloud of plant cotton. Then, multi-temporal point clouds of plants were registered, and organ correspondence was constructed with the Hungarian method. Finally, dynamic quantification of phenotypic traits including plant volume, plant height, leaf length, leaf width, and leaf area were calculated and analyzed. The overall performance of the approaches achieved a matching rate through a series of experiments, and the traits extracted by using of point cloud showed high correlation with the manually measured ones. The relative error between plant height and manual measurement results did not exceed 1.0%. The estimated leaf length and width on point clouds were highly correlated with the manually measured ones, and the coefficient of determination was nearly 1.0. The proposed 3D phenotyping methodology can be introduced and used to other crops for phenotyping.
Key words: cotton phenotypic traits; LiDAR; stem extraction; leaf segmentation; point cloud registration; 3D phenotyping

