基于SNP標記的荷包紅鯉與興國紅鯉鑒別研究
王彥云 王濟世 劉曉夏 趙璐瑤 趙汝婷 楊曙明
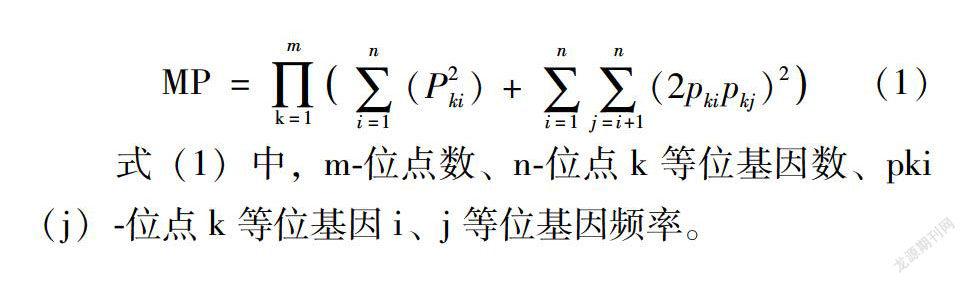


摘 要:興國紅鯉和荷包紅鯉是寶貴的紅鯉品種,作為優良雜交親本,廣泛應用于品種改良和新品種的選育。由于形態相近、差異較小,在選擇雜交親本的過程中,極易發生兩種紅鯉的混淆,影響新品種的養殖經濟效益。通過閱讀參考文獻,使用數據庫位點挑選和性狀基因位點挑選,獲得了8個SNP候選標記,理論上使用7個SNP標記,即可實現兩種紅鯉的鑒別,且鑒別錯誤率為6.30E-05,這對以兩種紅鯉為雜交親本的新品種選育及養殖效益的提升意義重大,也有利于兩種紅鯉的種質資源保護。
關鍵詞:興國紅鯉;荷包紅鯉;SNP標記;鑒別
紅鯉是極為重要的魚類育種材料之一,興國紅鯉和荷包紅鯉是中國江西省兩個傳統的地方鯉品種,其既可觀賞、又可食用,具有極高的經濟價值[1]。由于具有生長速度快、繁殖率高、抗逆性強和雜交親和力強等特點,兩種紅鯉被廣泛應用于品種改良和新品種的選育[2-3]。我國鯉魚養殖品種主要為德國鏡鯉選育系、荷包紅鯉抗寒系、豫選黃河鯉新品系等“三大系列”,其中,荷包紅鯉抗寒系是以荷包紅鯉為雜交親本,通過系統選育得到的一系列生長性狀更優、抗逆性更強鯉魚新品種。此外,興國紅鯉在育種方法亦有諸多應用,例如,豐鯉、芙蓉鯉、興德鯉等均為以興國紅鯉為親本雜交選育而得的優良鯉魚品種。到目前,通過這兩種紅鯉魚和其他當地普通鯉魚品種雜交,已經繁育出了10多個優良的雜交品種,對促進我國水產養殖生產的發展起到了很大的作用。兩種紅鯉形態特征較為相似,這給選育工作者選擇雜交親本造成了困難;同時,由于荷包紅鯉具有更高的經濟價值,也會有興國紅鯉假冒荷包紅鯉之名銷售的情況。因此,對兩種紅鯉的鑒定十分必要,這對在鯉魚新品種資源的開發和鯉魚養殖產業經濟效益的提升具有重大的意義[2,4]。興國紅鯉與荷包紅鯉均產自江西省長江中下游水域,從形態學角度來看,荷包紅鯉體粗短,體色呈純紅或橘橙色,體長/體高為2.0~2.3,而興國紅鯉呈紡錘形,體紅色,體長/體高為2.55~3.55[5];從核型學特征來看,二者染色體組型一樣[6]。兩種紅鯉形態上雖有差別,但這種差別屬于一個種內不同品種或生態種群間的細微差異[7]。由于地理因素和人為因素的影響,兩個品種極易混淆,這既不利于雜交親本的選擇,也不利于兩種紅鯉種質資源的保護。單核苷酸多態性(SNP)是基因組水平上單個堿基的變異所引起的DNA序列的多態性,屬于第三代遺傳標記技術。由于SNP標記具有存在廣泛、富有代表性、遺傳穩定性以及易實現分析的自動化等特點,被廣泛應用于種質鑒定、產品溯源等方面[8-11]。
1 材料與方法
1.1 DNA樣品的準備
實驗魚分別取自國家級婺源荷包紅鯉良種場和興國縣,兩個紅鯉品種各50條,各樣品取自不同池塘,相互之間不存在親屬關系。所有樣品均通過國標方法進行鑒定。所有樣本都用于SNP標記的挑選。DNA樣本的提取使用組織DNA提取試劑盒,提取后的樣本使用紫外分光光度計(NanoDrop 2000c)測定濃度和純度,DNA樣本的完整性采用電泳的方法考察。符合標準的樣品于-20 ℃儲存備用。
1.2 多態性位點驗證
位點的挑選基于之前研究獲得的大量鯉多態性位點,所選位點盡可能分布在鯉魚50條染色體鯉魚性狀基因上[12-17],且位于基因非編碼區[18-20]。挑選后的位點使用二代測序技術進行基因分型,二代測序由上海翼合應用生物技術有限公司提供支持。
1.3 統計分析
各位點在樣本群體中的等位基因頻率、基因型頻率通過直接計數方式獲得[21]。偶合概率(MP)計算公式為式(1)[22]:
MP=∏mk=1∑ni=1(P2ki)+∑ni=1∑nj=i+1(2pkipkj)2(1)
式(1)中,m-位點數、n-位點k等位基因數、pki(j)-位點k等位基因i、j等位基因頻率。
2 結果與分析
2.1 多態性位點驗證
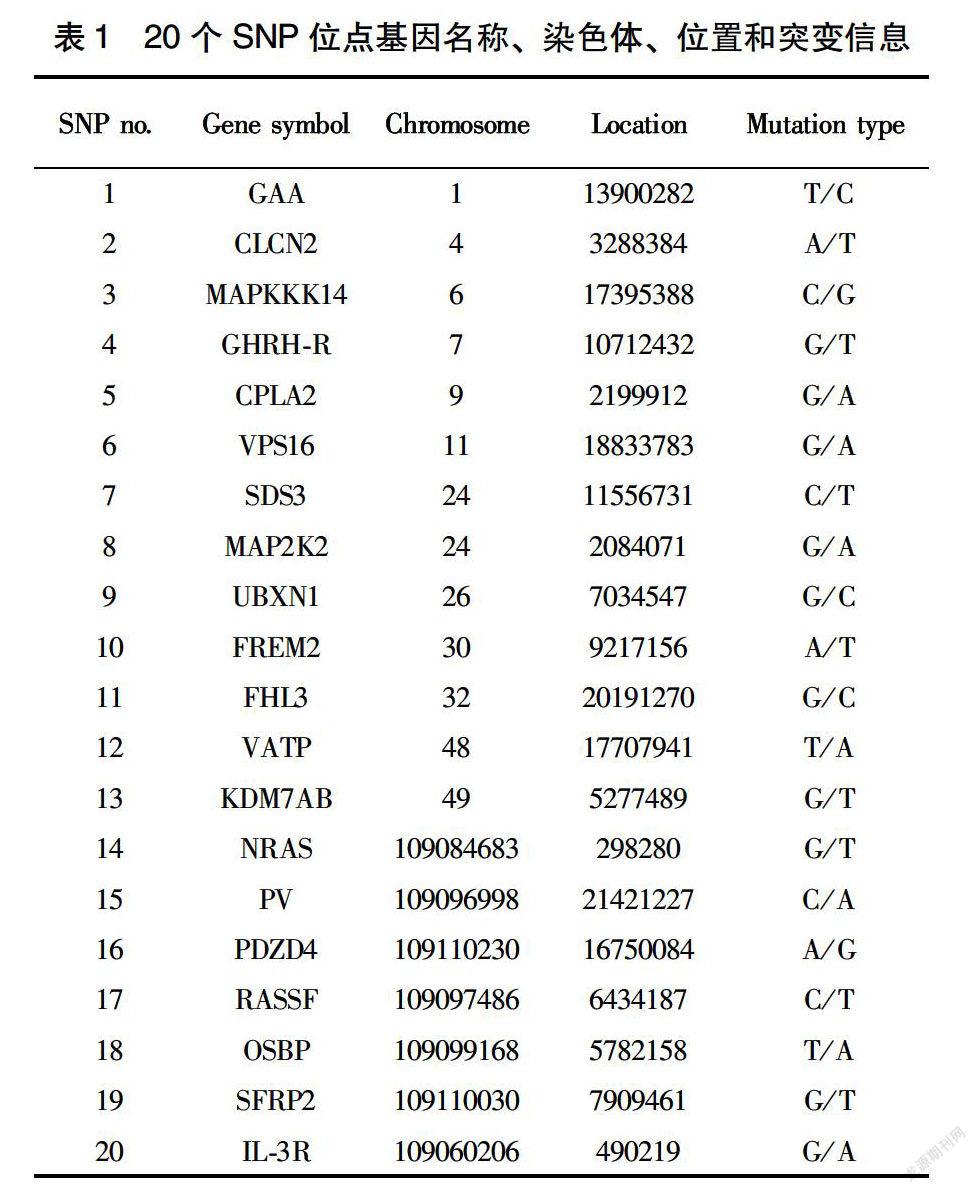
通過大量位點的挑選,共得多態性位點20個,位點基因名稱、染色體、位置和突變信息如表1。對兩個品種紅鯉共計100個樣本,測定上面20個多態性位點基因型,分型后對20個多態性位點在不同紅鯉品種中的基因型以及等位基因頻率進行分析,發現有一部分位點在兩個紅鯉品種間存在較大多態性差異,這些位點在一個紅鯉品種中兼具三種基因型,而在另一紅鯉品種中只有一種純合子基因型。我們共獲得這樣的候選標記11個,用于2個紅鯉品種的鑒別。由表2可知,11個候選標記均與鯉魚頭部和體型等性狀基因相關,其中,與頭部、體高性狀基因相關的標記就有一半,如SNP14、SNP15、SNP16與鯉魚頭長性狀基因有關,SNP17、SNP18與鯉魚眼徑、體高和體厚等性狀基因相關,這與明俊超等[23]對6 個不同鯉群體的形態差異分析結果一致,荷包紅鯉和興國紅鯉群體在形態上存在一定差異和分化,主要體現在體型、頭部和眼部特征上。
2.2 兩種紅鯉鑒定效果評估
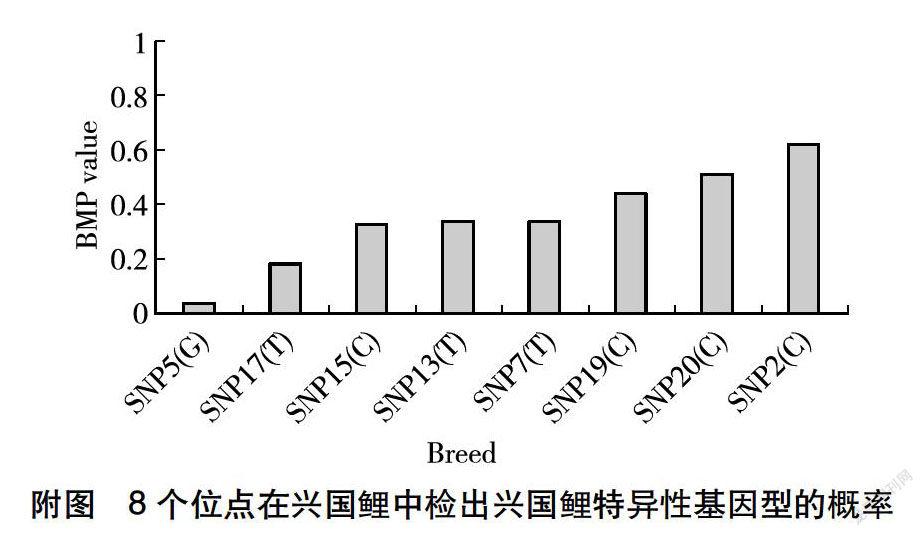
由表3可知,所有11個候選多態性位點,除SNP14、SNP16和SNP18外,其余8個位點在興國紅鯉中均表現出較高的多態性,兼具兩種純合子和一種雜合子,而在荷包紅鯉中保守,只有一種純合子。這8個標記均能用于兩種紅鯉的鑒定,若檢測這些位點上黃河鯉特有的等位基因,則單個標記使用時的品種耦合概率(BMP)如附圖所示。所有8個標記單獨使用的品種耦合概率(BMP)在0.040~0.735不等,以耦合概率最小的標記SNP5為例,荷包紅鯉群體只有一種純合子“AA”,而興國紅鯉群體兼具兩種純合子“AA”、“GG”以及雜合子“AG”,即等位基因“G”為興國鯉所特有。若未知樣品于標記SNP5上檢測出“G”等位基因,則判定樣品為興國紅鯉,耦合概率為0.04,即單獨使用SNP5標記對兩種紅鯉進行個體鑒定,每鑒定100個興國鯉個體,僅有4個被誤判為荷包紅鯉。
在實際應用中,僅使用單個標記遠不能滿足品種鑒定時群體數量和準確度的要求,為了達到更好的鑒定效果,通常需要增加標記的數目。本研究通過鑒定只在興國紅鯉中出現的等位基因來對兩個紅鯉品種進行鑒定,通過多個標記共同使用,降低品種耦合概率。按照區分能力大小對標記進行排序,并計算不同數量標記組合時品種鑒定耦合概率。從表3我們可以看到,隨著標記數目的增加,兩個品種紅鯉鑒定的耦合概率值在不斷下降,當使用區別能力最好的6個SNP標記SNP5、SNP17、SNP15、SNP13、SNP7、SNP19時,MP值僅為萬分之1.23,兩種紅鯉鑒定錯誤的概率不超過萬分之1.23;當使用全部7個標記時,MP值僅為10萬分之6.30,即兩種紅鯉鑒定錯誤的概率不超過10萬分之6.30。
3 結論
通過研究我們共獲得了8個SNP候選標記,結合兩種紅鯉在我國的養殖情況和分布范圍,使用5~7個SNP標記即可實現兩種紅鯉不同群體數量的鑒定。據中國漁業統計年鑒,2019年江西省鯉魚總產量近15萬t,只需使用7個SNP標記就能基本滿足荷包紅鯉和興國紅鯉的鑒定需求。普通小型實驗室使用等位特異性PCR結合毛細管電泳的方法[23-24],即可實現對興國紅鯉和荷包紅鯉的快速鑒別。本研究為兩種紅鯉快速、準確鑒別提供了新思路、新方法,有利于提高選育工作的效率和可靠性。◇
參考文獻
[1]Zhou J,Wu Q,Wang Z,et al.Genetic variation analysis within and among six varieties of common carp(Cyprinus carpio L.)in China using microsatellite markers[J].Russian Journal of Genetics,2004,40(10):1389-1393.
[2]Wang C H,et al.Developmental quantitative genetic analysis of body weight and morphological traits in red common carp,Cyprinus carpio L[J].Aquaculture,2006,251(2):219-230.
[3]樓允東.荷包紅鯉史話[J].科學養魚,2009(4):72-73.
[4]Li S F,Wang C H.Genetic diversity and selective breeding of red common carps in China[J].Naga,2001,24(3):56-61.
[5]樓允東.魚類育種學[M].北京:中國農業出版社,1999.
[6]王蕊芳,施立明,賀維順.幾種鯉魚染色體核仁組織者的銀染觀察[J].動物學研究,1985,6(4):391-398.
[7]張平.不同鯉群體的形態差異比較及RAPD擴增分析[D].南京:南京農業大學,2009.
[8]黃樹文,張哲,陳贊謀,等.廣東省現有5個地方豬種基于SNP芯片的遺傳多樣性分析[J].中國畜牧雜志,2018,54(6):33-37.
[9]魏中艷,等.應用SNP精準鑒定大豆種質及構建可掃描身份證[J].作物學報,2018,44(3):315-323.
[10]趙杰,游新勇,徐貞貞,等.SNP檢測方法在動物研究中的應用[J].農業工程學報,2018,34(4):299-305.
[11]Kawaguchi F,Wkitamura Y,Nakajima R,et al. Application of DNA markers for discrimination between Japanese and Australian Wagyu beef[J].Animal Science Journal,2017.
[12]Xinhua Wang,Beide Fu,Xiaomu Yu,et al.Fine mapping of growth-related quantitative trait loci in Yellow River carp(Cyprinus carpio haematoperus)[J].Aquaculture,2018(484):277-285.
[13]Deyana G,Stefka S.Polymorphisms of the candidate genes associated with the growth traits in common carp(Cyprinus carpio L)[J].Agricultural Sciences,2018,10(23):27-32.
[14]Song F,et al.A novel igf3 gene in common carp(Cyprinus carpio):evidence for its role in regulating gonadal development[J].PLoS One,2016,11(12).
[15]Xiangjiang Liu,Hongwei Liang,Yongjun Liang,et al. Significant associations of polymorphisms in the growth hormone gene with growth traits in common carp(Cyprinus carpio)[J].Meta Gene,2017(14):38-41.
[16]Li H,et al.Genetic diversity and association analysis of two duplicated ODC genes polymorphisms with weight gain in Cyprinus carpio L[J].Aquaculture,2016,(459):14-18.
[17]Feng,Xiu,Liu,et al.Molecular characterization and expression of three preprosomatostatin genes and their association with growth in common carp(Cyprinus carpio)[J].Comparative Biochemistry and Physiology Part B:Biochemistry & Molecular Biology,2015(182B):37-46.
[18]Kim E-S,Wkirkpatrick B W.Linkage disequilibrium in the North American Holstein population[J].Animal Genetics,2009,40(3):279-288.
[19]Zhao J,Chen A,You X,et al.A panel of SNP markers for meat traceability of Halal beef in the Chinese market[J].Food Control,2017(87):94-99.
[20]Kitts A,Sherry S.The single nucleotide polymorphism database(dbSNP)of nucleotide sequence variation[J].National Center for Biotechnology Information,2003(5):1-30.
[21]L Orrù,Catillo G,Napolitano F,et al.Characterization of a SNPs panel for meat traceability in six cattle breeds[J].Food Control,2009,20(9):856-860.
[22]Weir B S.Genetic Data Analysis II[J].Trends in Genetics,1996,13(9):379.
[23]陳吉寶,等.等位基因特異PCR技術的研究與應用[J].植物遺傳資源學報,2005(4):469-473.
[24]顧琳,等.PCR檢測食品中金黃色葡萄球菌腸毒素基因[J].中國食物與營養,2015,21(2):17-19.
Identification of Purse Red Carp and Xingguo Red Carp Based on SNP Markers
WANG Yan-yun,WANG Ji-shi,LIU Xiao-xia,ZHAO Lu-yao,ZHAO Ru-ting,YANG Shu-ming
(Institute of Agricultural Quality Standards and Testing Technology,Chinese Academy of Agricultural Sciences/Key Laboratory of Quality and Safety of Agricultural Products,Ministry of Agriculture and Rural Affairs,Beijing 100081,China)
Abstract:Xingguo red carp(C.carpio var.xingguonensis)and Purse red carp(C.carpio var.wuyuanensis)are precious red carp varieties.As excellent hybrid parents,they are widely used in variety improvement and breeding of new varieties.Due to the small morphological difference,the confusion of two varieties of red carp is common to occur in the selection of hybrid parents.By reading a large number of references and using database site selection and trait loci selection,we obtained 8 candidate SNP markers.Using 7 SNP markers,the identification error rate of two red carp in China was 6.30E-05,which is of great significance to the conservation and breeding of the germplasm resources of the two breeds.
Keywords:Xingguo red carp;Purse red carp;SNP marker;identification

