轉(zhuǎn)錄組測(cè)序分析氯化鈉對(duì)普魯蘭生物合成的影響
張高川,何超永,王崇龍,王大慧*,衛(wèi)功元*
(蘇州大學(xué)基礎(chǔ)醫(yī)學(xué)與生物科學(xué)學(xué)院,江蘇 蘇州 215123)
普魯蘭是一種由出芽短梗霉(Aureobasidium pullulans)合成的水溶性直鏈高分子物質(zhì)[1]。作為一種微生物胞外多糖,普魯蘭本身安全無毒,同時(shí)具有良好的可塑性、成膜性和穩(wěn)定性,以及耐熱、耐酸堿、耐鹽等特性,因而可以廣泛地應(yīng)用于食品加工、臨床醫(yī)藥、環(huán)境保護(hù)和輕工化工等諸多領(lǐng)域[2-4]。美國食品藥品監(jiān)督管理局已經(jīng)將普魯蘭認(rèn)證為GRAS(Generally Recognized As Safe)微生物多糖,我國也于2006年將普魯蘭列為新增的4 種食品添加劑之一[5]。此后,普魯蘭在市場(chǎng)上的需求量日益增長。
在普魯蘭的發(fā)酵生產(chǎn)中,普魯蘭產(chǎn)量和分子質(zhì)量是兩個(gè)重要的過程參數(shù),前者決定普魯蘭生產(chǎn)的得率和效率,而后者關(guān)系到普魯蘭的應(yīng)用范圍[6]。影響普魯蘭產(chǎn)量和分子質(zhì)量的因素有很多,既包括培養(yǎng)基中各種營養(yǎng)成分及其配比,又涉及生產(chǎn)菌株培養(yǎng)的環(huán)境條件[7-8]。其中,培養(yǎng)基中無機(jī)鹽及其含量在普魯蘭的生產(chǎn)中往往具有重要的作用。Badr-Eldin等[9]發(fā)現(xiàn)發(fā)酵培養(yǎng)基中磷酸鹽水平偏低不利于菌體生長,偏高則抑制普魯蘭合成。Reeslev等[10]發(fā)現(xiàn)當(dāng)培養(yǎng)基中Zn2+濃度高于0.45 μmol/L時(shí),出芽短梗霉的形態(tài)即從酵母狀細(xì)胞向菌絲狀轉(zhuǎn)變,普魯蘭的合成隨之受到不利影響[6]。鞠寶等[11]分析了二價(jià)陽離子在普魯蘭和黑色素形成中的作用,結(jié)果表明合適的金屬離子種類和濃度抑制了黑色素的形成,促進(jìn)了普魯蘭的合成。Gao Wa等[12]在單因素試驗(yàn)的基礎(chǔ)上,采用正交試驗(yàn)優(yōu)化了培養(yǎng)基中無機(jī)鹽含量,提高了普魯蘭產(chǎn)量。與此同時(shí),還發(fā)現(xiàn)影響出芽短梗霉細(xì)胞生長的最顯著因素是磷酸氫二鉀,而氯化鈉(NaCl)則是影響普魯蘭合成的最顯著因素[13]。以上結(jié)果均是基于發(fā)酵過程優(yōu)化而得到的,這些研究并沒有對(duì)無機(jī)鹽影響普魯蘭生物合成的內(nèi)在機(jī)制進(jìn)行深入分析。
在前期優(yōu)化普魯蘭發(fā)酵培養(yǎng)基的研究中,發(fā)現(xiàn)培養(yǎng)基中NaCl的存在有利于提高普魯蘭產(chǎn)量,但卻顯著降低了普魯蘭的分子質(zhì)量[6]。通過對(duì)普魯蘭合成關(guān)鍵酶(α-磷酸葡萄糖異構(gòu)酶、尿苷二磷酸葡萄糖焦磷酸化酶、葡萄糖基轉(zhuǎn)移酶)的活性、物質(zhì)和能量代謝進(jìn)行測(cè)定,發(fā)現(xiàn)NaCl同時(shí)提高了與普魯蘭生物合成和降解相關(guān)酶的活性,增加了能量物質(zhì)ATP的供給,結(jié)果部分揭示了NaCl在提高普魯蘭產(chǎn)量的同時(shí)卻降低了普魯蘭分子質(zhì)量的生理機(jī)制[14]。盡管如此,NaCl如何在分子水平影響出芽短梗霉胞內(nèi)相關(guān)基因的表達(dá),并體現(xiàn)在普魯蘭的產(chǎn)量和分子質(zhì)量上,這些問題至今仍然不得而知。
轉(zhuǎn)錄組測(cè)序(RNA-Seq)技術(shù)是通過對(duì)細(xì)胞轉(zhuǎn)錄產(chǎn)物進(jìn)行測(cè)序,統(tǒng)計(jì)測(cè)得的每條序列,獲得每個(gè)特定轉(zhuǎn)錄本的表達(dá)量,可更加精確地評(píng)估細(xì)胞表型,加深對(duì)細(xì)胞代謝的理解,也有助于對(duì)目標(biāo)基因進(jìn)行改良[15]。近年來,RNA-Seq因檢測(cè)動(dòng)態(tài)范圍寬、數(shù)據(jù)重復(fù)性好等優(yōu)勢(shì),逐漸成為研究基因表達(dá)差異、新基因挖掘與功能注釋的重要手段[16]。RNA-Seq技術(shù)和生物信息學(xué)分析手段已經(jīng)廣泛運(yùn)用于微生物過程分析中,為深入理解微生物現(xiàn)象中蘊(yùn)含的分子機(jī)制提供了可信的證據(jù)[17-20]。為此,本實(shí)驗(yàn)研究NaCl對(duì)出芽短梗霉細(xì)胞轉(zhuǎn)錄組的影響,采用生物信息學(xué)方法篩選與分析差異表達(dá)基因(differentially expressed genes,DEGs),并對(duì)這些DEGs進(jìn)行注釋與富集分析。
1 材料與方法
1.1 材料與試劑
出芽短梗霉CCTCC M 2012259,由蘇州大學(xué)微生物生理與代謝調(diào)控研究室保藏。
種子培養(yǎng)基:馬鈴薯200 g/L,葡萄糖20 g/L,自然pH值;發(fā)酵培養(yǎng)基:葡萄糖50 g/L,酵母粉3 g/L,(NH4)2SO40.6 g/L,K2HPO45 g/L,MgSO4·7H2O 0.2 g/L,pH 3.8,NaCl質(zhì)量濃度根據(jù)具體實(shí)驗(yàn)設(shè)計(jì)而定。
所有試劑均為國產(chǎn)分析純,購自生工生物工程(上海)股份有限公司。
1.2 儀器與設(shè)備
Minifors實(shí)驗(yàn)室臺(tái)式5 L發(fā)酵罐 瑞士Infors公司;HZ-2010K恒溫?fù)u瓶柜 太倉市華利達(dá)實(shí)驗(yàn)儀器設(shè)備有限公司;J6-MI型冷凍離心機(jī) 美國Beckman公司;T6新世紀(jì)紫外-可見分光光度計(jì) 北京普析通用儀器有限責(zé)任公司;LDZX-50KBS型立式壓力蒸汽滅菌器 上海申安醫(yī)療器械廠;1836稀釋型烏式毛細(xì)管黏度計(jì) 浙江臺(tái)州市椒江玻璃儀器廠;2100生物分析儀 美國安捷倫科技公司;Qubit 2.0熒光分光光度計(jì) 美國Invitrogen公司。
1.3 方法
1.3.1 種子培養(yǎng)
將在-70 ℃超低溫冰箱中保藏的菌種(1 mL)接種至50 mL種子培養(yǎng)基中,于30 ℃、200 r/min搖床中培養(yǎng)24 h。
1.3.2 發(fā)酵培養(yǎng)
搖瓶發(fā)酵培養(yǎng):以10%(V/V)的接種量,將種子液接種至裝有50 mL發(fā)酵培養(yǎng)基的500 mL三角瓶中,在200 r/min搖床中培養(yǎng)72 h,溫度30 ℃。
分批發(fā)酵培養(yǎng):以10%(V/V)的接種量,將種子液接種至裝有3 L發(fā)酵培養(yǎng)基的5 L發(fā)酵罐中,溫度30 ℃,攪拌轉(zhuǎn)速400 r/min,通氣量3 L/min。pH值采用梅特勒電極在位監(jiān)測(cè),通過自動(dòng)流加3 mol/L H2SO4或3 mol/L NaOH溶液控制pH值為3.80±0.02。
1.3.3 指標(biāo)測(cè)定
細(xì)胞干質(zhì)量和普魯蘭產(chǎn)量的測(cè)定方法參見文獻(xiàn)[5];普魯蘭分子質(zhì)量的測(cè)定采用黏度法[6];葡萄糖質(zhì)量濃度的測(cè)定采用3,5-二硝基水楊酸法[21];分批發(fā)酵過程參數(shù)的計(jì)算方法參見文獻(xiàn)[8]。
1.3.4 出芽短梗霉RNA-Seq
1.3.4.1 RNA提取
將分批發(fā)酵培養(yǎng)至36 h的出芽短梗霉細(xì)胞8 000 r/min、4 ℃離心10 min,液氮猝滅2 min;利用TRIzol試劑盒提取總RNA。采用Agilent 2100生物分析儀、NanoDrop和1%瓊脂糖凝膠對(duì)總RNA進(jìn)行品質(zhì)和定量分析。選擇1 μg RNA完整值(RNA integrity number,RIN)大于7的總RNA用于后續(xù)文庫制備。
1.3.4.2 cDNA文庫構(gòu)建
根據(jù)NEBNext?UltraTMRNA文庫制備試劑盒的操作步驟進(jìn)行測(cè)序文庫制備。首先使用NEBNext Poly(A) mRNA Magnetic Isolation Module從總RNA中分離Poly(A) mRNA,然后利用NEBNext First Strand Synthesis Reaction Buffer和NEBNext Random Primers對(duì)mRNA進(jìn)行破碎(約300 bp)和引物配對(duì)。以此為模板,利用ProtoScript II逆轉(zhuǎn)錄酶合成第1條cDNA鏈,利用Second Strand Synthesis Enzyme Mix合成第2條cDNA鏈。之后用AxyPrep Mag PCR Clean-up純化雙鏈cDNA產(chǎn)物,使用Agilent 2100生物分析儀評(píng)估其品質(zhì),使用Qubit 2.0熒光分光光度計(jì)對(duì)其進(jìn)行定量。最后使用End Prep Enzyme Mix修復(fù)純化后的雙鏈cDNA產(chǎn)物,添加dA尾巴,并通過T-A連接在其末端添加接頭。
1.3.4.3 RNA-Seq數(shù)據(jù)分析
利用Illumina HiSeqTM2000對(duì)上述構(gòu)建的cDNA文庫進(jìn)行150 bp讀長的雙末端測(cè)序,獲得的RNA-Seq數(shù)據(jù)經(jīng)FASTQC(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)質(zhì)控分析后,使用Trimmomatic(v0.30)軟件刪除測(cè)序質(zhì)量偏低的末端序列片段[22],處理后的干凈讀段(clean reads)用于后續(xù)分析。將RNA-Seq數(shù)據(jù)上傳至NCBI數(shù)據(jù)庫,登記號(hào)為PRJNA350822(https://dataview.ncbi.nlm.nih.gov/object/PRJNA350822)。
雖然已經(jīng)對(duì)出芽短梗霉全基因組進(jìn)行測(cè)序[23],但缺少完善的注釋信息,所以RNA-Seq數(shù)據(jù)分析采用基于Trinity(v2.6.6)從頭組裝的無參分析流程[24]。利用Trinity軟件對(duì)RNA-Seq數(shù)據(jù)進(jìn)行從頭組裝,利用BLASTX(2.7.1+)程序[25],將Trinity組裝獲得的所有轉(zhuǎn)錄本與來自UniprotKB數(shù)據(jù)庫(https://www.uniprot.org/)的34 084 個(gè)真菌已知蛋白進(jìn)行比對(duì)(截止日期2019-05-14),鑒別這些組裝轉(zhuǎn)錄本可能編碼的蛋白(統(tǒng)計(jì)期望閾值為1×10-5)。
使用RSEM(v1.3.1)軟件的RSEM-prepare-reference程序,根據(jù)Trinity組裝結(jié)果創(chuàng)建參考轉(zhuǎn)錄組;使用RSEM-calculate-expression工具計(jì)算Trinity組裝轉(zhuǎn)錄本在每個(gè)樣本中的表達(dá)水平;利用RSEM-generate-datamatrix程序,將所有樣本的基因表達(dá)數(shù)據(jù)合并為一個(gè)數(shù)據(jù)矩陣[26],用于DEGs分析。
1.3.4.4 DEGs的鑒別
利用R語言包edgeR(v3.20.9)對(duì)上述基因表達(dá)水平數(shù)據(jù)矩陣進(jìn)行計(jì)算分析[27],鑒別因培養(yǎng)基中添加NaCl而產(chǎn)生的DEGs。將基因差異表達(dá)倍數(shù)(fold change,F(xiàn)C)閾值設(shè)為2 倍,統(tǒng)計(jì)顯著性閾值設(shè)為0.05,作為DEGs的篩選標(biāo)準(zhǔn)[28]。對(duì)篩選結(jié)果進(jìn)行可視化分析,繪制火山圖。利用R語言包pheatmap(v1.0)對(duì)DEGs進(jìn)行聚類和可視化分析,用來評(píng)估平行樣本的一致性。
1.3.4.5 DEGs的功能注釋與富集分析
利用UniProt Retrieve/ID mapping tool(http://www.uniprot.org/uploadlists)在線工具,從Uniprot數(shù)據(jù)庫中獲取所有DEGs對(duì)應(yīng)蛋白質(zhì)的基因本體論(Gene Ontology,GO)注釋信息和序列信息。利用WEGO(http://wego.genomics.org.cn/)對(duì)其進(jìn)行GO注釋的富集分析[29],使用R語言包ggplot2對(duì)富集結(jié)果進(jìn)行可視化。利用京都基因和基因組百科全書(Kyoto Encyclopedia of Genes and Genome,KEGG)數(shù)據(jù)庫的Blast-KOALA工具(https://www.kegg.jp/blastkoala),分別對(duì)上調(diào)和下調(diào)的DEGs進(jìn)行通路富集分析[30],并利用KEGG mapper工具對(duì)其中的通路模塊進(jìn)行深入挖掘。
1.4 數(shù)據(jù)統(tǒng)計(jì)
RNA-Seq設(shè)置2 個(gè)平行樣品,其他所有實(shí)驗(yàn)數(shù)據(jù)均來自于3 組平行樣品,應(yīng)用SPSS 17.0軟件對(duì)數(shù)據(jù)進(jìn)行t檢驗(yàn)分析,P<0.05,差異顯著。
2 結(jié)果與分析
2.1 NaCl對(duì)普魯蘭生物合成的影響
在搖瓶發(fā)酵條件下,考察發(fā)酵培養(yǎng)基中NaCl質(zhì)量濃度對(duì)出芽短梗霉細(xì)胞生長和普魯蘭合成的影響,結(jié)果見圖1。可以看出,與對(duì)照(0 g/L NaCl)相比,NaCl的添加提高了普魯蘭產(chǎn)量,但降低了細(xì)胞干質(zhì)量和普魯蘭分子質(zhì)量。當(dāng)NaCl質(zhì)量濃度為3 g/L時(shí),普魯蘭產(chǎn)量達(dá)到最大值23.17 g/L,比對(duì)照提高了22.9%;而細(xì)胞干質(zhì)量和普魯蘭分子質(zhì)量在此質(zhì)量濃度下均處于最低水平,分別比對(duì)照降低了12.7%和44.9%。以上結(jié)果表明,培養(yǎng)基中NaCl的存在有利于普魯蘭產(chǎn)量的提高,但不利于將普魯蘭分子質(zhì)量維持在較高的水平。
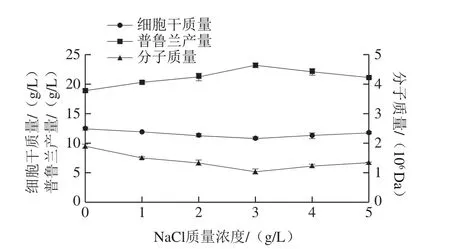
圖1 NaCl質(zhì)量濃度對(duì)普魯蘭生物合成的影響Fig.1 Effect of NaCl at different concentrations on pullulan biosynthesis
2.2 普魯蘭分批發(fā)酵過程參數(shù)比較
在5 L發(fā)酵罐中對(duì)出芽短梗霉CCTCC M 2012259進(jìn)行分批培養(yǎng),檢測(cè)了3 g/L NaCl和對(duì)照條件下的葡萄糖消耗、細(xì)胞生長和普魯蘭合成情況,結(jié)果見圖2和表1。通過對(duì)各個(gè)發(fā)酵過程參數(shù)進(jìn)行比較可以發(fā)現(xiàn),NaCl加快了葡萄糖的消耗速率,但卻降低了細(xì)胞生長的速率。因此,當(dāng)培養(yǎng)基中含有NaCl時(shí),細(xì)胞并沒有將多消耗的葡萄糖用于細(xì)胞生長,而是用于普魯蘭的合成,最終細(xì)胞干質(zhì)量比對(duì)照降低了16.5%,普魯蘭產(chǎn)量卻比對(duì)照提高17.6%。需要特別指出的是,在NaCl存在的情況下,普魯蘭最終分子質(zhì)量顯著下降55.8%。綜合表1中分批發(fā)酵其他過程參數(shù),可以得出結(jié)論,與對(duì)照相比,3 g/L NaCl顯著提高了普魯蘭產(chǎn)量,卻同時(shí)顯著降低了細(xì)胞干質(zhì)量和普魯蘭分子質(zhì)量。以下將采用RNA-Seq技術(shù)和生物信息學(xué)分析手段對(duì)NaCl影響普魯蘭生物合成的分子機(jī)制進(jìn)行解析。
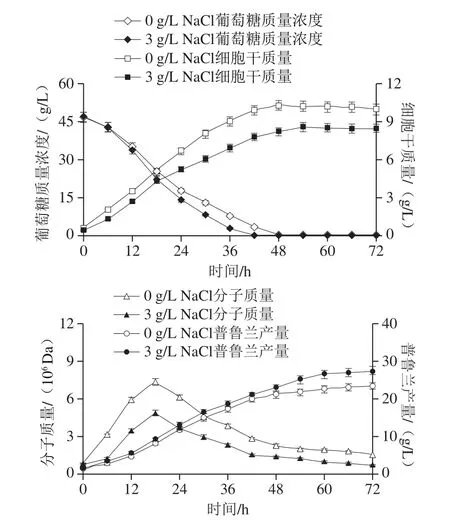
圖2 不同NaCl質(zhì)量濃度下的普魯蘭分批發(fā)酵過程Fig.2 Time-course curve of pullulan production in batch fermentation at different concentrations of NaCl
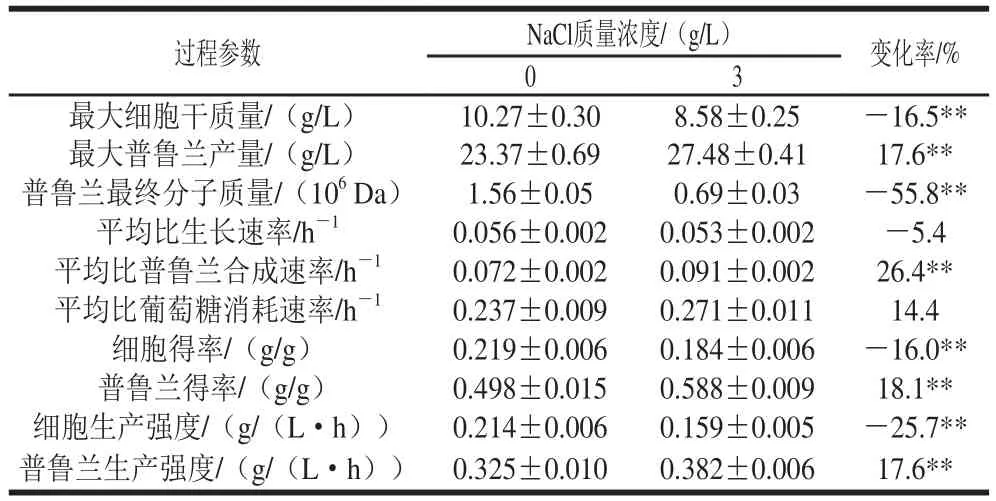
表1 普魯蘭分批發(fā)酵過程參數(shù)比較Table 1 Comparison of cell growth and pullulan production in batch fermentation in the presence and absence of NaCl
2.3 RNA-Seq分析
2.3.1 RNA-Seq數(shù)據(jù)分析
對(duì)來自0 g/L和3 g/L NaCl培養(yǎng)條件下的出芽短梗霉細(xì)胞樣本進(jìn)行RNA-Seq,共獲得近9 240萬 條讀段(reads),總長度約27.72 G堿基。4 個(gè)轉(zhuǎn)錄組樣本的Q30(0.1%堿基識(shí)別錯(cuò)誤率)均在93%左右(表2),表明這些RNA-Seq數(shù)據(jù)具有較高的質(zhì)量,能夠滿足后續(xù)生物信息學(xué)分析的要求。通過FASTQC質(zhì)控分析和trimmomatic處理后,使用Trinity(v2.6.6)對(duì)這些測(cè)序結(jié)果進(jìn)行從頭組裝,共獲得16 204 個(gè)基因和29 523 條轉(zhuǎn)錄本。通過與真菌已知蛋白進(jìn)行比對(duì),BLASTX程序成功鑒別出其中的16 130 條轉(zhuǎn)錄本。

表2 RNA-Seq數(shù)據(jù)Table 2 Overview of RNA-Seq data for A.pullulans
2.3.2 DEGs分析
利用edgeR對(duì)RSEM軟件計(jì)算獲得的Trinity組裝轉(zhuǎn)錄本表達(dá)水平數(shù)據(jù)矩陣進(jìn)行篩選,除去低表達(dá)基因后,在剩下的14 098 個(gè)轉(zhuǎn)錄本中鑒別DEGs。將統(tǒng)計(jì)顯著性閾值(P值)設(shè)為0.05,則有659 個(gè)DEGs被鑒別出,DEGs分析的火山圖見圖3A,其中227 個(gè)DEGs表達(dá)水平在NaCl存在時(shí)顯著上調(diào),432 個(gè)DEGs表達(dá)水平顯著下調(diào),表明培養(yǎng)基中的NaCl對(duì)出芽短梗霉的基因轉(zhuǎn)錄水平產(chǎn)生了顯著影響。利用pheatmap對(duì)這些DEGs進(jìn)行聚類分析(圖3B),結(jié)果顯示,該RNA-Seq數(shù)據(jù)分析結(jié)果可用于后續(xù)的基因功能注釋和代謝通路分析。
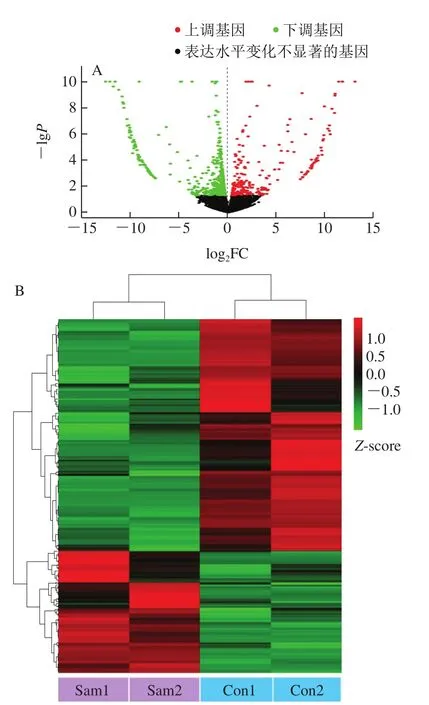
圖3 NaCl存在時(shí)DEGs分析可視化圖譜Fig.3 Volcano map and heatmap of DEGs in the presence of NaCl
2.3.3 DEGs功能注釋和富集分析
利用WEGO對(duì)在NaCl組中表達(dá)水平顯著上調(diào)的227 個(gè)基因和顯著下調(diào)的432 個(gè)基因分別進(jìn)行GO功能注釋及富集分析,共有169 個(gè)上調(diào)基因和360 個(gè)下調(diào)基因獲得GO功能注釋信息。利用R語言包ggplot2對(duì)富集結(jié)果進(jìn)行可視化分析,結(jié)果見圖4。可以看出,在NaCl組中顯著上調(diào)的基因,主要富集在初級(jí)代謝、氮化合物代謝、生物合成等生物學(xué)過程,具有離子綁定、水解酶活性、小分子綁定等分子功能,且大多在胞內(nèi)細(xì)胞器尤其是具有膜結(jié)構(gòu)的細(xì)胞器中發(fā)揮作用;在NaCl組中顯著下調(diào)的基因,其富集的生物學(xué)過程、分子功能和細(xì)胞組分均與上調(diào)基因相似。由此可見,培養(yǎng)基中NaCl的存在,影響了細(xì)胞中的一些水解酶,以及具有離子和小分子綁定功能的蛋白編碼基因的表達(dá)水平,這些均可能與普魯蘭產(chǎn)量和分子質(zhì)量的改變有關(guān)。
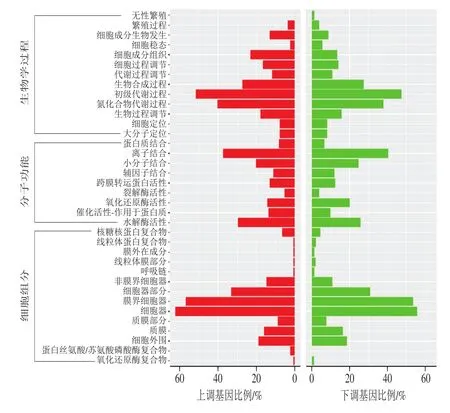
圖4 NaCl存在時(shí)DEGs的GO功能注釋及富集分析Fig.4 GO functional annotation and enrichment analysis of DEGs in the presence of NaCl
2.3.4 DEGs的KEGG代謝通路分析
使用KEGG的Blast-KOALA工具對(duì)NaCl組中表達(dá)水平顯著上調(diào)的227 個(gè)基因和下調(diào)的432 個(gè)基因進(jìn)行代謝通路分析,分別有109 個(gè)上調(diào)基因和224 個(gè)下調(diào)基因被KEGG注釋(表3)。結(jié)果表明,上調(diào)基因主要富集于遺傳信息過程、碳水化合物代謝、氨基酸代謝等相關(guān)通路,而下調(diào)基因則主要富集于碳水化合物代謝、遺傳信息過程、信號(hào)與細(xì)胞過程等相關(guān)通路。在此基礎(chǔ)上,利用KEGG mapper工具對(duì)其中的通路模塊進(jìn)行深入挖掘,發(fā)現(xiàn)分別有58 個(gè)上調(diào)基因和112 個(gè)下調(diào)基因編碼各類酶(表4),其中,上調(diào)基因編碼的酶參與的代謝通路主要包括次級(jí)代謝物生物合成、抗生素生物合成、不同環(huán)境下的微生物代謝、淀粉和蔗糖代謝、糖酵解/糖異生等代謝過程,而下調(diào)基因編碼的酶參與的代謝過程除了上述通路以外,還參與了碳代謝、氨基酸生物合成、丙酮酸代謝、氧化磷酸化等代謝過程。
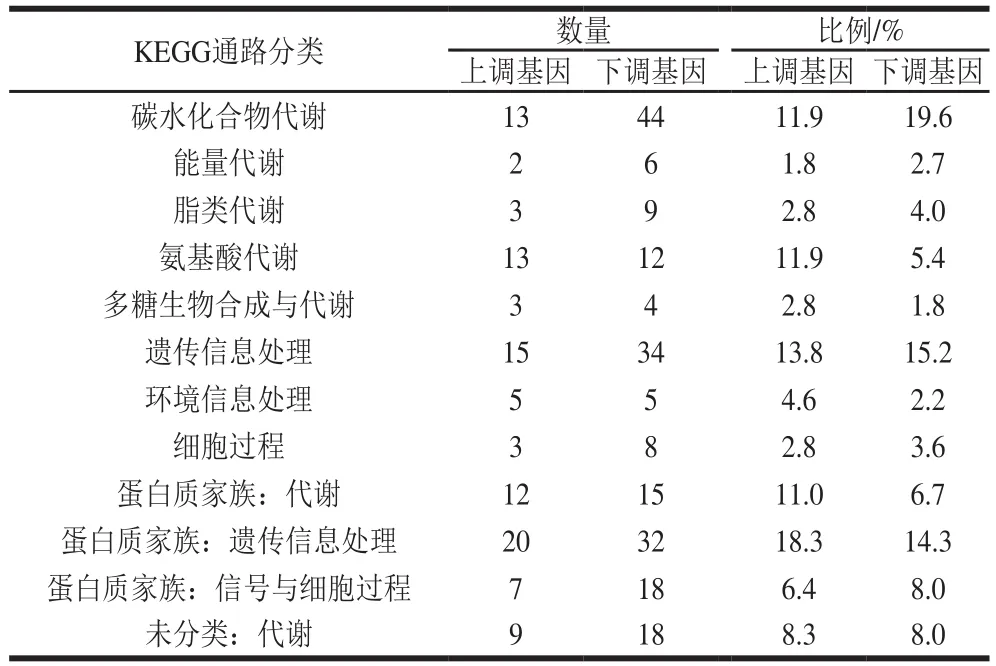
表3 DEGs的KEGG通路富集分析Table 3 KEGG pathway enrichment analysis of DEGs

表4 DEGs的KEGG通路功能挖掘Table 4 KEGG pathway functional annotation of DEGs
需要特別說明的是,直接參與普魯蘭生物合成的關(guān)鍵酶基因(pgm1、pgm2、ugp、fks)和降解酶基因(amy1)在本研究中均能被注釋到,其表達(dá)水平與前期研究[14]中real-time PCR所得到的結(jié)果一致。雖然這些基因的轉(zhuǎn)錄水平在NaCl存在時(shí)均有所上調(diào),但基因FC均小于2,因此不在鑒別出的659 個(gè)DEGs范圍之內(nèi)。盡管如此,針對(duì)RNA-Seq數(shù)據(jù)的分析結(jié)果,仍然可以為今后出芽短梗霉的遺傳改造提供可信的理論依據(jù)。
3 結(jié) 論
本研究考察NaCl在普魯蘭生物合成中的作用,發(fā)現(xiàn)3 g/L NaCl最有利于普魯蘭的過量合成,但培養(yǎng)基中NaCl的存在顯著降低了普魯蘭的分子質(zhì)量。在分批發(fā)酵的基礎(chǔ)上,采用生物信息學(xué)方法對(duì)出芽短梗霉RNA-Seq數(shù)據(jù)進(jìn)行分析,共鑒別出659 個(gè)DEGs,其中NaCl組中有227 個(gè)基因表達(dá)上調(diào),432 個(gè)基因表達(dá)下調(diào)。通過對(duì)這些DEGs進(jìn)行GO功能注釋和KEGG通路富集分析,發(fā)現(xiàn)NaCl影響了參與碳水化合物代謝、淀粉和蔗糖代謝、糖酵解/糖異生等代謝過程的基因的表達(dá)水平,最終在提高了普魯蘭產(chǎn)量的同時(shí)降低了普魯蘭分子質(zhì)量。研究結(jié)果有助于深入理解NaCl在普魯蘭生物合成中的作用機(jī)制,同時(shí)也為出芽短梗霉遺傳改造以實(shí)現(xiàn)高分子質(zhì)量普魯蘭的高效合成提供理論依據(jù)。

