基于RAD測序技術(shù)的瓦氏黃顙魚(Pelteobagrus vachellii)微衛(wèi)星標記的開發(fā)
王青云 郭穩(wěn)杰 成為為 鄧國喬 徐洪亮 夏儒龍
(1 武漢市農(nóng)業(yè)科學院,武漢 430207;2 華中農(nóng)業(yè)大學水產(chǎn)學院,武漢 430070)
瓦氏黃顙魚(Pelteobagrusvachellii)隸屬于鲇形目(Siluriformes)、鲿科(Bagridae)、黃顙魚屬(Pelteobagrus),是黃顙魚屬中個體最大、生長最快的類群,主要分布于長江及其支流中[1]。該魚因肉質(zhì)細嫩、味道鮮美、少肌間刺、營養(yǎng)價值高等優(yōu)點而備受消費者的青睞[2]。但是近些年,由于生境惡化和過度捕撈等原因,瓦氏黃顙魚野生資源遭到嚴重破壞,其種質(zhì)資源也面臨巨大威脅[3]。良種選育是我國瓦氏黃顙魚養(yǎng)殖產(chǎn)業(yè)健康與可持續(xù)發(fā)展亟待解決的重要內(nèi)容之一,而在良種選育過程中,對其遺傳多樣性和系譜信息的掌握是至關(guān)重要的。
微衛(wèi)星標記(microsatellite)又稱為簡單序列重復(simple sequence repeats,SSR),因具有共顯性、多態(tài)性豐富、遵循孟德爾遺傳、易于檢測等優(yōu)點,現(xiàn)已被廣泛用于群體遺傳學[4]、親子鑒定[5]、遺傳圖譜構(gòu)建[6]和基因定位研究[7]等領(lǐng)域。近年來,新一代高通量測序技術(shù)和生物信息學分析方法的發(fā)展,使得低廉且高效地獲得SSR標記序列成為可能。目前已利用高通量測序技術(shù)對黃顙魚 (Pelteobagrusfulvidraco)[8]、莖柔魚(Dosidicusgigas)[9]、大刺鰍 (Mastacembelusarmatus)[10]等物種進行了SSR標記的開發(fā)。本研究采用RAD(restriction association site DNA)簡化基因組測序技術(shù)開發(fā)瓦氏黃顙魚的SSR標記,以期為瓦氏黃顙魚分子標記輔助育種提供可用的分子標記。
1 材料和方法
1.1 瓦氏黃顙魚樣本采集
研究所用的30個瓦氏黃顙魚樣本全部采自長江武漢段,剪取部分魚的尾鰭后將其放生,尾鰭樣本用無水酒精固定后于-20 ℃冷凍保存?zhèn)溆谩;蚪MDNA采用TIANGEN[天根生化科技(北京)有限公司]試劑盒提取。
1.2 RAD文庫構(gòu)建和測序
隨機選取8個瓦氏黃顙魚樣本送交廣州基迪奧生物科技有限公司構(gòu)建RAD簡化基因組文庫,采用Illumina Hiseq 2500進行測序。
1.3 SSR位點鑒別
將8個樣品的高質(zhì)量測序數(shù)據(jù)進行聚類,拼接組裝獲得contig序列后,使用軟件MISA(http://pgrc.ipk-gatersleben.de/misa/)對所有contig序列進行SSR鑒別,鑒別標準為2、3、4、5、6堿基的重復次數(shù)至少為6、5、4、4、4。
1.4 SSR引物設(shè)計與多態(tài)性篩選
鑒定出的SSR位點采用primer 3引物設(shè)計工具(http://bioinfo.ut.ee/primer3/)進行批量設(shè)計。隨機挑選25對SSR重復次數(shù)相對較多的引物由武漢天一輝遠生物科技有限公司進行合成。
用8個瓦氏黃顙魚DNA模板對合成引物進行初步篩選和條件優(yōu)化,優(yōu)化后的引物進行熒光標記后以30個個體DNA為模板進行PCR擴增,擴增后的PCR產(chǎn)物采用毛細管電泳進行多態(tài)性篩選。PCR反應(yīng)體系包括:2×PCR Mix 10 μL,DNA模板1 μL(50 ng/μL),5 mmol/L引物對各0.5 μL,超純水8 μL。PCR反應(yīng)程序:95 ℃預變性4 min;95 ℃變性30 s,退火30 s,72 ℃延伸40 s,循環(huán)34次;72 ℃延伸7 min。PCR產(chǎn)物送武漢天一輝遠生物科技有限公司采用ABI 3730XL進行基因型分析。
1.5 數(shù)據(jù)統(tǒng)計及分析
根據(jù)統(tǒng)計的基因型數(shù)據(jù),用PopGen 32軟件[11]計算微衛(wèi)星位點等位基因數(shù)(Na);采用MS-TOOLS[12]分析觀測雜合度(Ho)、期望雜合度(He)及多態(tài)信息含量 (polymorphic information content,PIC);用ARLEQUIN version 3.1 軟件[13]檢測各位點是否偏離哈迪-溫伯格平衡(Hardy-Weinberg equilibrium,HWE)。
2 結(jié)果
2.1 瓦氏黃顙魚RAD簡化基因組SSR分析
序列組裝和拼接后共獲得2 275 778條contig序列,利用MISA對所得的contig序列進行微衛(wèi)星片段搜索,共檢測到466 983個SSR位點,其中二堿基重復位點最多(335 095個),主要是AC/GT(占49.8%),其次是四堿基重復位點(84 703個),主要是AATG/CATT(占3.9%);剩余依次是三堿基重復位點(31 271個)、五堿基重復位點(13 661個)和六堿基重復位點(2 253個)。相關(guān)信息詳見圖1和表1。
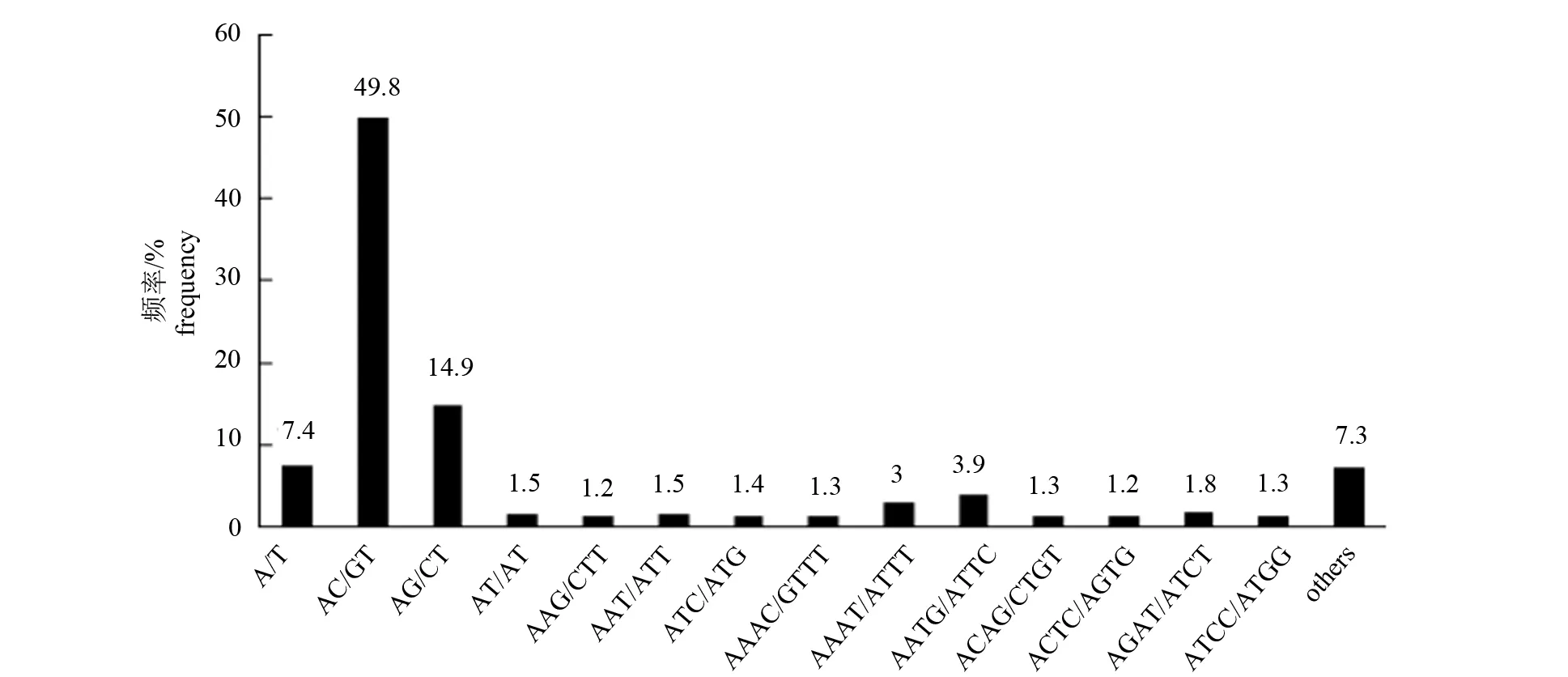
圖1 SSR重復單元分類和分布頻率統(tǒng)計Fig.1 Characterization and frequency of microsatellites with different motif sizes
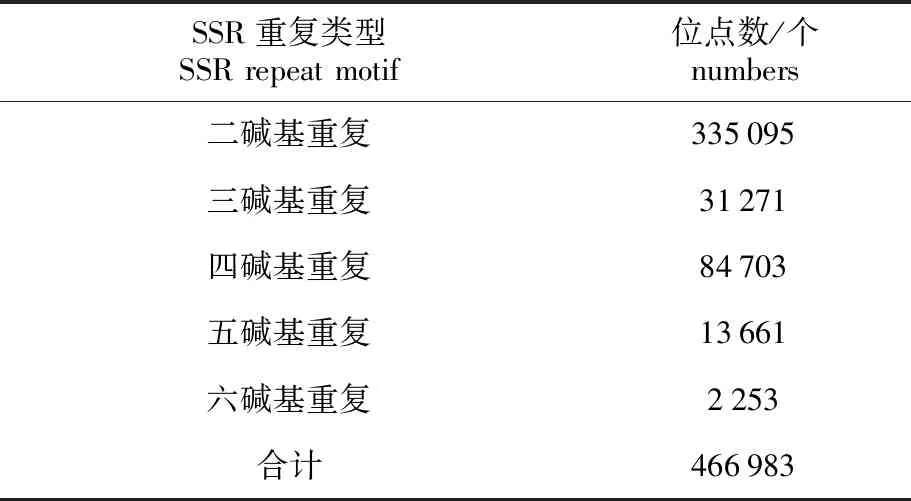
表1 微衛(wèi)星重復類型統(tǒng)計表Tab.1 Statistics information of microsatellites with different motif sizes
2.2 瓦氏黃顙魚SSRs多態(tài)性篩選和檢測
本研究從被篩選出的結(jié)果中隨機選取SSR重復次數(shù)相對較多的25對引物,對武漢江段8個瓦氏黃顙魚DNA樣本進行PCR擴增,經(jīng)優(yōu)化后,有19對引物能較好地擴增出產(chǎn)物,且產(chǎn)物大小與預期擴增目的片段大小一致。用30個瓦氏黃顙魚樣本對擴增出目標產(chǎn)物的引物進行多態(tài)性驗證,其中13個位點呈現(xiàn)出多態(tài)性(見表2)。
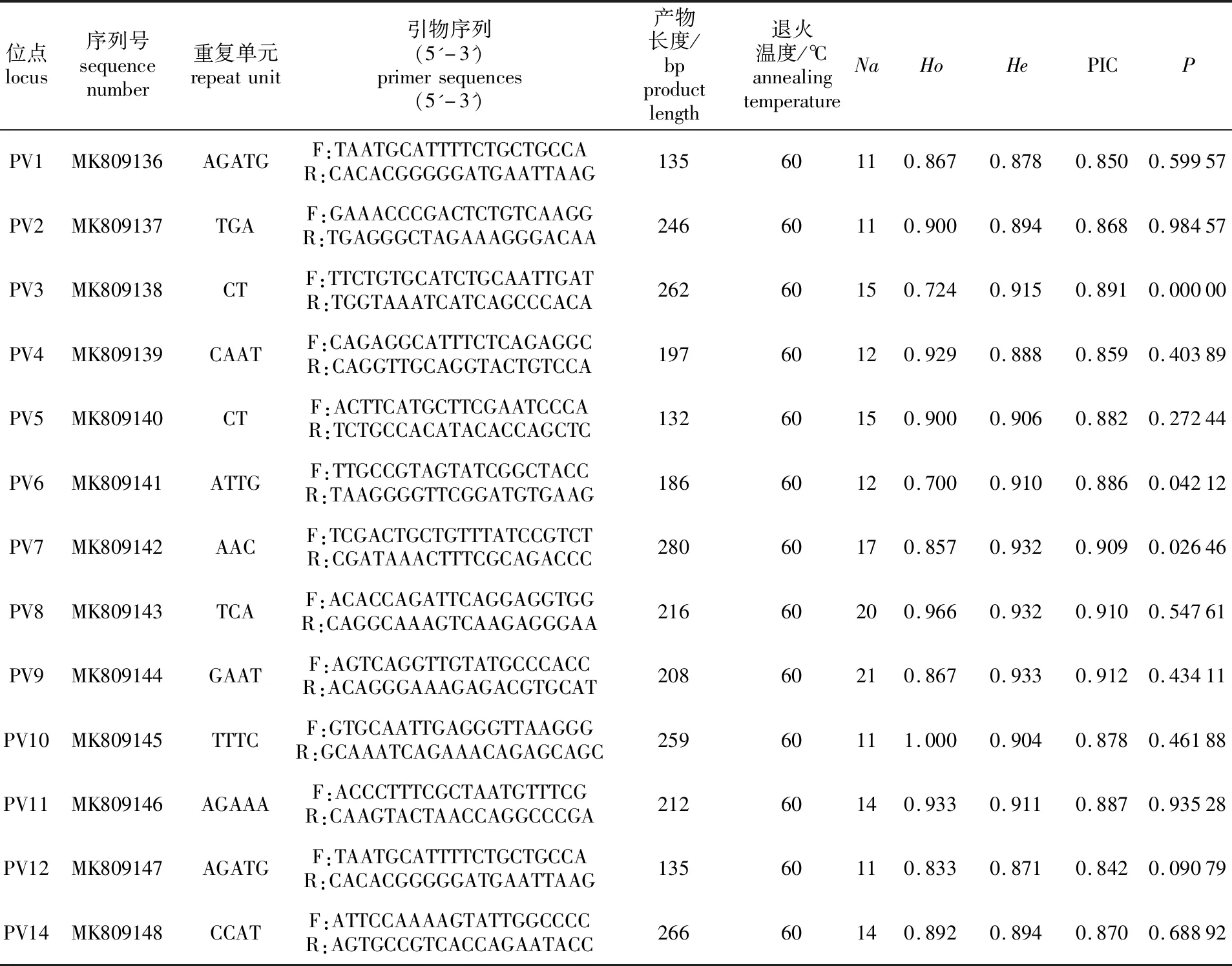
表2 瓦氏黃顙魚多態(tài)性微衛(wèi)星引物序列信息Tab.2 Information of polymorphic microsatellite markers isolated from Pelteobagrus vachelli
13個位點共檢測到184個等位基因,各位點等位基因數(shù)(Na)在11~21個,平均為14.2個;各位點觀察雜合度(Ho)在0.700~1.00,平均為0.874;期望雜合度(He)在0.871~0.933,平均為0.905;平均多肽信息含量(PIC)為0.880,所有位點均屬于高多態(tài)性位點(PIC>0.5)。除位點PV6和PV7顯著偏離Hardy-Weinberg平衡(0.01 傳統(tǒng)SSR開發(fā)方法往往需要構(gòu)建基因組富集文庫、雜交篩選和測序等步驟,過程比較耗時費力,如今高通量測序技術(shù)的飛速發(fā)展以及成本的急劇降低為SSR標記的開發(fā)創(chuàng)造了較好的條件,該技術(shù)具有開發(fā)周期短、得率高、靈活性好等優(yōu)點[14]。近年來,利用RAD簡化基因組測序開發(fā)SSR標記的技術(shù)日益成熟,相關(guān)研究報道也越來越多。劉連為等[9]對莖柔魚(D.gigas)進行了RAD簡化基因組測序,覆蓋度為2X,結(jié)果表明,可進行引物設(shè)計的SSR位點有5 177個,在隨機合成的100對引物中,有60對引物擴增出清晰條帶,其中39對引物擴增出的條帶呈現(xiàn)多態(tài)性。屈政委等[15]采用RAD-seq對印尼虎魚(Datnioidespulcher)進行簡化基因組測序,經(jīng)序列分析,共檢測到26 359個SSR位點,并成功開發(fā)出20個多態(tài)性的SSR位點。本研究通過分析瓦氏黃顙魚RAD簡化基因組測序數(shù)據(jù),共鑒定出466 983個SSR位點,在此基礎(chǔ)上,后續(xù)所要做的工作相對比較簡單,只需要進行PCR擴增以檢驗其是否具有多態(tài)性即可。因此,基于RAD簡化基因組測序進行SSR標記開發(fā)具有很好的應(yīng)用前景。 目前已報道的瓦氏黃顙魚SSR標記數(shù)量很少,均來自同屬黃顙魚的跨種擴增[16],尚未見到基于高通量測序技術(shù)大量開發(fā)瓦氏黃顙魚SSR標記的報道。本研究通過對瓦氏黃顙魚RAD簡化基因組序列進行分析,共設(shè)計出了414 484對符合引物開發(fā)條件的SSR候選引物。為了進一步檢驗引物的開發(fā)質(zhì)量及開發(fā)效率,利用熒光PCR毛細管電泳對其中25對引物進行了電泳檢測。檢測結(jié)果表明,在25對引物中,有13對為多態(tài)性引物,且均為高多態(tài)性引物(PIC>0.5)[17],這可能與選擇的SSR重復次數(shù)相對較多有關(guān)。這13個具有高度多態(tài)性的位點今后可直接應(yīng)用于瓦氏黃顙魚群體遺傳學、親子鑒定、分子標記輔助育種等方面研究中。3 討論

