楓涇豬、梅山豬和沙烏頭豬全基因組ROH檢測及候選基因鑒定
趙旭庭 徐盼 馬政 仲德 劉林雨 倪黎綱 吳井生


摘要:? 通過檢測楓涇豬、梅山豬和沙烏頭豬的長純合片段(Runs of homozygosity, ROH),鑒定其與3個豬種經濟性狀相關的候選基因。利用全基因組單核苷酸多態性(Single Nucleotide Polymorphisms, SNP)芯片數據對這3個豬群體進行全基因組ROH檢測,統計ROH數量、長度及分布,計算基于ROH的近交系數( F?? ROH ),并在高頻ROH區域鑒定候選功能基因。結果顯示,從楓涇豬、梅山豬和沙烏頭豬群體中分別檢測到 1 588 個、 3 314 個和 4 419 個ROH。楓涇豬的ROH平均長度為12.84 Mb,平均 F?? ROH 為0.084;梅山豬的ROH平均長度為10.55 Mb,平均 F?? ROH 為0.164;沙烏頭豬的ROH平均長度為10.52 Mb,平均 F?? ROH 為0.141。在高頻ROH區域共鑒定到9個楓涇豬候選功能基因和8個梅山豬候選功能基因。研究結果為楓涇豬、梅山豬和沙烏頭豬的資源保護和分子標記輔助選擇奠定了基礎。
關鍵詞:? 豬; 長純合片段; 近交系數; 候選基因
中圖分類號:? S813.1??? 文獻標識碼: A??? 文章編號:? 1000-4440(2021)05-1234-10
Genome-wide scan for run of homozygosity and identification of corresponding candidate genes in Fengjing pigs, Meishan pigs and Shawutou pigs
ZHAO Xu-ting? 1 , XU Pan? 1 , MA Zheng? 1 , ZHONG De? 1 , LIU Lin-yu? 1 , NI Li-gang? 1 , WU Jing-sheng? 2
(1.Jiangsu Agri-animal Husbandry Vocational College, Taizhou 225300, China; 2.Jiangsu Vocational College of Agriculture and Forestry, Jurong 212400, China)
Abstract:? By detecting runs of homozygosity (ROH) of Fengjing pig, Meishan pig and Shawutou pig, the candidate genes related to the economic traits of the three pig breeds were identified. The genome-wide single nucleotide polymorphisms (SNP) chip data were used to perform genome-wide ROH scan for the three pig breeds. The number, length and distribution of ROH were counted, and the inbreeding coefficient ( F?? ROH ) was calculated based on ROH. In addition, the functional candidate genes were identified in the high frequency ROH region. The results showed that ?1 588 , 3 314 and 4 419 ROH were detected in the three pig populations, respectively. The average length of ROH was 12.84 Mb and the average ?F?? ROH ?was 0.084 in Fengjing pigs. The average length of ROH was 10.55 Mb and the average ?F?? ROH ?was 0.164 in Meishan pigs. The average length of ROH was 10.52 Mb and the average ?F?? ROH ?was 0.141 in Shawutou pigs. Nine functional candidate genes of Fengjing pig and eight functional candidate genes of Meishan pig were identified in high frequency ROH region. The results lay a foundation for resource conservation and molecular marker-assisted selection of Fengjing pig, Meishan pig and Shawutou pig.
Key words:? pig; runs of homozygosity (ROH); inbreeding coefficient; candidate gene
長純合片段(Runs of homozygosity, ROH)是基因組上一定數量、一定密度的單核苷酸多態性(Single nucleotide ploymorhphisms, SNPs)表現為純合的一段區域? [1] 。在人工選擇的過程中,畜禽群體的基因組被重塑,所選區域中的ROH頻率會提高,甚至產生“ROH島”。ROH信息在畜禽親緣關系鑒定、遺傳多樣性分析、近交衰退評估、選擇信號鑒定和功能基因篩選等方面發揮著重要作用。Keller等? [2] 在比較基于ROH的近交系數( F?? ROH )、基于基因組雜合度的近交系數( F?? HOM )、基于基因組關系矩陣的近交系數( F?? GRM )和基于系譜信息的近交系數( F?? PED )后發現, F?? ROH 是最有效的估計近交的方法。Mastrangelo等? [3] 在Pontremolese、Varzese-Ottonese和Mucca Pisana群體中檢測到高水平的ROH,針對這些群體,在實施配種計劃中應盡量增加種公畜血統,減少其遺傳多樣性的損失,維持或者增加其群體有效含量。Saura等? [4] 分析了近交對伊比利亞豬繁殖性狀的影響,結果表明,近交系數每增加0.1,其仔豬初生后的存活率、仔豬出生后的總數量有下降趨勢。Zhong等? [5] 利用簡化基因組測序數據在金華豬群體的ROH島中鑒別到與繁殖、肉質、食欲及抗病性狀相關的基因。
楓涇豬、梅山豬和沙烏頭豬是江蘇省優良的地方豬遺傳資源,以產仔數高、肉質優良、耐粗飼和環境適應力強等特點而聞名。本研究利用中芯一號育種芯片數據對這3個豬群體進行全基因組ROH檢測,統計ROH數量、長度及分布,計算基于ROH的近交系數,并在高頻ROH區域鑒定候選功能基因,以期為楓涇豬、梅山豬和沙烏頭豬的資源保護和分子標記輔助選擇奠定基礎。
1 材料與方法
1.1 試驗動物
試驗動物為來自鎮江牧苑動物科技開發有限公司省級楓涇豬保種場的107頭楓涇豬(FJ)、江蘇農林職業技術學院國家級梅山豬保種場的94頭梅山豬(MS)和江蘇興旺農牧科技發展有限公司國家級沙烏頭豬保種場的146頭沙烏頭豬(SWT)。采集豬耳組織樣后保存于 -20 ℃ 冰箱內待用。
1.2 SNP芯片分型及質量控制
委托北京康普森生物技術有限公司使用中芯一號育種芯片對107頭楓涇豬、94頭梅山豬和146頭沙烏頭豬進行基因分型。用plink軟件分別對3個豬群體的芯片數據進行質量控制,質量控制標準:1)個體檢出 率≥ 90%;2)SNP檢出 率≥ 90%;3)次等位基因頻率( MAF )≥0.01;4)哈迪溫伯格平衡? P ≥? 1× 10? -5 ;5)剔除性染色體。
1.3 全基因組ROH檢測
ROH檢測使用Plink軟件,使用滑動窗口的方法對常染色體進行檢測,具體的檢測參數:1)ROH長度大于1 Mb;2)至少連續50個SNPs;3)連續SNP間的距離小于1 Mb;4)最多允許ROH中有5個缺失和1個雜合;5)窗口閾值為0.01;6)密度為0.01 ?SNP/kb 。
1.4 基因組近交系數的計算
利用ROH計算近交系數( F?? ROH ),公式如下:
F?? ROH? =∑? L?? ROH? /? L?? auto
其中:∑? L?? ROH? 為常染色體上ROH片段的長度之和, L?? auto 為常染色體的物理總長度。
根據上式計算 F?? ROH 1.0~5.0 Mb 、 F?? ROH 5.1~10.0 Mb 、 F?? ROH>10.0 Mb 和 F?? ROH total ?4種近交系數之間的Pearson相關系數,其中 F?? ROH total 為基于全部長度的ROH計算的近交系數。
1.5 高頻ROH區域的檢測及候選基因的鑒定
統計每個SNP在各自豬群體內參與組成ROH的次數占樣本數的比例,并將高于45%的SNP區域作為高頻ROH區域,利用RCircos包繪制3個豬群體ROH頻率在染色體上分布的Circos圖。基于高頻ROH區段的物理位置,在豬基因組注釋文件(ftp://ftp.ensembl.org/pub/release-100/gtf/sus_scrofa/Sus_scrofa.Sscrofa11.1.100.chr.gtf.gz)中檢索基因。
利用DAVID數據庫(https://david.ncifcrf.gov/)對高頻ROH區域注釋到的基因進行GO(Gene ontology,基因本體)功能富集和KEGG(Kyoto Encyclopedia of Genes and Genomes,京都基因與基因組百科全書)Pathway富集分析,當? P ≤ 0.05時,則表示顯著富集。將顯著富集到生物學過程和信號通路的基因與豬數量性狀基因座(Quantitative Trait Locus, QTL)數據庫(https://www.animalgenome.org/cgi-bin/QTLdb/SS/index)進行比對,獲得候選功能基因。
2 結果與分析
2.1 基因型的質量控制
中芯一號育種芯片含有 51 315 個SNP位點,經過質量控制后,對剩余107頭楓涇豬的 19 895 個SNPs、94頭梅山豬的 24 526 個SNPs和146頭沙烏頭豬的 32 275 個SNPs進行后續分析。
2.2 ROH數量、長度及分布統計
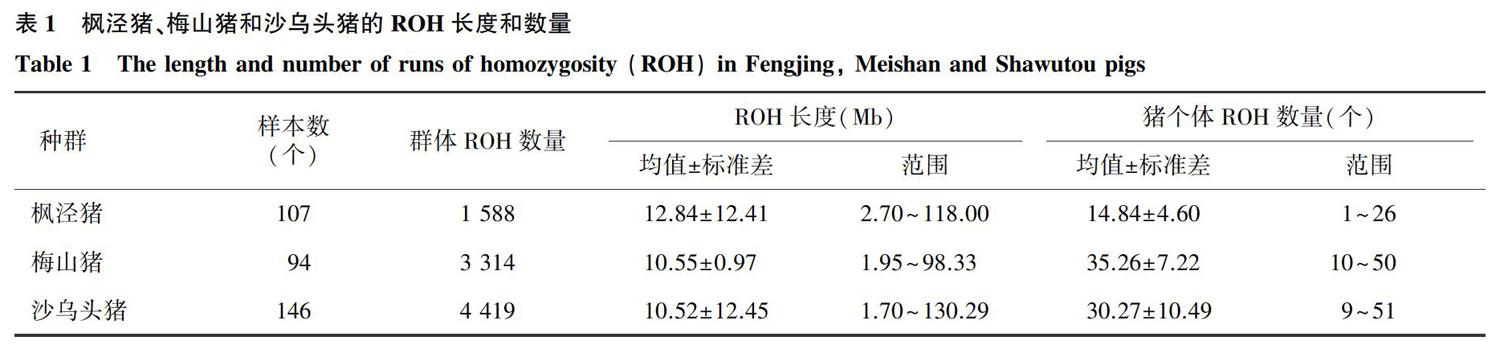
由表1、圖1、圖2可以看出,從楓涇豬、梅山豬和沙烏頭豬群體中分別檢測到 1 588 個、 3 314 個和 4 419 個ROH,其長度主要集中在40.00 Mb以內。楓涇豬ROH的平均長度為12.84 Mb;ROH的最長長度為118.00 Mb,含有 1 328 個SNPs,位于第15號染色體上;ROH的最短長度為2.70 Mb,含有50個SNPs,位于第13號染色體上;楓涇豬個體的平均ROH數量為14.84個,以15號染色體上的ROH最多,18號染色體上的ROH最少。梅山豬ROH的平均長度為10.55 Mb;ROH的最長長度為98.33 Mb,含有 1 313 個SNPs,位于第14號染色體上;ROH的最短長度為1.95 Mb,含有59個SNPs,位于第6號染色體上;梅山豬個體ROH的平均數量為35.26個,14號染色體上的ROH最多,12號染色體上的ROH最少。沙烏頭豬ROH的平均長度為10.52 Mb;ROH的最長長度為130.29 Mb,含有 1 727 個SNPs,位于第1號染色體上;ROH的最短長度為1.70 Mb,含有50個SNPs,位于第6號染色體上;沙烏頭豬個體ROH的平均數量為30.27個,1號染色體上的ROH最多,10號染色體上的ROH最少。
2.3 近交系數
由表2可以看出,楓涇豬基因組 F?? ROH total 的最大值為0.171,最小值為0.003,平均值為0.084;梅山豬基因組 F?? ROH total 的最大值為0.305,最小值為0.057,平均值為0.164;沙烏頭豬基因組 F?? ROH total 的最大值為0.501,最小值為0.022,平均值為0.141;3個群體中梅山豬的平均 F?? ROH total 最高。對比表2中3種豬群體的4種近交系數發現,楓涇豬群體中 F?? ROH 5.1~10.0 Mb 與 F?? ROH >10.0 Mb 、 F?? ROH total 分別在0.05、 0.001 水平顯著相關, F?? ROH >10.0 Mb 與 F?? ROH total 在0.001水平顯著相關;梅山豬群體中 F?? ROH 5.1~10.0 Mb 與 F?? ROH 1.0~5.0 Mb 、 F?? ROH total 及 F?? ROH >10.0 Mb 與 F?? ROH total 均在0.001水平顯著相關;沙烏頭豬群體中 F?? ROH 1.0~5.0 Mb 與 F?? ROH total 在0.01水平顯著相關, F?? ROH 1.0~5.0 Mb 與 F?? ROH 5.1~10.0 Mb 、 F?? ROH 5.1~10.0 Mb 與 F?? ROH >10.0 Mb 、 F ??ROH 5.1~10.0 Mb 與 F?? ROH total 、 F?? ROH >10.0 Mb 與 F?? ROH total 均在0.001水平顯著相關(表3)。
2.4 高頻ROH區域及候選基因的鑒定
本研究選取45%作為高頻ROH區域的閾值,在楓涇豬、梅山豬和沙烏頭豬群體中分別檢測到7個、20個和9個高頻ROH區域,分別注釋到414個、547個和151個基因(表4)。楓涇豬、梅山豬和沙烏頭豬沒有共同的高頻ROH區域,楓涇豬和梅山豬在第14號染色體上有共同高頻ROH區域,梅山豬、沙烏頭豬在第8號、11號染色體上有共同高頻ROH區域(圖3)。
利用DAVID數據庫分別對楓涇豬、梅山豬和沙烏頭豬群體中注釋到的414個、547個和151個基因進行GO功能富集分析和KEGG Pathway富集分析,結果分別見表5、表6和表7。分析得出,楓涇豬群體的414個基因中的136個基因顯著富集于谷胱甘肽代謝過程、肝臟發育、炎癥反應的負調控等22個GO條目和朊病毒病等4個信號通路;梅山豬群體的547個基因中的134個基因顯著富集于半胱氨酸型內肽酶活性的負調節、谷胱甘肽代謝過程、類黃酮葡萄糖醛酸化等22個GO條目和11個信號通路;沙烏頭豬群體的151個基因中的24個基因顯著富集于類黃酮葡萄糖醛酸化、類黃酮生物合成過程、生物礦物組織發育等11個GO條目和抗壞血酸和醛糖酸鹽代謝等9個信號通路。分別將楓涇豬群體的136個基因、梅山豬群體的134個基因和沙烏頭豬群體的24個基因與豬QTL數據庫進行比對,得到楓涇豬候選功能基因? HNF1A、CDH17、LIF、DECR1、ARL15、ACTN2、FABP5、FABP4和EWSR1? (表5),梅山豬候選功能基因? CTSH、MSTN、GC、VEGFA、ZDHHC2、MTMR7、CDC5L和LIF? (表6)。
3 討 論
ROH可以指示親緣的遠近和近交的歷史,ROH片段越長,家系內存在近世代近交的可能性越高? [2, 6] 。在0~5.0 Mb長度的ROH中,楓涇豬所占比例最低,沙烏頭豬所占比例最高;在大于5.0 Mb長度的ROH中,楓涇豬所占比例最高,沙烏頭豬所占比例最低。該結果與ROH平均長度的結果一致(楓涇豬ROH的平均長度最長,沙烏頭豬ROH的平均長度最短)。3個豬群中長度大于5.0 Mb的ROH所占比例都較高,表明3個豬群經歷了較高程度的近交,可能與3個豬群的群體規模有關。在過去幾十年里,歐洲現代品種豬(主要是大白豬、長白豬和杜洛克豬)因瘦肉率高、生長速度快、飼料轉化率高而不斷被引進中國,目前占據著中國養豬業的統治地位,導致中國地方豬的養殖數量顯著減少,楓涇豬、梅山豬和沙烏頭豬的種群規模也因此受到影響。在本研究中,楓涇豬、梅山豬和沙烏頭豬樣品采集于保種場,種群數量有限,從而導致近世代較高程度的近交。
每個環代表1個豬群體,從內到外的環分別為楓涇豬、梅山豬、沙烏頭豬,柱子代表ROH頻率。圖中數據表示染色體的位置信息。
研究發現,基于ROH的近交系數( F?? ROH )與基于系譜信息的近交系數( F?? PED )間的相關系數隨著ROH長度的增加而提高,在群體系譜信息不全的情況下, F?? ROH 可以替代 F?? PED 來評估近交程度? [7] 。楓涇豬、梅山豬和沙烏頭豬保種場早期的豬主要從主產區及周邊長期從事地方豬飼養的農戶收集而來,系譜信息不全,因此需要通過基于ROH的近交系數( F?? ROH )來評估其近交程度。本研究基于ROH計算了每個豬群的4種近交系數( F?? ROH 1.0~5.0 Mb 、 F ??ROH 5.1~10.0 Mb 、 F?? ROH >10.0 Mb 和 F?? ROH total ),結果表明,3個豬群中楓涇豬的近交程度最低,梅山豬的近交程度最高。與 F?? ROH 1.0~5.0 Mb 、 F?? ROH 5.1~10.0 Mb 相比,3個豬群的 F?? ROH >10.0 Mb 與 F?? ROH total 最接近,與4種近交系數間的相關性分析結果一致, F?? ROH >10.0 Mb 和 F?? ROH total 間的相關系數最高。Zhong等? [5] 利用簡化基因組測序數據計算了金華豬的 F?? ROH 1.0~5.0 Mb 、 F?? ROH 5.1~10.0 Mb 、 F?? ROH >10.0 Mb 和 F?? ROH all ,結果表明, F?? ROH >10.0 Mb 和 F?? ROH all 間的相關系數最高,與本研究結果一致。
本研究通過將富集到生物學過程和信號通路的基因與豬QTL數據庫進行比對,鑒定到9個楓涇豬候選功能基因和8個梅山豬候選功能基因,其中 LIF 基因是共同的候選功能基因, ?HNF1A? 基因是豬肉質和胴體性狀的候選基因? [8] , ?CDH17? 基因影響公豬睪丸中雄烯酮水平? [9] , LIF 基因影響豬的產活仔數和21日齡仔豬數? [10] , ?DECR1? 基因與杜洛克豬脂質組成性狀顯著相關? [11] , ?ARL15? 基因是豬的眼肌面積的候選基因? [12] , ?ACTN2? 基因與豬肉的烹飪損失有關? [13] , ?FABP5? 和? FABP4? 基因與豬的脂肪性狀相關? [14-15] , ?EWSR1? 基因影響豬肉肉色? [13] , ?CTSH? 基因與飼料增質量比相關? [16] , ?MSTN? 基因與豬日增質量和體質量等性狀顯著相關? [17] , ?GC? 基因與皮特蘭豬的pH? 24 h 相關? [18] , ?VEGFA? 基因影響豬后腿步態評分? [19] , ?ZDHHC2? 基因是魯萊黑豬肌內脂肪含量的候選基因? [20] , ?MTMR7? 基因與豬血液中平均紅細胞血紅蛋白含量有關? [21] , ?CDC5L? 基因與豬血細胞比容和血紅蛋白相關? [22] 。本研究鑒定到的候選功能基因與豬的生長發育、胴體性狀、肉質性狀和繁殖性狀相關,下一步試驗將利用候選基因法研究候選功能基因與相關性狀的關系,從而為解析豬生長發育、胴體性狀、肉質性狀和繁殖性狀的分子機制提供參考。
參考文獻:
[1]? GIBSON J, MORTON N E, COLLINS A. Extended tracts of homozygosity in outbred human populations[J]. Human Molecular Genetics, 2006, 15(5): 789-795.
[2] KELLER M C, VISSCHER P M, GODDARD M E. Quantification of inbreeding due to distant ancestors and its detection using dense single nucleotide polymorphism data[J]. Genetics, 2011, 189(1): 237-249.
[3] MASTRANGELO S, SARDINA M T, TOLONE M, et al. Genome-wide identification of runs of homozygosity islands and associated genes in local dairy cattle breeds[J]. Animal, 2018, 12(12): 2480-2488.
[4] SAURA M, FERNNDEZ A, VARONA L, et al. Detecting inbreeding depression for reproductive traits in Iberian pigs using genome-wide data[J]. Genetics Selection Evolution, 2015, 47(1): 1.
[5] ZHONG X, SUN H, ZHANG Z, et al. Assessment of autozygosity derived from runs of homozygosity in Jinhua pigs disclosed by sequencing data[J]. Frontiers in Genetics, 2019, 10: 274.
[6] HOWRIGAN D P, SIMONSON M A, KELLER M C. Detecting autozygosity through runs of homozygosity: a comparison of three autozygosity detection algorithms[J]. BMC Genomics, 2011, 12(1): 460.
[7] PERIPOLLI E, STAFUZZA N B, MUNARI D P, et al. Assessment of runs of homozygosity islands and estimates of genomic inbreeding in Gyr ( Bos indicus ) dairy cattle[J]. BMC Genomics, 2018, 19(1): 34.
[8] KAYAN A, UDDIN M J, KOCAMIS H, et al. Association and expression analysis of porcine HNF1A gene related to meat and carcass quality traits[J]. Meat Science, 2013, 94(4): 474-479.
[9] DRAG M H, KOGELMAN L J, MARIBO H, et al. Characterization of eQTLs associated with androstenone by RNA sequencing in porcine testis[J]. Physiological Genomics, 2019, 51(10): 488-499.
[10] MUCHA A, ROPKA-MOLIK K, PIRKOWSKA K, et al. Effect of ?EGF, AREG ?and ?LIF ?genes polymorphisms on reproductive traits in pigs[J]. Animal Reproduction Science, 2013, 137(1/2): 88-92.
[11] RAMREZ O, QUINTANILLA R, VARONA L, et al. DECR1 and ME1 genotypes are associated with lipid composition traits in Duroc pigs[J]. Journal of Animal Breeding nad Genetics, 2014, 131(1): 46-52.
[12] ZHUANG Z W, LI S Y, DING R R, et al. Meta-analysis of genome-wide association studies for loin muscle area and loin muscle depth in two Duroc pig populations[J]. PLoS One, 2019, 14(6): e0218263.
[13] LI X P, KIM S W, DO K T, et al. Analyses of porcine public SNPs in coding-gene regions by re-sequencing and phenotypic association studies[J]. Molecular Biology Reports, 2011, 38(6): 3805-3820.
[14] ESTELL J, PREZ-ENCISO M, MERCAD A, et al. Characterization of the porcine?? FABP5?? gene and its association with the?? FAT1?? QTL in an Iberian by Landrace cross[J]. Animal Genetics, 2006, 37(6): 589-591.
[15] MERCAD A, PREZ-ENCISO M, VARONA L, et al. Adipocyte fatty-acid binding protein is closely associated to the porcine locus on chromosome 4[J]. Journal of Animal Science, 2006, 84(11): 2907-2913.
[16] RUSSO V, FONTANESI L, SCOTTI E, et al. Single nucleotide polymorphisms in several porcine cathepsin genes are associated with growth, carcass, and production traits in Italian Large White pigs[J]. Journal of Animal Science, 2008, 86(12): 3300-3314.
[17] TU P A, SHIAU J W, DING S T, et al. The association of genetic variations in the promoter region of myostatin gene with growth traits in Duroc pigs[J]. Animal Biotechnology, 2012, 23(4): 291-298.
[18] CINAR M U, KAYAN A, UDDIN M J, et al. Association and expression quantitative trait loci (eQTL) analysis of porcine ?AMBP, GC ?and?? PPP1R3B?? genes with meat quality traits[J]. Molecular Biology Reports, 2012, 39(4): 4809-4821.
[19] GUO Y M, ZHANG X F, REN J, et al. A joint genomewide association analysis of pig leg weakness and its related traits in an F 2 population and a Sutai population[J]. Journal of Animal Science, 2013, 91(9): 4060-4068.
[20] WANG Y P, NING C, WANG C, et al. Genome-wide association study for intramuscular fat content in Chinese Lulai black pigs[J]. Asian Australasian Journal of Animal Sciences, 2019, 32(5): 607-613.
[21] WANG J Y, LUO Y R, FU W X, et al. Genome-wide association studies for hematological traits in swine[J]. Animal Genetics, 2013, 44(1): 34-43.
[22] ZHANG Z Y, HONG Y, GAO J, et al. Genome-wide association study reveals constant and specific loci for hematological traits at three time stages in a White Duroc×Erhualian F 2 resource population[J]. PLoS One, 2013, 8(5): e63665.
(責任編輯:徐 艷)

