微型生物群落eDNA-PFU監測
姜傳奇 熊 杰 黃 潔 馮偉松 龔迎春 繆 煒
(中國科學院水生生物研究所, 武漢 430072)
主要由藻類和原生動物等組成的微型生物群落在水生態系統中起著至關重要的作用, 是水生態系統食物鏈的關鍵環節。微型生物個體小, 對環境變化響應靈敏, 非常適合用于環境的生物監測。大量研究表明微型生物群落的結構和功能可以很好地反映不同生境的水質特征。
1991年發布的《水質 微型生物群落監測 PFU法》(GB/T 12990-91)[1]是我國自行制訂的首個生物監測國家標準。該標準應用聚氨酯泡沫塑料塊(PFU)作為人工基質采集微型生物, 并根據該生物群落的結構與功能參數評價水質。相比于直接采水樣, PFU法可采集到水體中85%以上的微型生物,同時還可避免河流等流水生境中采集的瞬時性和隨機性等問題[2]。標準頒布后, PFU法在全國范圍內對不同河流(包括長江、漢江、烏江、清江和沅江等)的水質進行評價, 被證明是一種快速、經濟、準確的監測方法[3]。
PFU法需要專業人員利用顯微鏡對微型生物進行種類鑒定, 對專業知識要求高、費時耗力且不易標準化。環境DNA(eDNA)技術的發展則突破了以往監測人員無法準確鑒定微型生物的瓶頸, 能更為高效和準確地鑒定出水環境中包括微型生物在內的各種水生生物[4—6]。eDNA方法在進行物種鑒定時依賴的參考序列庫具有通用性和標準性, 所獲鑒定數據更客觀且可比性強。尤其在萬種原生生物基因組計劃[7]支撐下, 微型生物基因組和條形碼數據庫不斷豐富和完善, 使得微型生物種類的鑒定更為準確; 同時由于DNA測序成本的降低和設備的國產化, 檢測快速、經濟, 易于推廣。
基于此提出微型生物群落(藻類和原生動物)eDNA-PFU法, 即基于eDNA技術改進的PFU法, 對微型生物群落的結構和功能進行監測, 并將藻類和原生動物種類數、指示種類和功能類群等微型生物群落的結構和功能參數作為湖泊河流水庫等水體的生態考核指標。為了對上述提議進行驗證, 我們針對武漢東湖水體中的原生動物群落開展了預實驗, 全面評價了eDNA-PFU法監測微型生物群落的準確性和有效性。
1 材料與方法
1.1 樣品采集與制備
2020年8月12日至9月12日, 本研究在湖北武漢東湖中設置了一個采樣點(30°53′95″N, 114°37′51″E;圖 1)。實驗方法參照《水質 微型生物群落監測PFU法》(GB/T 12990-91), 將PFU(孔徑為100—150 μm)制作成5 cm×6.5 cm×8 cm的小塊, 采用沉式掛放法將PFU塊掛放于距離岸邊20 m處, 掛放深度為距離水面30—50 cm。分別在放置3d、5d、7d、9d、11d、13d、15d、17d、19d、23d和30d后隨機取6塊PFU樣品, 采集的PFU塊放到密封袋中帶回實驗室, 獲取各PFU塊中的水樣, 同時在PFU周圍利用采水器采集3 L原位水環境樣品帶回實驗室。取PFU樣品和原位水環境樣品分別進行微型生物活體鑒定, 另取等量的PFU樣品和水環境樣品進行eDNA樣品制備(每組樣品設置3個平行)。
圖1 PFU在武漢東湖的樣點位置及PFU的放置方式Fig. 1 Location and PFU systems applied in East Lake, Wuhan
1.2 原生動物的形態學鑒定與豐度測定
吸取一定體積的PFU擠出液或原位水在倒置顯微鏡下進行物種鑒定和計數。
原生動物的鑒定和計數都是在顯微鏡(OLYMPUS BX53, 日本)下進行[3]。鏡檢時用微吸管從含PFU擠出液或原位水的燒杯底部吸3滴水樣進行觀察。第一片在高倍鏡下觀察, 第二、三片分別用中倍鏡和低倍鏡觀察。計數時先把燒杯中的水樣搖勻, 用微吸管吸0.1 mL水樣于0.1 mL的技術框內,全片進行活體計數。原生動物的物種鑒定主要參考《微型生物監測新技術》[8]和《原生動物學》[9]。
1.3 eDNA的提取、測序與數據分析
采用十六烷基三甲基溴化銨(Cetyltrimethylammonium Bromide, CTAB )法對樣本的DNA進行提取, 即將適量的樣品加入到CTAB裂解液中, 然后經酚-氯仿-異戊醇抽提除去蛋白質、多糖等雜質, 再加入乙醇沉淀分離出核酸[10]。用2%的瓊脂糖凝膠對DNA的純度和濃度進行檢測, 對于質量合格的DNA, 使用M220聚焦超聲儀(Covaris Inc., Woburn,MA, USA)將DNA破碎成大約300 bp的片段, 以建立文庫。在檢驗文庫質量后, 所有樣品均基于MGISEQ-T1進行測序。
對測序所得的原始序列用fastQC(v0.11.9)進行質量評估, 并移除低質量堿基, 得到測序質量較好的Reads(Clean data), 將經過質控的Reads, 通過BLASTN比對至SILVA數據庫[11], 設置e值小于1e-5, 篩選出符合要求的Reads, 使用Megahit (v1.1.3)將這些Reads組裝到一起得到全部樣本的 rDNA contigs。用nhmmer判定組裝得到的 rDNA contigs中屬于18S rDNA的部分并進行截取, 經97%相似度聚類后得到最終18S contigs用于物種鑒定。使用Bwa(0.7.17-r1188)將每個樣品的 Reads分別比對至18S contigs上, 得到比對結果文件, 并利用Featurecounts(v1.6.0)進行統計, 然后采用RPKM(Reads Per Kilobase per Million mapped reads)的方式進行標準化得到OTU(Operational Taxonomic Units)豐度表。
1.4 數據分析
本研究主要對eDNA-PFU法和傳統PFU法在物種鑒定的全面性和準確性等方面進行了比較, 包括兩種方法在不同天數所監測到的種數和具體物種組成等數據的比較。
2 結果與討論
2.1 eDNA-PFU法在物種鑒定多樣性方面的特征
經過持續30d共11次的樣品采集, 通過顯微鏡活體觀察分別從PFU塊和原位水樣檢測到153種(11門)和79種(9門)原生動物; 通過eDNA宏基因組測序法分別從PFU塊和原位水樣中獲得473個OTU(14門)和440個OTU(14門)的原生動物序列。eDNA-PFU法鑒定到原生動物的物種數遠遠高于傳統PFU法及原位水環境樣品中所檢出的數目, 監測到的多樣性更高。eDNA鑒定出的物種比傳統方法高這么多倍的原因主要是因為前者可以鑒定出許多豐度相對較低的物種及在形態上很難辨別的物種。
基于累積所檢出的原生動物進行比較分析, 發現eDNA-PFU法逐次累加檢測物種數目均顯著高于PFU法和水樣eDNA檢測方法, 通過可重復雙因素方差分析發現, eDNA-PFU法所檢測到的原生動物數目與PFU法和單次eDNA-水樣檢測到的物種數目均有顯著差異, 并且eDNA-PFU法檢測穩定性更高, 僅在前兩次采樣時存在部分偏差, 之后數次采樣偏差均較小(圖 2)。此外, eDNA-PFU法較傳統PFU法達到檢測飽和的速度更快, 第7天即檢測到原生動物總物種數的98.19%, 而傳統的PFU法僅可達到60.78%(圖 3)。
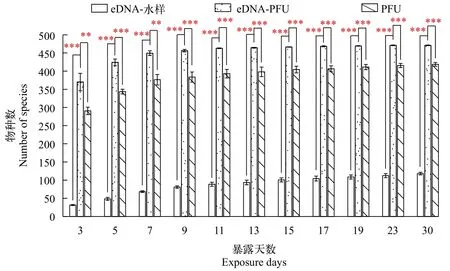
圖2 eDNA-PFU法與水樣eDNA法和傳統PFU法累積采集原生動物物種數比較分析Fig. 2 Comparative analysis of the number of protozoan species collected by eDNA-PFU and water eDNA and traditional PFU methods
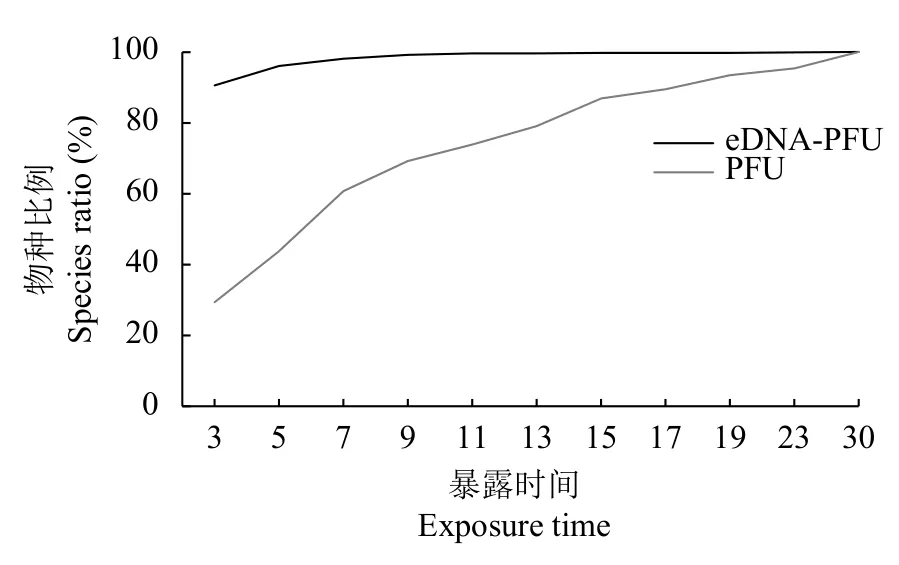
圖3 eDNA-PFU法和傳統PFU法累積采集原生動物物種數所占比例Fig. 3 The proportion of the number of protozoa species collected by eDNA-PFU and traditional PFU method
2.2 eDNA-PFU法在物種鑒定準確性方面的特征
將eDNA-PFU法和傳統PFU法所檢出的原生動物進行比較(表 1), 發現eDNA-PFU法鑒定到種級階元的有64.4%, 到屬級階元的有20.0%, 鑒定到科及以上階元的有15.6%; PFU法鑒定到種級階元的有62.4%, 到屬級階元的有34.8%, 鑒定到科及以上階元的有2.8%。隨后對兩種方法所鑒定的原生動物在各分類階元進行比較(圖 4), 發現在門水平100%覆蓋, 綱、目和科水平的覆蓋程度較好, 均大于70%,傳統PFU法所檢出的原生動物大部分包含在eDNAPFU法檢出的原生動物中, 且在屬水平的覆蓋度也較高, 可達到45%。但值得注意的是兩種方法在種類水平上匹配度很低, 鏡檢確定的70個種類, 僅有5種被eDNA監測到, 僅6%鑒定為同一物種(圖 4)。我們分析一方面可能是由于傳統PFU法是根據活體特征對原生動物進行物種鑒定, 而原生動物個體微小, 運動較快, 因此很難鑒定到種, 其種水平上的鑒定結果很難保證準確性, 從而造成兩種方法在種水平的鑒定結果偏差較大; 另一方面由于原生動物的多樣性極為豐富, 很多物種還沒有可以使用的分子序列信息, 從而影響了eDNA-PFU法在物種鑒定中的準確性。此外, 本研究加入內參(嗜熱四膜蟲細胞)作為鑒定參照, 發現eDNA-PFU法可以鑒定到種, 而傳統PFU法僅可鑒定到四膜蟲屬。因此, 整體而言, eDNA-PFU法與傳統PFU法比, 覆蓋度和靈敏度均更高。
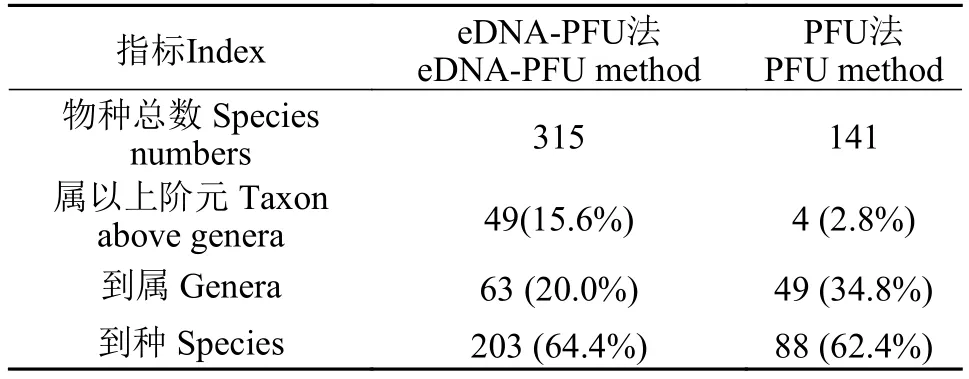
表1 eDNA-PFU法和傳統PFU法鑒定原生動物的精準度比較Tab. 1 Comparison of accuracy in identifying protozoa by eDNA-PFU and traditional PFU methods
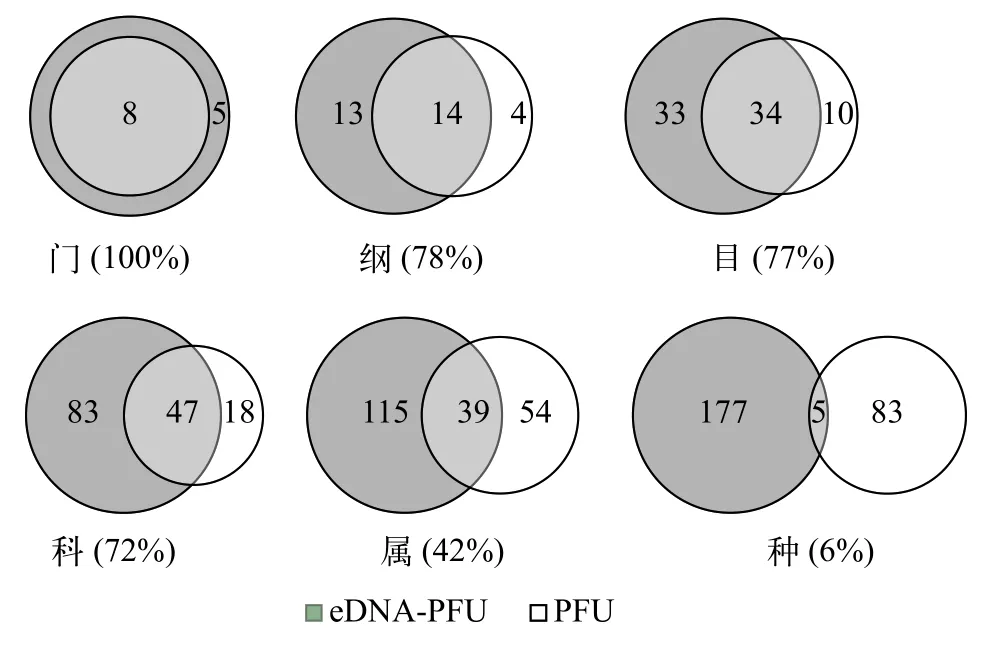
圖4 eDNA-PFU法和傳統PFU法所檢出的原生動物各階元比較圖Fig. 4 Comparison of each category of protozoa detected by eDNA-PFU and conventional PFU methods
3 結論與展望
本研究提出利用eDNA-PFU法對水體中的微型生物群落(藻類和原生動物)進行監測, 在東湖水體中對該方法監測微型生物群落的有效性進行了評估。結果表明eDNA-PFU法較傳統PFU法覆蓋度更高, 可以全面地揭示水體中微型生物群落結構特征。此外, 本文發現eDNA-PFU法鑒定到原生動物的物種數遠遠高于傳統PFU法以及原位水環境樣品中所檢出的數目, 檢測到的物種多樣性更高, 且與傳統PFU法相比, 基于eDNA-PFU法的檢測在科屬水平上具有較高的覆蓋度和準確性。因此, 我們的結果表明eDNA-PFU法可以全面、真實、準確地揭示水體中微型生物群落結構特征。
然而, eDNA-PFU法在水質監測的應用中仍有許多亟待解決和完善的地方。首先, 由于原生動物和藻類等微型生物的多樣性極為豐富, 其分子序列信息也十分多樣, 為了更好地對eDNA-PFU加以應用, 后續需要進一步豐富和完善本土原生動物等物種的DNA條形碼數據庫。再者, 基于eDNA-PFU法獲得的物種和豐度信息較之傳統PFU法在數據的呈現形式上不一樣, 例如傳統PFU法可以提供不同類群的絕對豐度(由于依據活體樣品對原生動物的定量難度非常大, 因此在實際應用中常常很難獲得這方面的數據), 而eDNA-PFU法只能提供相對豐度, 因此后續需要開展系統研究以制定和規范依據eDNA-PFU法的水質監測參數。最后, 由于原生動物和藻類等微型生物的分布與季節、地理位置, 及局部小環境特征都有非常緊密的關系, 本文僅用了1個調查點的數據, 雖然重復較多, 但仍不具備代表性。后續將增加調查樣點數, 涵蓋多個浮游生物多樣性高、中、低梯度的樣點。通過比較不同生物多樣性背景的水體監測結果的異同, 以對eDNAPFU法和傳統PFU方法做進一步的比較和評價, 并全面分析基于eDNA-PFU獲得的微型浮游生物種類數、指示種類和功能類群等微型生物群落的結構和功能參數對生態考核的支撐作用, 從而為湖泊河流水庫等水體的生態考核提供新一代的生物監測標準。總之, 通過進一步完善, eDNA-PFU法有望成為水體微型生物群落監測的新一代監測方法。
致謝:
感謝中國科學院水生生物研究所何舜平研究員對于eDNA評估水質方法的提出給予的指導, 感謝劉國祥研究員在藻類的eDNA監測評估中給予的建議, 感謝徐軍研究員在長江中下游水生態系統結構和功能方面提供的信息, 感謝水生生物數據分析管理平臺的曾宏輝高級工程師在水生生物DNA條形碼庫構建中給予的支持。

