吉爾吉斯斯坦傳統酸馬奶中乳酸菌的分離鑒定
叢琳,袁婧,劉文俊,孫志宏
(內蒙古農業大學 乳品生物技術與工程教育部重點實驗室 呼和浩特010010)
0 引言
酸馬奶在吉爾吉斯斯坦又稱為“Kymyz”,被當地人譽為國飲,通常用來招待遠方貴客。傳統酸馬奶以木桶、瓷缸或塑料桶為容器,新鮮馬奶為原料,前一天的酸馬奶作為發酵劑,通過攪打,低溫自然發酵而成。是一種主要產物為乳酸,酒精含量低的發酵乳飲品。相較于鮮馬乳,酸馬奶有更高的營養價值和保健效果。本研究采用純培養方法對吉爾吉斯斯坦地區酸馬奶中的乳酸菌進行分離純化,應用16S rRNA基因序列分析方法對分離株進行鑒定,結合PacBio SMRT三代測序技術對酸馬奶樣品中的細菌多樣性進行分析,以更好了解該地區自然發酵乳制品中乳酸菌的組成與種類,為乳酸菌的研究和開發利用提供菌株,為吉爾吉斯斯坦乳制品中微生物多樣性研究提供基礎數據。
1 材料與方法
1.1 樣品的采集
本研究10份酸馬奶樣品采集自吉爾吉斯斯坦境內,位于比什凱克、阿倫和奧什3個不同地區,所有樣品由當地牧民采用傳統自然發酵的方法家庭手工制作。現場將樣品混勻后采集,一式兩份。采集的樣品一部分置于裝有0.5 g滅菌緩沖劑(M淀粉:MCaCO3=50∶1)的2 mL無菌凍存管中,另一部分裝入無菌離心管中,加入RNA/DNA樣品保護液并充分混勻、標號。樣品運輸全程保持低溫(2~8℃),到達實驗室后立即進行乳酸菌分離培養實驗,宏基因組測序樣品于-80℃保存備用。
1.2 試劑及儀器
MRS培養基,青島高科技工業園海博生物技術有限公司;Agar Powder,北京康倍斯(chembase)科技有限公司);TIANamp Bacteria DNA Kit,天根生化科技(北京)有限公司;DneasyPowerFood Kit,德國QIAGEN公司;KAPA HiFi HotStartReadyMix PCR Kit,美國KAPA公司。
微量紫外分光光度計,美國Nanodrop公司;PCR儀,美國Life Technologies公司;電泳儀,北京六一儀器廠;凝膠成像儀,美國UVP公司;三代測序儀PacBio RS II,美國Pacific Biosciences公司。
1.3 乳酸菌分離與鑒定
自然發酵乳制品中乳酸菌計數采用傾注培養法[1]。將樣品充分混勻后用10倍梯度稀釋法[2]選取合適的濃度梯度(10-5,10-6,10-7)對自然發酵乳制品進行梯度稀釋。吸取1 mL樣品稀釋液于未凝固的MRS固體培養基中搖勻,待培養基徹底干透后置于37℃恒溫培養箱需氧培養48~72 h。同時,吸取與活菌計數相同稀釋倍數的樣品稀釋液涂布于MRS固體培養基上,待菌落形成后,挑取形態特征各異的單個菌落在相應的固體培養基上采用平板劃線法進行2~3次的劃線純化[3]。把鏡檢結果為單一菌落形態的菌落進行傳代培養,將經革蘭氏染色后呈陽性、過氧化氫酶試驗顯陰性、菌落無芽孢且形態均一的純培養物定為疑似乳酸菌,加入適量脫脂乳保護劑進行保藏并編號。
乳酸菌分離株DNA提取使用TIANamp Bacteria DNA Kit試劑盒,按照說明書的步驟嚴格操作。取得DNA后,用ND-1000紫外分光光度計檢測提取DNA的純度。將符合要求的DNA作為擴增模板進行擴增體積為50μL的PCR反應,具體包括模板DNA(100~300 ng/mL)2μL,細菌通用引物正向引物27F(5′-AGAGTTTGATCMTGGCTCAG-3′)1.5μL,反向引物1492R(5′-ACCTTGTTACGACTT-3′)1.5μL,引物濃度均為10μmol/L,10×PCR Buffer(含Mg2+離子)5μL,Taq DNA polymerase 0.5μL,dNTP 4μL,ddH2O 35.5μL。擴增條件為94℃預變性5 min,94℃變性1 min,58℃退火1 min,72℃延伸2 min,30個循環,72℃末端延伸10 min[4]。
上述PCR產物需在1.0%的瓊脂糖凝膠電泳中檢測,將滿足測序要求的PCR擴增產物在低溫條件下送往上海美吉生物公司進行純化和雙向測序。
1.4 同源性分析及系統發育樹的構建
運用DNAStar 5.01軟件中的SeqMan拼接經上海美吉生物公司測序的16S rRNA序列,將拼接好的序列在NCBI(National Center for Biotechnology Infornation)上進行BLAST(Basic Local Alignment Search Tool,https://blast.ncbi.nlm.nih.gov/Blast.cgi)同源性比對,將同源性大于99%且基因片段吻合的DNA序列認定為與模式菌株屬于同一菌種,運用MEGA 7.0軟件采用鄰接法(Neighbor-Joining)構建菌株序列進化樹,比較它們之間的親緣關系進化距離。
1.5 酸馬奶樣品中宏基因組DNA的提取
使用DneasyPowerFood Kit,2 mL tubes(100)抽提樣品宏基因組DNA,其步驟參照試劑盒中的說明書[5]。用ND-1000紫外分光光度計和1.0%瓊脂糖凝膠電泳檢驗提取DNA的濃度、純度與完整度,將DNA純度OD260/280處于1.8~2.0之間,濃度大于20 ng/μL,且片段化程度小的DNA置于-20℃低溫冰箱保存備用。
1.6 細菌16S rRNA基因序列全長擴增
將提取好的細菌基因DNA作為模板,通過16SrRNA基因序列通用引物(正向引物27F和反向引物1492R)進行擴增,通過在引物5′端加不同的核苷酸標簽(Barcode)來區分各樣品。PCR擴增所需的試劑盒為KAPA HiFi HotStart Ready Mix PCR Kit,擴增反應體系為50μL:10μmol/L正反向引物各1.5μL,模板DNA 1.5μL,模板濃度需小于100 ng/μL,KAPA Mix 25.0μL,最后用ddH2O補足體系至50μL。擴增條件為:95℃預變性3 min,98℃變性20 s,60℃退火15 s,72℃延伸30 s,30個循環,72℃末端延伸2 min[6]。
1.7 PacBio SMRT測序及數據分析
將經1.0%的瓊脂糖凝膠電泳檢測后符合要求的PCR產物純化后混樣,用SMRTbell Template Prep Kit 1.0試劑盒對混樣DNA進行DNA損傷修復和DNA雙鏈部分缺失堿基修復。修復純化后的DNA加接頭,以Qubit 2.0熒光計檢測制備文庫的濃度,再用PacBio RS II三代測序儀對構建完成的DNA文庫進行上機測序。對得到的原始數據進行質量控制,具體的條件為:測序的最小循環次數為5;測序的最小預測精確度為90%;測序時DNA插入片段應屬于1 400~1 800 nt之間。將滿足要求的高質量序列根據每個樣品的Barcode信息進行去除Barcode處理[7],用QIIME平臺對符合要求的高質量序列進行微生物組分析,通過柱形圖、折線圖、散點圖展現不同水平上不同自然發酵酸馬奶中的的菌群結構,用Mann-Whitny/Kruskal-Wallis檢驗統計吉爾吉斯斯坦不同地區酸馬奶菌落結構的差異性,應用R 3.5.0和Origin 2019等軟件對QIIME分析的微生物組數據進行可視化處理。
2 結果與分析
2.1 自然發酵酸馬奶中乳酸菌總數計數與分離
本研究中10份自然發酵酸馬奶通過純培養后的乳酸菌總數在6.80×105~3.04×109mL-1范圍之間,具體結果如表1所示。

表1 吉爾吉斯斯坦酸馬奶中乳酸菌計數結果 mL-1
乳酸菌總數能夠反映發酵乳制品的新鮮程度,展現發酵乳制品在運輸、儲藏等過程中菌株的生長狀態。孫天松等[1]曾研究哈薩克斯坦的不同地區傳統酸馬奶中的乳酸菌多樣性,揭示該地區酸馬奶樣品乳酸菌總數均大于1.00×106mL-1。由此可以推測吉爾吉斯斯坦地區自然發酵酸馬奶的新鮮程度較好,乳酸菌的存活能力較高。
本研究中10份酸馬奶樣品共分離得到109株菌,經革蘭氏染色呈陽性、過氧化氫酶試驗顯陰性結果篩選后得到疑似乳酸菌共91株。通過觀察菌落形態,其菌落邊緣呈整齊或不平,菌落表面呈粗糙或光滑,菌落中央有凸起或凹陷,菌落顏色呈白色、乳黃色或接近于透明。在顯微鏡下觀察發現,經革蘭氏染色后的疑似乳酸菌分離菌株形態呈均勻排列的桿狀、球狀或橢球狀,排列呈對、呈鏈、成片或呈環狀。
2.2 乳酸菌分離菌株鑒定結果
經同源性序列比對(BLAST)結果顯示,91株分離株均為乳酸菌,將其16S rRNA基因序列與模式菌株的16S rRNA基因序列利用MEGA7.0軟件繪制乳酸菌序列進化樹。通過圖1(比例尺為0.01)的系統發育樹分析可知,分離到的91株乳酸菌分離株為2個屬11個種,包括植物乳桿菌(Lb.plantarum)、馬乳酒樣乳桿菌(Lb.kefiranofaciens)、副干酪乳桿菌(Lb.paracasei)、干酪乳桿菌(Lb.casei)、哈爾濱乳桿菌(Lb.harbinensis)、發酵乳桿菌(Lb.fermentum),德氏乳桿菌(Lb.delbrueckii)和瑞士乳桿菌(Lb.helveticus)等。

圖1 部分乳酸菌分離株16S rRNA基因序列的系統發育樹
經純培養從吉爾吉斯斯坦地區的10份酸馬奶樣品中分離共91株乳酸菌,其中副干酪乳桿菌為優勢菌種,占所有分離株的29.67%,除KG12,KG15和KG26樣品外,其余樣品均有分離,具體結果如表2所示。
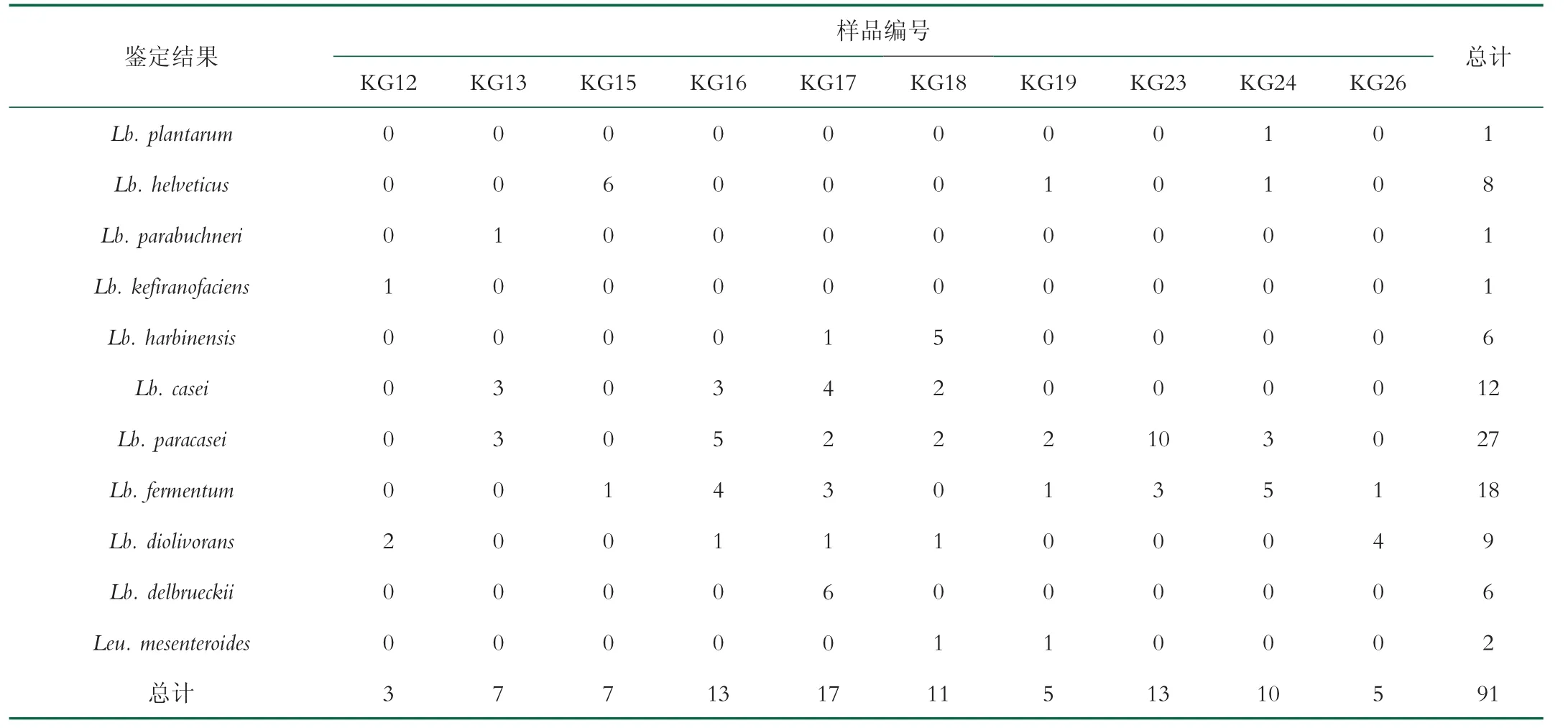
表2 吉爾吉斯斯坦酸馬奶中乳酸菌分離鑒定結果
2.3 吉爾吉斯斯坦自然發酵酸馬奶的細菌多樣性
2.3.1 α多樣性
經過SMRT三代測序,10份吉爾吉斯斯坦自然發酵酸馬奶共獲得32 553條高質量DNA序列,平均每個發酵乳樣品獲得3 255條DNA序列。其中KG12樣品獲得的代表性OTUs最少,為146條,KG17樣品獲得的代表性OTUs最多,為488條。吉爾吉斯斯坦比什凱克、阿倫和奧什3個地區酸馬奶的Chao1指數分別為(445.94±207.47)、(294.38±0.00)和(308.29±53.76);發現物種數分別為(206.50±163.00)、(163.00±0.00)和(144.67±31.64);香農指數分別為(4.22±1.39)、(3.11±0.00)、(5.08±0.25);辛普森指數分別為(0.74±0.19)、(0.58±0.00)、(0.93±0.02)。通過比較發現,奧什地區的酸馬奶細菌群落多樣性和種數較其他兩個地區更為豐富,具體如表3所示。
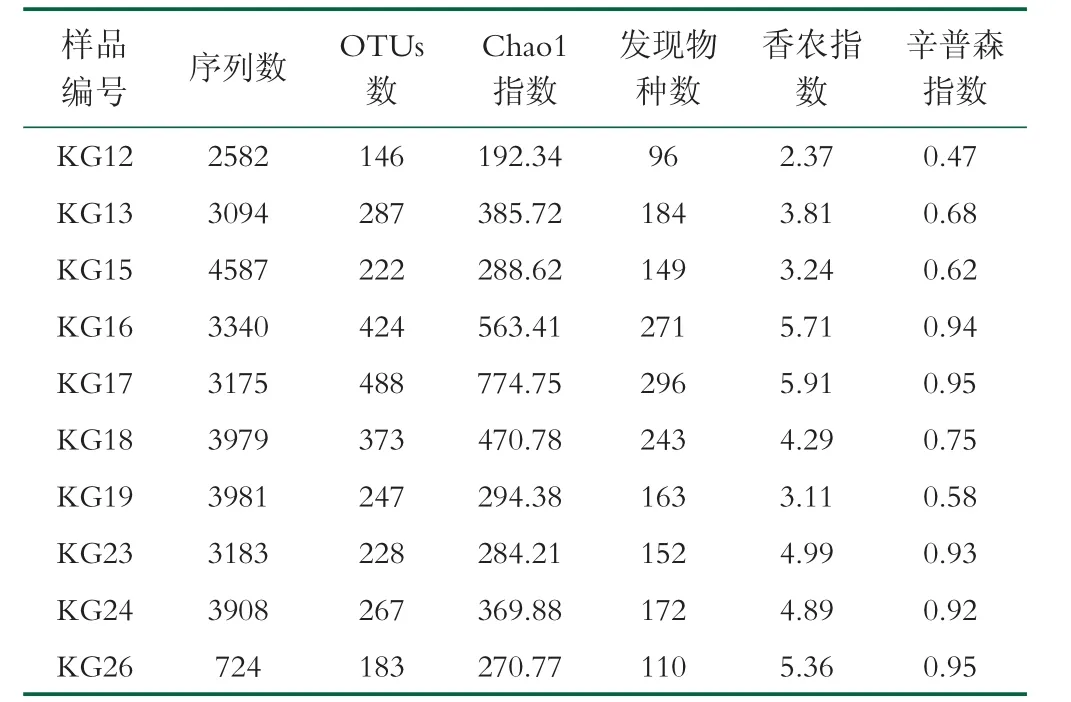
表3 吉爾吉斯斯坦酸馬奶的測序序列信息和α多樣性指數
2.3.2 β多樣性
分析吉爾吉斯斯坦不同地區酸馬奶的菌群多樣性變化,通過PC1(第一主成分)和PC2(第二主成分)進行主坐標分析,基于加權距離的主坐標分析PC1坐標和PC2坐標的貢獻率分別為77.5%和13.17%,基于非加權距離的主坐標分析PC1坐標和PC2坐標的貢獻率分別為29.92%和14.19%。從圖中可以看出,采集于吉爾吉斯斯坦不同地區的酸馬奶制品有明顯的分離狀態,同時存在一定的交疊,如圖2所示。
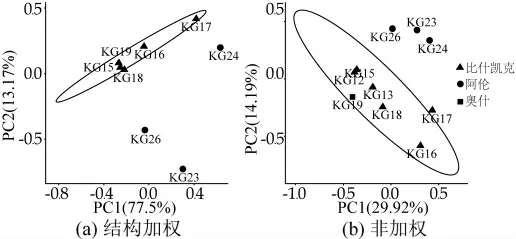
圖2 不同地區酸馬奶制品中菌群主坐標分析
2.3.3 細菌組成
經檢測,吉爾吉斯斯坦10份自然發酵酸馬奶細菌在細菌門水平的組成主要分為4個菌門,除厚壁菌門外,還有變形菌門、擬桿菌和極少量的藍藻細菌。其中,厚壁菌門是吉爾吉斯斯坦自然發酵酸馬奶制品中的主要菌門,相對占比的均值為98.38%,是絕對優勢菌門。除絕對優勢菌門厚壁菌門外,不同地區不同樣本的酸馬奶制品中細菌門的相對占比各不相同,不同樣本細菌門水平組成如表4所示。

表4 不同酸馬奶中細菌門水平組成 %
在細菌屬水平,吉爾吉斯斯坦10份自然發酵酸馬奶樣品中共檢測到20個細菌屬。主要由乳桿菌屬、乳球菌屬這2個乳酸菌屬構成。其中,KG12酸馬奶樣品中有相對占比最高的乳桿菌屬,達99.92%;KG23酸馬奶樣品中有相對占比最高的乳球菌屬,為12.97%。通過進一步分析可知,吉爾吉斯斯坦自然發酵酸馬奶中還有一些相對占比小于1%的細菌菌屬,如克呂沃爾菌屬、沙雷氏菌屬、土芽孢桿菌屬、魏斯特菌屬等,研究發現不同地區酸馬奶細菌屬種類及相對占比不同,不同地區酸馬奶制品細菌屬水平組成如圖3(a)所示。

圖3 不同酸馬奶細菌組成
在細菌種水平,10份樣品共檢測到30個細菌菌種,在所有樣品中相對占比大于1%的除了有瑞士乳桿菌(48.04%)和哈爾濱乳桿菌(11.68%)外,還有類谷糠乳桿菌(6.11%),德氏乳桿菌(5.69%),馬乳酒樣乳桿菌(4.52%),發酵乳桿菌(3.79%),食二酸乳桿菌(3.21%),橋乳桿菌(3.01%),乳酸乳球菌(2.06%),副干酪乳桿菌(1.20%),牛糞乳桿菌(1.14%)等。除含量較多的菌種外,吉爾吉斯斯坦自然發酵酸馬奶樣品中還檢測出一些低豐度(相對占比小于1%)的菌種,不同發酵乳制品細菌種水平組成見圖3(b)。
測序中發現的細菌菌種包含了純培養中獲得的所有分離菌株種類,其中瑞士乳桿菌為絕對優勢菌株,這與在純培養中優勢菌株為副干酪乳桿菌的結果并不相同。
2.3.4 核心微生物分析
核心微生物通常表示每個OTU在樣品中出現的頻率[8],通過核心微生物組成有助于分析不同發酵乳制品微生物群落的特征。通過分析可知,吉爾吉斯斯坦酸馬奶樣品種水平的核心微生物為瑞士乳桿菌,其次為哈爾濱乳桿菌、類谷糠乳桿菌、德氏乳桿菌、馬乳酒樣乳桿菌、發酵乳桿菌、食二酸乳桿菌、橋乳桿菌、乳酸乳球菌、副干酪乳桿菌、牛糞乳桿菌等,酸馬奶樣品的核心微生物組成如圖4所示。
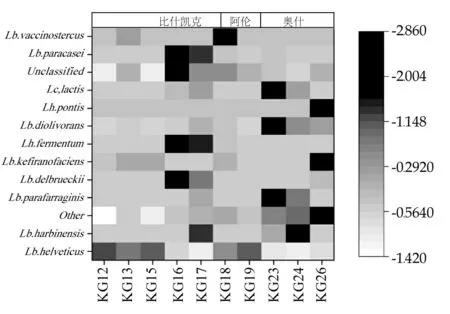
圖4 酸馬奶中核心微生物組成
2.3.5 OTU水平的菌群分析
通過Venn圖對吉爾吉斯斯坦不同地區采集的發酵酸馬奶制品OTU(代表性序列)的組成分析,根據不同OTU在酸馬奶制品中的豐度計算出不同樣品間的共有或特有OTU[9]。由圖5可知,比什凱克地區采集的酸馬奶有211個特有OTU,奧什地區有132個特有OTU,阿倫地區有125個特有OTU。比什凱克,奧什和阿倫3個地區采集的酸馬奶的共有OTU有30個,占總OTU的4.47%。其中,奧什和阿倫地區的共有OTU為2個,只占OTU總數的0.3%。結果表明不同地區的發酵乳制品之間菌群的共性各有不同,其中奧什地區與阿倫地區的差異最大。

圖5 不同采樣地區酸馬奶中的特有OTU分析
3 討論
通過傳統純培養方法從吉爾吉斯斯坦10份自然發酵酸馬奶中分離得到91株乳酸菌菌株,屬于2個屬,11個種。樣品的優勢菌種為副干酪乳桿菌,占所有酸馬奶樣品分離株的29.67%,這與喬健敏[10]等人研究的內蒙古地區自然發酵酸馬奶中乳酸菌組成結果符合。通過PacBio SMRT測序方法分析酸馬奶后,發現樣品中的細菌隸屬于4個門,20個屬,30個種。優勢細菌屬為乳桿菌(93.98%),優勢細菌種為瑞士乳桿菌(48.04%)和哈爾濱乳桿菌(11.68%)。與劉文俊等[11]研究的蒙古國的傳統酸馬奶中的乳酸菌組成結果類似。孫志宏等[12]通過高通量測序法研究tarag(自然發酵乳制品)中的細菌和真菌多樣性、種群結構等,發現目前研究中使用的瑞士乳桿菌分離物中存在3個亞群,且大多數來自特定生態起源的瑞士乳桿菌菌株都有特定的種群結構,這可能是酸馬奶樣品中的優勢菌種為瑞士乳桿菌的重要原因之一[13]。吉爾吉斯斯坦境內酸馬奶的細菌組成在3個地區之間也存在一定差異:瑞士乳桿菌是比什凱克與阿倫地區的優勢菌種,奧什地區的優勢菌種為哈爾濱乳桿菌。在3個地區中,奧什地區的細菌多樣性最高,阿倫地區的酸馬奶樣品中菌群豐富度最低。
4 結論
本文通過純培養和非培養技術研究了吉爾吉斯斯坦10份自然發酵酸馬奶中的乳酸菌與細菌組成,共分離得到91株乳酸菌。通過PacBio SMRT分析后,發現該地區優勢細菌種為瑞士乳桿菌(48.04%),酸馬奶中的細菌組成、細菌數量及優勢菌種存在地域差異。本研究保存的乳酸菌資源和報道的自然發酵酸馬奶細菌圖譜,有助于后續幫助當地傳統發酵乳的發展。此外,我們需要關注傳統發酵乳的發酵環境和工藝,避免一些條件致病菌帶來的食品安全問題。

