CsBLH7與CsKNAT4蛋白互作調節下胚軸細胞的伸長

摘要:CsBLH7與CsKNAT4分別屬于BELL類蛋白與KNOX類蛋白,可能與植物器官形態建成密切相關,但目前幾乎沒有關于CsBLH7與CsKNAT4蛋白互作的深入研究報道。采用酵母雙雜交初步確定CsBLH7與CsKNAT4能夠在細胞體內互作,形成BLH7-KNAT4蛋白復合物。雙分子熒光互補(BiFC)進一步確定了這種互作關系。原生質體瞬時轉染試驗結果表明,CsBLH7與CsKNAT4分別為轉錄抑制因子與轉錄激活因子。同時,針對35S:CsBLH7、35S:CsKNAT4材料的形態分析結果表明,下胚軸細胞長度分別縮短與伸長,這與它們的轉錄活性相適應。再者,35S:CsBLH7、35S:CsKNAT4遺傳材料的赤霉素合成酶基因分別下調與上調。研究結果表明,BLH7-KNAT4可能是通過調節赤霉素合成來調控下胚軸細胞長度,對揭示CsBLH7和CsKNAT4調控植物形態建成分子機理具有重要意義。
關鍵詞:CsBLH7;CsKNAT4;蛋白互作;BiFC;下胚軸細胞;轉錄活性;植物形態建成
中圖分類號: S563.301? 文獻標志碼: A
文章編號:1002-1302(2022)06-0041-04
收稿日期:2021-06-11
基金項目:黑龍江省自然科學基金(編號:LH2020C097);黑龍江省農業科學院國自然培育項目(編號:2019JJPY003);黑龍江省麻類科技創新基金(編號:MLCX-20);特色經濟作物綠色種植技術示范推廣項目(編號:KYBG-05WDL-2020001)。
作者簡介:張利國(1978—),男,黑龍江哈爾濱人,博士,助理研究員,主要從事麻類分子育種研究。E-mail:zlg86@aliyun.com。
BELL類蛋白幾乎存在于所有植物當中[1],大麻中目前發現有13個BELL蛋白[2]。現有研究揭示,BLH1能夠調節植株花基形態,在生殖生長與營養生長的轉換節點可能發揮重要負調控功能;在擬南芥中BLH3和BLH6的轉錄活性相反,同時還可能參與調節植株葉片的形態與花期[3]。
KNOX家族作為TALE蛋白的一個亞類,也能夠從多方面參與對植物形態建成的調控。不同物種的同源BELL和KNOX蛋白可能存在不同的蛋白互作關系[4-5]。研究的前期工作基礎初步表明,CsBLH7可能與CsKNOXs家族部分成員之間存在蛋白互作關系。對比大麻的全基因組測序數據,在大麻中發現6個KNOX蛋白和13個BELL蛋白,因此確定它們之間的互作關系、調節方式,對解析大麻器官形態發生的機制是十分必要的,將是揭示CsBLH7調控基因表達分子機理的基礎與關鍵。
1 材料與方法
1.1 試驗材料
本研究中植物材料為工業大麻新品種龍大麻4號,由黑龍江省農業科學院經濟作物研究所選育。龍大麻4號的THC (四氫大麻酚)含量低于千分之一,為高產高纖型工業大麻品種。
1.2 酵母雙雜交相關質粒構建與測試蛋白-蛋白互作
采用PCR方法通過對大麻cDNA進行克隆獲取BLH7和KNAT4的開放讀碼框片段。在酵母細胞中,轉入BLH7的同源異型結構域蛋白片段與 Bell-like 片段,在pvgDT3中插入BLH7與BLH10的cDNA。在缺乏Trp及缺乏Leu的培養基中篩選酵母細胞,以測試其蛋白互作關系,在缺少Ade與Leu,以及無Trp、無His的培養基中培養篩選后的株系。使用陰性對照Pgatd7SV(40)和Pgbkt (7)Lam,陽性對照是Pgatd7SV (40)與Pgbkt (7)p53。
1.3 采用雙分子熒光互補技術(BiFC)測試蛋白-蛋白互作
通過35S啟動子的調控,黃色熒光蛋白的兩端[4]分別連接BLH蛋白與KNOX蛋白形成融合蛋白,即C-KNAT4 與N-BLH7。擴增完成后,在pSAT6-nEYFP-N1中亞克隆KNAT4-YFPC、KNAT3-YFPC,在pSAT6-cEYFPN1中亞克隆BLH7-YFPN、BLH10-YFPN,參考Yoo等聚乙二醇轉染擬南芥原生質體的試驗方法[4]展開試驗,于Leica 2PS SSC 激光共聚焦顯微鏡下觀察熒光成像情況。
1.4 原生質體瞬時轉染試驗
在pUC19質粒中構建35S:GD-OIPs,用于質粒提取和擴增。反式強激活作用因子Ld-Vp16,以及報告基因2 LexA-2 Gal4:Gus和2Gal4:Gus也在本試驗中應用。本試驗采用無內毒素轉染級質粒試劑盒(Abcam)進行報告質粒和效應因子的分析,至少重復進行3次生物學試驗和設置2次技術性重復,數據分析處理采用t檢驗。研究采用Gal4 DNA-binding domain和LexA DNA-binding domain,對應GD-OIPs與VP16,其可靠性已被反復證實,例如AUX/IAA蛋白[6]、OFP4蛋白[7]。
2 結果與分析
2.1 酵母雙雜交測試CsBLH7與CsKNAT4之間的蛋白互作
通過酵母雙雜交試驗測試CsBLH7與KNAT4在體內的相互作用,CsBLH7與轉錄蛋白因子的結合域連接組成融合蛋白,KNAT4與轉錄激活域形成融合蛋白。為排除目標蛋白可能的自激活活性,測試結合域蛋白-Oips與激活域蛋白-Empty蛋白互作。試驗結果表明,CsBLH7、KNAT4均無自激活活性,CsBLH7與CsKNAT4在體內存在蛋白互作關系,形成BLH7-KNAT4蛋白復合物。CsBLH7與CsKNAT4酵母雙雜交(圖1)。在缺乏Leu與缺乏Trp的培養基質中,Yeast-cell首先被選擇,在缺Leu、缺Trp、無His與無Ade的培養基質中培養5~6 d,完成BLH-KNOX蛋白-蛋白互作測試。
2.2 雙分子熒光互補(BtFC)研究BLH7與KNAT4蛋白互作
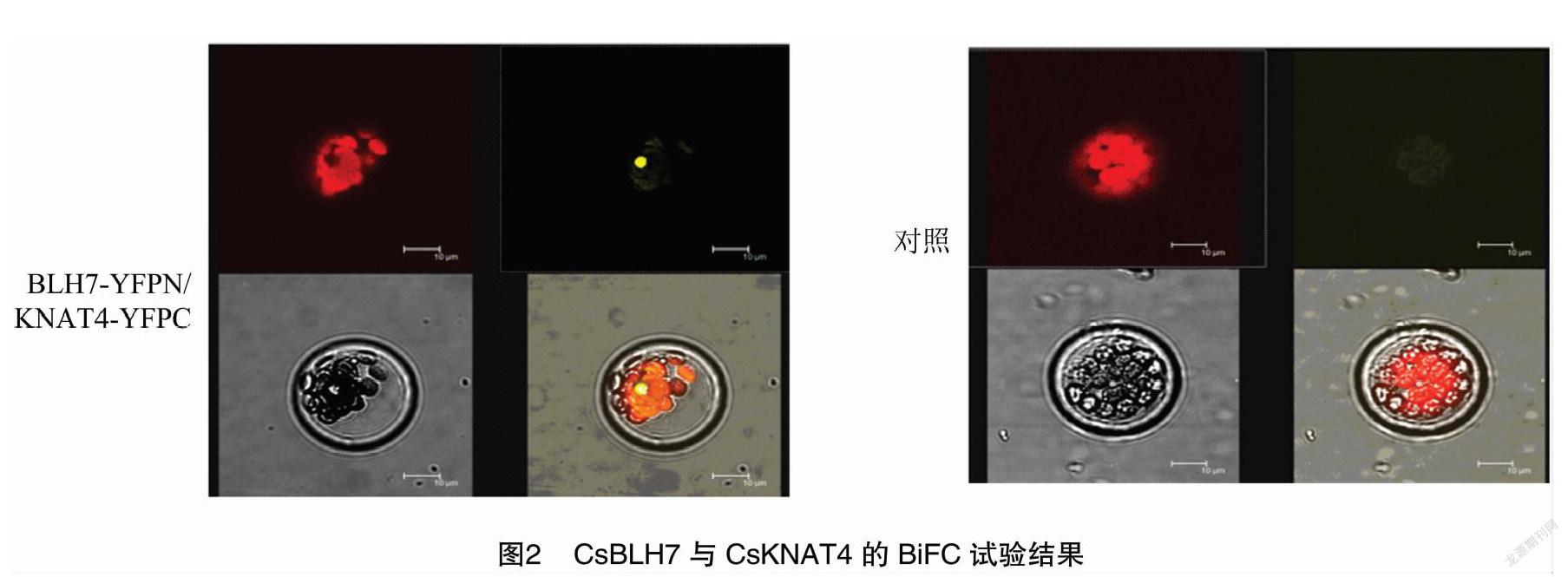
由于酵母雙雜交技術上的局限性,為進一步證實BLH7與KNAT4在細胞體內的蛋白互作,采用雙分子熒光互補方法進行測試。試驗采用擬南芥葉肉原生質體,共轉染YFPC-CsKNAT4與YFPN-CsBLH7,試驗體系采用RACK1-C-EYFP作為陰性對照。BiFC試驗結果顯示,BLH7與KNAT4蛋白互作發生在細胞核中(圖2)。
2.3 BLH7與KNAT4轉錄活性測試
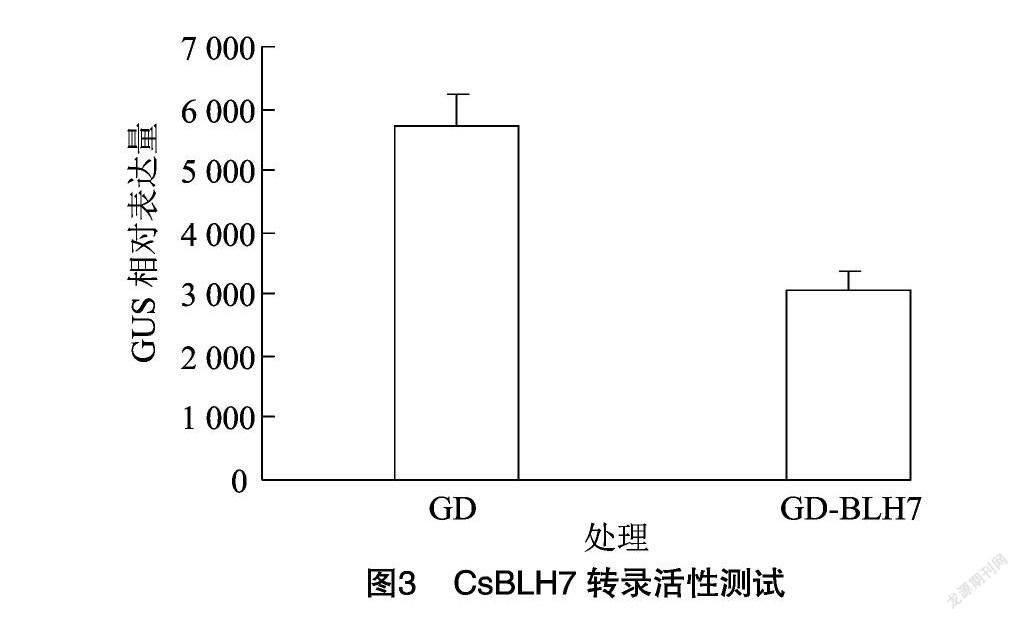
從原生質體瞬時轉染效應因子相對表達量可知,圖中GD-BLH7的GUS相對表達量明顯降低(圖3),GD-KNAT4的GUS相對表達量明顯上調(圖3、圖4),由此可知CsBLH7為轉錄抑制因子,CsKNAT4為激活因子,且CsBLH7能夠與CsKNAT4互作,并能夠抑制CsKNAT4的轉錄激活活性(圖5)。
2.4 CsBLH7-CsKNAT4調節下胚軸細胞的伸長
在過去針對BELL與KNOX類蛋白研究的結果表明,二者能夠調節植株的形態發育,包括葉片的形狀、莖的長度、花基發育等。對比野生型(Col),35S:CsBLH7下胚軸長度與下胚軸細胞長度明顯縮短,35S:CsKNAT4下胚軸長度明顯伸長(圖6、圖7)。
在水稻、擬南芥等植物中過去關于BELL與 KNOX蛋白的研究曾報道,BELL-KNOX通過調控赤霉素合成酶基因來調節莖與細胞的伸長。為進一步探究CsBLH7-CsKNAT4調節胚軸與莖細胞長度的機制,對不同遺傳背景材料的赤霉素合成酶基因做熒光定量分析。結果表明,35S:CsBLH7中CsGA20ox1表達量均降低,35S:CsKNAT4中CsGA20ox1表達量上升(圖8)。
3 討論
研究采用酵母雙雜交初步確定,CsBLH7與
CsKNAT4能夠在細胞體內互作,形成BLH7-KNAT4蛋白復合物,雙分子熒光互補(BiFC)進一步證實了這種蛋白互作。以擬南芥原生質體為材料,為確定轉錄蛋白的作用開展瞬時轉染,通過酶標儀以GUS基因相對表達量,確定CsBLH7為轉錄抑制因子,35S:CsKNAT4為轉錄激活因子。材料35S:CsBLH7下胚軸與細胞長度降低,材料35S:CsKNAT4下胚軸與細胞長度增加。BLH7與KNAT4互作形成BLH7-KNAT4蛋白復合物,BLH7能夠抑制莖與胚軸細胞的伸長,KNAT4能夠促進莖與胚軸細胞的發育,這與BLH7作為一個轉錄抑制因子,KNAT4作為一個轉錄激活因子的遺傳特性相適應。結合不同材料CsGA20ox1的表達量,在BLH7與KNAT4蛋白互作基礎上,暗示BLH7-KNAT4可能是通過調節赤霉素合成來調控下胚軸與莖細胞長度,從而影響下胚軸的發育。
同時,在過去的報道中,OFP等其他轉錄蛋白可能與BELL-KNOX蛋白復合物發生互作來發揮調控作用,形成OFP-BELL-KNOX復合蛋白[8-9],本研究中KNAT4與BLH7的蛋白-蛋白互作,也可能并不是直接形成的,并不能排除是通過其他蛋白間接形成的互作,這需要通過體外蛋白互作試驗進一步來確定。
參考文獻:
[1]Arnaud N,Pautot V. Ring the BELL and tie the KNOX:roles for TALEs in gynoecium development[J]. Frontiers in Plant Science,2014,5:93.
[2]Kumar R,Kushalappa K,Godt D,et al. The Arabidopsis BEL1-LIKE HOMEODOMAIN proteins SAW1 and SAW2 act redundantly to regulate KNOX expression spatially in leaf margins[J]. Plant Cell,2007,19(9):2719-2735.
[3]Gomez-Mena C,Sablowski R. ARABIDOPSIS THALIANA HOMEOBOX GENE1 establishes the basal boundaries of shoot organs and controls stem growth[J]. Plant Cell,2008,20(8):2059-2072.
[4]Yoo S D,Cho Y H,Sheen J. Arabidopsis mesophyll protoplasts:a versatile cell system for transient gene expression analysis[J]. Nature Protocols,2007,2(7):1565-1572.
[5]Furumizu C,Alvarez J P,Sakakibara K,et al. Antagonistic roles for KNOX1 and KNOX2 genes in patterning the land plant body plan following an ancient gene duplication[J]. PLoS Genetics,2015,11(2):e1004980.
[6]Oh E,Zhu J Y,Bai M Y,et al. Cell elongation is regulated through a central circuit of interacting transcription factors in the Arabidopsis hypocotyl[J]. eLife,2014,3:e03031.
[7]Giacomo E D,Sestili F,Iannelli M A,et al. Characterization of KNOX genes in Medicago truncatula[J]. Plant Molecular Biology,2008,67(1/2):135-150.
[8]Zhou H B,Liu B,Weeks D P,et al. Large chromosomal deletions and heritable small genetic changes induced by CRISPR/Cas9 in rice[J]. Nucleic Acids Research,2014,42(17):10903-10914.
[9]Tzfira T,Tian G W,Lacroix B,et al. pSAT vectors:a modular series of plasmids for autofluorescent protein tagging and expression of multiple genes in plants[J]. Plant Molecular Biology,2005,57(4):503-516.

