融合邊緣增強注意力機制和U-Net網絡的醫學圖像分割
李翠云,白 靜,2,鄭 涼
融合邊緣增強注意力機制和U-Net網絡的醫學圖像分割
李翠云1,白 靜1,2,鄭 涼1
(1. 北方民族大學計算機科學與工程學院,寧夏 銀川 750021;2. 國家民委圖像圖形智能處理實驗室,寧夏 銀川 750021)
醫學圖像分割是醫學圖像處理領域中的關鍵步驟,隨著深度學習技術的逐步深入,圖像分割技術有了突飛猛進的發展。然而,在分割過程中,病灶特征的邊緣像素點劃分仍存在模糊、不準確的問題。為此,提出一種邊緣增強的注意力模塊(CEA),分別進行水平和垂直2個不同方向的特征編碼捕獲位置信息,并通過計算位置特征和輸入特征之間的偏移量加強邊緣信息。將該模塊應用基于U-Net的醫學圖像分割網絡中,可突破卷積核的空間限制,捕獲具有位置感知的跨通道信息及更加明確的邊緣輪廓信息,從而提高分割的準確性。在公開數據集Kvasir-SEG上的定量對比實驗表明,加入注意力模塊的網絡在Dice、精確度、召回率等指標上均取得了更好的結果,可有效改善醫學圖像分割效果。
醫學圖像分割;位置信息;注意力機制;邊緣輪廓;邊界差異
近年來,隨著深度學習算法在醫學圖像分割中的應用,醫學圖像分割技術取得了顯著的進展。精確地分割結果可為醫療提供輔助依據,有效減少因醫生個人水平差異對最終診斷的主觀影響,具有十分重要的研究意義。
醫學圖像具有對比度不明顯、邊界信息模糊、形狀大小不統一等獨特的數據屬性,在分割任務中具有一定挑戰性。現有網絡通常利用目標特征屬性及其相關位置信息對目標區域進行分割,改善醫學圖像的分割效果。如FCN[1]將卷積層替換全連接層直接獲取每個像素的分割結果,獲取最終目標特征。MU-Net[2]將殘差網絡特征與跳躍連接合并,增強目標全局信息獲取分割結果。U-Net3+[3]利用全尺度跳躍連接直接獲取目標特征。而病灶特征具有其特定的結構屬性,以上方法均取得一定的效果,但也更強調空間位置信息,忽略了特征的空間結構信息,導致對目標區域的分割產生一定誤差。另一方面,病灶的邊緣信息同樣影響著分割的準確性,但大多數網絡僅利用CNN[4]隱式地對目標區域進行分割,間接地完成邊界輪廓信息的提取[5]。因此以上2個因素相互作用,影響了對病灶目標區域分割的準確性。
針對上述問題,本文以經典的U-Net網絡為基本框架,提出邊緣增強的注意力模塊(contour enhanced attention,CEA),同時捕捉輸入圖像的位置信息和結構信息,進一步提高醫學圖像分割準確率。
1 相關工作
1.1 圖像分割網絡
近年來,深度卷積神經網絡因其可實現端到端的自動化分割,受到了廣泛地關注和應用[6]。2015年U-Net[7]卷積網絡被提出,采用編碼器、解碼器以及跳躍連接的拓撲結構,成為醫學圖像分割中的主流方法。SegNet[8]直接對目標特征進行提取,取得了良好的分割表現。UNet++[9]在跳躍連接的過程中采用密集連接的方式實現對目標的分割。R2UNet[10]將殘差連接和循環卷積結合起來,替換U-Net中的子模塊,改善醫學圖像的分割效果。但大多數醫學圖像分割技術通過特征區域位置或其特征本身屬性完成最終分割,間接地對邊界輪廓特征進行利用,且對特征位置信息利用不充分,影響最終的分割效果。
1.2 注意力機制
隨著注意力機制的不斷增強[11],使得網絡既能獲取特征的結構信息又能獲取位置信息,在醫學圖像分割領域應用愈加廣泛。壓縮激勵網絡(squeeze-and-excitation networks,SENet)[12]通過信息的壓縮激勵調整每個通道的權重,將不同權重應用于特征圖的目標區域,促使網絡關注目標特征,增強網絡的特征提取能力,有效提升模型的分割效果。從瓶頸注意力模型(bottleneck attention module,BAM)[13]和卷積塊注意力模型(convolutional block attention module,CBAM)[14]中受到啟發,通過將空間和通道信息進行融合,獲取空間上特征位置信息,建立通道之間的相互依賴關系,更好地獲取對象識別能力。Attention U-Net[15]使用注意力機制關注目標區域,抑制無關特征,出色地完成了對病灶的分割。Spatial Attention[16]通過獲取空間上的特征信息提升網絡模型的分割能力。Coordinate Attention[17]可以同時獲取特征的結構性和位置信息,使得最終輸出的特征圖隱式含有豐富的位置信息。
2 本文方法
針對現有方法不能充分利用特征位置信息,并且對病灶特征的邊緣像素點劃分不準確,進而導致邊界模糊、圖像分割效果不理想的問題,提出一種CEA。該方法以經典的U-Net網絡為基本結構,將注意力模塊應用于解碼器階段的上采樣中,使其充分捕捉位置信息的同時增強邊緣輪廓特征,實現對目標區域的精準分割。
整體網絡結構設計如圖1所示,其中左半支為編碼器階段,進行下采樣操作,提取目標的特征信息。右半支為解碼器階段[18],進行上采樣操作完成特征信息的解碼過程。在上采樣過程中引入CEA,加強對特征位置信息利用的同時有效捕捉邊界的輪廓信息,幫助網絡提取到邊緣信息更加明確的病灶特征,提高分割結果的準確性。

圖1 整體網絡架構
在解碼器網絡中,CEA的整體設計如圖2所示,將經過上采樣的每一層輸入該模塊。首先對輸入特征進行2個方向的特征編碼,讓其充分獲取病灶的位置信息,并在此基礎上加強邊界差異,最終輸出的特征圖包含豐富的位置感知和較強的邊界輪廓,改善醫學圖像的最終分割效果。具體步驟如下:
步驟1.初始化。給定尺寸大小為×的輸入特征,分別沿著水平方向和垂直方向進行平均池化操作,獲得輸入特征垂直結構和水平結構上的信息編碼,即
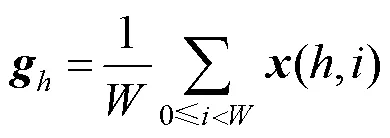
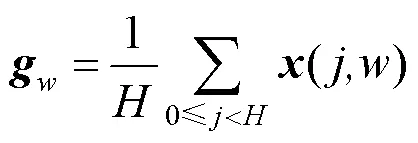
步驟2. 特征壓縮。將2個方向的特征進行拼接操作后,送入卷積中,得到2個方向空間信息的特征圖;為加快模型的收斂速度并且提高對目標區域的關注度,對融合后的特征圖進行非線性歸一化操作,即
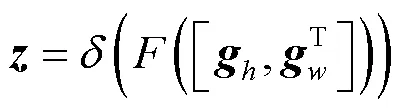
其中,T為轉置操作;[ ,]為拼接操作;為卷積操作;為非線性激活函數。



步驟4.計算加權特征圖。通過矩陣相乘操作,綜合水平垂直方向權重信息,獲得空間中每一個位置的權重;將其作用于原始圖像中,以獲得空間加權后特征注意力,即

步驟5. 計算邊緣增強特征。為捕捉差異、加強邊緣特征信息,進一步地計算原始特征圖和加權特征注意力圖之間的差值,得到邊界偏移量,即

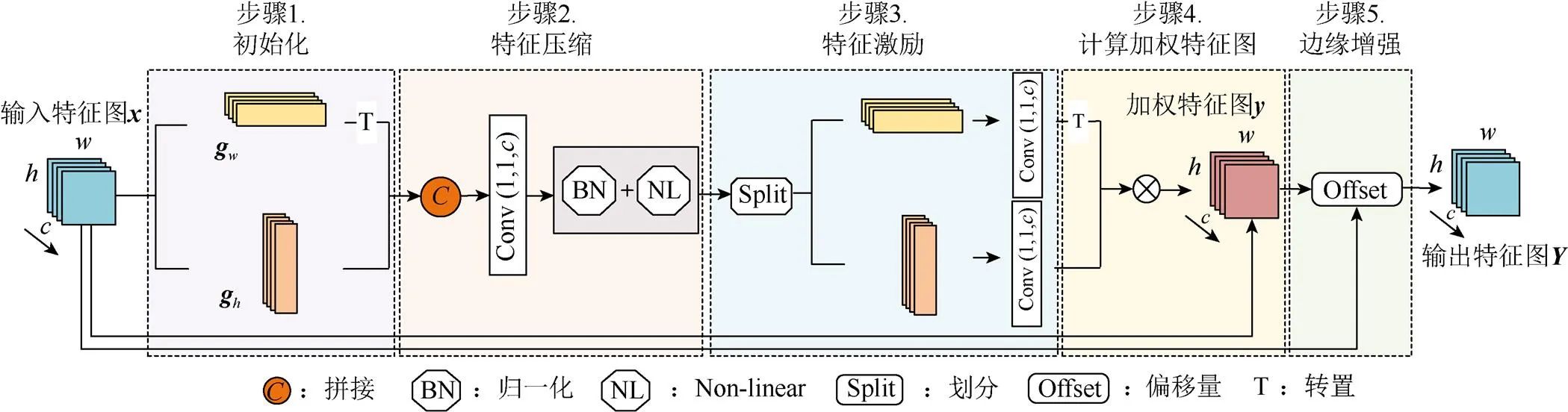
圖2 邊緣增強注意力模塊
本文采用的CEA首先從2個方向出發計算加權特征圖,同時獲取特征的結構和位置信息,有效捕捉醫學圖像中的全局結構和局部細節特征;再通過offset方式,計算輸入特征圖與加權特征圖的一階差分,增強邊界輪廓信息,突出病灶的邊界信息,獲取特征區域與背景信息的差異性,從而達到更精確的分割效果。
3 實驗結果
3.1 數據集及評價指標
3.1.1 數據集
本文采用具有挑戰性的Kvasir-SEG[19]息肉數據集作為實驗數據,該數據集由1 000個息肉圖像及標簽組成。訓練過程采用PraNet等[20-22]相同的通用數據劃分方式,其中,88%作為訓練數據集,12%作為測試數據集。
該數據集中包含原始圖像與其對應的真實標簽,且具有病灶特征差異大、形狀不統一等特性。其中息肉數據的病灶邊界與背景信息難以區分,因而該數據集在醫學圖像分割任務中更具挑戰性。
3.1.2 評價指標
相似系數(Dice)。衡量2個樣本的重疊部分,指標范圍從0到1,其中1代表完全重疊的狀態,0代表完全不重疊的狀態,即
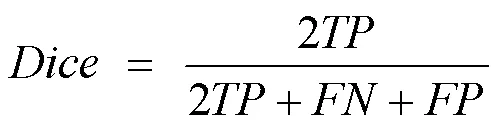
平均交并比(mIoU)。利用分割區域重疊面積的比值測量真值和預測值之間的相關度,即
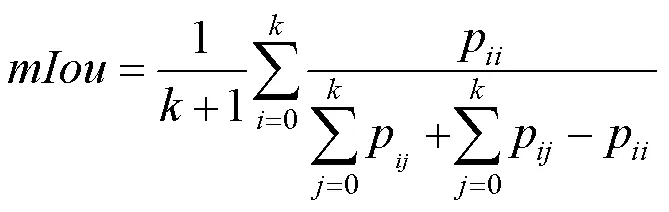
其中,為真值;為預測值;p為將預測為。
查準率(Precision)。為預測正確的實例個數占總的正例預測個數的比例,即
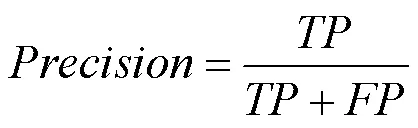
查全率(Recall)。為預測正確的正例占真實正樣本的百分比,即

F1分數。為模型查準率和查全率的一種加權平均,即

整體實驗結果以Dice為主要評價指標(其他指標作為輔助參考),其中以上所有指標取值越大代表分割精度越高,實驗效果越好。實驗過程中使用了水平翻轉、旋轉等數據增強方法。
3.2 實驗設置
軟硬件環境:實驗硬件環境為Intel Core i7 2600+GTX 2070 super 8 GB+16 GB RAM;軟件環境為CUDA10.0+Pytorch1.3.0+python3.6+VS Code。
實驗細節:訓練中采用Dice Loss計算預測圖像與真實圖像間的損失。網絡的初始學習率為10-4,batchsize為4,epoch為200。原始輸入圖像尺寸為512×512,初始通道數為3;整個網絡采用端到端的方式進行訓練,并使用early-stop機制提高網絡的泛化性能。
3.3 對比實驗
為了驗證提出的CEA的有效性,實驗以經典的U-Net醫學圖像分割網絡為基準,完成醫學圖像分割任務。實驗結果見表1。
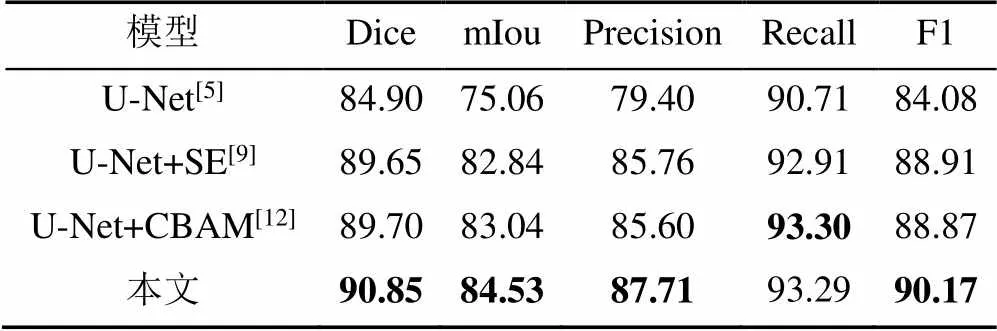
表1 CEA模塊實驗對比(%)
注:加粗數據為最優值
由表1可知,針對U-Net網絡,添加SE模塊后Dice得分提高了4.75%,添加CBAM模塊后提高了4.80%,而本文方法在Dice得分上提高了5.95%。由對比可知,各個模塊的添加均有效加強了醫學圖像的分割效果,其中本文提出的CEA比CBAM和SE模塊分別提高了1.15%和1.20%。結合Dice,mIou和F1綜合指標分析,本文方法均取得最優成績。而Precision和Recall在數據統一情況下具有一定相關性,當其中一個值較高時,另一個指標值較低。綜合所有指標數據,本文方法在醫學圖像分割任務中具有一定的積極作用。
圖3為其他方法與本文方法的可視化對比結果。由對比結果可見,U-Net網絡在分割目標病灶時出現明顯偏差,包含大量冗余區域,且無法保證分割結果的完整性;SENet雖然改進了U-Net存在的冗余問題,整體結構也相對完整,但仍出現分割區域不連續的情況(如第3行所示);CBAM網絡在分割完整性上具有進一步的提升,但仍包含冗余區域;相比較而言,本文方法可較好預測病灶形狀和位置,有效改善了缺失或多余分割的現象。綜上,該方法在分割完整性、準確性上均具有顯著優勢。
綜合對比實驗數據及可視化結果,表明目標特征的位置信息及邊界輪廓信息的充分利用可幫助網絡捕獲更精確的目標特征,使分割的準確率得到顯著提升。
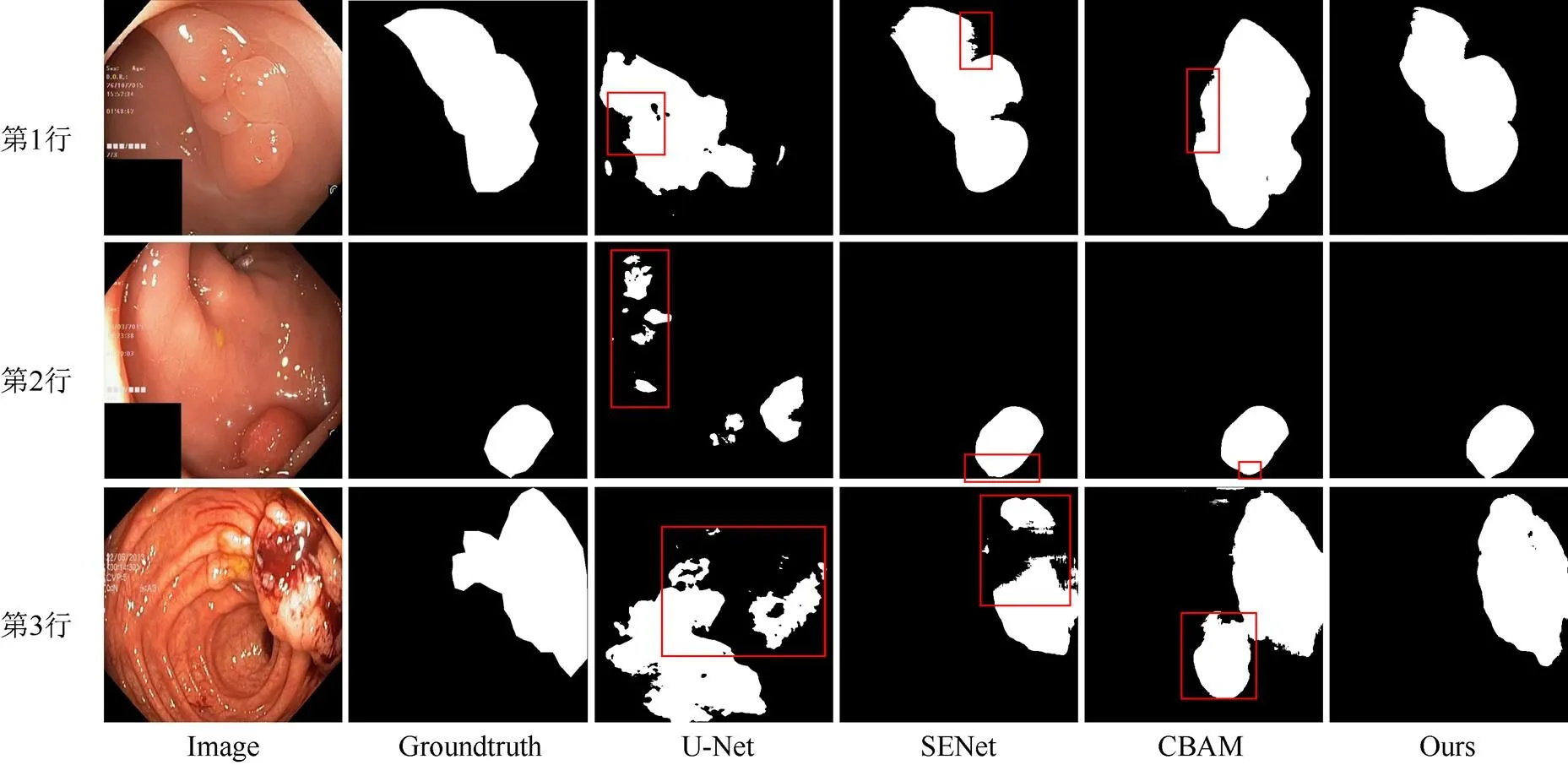
圖3 各種算法分割結果可視化比較
3.4 消融實驗
3.4.1 不同模塊數量對實驗結果的影響
實驗中針對CEA模塊設計了不同數量的連接方式。具體的,考慮特征信息進行上采樣操作過程中信息的逐層傳遞,為獲取更清晰的邊緣輪廓信息,始終保留下層的CEA模塊連接結構。對上層模塊進行不同程度的刪減,形成不同的連接方式,實驗結果見表2。
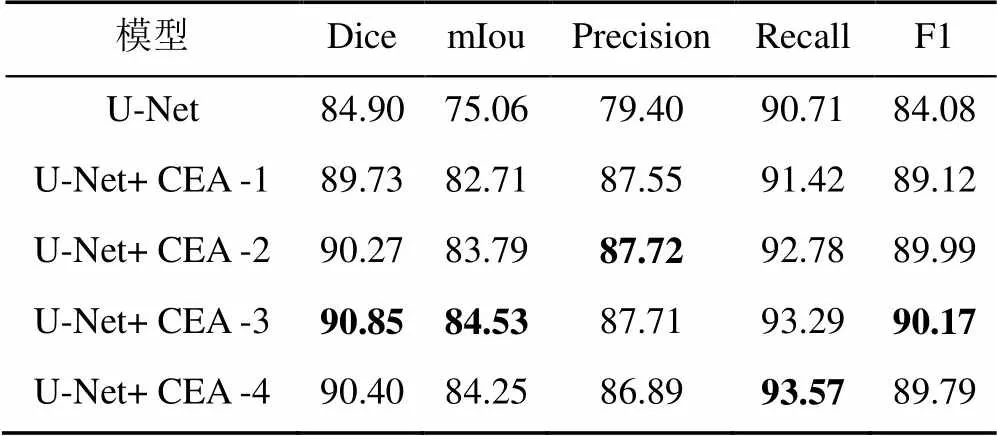
表2 不同CEA模塊數量對實驗結果的影響(%)
注:加粗數據為最優值
由表2可發現,1中加入不同數量的CEA模塊后,網絡的分割性能均有不同程度的提升。2中加入3塊CEA模塊后取得最優效果,比原始U-Net的Dice得分提高5.95%。
通過數據分析可知,目標區域的邊界信息對分割任務具有十分重要的作用。隨著上采樣次數的增加,特征位置信息和邊界信息經過不斷的提取,對分割結果起到了積極的作用。但添加模塊數量過多,造成特征信息冗余,反而起到抑制作用。綜合來看,對于U-Net網絡,當添加3個CEA模塊時,取得醫學圖像分割的效果最好。
3.4.2 邊緣輪廓信息對實驗結果的影響
本節將探究病灶的邊界輪廓信息對圖像分割任務的影響,所列的實驗結果均選取效果最佳時的模塊數量進行對比,同時采用多種評價指標全面衡量病灶的分割效果。其中w/o Contour表示未獲取邊界信息的模塊,w Contour表示獲取邊界信息的模塊,實驗結果見表3。
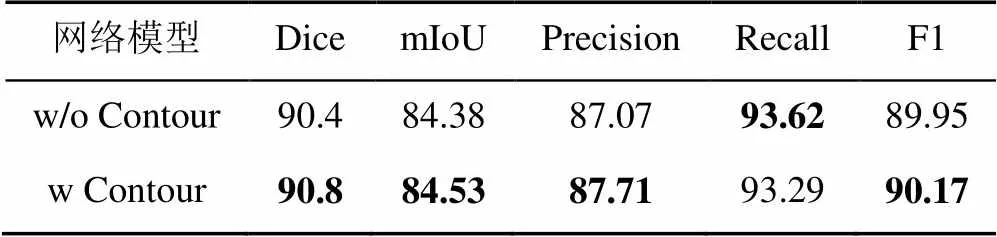
表3 邊緣輪廓信息對模型的影響(%)
注:加粗數據為最優值
由表3對比數據可發現,加入邊界信息增強后,網絡的分割性能均有所提升。以Dice為主要評價指標可以看出,加入邊界增強后比原始提升了0.40%,可見邊緣特征的提取對圖像分割起到了十分重要的作用。
4 總 結
針對醫學圖像的分割任務,提出一種CEA,首先分別對特征圖進行2個方向的特征編碼,獲取特征的空間結構信息和位置信息,捕獲長距離依賴關系;其次將其進行融合,得到具有豐富位置信息的特征注意力圖,增強對目標區域的關注;最后在此基礎上增強邊界信息與背景信息的差異性,使得病灶特征的邊緣輪廓更加明顯,提高分割的準確性。在相關息肉數據集上的實驗表明,本文所提CEA模塊在醫學圖像分割方面性能突出,具有一定先進性。
[1] SHELHAMER E, LONG J, DARRELL T. Fully convolutional networks for semantic segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(4): 640-651.
[2] SEO H, HUANG C, BASSENNE M, et al. Modified U-net (mU-net) with incorporation of object-dependent high level features for improved liver and liver-tumor segmentation in CT images[J]. IEEE Transactions on Medical Imaging, 2020, 39(5): 1316-1325.
[3] HUANG H M, LIN L F, TONG R F, et al. UNet 3+: a full-scale connected UNet for medical image segmentation[C]//ICASSP 2020 - IEEE International Conference on Acoustics, Speech and Signal Processing. New York: IEEE Press, 2020: 1055-1059.
[4] Kim Y. Convolutional neural networks for sentence classification[EB/OL]. (2014-08-25) [2021-06-30]. https:// arxiv.org/abs/1408.5882.
[5] 孫文燕, 董恩清, 曹祝樓, 等. 一種基于模糊主動輪廓的魯棒局部分割方法[J]. 自動化學報, 2017, 43(4): 611-621.
SUN W Y, DONG E Q, CAO Z L, et al. A robust local segmentation method based on fuzzy-energy based active contour[J]. Acta Automatica Sinica, 2017, 43(4): 611-621 (in Chinese).
[6] VALPOLA HARRI. From neural PCA to deep unsupervised learning[EB/OL]. (2014-11-28) [2021-06-30]. http://arxiv.org/ abs/1411.7783v2.
[7] RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation[C]// Medical Image Computing and Computer-Assisted Intervention. Heidelberg: Springer, 2015: 234-241.
[8] BADRINARAYANAN V, KENDALL A, CIPOLLA R. SegNet: a deep convolutional encoder-decoder architecture for image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2017, 39(12): 2481-2495.
[9] ZHOU Z W, RAHMAN SIDDIQUEE M M, TAJBAKHSH N, et al. UNet++: a nested U-net architecture for medical image segmentation[C]//Deep Learning in Medical Image Analysis and Multimodal Learning for Clinical Decision Support. Heidelberg: Springer, 2018: 3-11.
[10] ALOM M Z, YAKOPCIC C, TAHA T M, et al. Nuclei segmentation with recurrent residual convolutional neural networks based U-net (R2U-net)[C]//NAECON 2018 - IEEE National Aerospace and Electronics Conference. New York: IEEE Press, 2018: 228-233.
[11] 萬加龍, 金煒東, 唐鵬, 等. 基于視覺注意力增強CBAM-U-Net模型的視網膜血管分割[J]. 計算機應用研究, 2020, 37(S2): 321-323.
WAN J L, JIN W D, TANG P, et al. Retinal vascular segmentation based on visual attention enhanced CBAM-U-Net model[J]. Application Research of Computers, 2020, 37(S2): 321-323 (in Chinese).
[12] HU J, SHEN L, ALBANIE S, et al. Squeeze-and-excitation networks[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(8): 2011-2023.
[13] PARK J, WOO S, LEE J Y, et al. BAM: bottleneck attention module[EB/OL]. (2018-08-13) [2021-06-21]. http://arxiv.org/ abs/1807.06514.
[14] WOO S, PARK J, LEE J Y, et al. CBAM: convolutional block attention module[M]//Computer Vision – ECCV 2018. Cham: Springer International Publishing, 2018: 3-19.
[15] OKTAY O, SCHLEMPER J, FOLGOC L L, et al. Attention U-net: learning where to look for the pancreas[EB/OL]. (2018-05-13) [2021-06-21]. https://arxiv.org/abs/1804.03999.
[16] WANG H R, FAN Y, WANG Z X, et al. Parameter-free spatial attention network for person Re-identification[EB/OL]. (2018-11-30) [2021-06-21]. https://arxiv.org/abs/1811.12150.
[17] HOU Q B, ZHOU D Q, FENG J S. Coordinate attention for efficient mobile network design[C]//2021 IEEE/CVF Conference on Computer Vision and Pattern Recognition. New York: IEEE Press, 2021: 13708-13717.
[18] 李天培, 陳黎. 基于雙注意力編碼-解碼器架構的視網膜血管分割[J]. 計算機科學, 2020, 47(5): 166-171.
LI T P, CHEN L. Retinal vessel segmentation based on dual attention and encoder-decoder structure[J]. Computer Science, 2020, 47(5): 166-171 (in Chinese).
[19] JHA D, SMEDSRUD P H, RIEGLER M A, et al. Kvasir-SEG: A segmented polyp dataset[M]//MultiMedia Modeling. Cham: Springer International Publishing, 2019: 451-462.
[20] FAN D P, JI G P, ZHOU T, et al. PraNet: parallel reverse attention network for polyp segmentation[C]//Medical Image Computing and Computer Assisted Intervention - MICCAI 2020. Heidelberg: Springer, 2020: 263-273.
[21] TOMAR N K, JHA D, ALI S, et al. DDANet: dual decoder attention network for automatic polyp segmentation[M]// Pattern Recognition. ICPR International Workshops and Challenges. Cham: Springer International Publishing, 2021: 307-314.
[22] HUANG C H, WU H Y, LIN Y L. HarDNet-MSEG: a simple encoder-decoder polyp segmentation neural network that achieves over 0.9 mean dice and 86 FPS[EB/OL]. (2021-01-18) [2021-01-20]. https://arxiv.org/abs/2101.07172.
A U-Net based contour enhanced attention for medical image segmentation
LI Cui-yun1, BAI Jing1,2, ZHENG Liang1
(1. School of Computer Science and Engineering, North Minzu University, Yinchuan Ningxia 750021, China; 2. The Key Laboratory of Images & Graphics Intelligent Processing of State Ethnic Affairs Commission, Yinchuan Ningxia 750021, China)
Medical image segmentation is vital for medical image processing. With the development of deep learning, image segmentation techniques have achieved remarkable development. However, there remain fuzzy and inaccurate problems in the discrimination of contour pixels for lesion features. To address the problems, we proposed a contour enhanced attention (CEA) module. It can obtain rich location information by feature encoding in two different directions and strengthen contours by calculating the offset between location features and input features. Furthermore, we constructed a U-Net for medical image segmentation based on the proposed module, it can break through the space limitation of convolution kernel, thus capturing position-aware cross-channel information and clearer edge contour information. In doing so, the accuracy of segmentation can be improved. Experiments on the public Kvasir-SEG dataset demonstrates that the network with CEA module achieves better results in Dice, precision, recall rate, and other evaluation indexes in medical segmentation.
medical image segmentation; location information; attention mechanism; edge contour; contour differences
TP 391
10.11996/JG.j.2095-302X.2022020273
A
2095-302X(2022)02-0273-06
2021-07-22;
2021-09-18
國家自然科學基金項目(61762003,62162001);中國科學院“西部之光”人才培養引進計劃(JF2012c016-2);寧夏優秀人才支持計劃;寧夏自然科學基金項目(2022AAC02041)
李翠云(1996–),女,碩士研究生。主要研究方向為數字圖像處理與模式識別。E-mail:1107893490@qq.com
白 靜(1982–),女,教授,博士。主要研究方向為CAD&CG、計算機視覺和機器學習等。E-mail:baijing@nun.edu.cn
22 July,2021;
18 September,2021
NationalNaturalScienceFoundationofChina (61762003, 62162001); “Light of the West” Talent Training and Introduction Program of Cas (JF2012c016-2); Ningxia Excellent Talents Support Program; Ningxia Natural Science Foundation Project (2022AAC02041)
LI Cui-yun (1996–), master student. Her main research interests cover digital image processing and pattern recognition. E-mail:1107893490@qq.com
BAI Jing (1982–), professor, Ph.D. Her main research interests cover CAD&CG, computer vision, and machine learning, etc. E-mail:baijing@nun.edu.cn

