WOX轉錄因子在植物發育中的作用
曾丹琦, 何春梅, 俞振明, 司燦, 陳璟,黃磊, 李冬妹,段俊*
WOX轉錄因子在植物發育中的作用
曾丹琦1,2, 何春梅1, 俞振明1, 司燦1,2, 陳璟1,黃磊1,2, 李冬妹3,段俊1*
(1. 中國科學院華南植物園,廣東省應用植物學重點實驗室,中國科學院華南農業植物分子分析與遺傳改良重點實驗室,廣州 510650;2. 中國科學院大學,北京 100049;3. 順德職業技術學院,廣東 佛山 528300)
WOX (WUSCHEL-related homeobox)轉錄因子與植物發育密切相關,包括植物胚胎發育和體胚發生、花和根發育、愈傷組織的形成和維持,以及干細胞維持等過程。越來越多的研究表明WOX在植物發育過程中扮演著極其重要的角色。WOX調控植物發育的機理研究在促進植物發育以及構建植物良好表型等研究提供了突破口。主要對WOX調控植物發育的相關研究進行綜述,并結合表觀遺傳學調控,探討了WOX調控植物發育的過程,以期為WOX轉錄因子調控植物的作用機制提供啟示。
WOX轉錄因子;植物發育;轉錄調控;綜述
轉錄因子(transcription factors, TFs)是指能結合基因啟動子從而調控基因表達的一類蛋白質分子。轉錄因子的調節是一個十分復雜的過程,在調控真核生物生長發育中起著重要作用。1996年,Laux等[1]首次從擬南芥()中鑒定到WUSCHEL (WUS)轉錄因子,該轉錄因子在頂端分生組織維持起到關鍵作用[2]。隨后,越來越多的植物WUSCHEL- related轉錄因子被克隆和分析,均含有同源異形盒(homeobox, HB)的保守結構域, 屬于homeobox超家族的成員,被命名為WOX轉錄因子[1,3]。
WOX轉錄因子在植物發育過程中扮演的角色非常多樣化,且其調控作用非常重要。以模式植物擬南芥為例,目前發現擬南芥共有15個WOX成員,功能涉及從干細胞的維持到胚胎模式的形成,包括枝條、花分生組織形成、根形成等生長發育過程[1,4–5],均被證明發揮著廣泛的作用;水稻()中共有13個WOX成員[6–9],如影響水稻花發育和抗旱性[6],調控不定根生長發育[7],影響莖尖分生組織和葉片發育[8]等。在鐵皮石斛()中,不同基因在原球莖發育過程中表達具有差異性,有研究表明基因可能與頂端分生組織發育相關,其在頂端分生組織形成期表達量高于其他[10],這為蘭科草本植物的WOX家族成員研究添上了重要一筆。在茉莉花()中,有研究證明4和分別與愈傷組織增殖和根再生有關[11],為基因在木本植物器官發生的功能研究提供了一條引領性的線索。在矮牽牛()和金魚草()中,有研究表明WUS直系同源物突變體導致了相關功能的缺失[12–13],這證實了基因在維持莖尖分生組織(shoot apical meri- stem, SAM)起著不可或缺的作用。在多種植物中,基因參與調控植物的花發育和花結構的形成[14]。不同物種中WOX功能的相關研究取得了重要進展,為解釋WOX轉錄因子在植物發育相關過程和調控機理奠定基礎。
在植物發育過程中,胚胎發育是保證植物正常生長發育的起源和核心[15],WOX轉錄因子在植物胚胎發育的啟動區域特異性轉錄程序過程中有著重要的作用[4],是胚胎發育早期表達的分生組織的基因[16]。盡管胚胎發育這一過程至關重要,但是其相關機制仍不清晰,因此,對調控胚胎發育的重要轉錄因子的研究進而解釋其相關機制充滿機遇和挑戰。本文對WOX家族不同成員、不同物種中WOXs成員的表達調控進行綜述,將植物表觀調控對植物發育的潛在影響和WOX家族成員對植物發育的影響聯系起來,旨在為揭示其相關機理提供依據,并為相關研究提供思路。
1 WOX家族成員概述
最早發現于黑腹果蠅中[17],其結構特征是有一小段氨基酸(60~66個殘基),這些氨基酸折疊成一個稱為同源異形結構域的DNA結合域,該結構域由HB DNA序列編碼[18]。動物HOX蛋白是典型的HB調節因子,在黑腹果蠅中最初是通過同源異型突變導致身體片段的轉變被鑒定發現的[19]。更多的研究表明,HB轉錄因子也在植物中發揮廣泛作用。1996年,Laux等[1]從擬南芥中首次鑒定到一個在頂端分生組織維持起到關鍵作用的()。WOX轉錄因子家族在植物發育過程的地位至關重要,參與植物的多種發育過程,如頂端分生組織的維持、花序發育、根發育和胚胎發育等。
關于WOX家族成員分類的研究主要集中在進化起源上。WOX家族成員的進化情況可以根據HB同源異形結構域蛋白序列的系統發育重建進行推測,有研究表明DNA-binding motif可能起源于真核生物分化之前[20]。以HB蛋白質的進化發育情況為基礎,植物WOX的系統發育樹可以分為3個分支,分別是WUS clade、Intermediate clade和Ancient clade[21–22]。在擬南芥中,WUS和WOX1~WOX7及其在種子植物中的直系同源蛋白屬于WUS clade,WOX8、WOX9、WOX11和WOX12及其在維管植物中的直系同源蛋白屬于Intermediate clade, WOX10、WOX13和WOX14及其在維管和非維管植物中的直系同源蛋白屬于Ancient clade[4,23]。研究表明只有Ancient clade包含來自綠藻和非維管苔蘚小立碗蘚()的WOX序列[24]; Intermediate clade除開花植物外還包括維管植物卷柏()[24];WUS clade僅包含開花植物的蛋白質序列[24]。這說明引起Intermediate clade的旁系同源基因必須已經存在于維管植物的最后共同祖先中。
編碼的蛋白結構的典型特征是存在同源異形結構域(homeodomain, HD)(圖1),該結構域通過形成螺旋-轉角-螺旋(helix-turn-helix, HTH)結構與DNA結合,調控下游基因的轉錄。植物WOX同源異形結構域通常在高度保守的結構中有2個延伸的環, 該環在生物發育中具有重要意義[7],且在HB其他家族如TALE家族也被發現[25]。此外,研究表明WUS box是WUS clade成員特有的結構域[4], 在WUS和其他3個成員(即WOX1、WOX4和WOX5)的WUS- box上游大約10個殘基處有1個酸性結構域,可能行使轉錄激活作用。而ERF-associated amphiphilic repression (EAR)結構域可能具有轉錄抑制功能,在擬南芥中只有WUS、WOX5和WOX7包含嚴格意義上的羧基末端EAR motif[24]。有趣的是,在WOX2的同源異形結構域的下游還有1個假定的鋅指結構域[4](圖1),這是WOX2成員特有的。
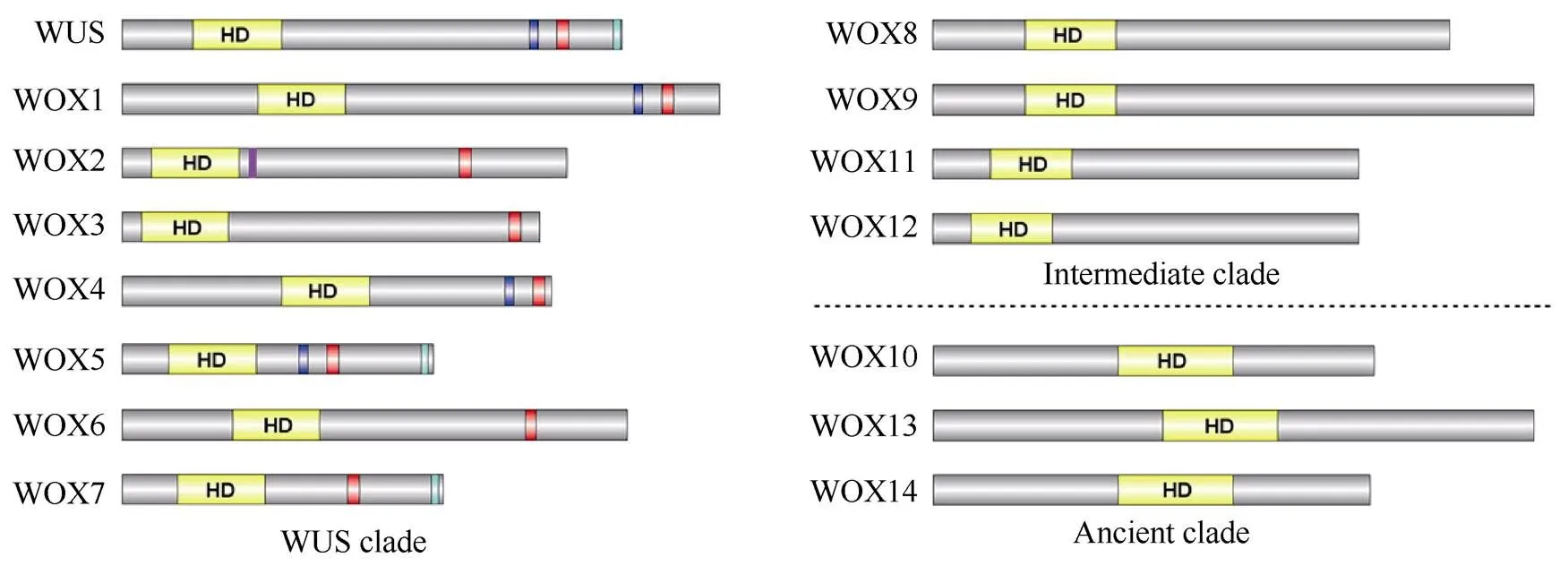
圖1 擬南芥AtWOX家族蛋白結構示意圖。同源域(HD)(黃色)是該家族最突出和最明確的特征;WUS box motif (紅色)在嚴格意義上被定義為 T-L- [DEQP]-L-F-P-[GITVL]-[GSKNTCV];酸性結構域(藍色)位于WUS-box上游大約10個殘基處;EAR結構域(綠色)位于羧基末端,其結構特征是[LVI]- X-[LVI]-X-[LVI];1個假定的鋅指結構域(紫色)位于HD下游,是WOX2特有的。
2 WOX轉錄因子的表達模式
WOX轉錄因子的表達模式具有組織特異性。具體表現為有些WOXs成員可能在某個特定器官中高表達,但在其他器官基本檢測不到,如在擬南芥的根尖分生組織的靜止中心細胞和側根原基中表達[5,26–27],但在其他部位的表達量低或者檢測不到[27]。除了特異表達外,WOX家族成員也存在一定的相關性,也可以同時在多個器官表達進而調控植物的生長發育,如WUS不僅在擬南芥的莖中表達[5],而且在擬南芥和禾本科植物的胚珠和花藥中表達[28]。盡管WOX家族成員間在結構上存在很大的差異,如同源異形結構域、保守肽等,但是從進化角度看,這些WOX之間似乎會存在從祖先而來的共同功能[29],這為尋找WOX家族成員的共性提供線索。早前研究表明,WOX基因()和的表達模式是重疊或相似的[29],且通過雙突變體的表型證明了他們在功能上是冗余的;在玉米()中的同源基因是和,對葉緣發育具有重要調控作用[30],進而說明單子葉和雙子葉植物的花發育過程中基因具有功能保守性[14]。
WOX除了在重疊的表達模式下具有功能冗余外, 也存在表達模式不同但功能具有冗余性的成員。WUS clade具有特殊的WUS box,有研究表明可以彌補在不同啟動子表達下和突變表型[31],且擬南芥中的大部分WUS clade成員均可回補煙草()同源突變體的缺陷[32],但非WUS clade則不能回補突變體的表型[14]。這證明了不同WUS clade成員在不同表達模式下功能仍具有重疊性,且說明了WUS clade特有的WUS box具有一定特殊性和重要性。此外,有研究表明,與WUS box或SRDX阻遏物域融合的嵌合WOX7、WOX9和WOX13蛋白(非WUS clade)可以回補的表型,這進一步證實了WUS box的功能重要性[32]。這暗示了WUS clade成員功能上的重疊可能是存在某種共性,如均含有功能特殊的WUS box,相較于更古老的Ancient clade和Inter- mediate clade,WUS clade存在的1個或多個轉錄抑制因子結構域[14],或許在植物的某個生長發育階段扮演著核心的角色。
綜上所述,WOX轉錄因子表達模式的不同或相似導致了它們在功能上的多樣性或冗余性。同時,功能的多樣性在不同植物不同發育過程所起的作用也得到了充分體現,進一步為WOX在植物發育扮演多樣化角色奠定了基礎;此外,功能的冗余性增加了WOX的穩定性,例如某個WOX轉錄因子突變導致的缺陷或許可以由其他具有相同或相似功能的WOX彌補,保障了植物的正常生長發育。
3 WOX參與植物發育
許多同源框基因在植物發育中發揮了重要作用。WOX家族作為植物特定的轉錄因子家族,在植物的生長發育扮演了許多重要的角色,如植物胚胎發育和體胚發生、花發育、根發育、愈傷組織的形成和維持以及干細胞維持等。
3.1 參與植物胚胎發育和體胚發生
植物胚胎發育是一個十分復雜的過程。目前有關早期胚胎發生的分子機理的研究進展緩慢,早期胚胎小且被母體細胞包圍導致難以跟蹤觀察[33]。體細胞胚胎發生(簡稱體胚發生)是指在沒有受精的情況下從體細胞發育出胚,進而發育成為獨立個體的過程[34–35],在很大程度上克服了研究胚胎發育的局限性。此外,體胚類似于合子胚,兩者經歷幾乎相同的發育階段[36–37],很好詮釋了植物細胞的全能性表達。近年來該方面的研究發展迅速。有研究表明,體胚發生過程涉及的發育轉換(誘導體細胞形成全能胚胎進而再生為完整植株)與一系列識別內部信號和外部刺激的分子密切相關[38]。其中轉錄因子在胚胎發育和體胚發生等復雜的生物過程發揮著至關重要的調節功能。
大量研究表明,WOX轉錄因子在植物的胚胎發育中起著關鍵的調控作用[4,14,39–40],的差異表達影響胚胎早期細胞的分化命運。在擬南芥中,不同WOX家族成員的表達動態影響了胚胎發育中的區域特異性轉錄,為揭示擬南芥胚胎發育模式的建成機理起到了很好的助推作用,如和在卵細胞和受精卵以共表達和不對稱分裂模式分別局限于受精卵的頂子細胞和基部子細胞,在胚胎發育的中心胚區啟動發揮重要作用,在大多數早期球形胚胎的垂體中特異表達等[4],在胚胎發生被激活重新表達[41];在番茄()中,是胚胎和胚后發育期間維持細胞分裂活性的必需基因[42];在水稻中,在調控胚胎發育的區域特異性轉錄扮演著重要角色[43]。此外,研究表明,突變后,胚胎的頂端分生組織無法正常形成,且突變植株中莖頂端分生組織形成受到影響[1]。植物的胚胎發育是復雜的,胚胎模式不僅受到生長素反應的影響[44],而且細胞之間的分子差異在胚胎發育中相對較早地建立[45–46]。
WOX轉錄因子在植物體胚發生扮演著重要的角色。有研究表明,WUS同源異形結構域蛋白的異位表達可以促進幼苗的體細胞胚發育[47–48]。WUS不直接決定胚胎命運,而是主要維持未分化細胞狀態,以響應不同的刺激,從而改變植物體內組織的發育命運[48–49],說明其可能在體胚發生等未分化狀態具有重要調控作用。在擬南芥中,體胚發生可通過單個轉錄因子如()的異位過表達[48]來實現。有研究表明,WOX2和WOX3是擬南芥體胚發生必不可少的關鍵轉錄因子[50];在云杉()中,與體胚發生過程存在相關性,例如在早期體細胞胚胎發育過程中高度表達,但隨著胚胎的成熟而下降[22];在火炬松()中,STIMPY/WOX9通過調節WUS的活性來維持干細胞群[51–52],暗示其在胚胎發生過程中扮演重要角色。且遺傳學研究表明, WUS/ CLAVATA (CLV)自我調節回路對于維持干細胞的特性非常重要[53–55]。因此,WUS-CLV是一個極其復雜的調控網絡,對于體胚發生機理的進一步闡明是一個挑戰。
此外,關于胚胎發育的機理,有研究表明通過調控運輸和局部生長素反應可以影響胚胎發育模式[4]。例如,胚胎中特定的生長素反應作為信號分子的調控可以保證根發育正常[56],但是相關胚胎模式的形成機制目前尚不太清楚,其中作為胚胎發育的關鍵調控基因——WOX家族成員是否與生長素調控胚胎發育存在一定聯系,或許可以為揭示胚胎發育調控的網絡機制提供突破口。慶幸的是,目前胚胎發育的研究跳出了以往傳統的思維,全基因組的表觀遺傳分析揭示了siRNA在表觀遺傳學層面上維持胚胎基因組穩定的作用[57],這是一個巨大突破。然而,目前尚未直接有從表觀遺傳角度研究以揭示WOX調控胚胎發育機理的相關報道,因此WOX對胚胎發育的研究仍需進一步探討。
3.2 參與植物花發育
花作為植物的重要功能器官,在植物繁殖和植物進化過程中發揮著關鍵的作用。研究表明WOX在植物的器官發育尤其是花器官發育具有重要作用[14,58]。在擬南芥中,WUS是花中的干細胞維持所必需的, WOX13和WOX14影響花的發育[59],可以調控擬南芥花的側軸發育和影響花的外觀[60];在矮牽牛中,WOX1可以維持花發育中的正常表型, WOX8是花序發育和體系結構所必需的[61];在紫花苜蓿()和煙草中,花的表型受到了WOX1的影響[32];在玉米中,NARROW SHEATH1 (NS1)和NS2 (WOX3的同源物)保證葉片和花器官的正常表型[62]。這說明WOX轉錄因子有助于維持花卉結構和形態,是植物花發育不可或缺的角色。
盡管不同的WOX家族成員在結構上差異很大,但部分成員在功能上存在冗余。研究表明, WOX1在調控不同植物如矮牽牛、紫花苜宿、煙草和豌豆等的花發育中是功能冗余的[29];(,WOX9亞家族代表之一)是一個復制型基因,它在花序發育過程中獲得了新的功能[63];在玉米葉片的側結構域發育中,窄鞘復制型基因可以執行祖先保守的冗余功能[62]。此外,花其實是經過修飾的葉片,WOX在影響花發育的同時會影響營養生長,如擬南芥中基因的無效突變不僅會導致葉片上的側托葉缺失,還會使花朵上的側萼片和雄蕊缺失[31],因此,關注植物的葉片生長或許可以填補植物花發育研究的部分空白領域。
3.3 參與根的發育
根尖分生組織是根生長發育的細胞源泉[64]。植物根系發育是復雜的,不僅包括胚胎期間的初生根,而且包括萌發后產生的側根和不定根,因此了解其相關的分子機理非常必要。目前有關WOX轉錄因子調控植物根發育的研究較多。在水稻中,是調控不定根生長發育的關鍵基因,很大程度上可以影響冠狀根發育[7,65];在擬南芥中,根的發育不僅受到WOX13和WOX14的影響[59]以及WOX11和WOX12的共同調控[66],且WOX5可以在根分生組織中發揮調控作用[5],WOX4在形成層(根和莖中的一種分生組織)中也發揮了重要作用[26,67];在茉莉花中,WOX1對根的影響主要體現在根原基起始和分化[11];在小麥()中,WOX5在根中大量表達[68]。這說明WOX家族成員在不同植物根發育中扮演的角色重要且多樣。
高等植物的根發育過程由多種信號因子調控,如光、植物激素、環境信號等多種信號調控。植物激素對根系的調節起著重要的作用,其中WOX家族成員從植物激素角度調控植物根系發育的相關研究[69–71]較多。正常的根發育需要在胚胎模式中有特定的生長素反應,起著發出信號的作用。研究表明,WOX11可能是生長素和細胞分裂素的整合子,它可以通過對生長素和細胞分裂素信號協調來影響水稻根的細胞分裂[65],這為WOX11參與植物根的轉錄程序機制揭開了面紗,進而補充了WOX轉錄因子調控植物根發育的相關研究。
3.4 參與植物愈傷組織的形成和維持
植物具有強大的發育可塑性,愈傷組織可以使植物獲得再生。WOX家族成員對植物愈傷組織的形成和維持起著關鍵的調控作用。研究表明,B型ARR-HD-ZIPⅢ復合體可以誘導在植物愈傷組織表達[41]。愈傷組織轉變為綠色或黃綠色后,需要重新編程為芽祖細胞以形成不定芽。研究表明, 芽祖細胞的形成需要細胞分裂素誘導的表達[41,72]。在擬南芥中,擬南芥轉錄因子LBD16被WOX11激活后在CIM (愈傷組織培養基)中特異表達, WOX11- LBD16途徑是愈傷組織獲得多能性的保證[73]。愈傷組織命運轉變(再生能力細胞轉變為基源細胞)的首步需要生長素激活WOX11和WOX12,第二步是WOX11/WOX12激活WOX5/WOX7和LBD16的表達[66];水稻愈傷組織中含有WOX11轉錄本[7], 暗示WOX11可能影響水稻愈傷組織的生長;在茉莉花中,WOX4、WOX13和一個特異的同源異形盒基因對愈傷組織的誘導和增殖起調控作用,進一步表明了WOX家族成員在愈傷組織誘導中存在多方面的遺傳相互作用[11]。因此,了解WOX家族成員在愈傷組織的激活,有助于揭示植物發育中調控植物愈傷組織的機制。
盡管對植物細胞全能性的研究歷史很悠久,但其分子機制在很大程度上仍然是模糊的。目前關于WOX家族影響植物愈傷組織的研究不多,其中比較熱門的是結合組學技術的研究。選擇WOX轉錄因子作為切入點進行分析進而實現精準定位的組學技術,或許可以解析WOX在愈傷組織表達情況,進而揭示調控愈傷組織的相關機制。
3.5 WOX參與植物干細胞發育
干細胞是一類未分化和持續分裂的細胞群,植物干細胞實為莖尖分生組織(SAM)、根尖分生組織(RAM)等各類分生組織,可以高效率地利用生物反應器生產活性物質,在藥品(下游制藥)、食品(功能性食品)以及化妝品行業具有廣闊的應用前景[74]。
大量研究發現WOX家族成員在植物未分化細胞的發生和維持具有重要的作用。WUS是WOX家族的創始成員,可以促進干細胞的識別和維持[1]。在擬南芥中,WUS在莖尖分生組織(SAM)中的干細胞維持扮演著關鍵調控因子的重要角色[1,75],同時它也對莖尖頂端分生組織的維持和形成具有調控作用[48],因此可以認為WUS是干細胞維持的中央調節劑,是植物發育調控網絡的一個密不可分的角色。此外,有研究表明細胞分裂素反應調節因子被WUS直接調節,從而控制分生組織功能[76]。有趣的是,WUS特有結構WUS box motif調控著植物發育中芽再生過程的干細胞生態位的建立[2],暗示WUS box motif或許是植物干細胞發育調控網絡的關鍵因子,但仍需要進一步的實驗驗證。
植物中干細胞群體的自我更新是動態的,涉及了復雜的調控網絡。WUS蛋白是調控植物莖端干細胞的核心因子,以WOX轉錄因子功能為中心的干細胞生態位的遺傳機制的本質一直難以捉摸,目前有關干細胞生態位中保守的WOX功能的分子聯系以及WOX如何與其他因子相關作用形成調控網絡只有少量研究。因此,深入研究WOX蛋白及其互作因子,對于解析植物干細胞發育過程很關鍵。有研究表明,擬南芥()家族轉錄調控因子與WOX蛋白相互作用,以促進干細胞的增殖,同時,形成不同干細胞生態位的基礎之一是WOX和HAM家族成員重疊表達模式的差異,為植物發育過程中干細胞生成的調控機理建立了一個新的框架[77]。在擬南芥中,WUS和CLV相互作用共同參與芽和花分生組織的發育過程,是一個CLV抑制WUS轉錄水平、WUS表達誘導CLV3的動態反饋回路[54]。WUS、STM和CLV這3大因子在植物干細胞的調控關系是其解析調控網絡的重要突破。研究表明,WUS與STM直接相互作用形成異源二聚體并結合到CLV3的啟動子上,共同調控植物的莖端干細胞[78]。此外,WUS和其他WOXs成員功能常常可以替代,如和基因在調節莖和根的干細胞維持的調控功能是可以互換的[5],說明基因間具有冗余性。生長素可以促進形成層活動,離不開的參與[26–67]。研究表明,在形成層中表達豐富,對于形成層干細胞具有非常重要的調節作用。此外,在根分生組織中的靜止中心特異表達,通過抑制干細胞的分化進而發揮調控作用[5]。其中,負責莖和根干細胞維持的作用機制與CLV3-WUS途徑的負反饋環機制類似,通過CLE40 (一種與CLV3密切相關的肽)與ACR4受體結合從而抑制WOX5的轉錄活性,形成了一條CLE40-WOX5信號轉導途徑[79],表明分生組織的維持機制在進化上可能具有保守性。
綜上所述,WOX轉錄因子可以調控植物的干細胞命運。WOX具有保守性功能[80],有研究表明,相關機制可能是WOXs的非細胞自主性賦予了其在干細胞的功能特性[31],可移動的WOX蛋白與其他關鍵蛋白互作或結合,共同發揮作用,并構成完整的胞間信號網絡。
3.6 WOX與表觀遺傳共同調控植物發育
基因是植物發育的關鍵調控因子(圖2)。在植物發育中,重編程基因表達的染色質機制在很大程度上是未知的。截至目前,已有多個WOX與DNA甲基化、組蛋白修飾等表觀遺傳相關的報道。初步研究表明的調節會受到表觀遺傳機制的影響[81]。其中,DNA甲基化和基因表達之間存在潛在的相關性[82],有研究表明,在蘋果()中WOX轉錄因子在花芽中高表達,這與基因體區域的低甲基化水平有關[83];海棠()成葉中2個基因的甲基化水平高于幼葉[84]; 有研究表明,基因的表達受DNA甲基化的調控[85],且水稻和擬南芥的啟動子具有相同的DNA甲基化的特征性表觀遺傳標記[86], 暗示了可能在進化過程中具有表觀遺傳甲基化修飾以調節其表達。此外有研究表明,擬南芥組蛋白乙酰轉移酶對/基因有負作用,他們共同調控花分生組織[87];WOX11通過與組蛋白H3K27me3去甲基化酶JMJ705相互作用,使得水稻莖頂端發育基因發生表觀重編程以刺激水稻莖端生長[9];通過觀測其組蛋白乙酰化修飾水平與其他基因的相關性進一步解析了向日葵()胚胎發育的調控機理[88]; WOX5通過組蛋白去乙酰化抑制分化因子CDF4以抑制擬南芥根中干細胞的分化[89]。綜上所述,本文從表觀遺傳角度對不同基因、不同植物中基因的表達調控等方面進行了綜述,將植物表觀基因組對植物發育的影響和WOX家族對植物發育的調控聯系起來,旨在為進一步揭示WOX調控植物發育的相關機理提供理論依據。

圖2 WOX轉錄因子基因在植物生長發育過程中的調控功能。Auxin: 生長素;CK: 細胞分裂素; ARRs: 擬南芥反應調節因子;LBD16: 側器官邊界-結構域16。實線箭頭表示直接調節,虛線箭頭表示間接調節,其機制尚不清楚。
4 結語
WOX轉錄因子參與植物的發育如胚胎發育、體胚發生,以及植物花、葉、根的發育,同時在愈傷組織形成中發揮重要作用。盡管大多數WOX亞家族的結構明顯不同(如外顯子數差異、每個亞家族特有的保守肽基序等),但這些亞家族中許多蛋白質確實存在共享一些祖先的共同功能現象,如功能重疊、功能互補等,這或許是研究WOX家族成員進化歷程的突破口。其中,有些WOX家族成員盡管表達模式不同,但它們的蛋白質序列保持著相似的能力即功能重疊,說明了WOX家族成員功能的進化可能主要通過表達模式的改變。然而,WOX家族不同成員的功能互補主要通過功能多樣性的多種成分調控元件實現,暗示WOX功能的進化可能是氨基酸序列變化起作用。可以補充擬南芥根分生組織中功能的缺失[5],它們主要通過不同啟動子的特異性實現WUS1和PRS1/WOX3等價的蛋白-蛋白相互作用。
植物和動物最大的區別之一是植物不能移動,因此,為了應對復雜多變的生長環境,植物必須進化出獨特的信號機制來完成器官形成和發育,主要方式之一是通過胞間連絲進行細胞間運輸,相關的移動分子包括蛋白、植物激素等。其中,蛋白大多數是非細胞自主性轉錄因子,植物中非細胞自主性轉錄因子參與了植物發育調控網絡。在植物生長發育的過程中,WOX家族成員作為植物特有的轉錄因子家族,可以調控植物生長發育的多個環節,在調控過程中也涉及到了細胞分裂素、生長素等生長素的信號傳導以及反應調節因子等多種因素,是一個極其復雜的調控網絡。雖然前人已經進行了大量的相關研究,但是很多機理如這些因素在植物發育過程中“何時”、“何地”進行相互作用至今沒有定論。
此外,從表觀遺傳角度研究基因對植物發育的影響具有巨大的潛在研究價值。前人研究表明,植物為了防止異位表達,具有重要發育作用的轉錄因子在編碼過程中往往會受到表觀遺傳抑制, 如組蛋白修飾和DNA甲基化[90]。植物WOX轉錄因子基因家族是重要的且特異的,有研究表明,轉錄因子WUS在組織培養中起關鍵作用,表觀遺傳調控缺陷的突變體在愈傷組織的表型差異與重編程調節子的差異表達有關,這主要通過調節表達和生長素信號傳導來實現[85]。綜上所述,未來的研究或許可以從表觀遺傳層面對WOX家族成員參與植物發育的調控展開研究,并結合最新的組學技術和CRIPSR技術等工具,研究在不同物種的WOX家族成員在進化過程中出現的亞功能化和新功能化等相關機制,相信未來我們可以更加清晰地解析WOX家族成員調控不同植物不同發育過程涉及的作用機制。
[1] LAUX T, MAYER K F, BERGER J, et al. The WUSCHEL gene is required for shoot and floral meristem integrity in[J]. Development, 1996, 122(1): 87–96. doi: 10.1242/dev.122.1.87.
[2] IKEDA M, MITSUDA N, OHME-TAKAGI M.WUS- CHEL is a bifunctional transcription factor that acts as a repressor in stem cell regulation and as an activator in floral patterning [J]. Plant Cell, 2009, 21(11): 3493–3505. doi: 10.1105/tpc.109.069997.
[3] LIU X G, KIM Y J, MüLLER R, et al.terminates floral stem cell maintenance inby directly repressingthrough recruitment of polycomb group proteins [J]. Plant Cell, 2011, 23(10): 3654–3670. doi: 10.1105/tpc.111.091538.
[4] HAECKER A, GRO?-HARDT R, GEIGES B, et al. Expression dyna- mics ofgenes mark cell fate decisions during early embryonic patterning in[J]. Development, 2004, 131(3): 657–668. doi: 10.1242/dev.00963.
[5] SARKAR A K, LUIJTEN M, MIYASHIMA S, et al. Conserved factors regulate signalling inshoot and root stem cell organizers [J]. Nature, 2007, 446(7137): 811–814. doi: 10.1038/nature 05703.
[6] MINH-THU P T, KIM J S, CHAE S, et al. A WUSCHEL homeobox transcription factor, OsWOX13, enhances drought tolerance and triggers early flowering in rice [J]. Mol Cells, 2018, 41(8): 781–798. doi: 10.14348/molcells.2018.0203.
[7] ZHAO Y, HU Y, DAI M, et al. The WUSCHEL-related homeobox gene WOX11 is required to activate shoot-borne crown root development in rice [J]. Plant Cell, 2009, 21(3): 736–748. doi: 10.1105/tpc.108.061 655.
[8] DAI M, HU Y, ZHAO Y, et al. Agene represses agene expression required for rice leaf development [J]. Plant Physiol, 2007, 144(1): 380–390. doi: 10.1104/pp.107.095737.
[9] CHENG S, TAN F, LU Y, et al. WOX11 recruits a histone H3K27me3 demethylase to promote gene expression during shoot development in rice [J]. Nucl Acid Res, 2018, 46(5): 2356–2369. doi: 10.1093/nar/ gky017.
[10] LIANG Y. Expression analysis ofandinduring protocorm development [D]. Chengdu: South- west Jiaotong University, 2018.
梁易. 鐵皮石斛原球莖發育過程中與的表達分析 [D]. 成都: 西南交通大學, 2018.
[11] LU Y, LIU Z Y, LYU M, et al. Characterization ofandduring callus and root induction in the shrub species[J]. Plants (Basel), 2019, 8(4): 79. doi: 10.3390/plants804 0079.
[12] STUURMAN J, JAGGI F, KUHLEMEIER C. Shoot meristem main- tenance is controlled by a-gene mediated signal from differen- tiating cells [J]. Genes Dev, 2002, 16(17): 2213–2218. doi: 10.1101/ gad.230702.
[13] KIEFFER M, STERN Y, COOK H, et al. Analysis of the transcription factor WUSCHEL and its functional homologue inreveals a potential mechanism for their roles in meristem maintenance [J]. Plant Cell, 2006, 18(3): 560–573. doi: 10.1105/tpc.105.039107.
[14] COSTANZO E, TREHIN C, VAN DEN BUSSCHE M. The role ofgenes in flower development [J]. Ann Bot, 2014, 114(7): 1545– 1553. doi: 10.1093/aob/mcu123.
[15] West M, Harada J J. Embryogenesis in higher plants: An overview [J]. Plant Cell, 1993, 5(10): 1361–1369. doi: 10.1105/tpc.5.10.1361.
[16] LENHARD M, JURGENS G, LAUX T. Theandgenes fulfil complementary roles inshoot meristem regulation [J]. Development, 2002, 129(13): 3195– 3206. doi: 10.1242/dev.129.13.3195.
[17] CARROLL S B. Homeotic genes and the evolution of arthropods and chordates [J]. Nature, 1995, 376(6540): 479–485. doi: 10.1038/376479 a0.
[18] GEHRING W J, MüLLER M, AFFOLTER M, et al. The structure of the homeodomain and its functional implications [J]. Trends Genet, 1990, 6: 323–329. doi: 10.1016/0168-9525(90)90253-3.
[19] GEHRING W J. Exploring the homeobox [J]. Gene, 1993, 135(1/2): 215–221. doi: 10.1016/0378-1119(93)90068-e.
[20] ARIEL F D, MANAVELLA P A, DEZAR C A, et al. The true story of the HD-Zip family [J]. Trends Plant Sci, 2007, 12(9): 419–426. doi: 10. 1016/j.tplants.2007.08.003.
[21] RICHARDT S, LANG D, RESKI R, et al. PlanTAPDB, a phylogeny- based resource of plant transcription-associated proteins [J]. Plant Physiol, 2007, 143(4): 1452–1466. doi: 10.1104/pp.107.095760.
[22] PALOVAARA J, HAKMAN I. Conifer-related homeodomain transcription factors, developmental consideration and expression dynamic ofduringsomatic embryogenesis [J]. Plant Mol Biol, 2008, 66(5): 533–549. doi: 10.1007/s11103-008-9289-5.
[23] NARDMANN J, WERR W. The invention of WUS-like stem cell- promoting functions in plants predates leptosporangiate ferns [J]. Plant Mol Biol, 2012, 78(1/2): 123–134. doi: 10.1007/s11103-011-9851-4.
[24] VAN DER GRAAFF E, LAUX T, RENSING S A. The WUS homeo- box-containing (WOX) protein family [J]. Genome Biol, 2009, 10(12): 248. doi: 10.1186/gb-2009-10-12-248.
[25] MUKHERJEE K, BüRGLIN T R. Comprehensive analysis of animal TALE homeobox genes: New conserved motifs and cases of accelerated evolution [J]. J Mol Evol, 2007, 65(2): 137–153. doi: 10.1007/s00239- 006-0023-0.
[26] JI J B, SHIMIZU R, SINHA N, et al. Analyses oftransgenics provide further evidence for the evolution of thegene family during the regulation of diverse stem cell functions [J]. Plant Signal Behav, 2010, 5(7): 916–920. doi: 10.4161/psb.5.7.12104.
[27] DITENGOU F A, TEALE W D, KOCHERSPERGER P, et al. Mecha- nical induction of lateral root initiation in[J]. Proc Natl Acad Sci USA, 2008, 105(48): 18818–18823. doi: 10.1073/ pnas.0807814105.
[28] DEYHLE F, SARKAR A K, TUCKER E J, et al.regulates cell differentiation during anther development [J]. Dev Biol, 2007, 302 (1): 154–159. doi: 10.1016/j.ydbio.2006.09.013.
[29] VAN DENBUSSCHE M, HORSTMAN A, ZETHOF J, et al. Diffe- rential recruitment oftranscription factors for lateral develop- ment and organ fusion in petunia and[J]. Plant Cell, 2009, 21(8): 2269–2283. doi: 10.1105/tpc.109.065862.
[30] NARDMANN J, JI J B, WERR W, et al. The maize duplicate genesandencode a conserved homeobox gene function in a lateral domain of shoot apical meristems [J]. Development, 2004, 131(12): 2827–2839. doi: 10.1242/dev.01164.
[31] SHIMIZU R, JI J B, KELSEY E, et al. Tissue specificity and evolution of meristematic WOX3 function [J]. Plant Physiol, 2009, 149(2): 841– 850. doi: 10.1104/pp.108.130765.
[32] LIN H, NIU L F, MCHALE N A, et al. Evolutionarily conserved repressive activity of WOX proteins mediates leaf blade outgrowth and floral organ development in plants [J]. Proc Natl Acad Sci USA, 2013, 110(1): 366–371. doi: 10.1073/pnas.1215376110.
[33] Zhou J, Sun J b, Peng M. The research progresses of somatic embryogenesis in higher plant [J]. Chin Agric Sci Bull, 2008, 24(2): 129–133.
周鍵, 孫建波, 彭明. 高等植物體細胞胚胎發生的研究進展 [J]. 中國農學通報, 2008, 24(2): 129–133.
[34] SCHMIDT E D, GUZZO F, TOONEN M A, et al. A leucine-rich repeat containing receptor-like kinase marks somatic plant cells competent to form embryos [J]. Development, 1997, 124(10): 2049–2062. doi: 10. 1242/dev.124.10.2049.
[35] BIRNBAUM K D, ALVARADO A S. Slicing across kingdoms: Regeneration in plants and animals [J]. Cell, 2008, 132(4): 697–710. doi: 10.1016/j.cell.2008.01.040.
[36] DODEMAN V L, DUCREUX G, KREIS M. Zygotic embryogenesis versus somatic embryogenesis [J]. J Exp Bot, 1997, 48(313): 1493– 1509.
[37] MORDHORST A P, TOONEN M A J, DE VRIES S C. Plant embryo- genesis [J]. Crit Rev Plant Sci, 1997, 16(6): 535–576. doi: 10.1080/ 07352689709701959.
[38] CHUGH A, KHURANA P. Gene expression during somatic embryo- genesis: Recent advances [J]. Curr Sci, 2002, 83(6): 715–730.
[39] BREUNINGER H, RIKIRSCH E, HERMANN M, et al. Differential expression ofgenes mediates apical-basal axis formation in theembryo [J]. Dev Cell, 2008, 14(6): 867–876. doi: 10. 1016/j.devcel.2008.03.008.
[40] WU X L, CHORY J, WEIGEL D. Combinations ofactivities regulate tissue proliferation duringembryonic develop- ment [J]. Dev Biol, 2007, 309(2): 306–316. doi: 10.1016/j.ydbio.2007. 07.019.
[41] ZHANG T Q, LIAN H, ZHOU C M, et al. A two-step model for de novo activation ofduring plant shoot regeneration [J]. Plant Cell, 2017, 29(5): 1073–1087. doi: 10.1105/tpc.16.00863.
[42] LIPPMAN Z B, COHEN O, ALVAREZ J P, et al. The making of a compound inflorescence in tomato and related nightshades [J]. PLoS Biol, 2008, 6(11): e288. doi: 10.1371/journal.pbio.0060288.
[43] CHENG S F, HUANG Y L, ZHU N, et al. The rice-related homeobox genes are involved in reproductive organ development, hormone signaling and abiotic stress response [J]. Gene, 2014, 549(2): 266–274. doi: 10.1016/j.gene.2014.08.003.
[44] JüRGENS G. Apical-basal pattern formation inembryo- genesis [J]. EMBO J, 2001, 20(14): 3609–3616. doi: 10.1093/emboj/20. 14.3609.
[45] LU P, PORAT R, NADEAU J A, et al. Identification of a meristem L1 layer-specific gene inthat is expressed during embryonic pattern formation and defines a new class of homeobox genes [J]. Plant Cell, 1996, 8(12): 2155–2168. doi: 10.1105/tpc.8.12.2155.
[46] WETERINGS K, APUYA N R, BI Y P, et al. Regional localization of suspensor mRNAs during early embryo development [J]. Plant Cell, 2001, 13(11): 2409–2425. doi: 10.1105/tpc.010326.
[47] ZUO J R, NIU Q W, FRUGIS G, et al. Thegene promotes vegetative-to-embryonic transition in[J]. Plant J, 2002, 30(3): 349–359. doi: 10.1046/j.1365-313X.2002.01289.x.
[48] GALLOIS J L, NORA F R, MIZUKAMI Y, et al. WUSCHEL induces shoot stem cell activity and developmental plasticity in the root meristem [J]. Genes Dev, 2004, 18(4): 375–380. doi: 10.1101/gad.29 1204.
[49] GALLOIS J L, WOODWARD C, REDDY G V, et al. Combined SHOOT MERISTEMLESS and WUSCHEL trigger ectopic organo- genesis in[J]. Development, 2002, 129(13): 3207–3217. doi: 10.1242/dev.129.13.3207.
[50] WANG F X, SHANG G D, WU L Y, et al. Chromatin accessibility dynamics and a hierarchical transcriptional regulatory network structure for plant somatic embryogenesis [J]. Dev Cell, 2020, 54(6): 742–757.E8. doi: 10.1016/j.devcel.2020.07.003.
[51] CAIRNEY J, ZHENG L, COWELS A, et al. Expressed sequence tags from loblolly pine embryos reveal similarities with angiosperm embryogenesis [J]. Plant Mol Biol, 2006, 62(4–5): 485–501. doi: 10. 1007/s11103-006-9035-9.
[52] WU X L, DABI T, WEIGEL D. Requirement of homeobox geneformeristem growth and maintenance [J]. Curr Biol, 2005, 15(5): 436–440. doi: 10.1016/j.cub.2004.12.079.
[53] BRAND U, FLETCHER J C, HOBE M, et al. Dependence of stem cell fate inon a feedback loop regulated byactivity [J]. Science, 2000, 289(5479): 617–619. doi: 10.1126/science.289.5479. 617.
[54] SCHOOF H, LENHARD M, HAECKER A, et al. The stem cell popu- lation ofshoot meristems is maintained by a regulatory loop between theandgenes [J]. Cell, 2000, 100 (6): 635–644. doi: 10.1016/s0092-8674(00)80700-x.
[55] WAITES R, SIMON R. Signaling cell fate in plant meristems: Three clubs on one tousle [J]. Cell, 2000, 103(6): 835–838. doi: 10.1016/ s0092-8674(00)00186-0.
[56] HADFI K, SPETH V, NEUHAUS G. Auxin-induced developmental patterns inembryos [J]. Development, 1998, 125(5): 879–887. doi: 10.1242/dev.125.5.879.
[57] HE G M, ELLING A A, DENG X W. The epigenome and plant deve- lopment [J]. Annu Rev Plant Biol, 2011, 62: 411–435. doi: 10.1146/ annurev-arplant-042110-103806.
[58] ZHANG C L, WANG J F, WANG X, et al. UF, a WOX gene, regulates a novel phenotype of un-fused flower in tomato [J]. Plant Sci, 2020, 297: 110523. doi: 10.1016/j.plantsci.2020.110523.
[59] DEVEAUX Y, TOFFANO-NIOCHE C, CLAISSE G, et al. Genes of the most conserved WOX clade in plants affect root and flower development in[J]. BMC Evol Biol, 2008, 8: 291. doi: 10. 1186/1471-2148-8-291.
[60] MATSUMOTO N, OKADA K. A homeobox gene,, regulates lateral axis-dependent development offlowers [J]. Genes Dev, 2001, 15(24): 3355–3364. doi: 10.1101/gad. 931001.
[61] SOUER E, VAN DER KROL A, KLOOS D, et al. Genetic control of branching pattern and floral identity duringinflorescence development [J]. Development, 1998, 125(4): 733–742. doi: 10.1242/ dev.125.4.733.
[62] SCANLON M J, FREELING M. The narrow sheath leaf domain deletion: A genetic tool used to reveal developmental homologies among modified maize organs [J]. Plant J, 1998, 13(4): 547–561.
[63] REBOCHO A B, BLIEK M, KUSTERS E, et al. Role of EVER- GREEN in the development of the cymose petunia inflorescence [J]. Dev Cell, 2008, 15(3): 437–447. doi: 10.1016/j.devcel. 2008.08.007.
[64] GALUN E. Patterning of flowers and their members [M]// GALUN E. Plant Patterning. Singapore: World Scientific, 2007: 383–440. doi: 10. 1142/9789812778840_0012.
[65] ZHANG T, LI R N, XING J L, et al. The YUCCA-Auxin-WOX11 module controls crown root development in rice [J]. Front Plant Sci, 2018, 9: 523. doi: 10.3389/fpls.2018.00523.
[66] LIU J C, SHENG L H, XU Y Q, et al.andare involved in the first-step cell fate transition duringroot organogenesis in[J]. Plant Cell, 2014, 26(3): 1081–1093. doi: 10.1105/tpc. 114.122887.
[67] SUER S, AGUSTI J, SANCHEZ P, et al.imparts auxin respon- siveness to cambium cells in[J]. Plant Cell, 2011, 23(9): 3247–3259. doi: 10.1105/tpc.111.087874.
[68] ZHAO S, JIANG Q T, MA J, et al. Characterization and expression analysis ofgenes from wheat and its relatives [J]. Gene, 2014, 537(1): 63–69. doi: 10.1016/j.gene.2013.12.022.
[69] IOIO R D, LINHARES F S, SABATINI S. Emerging role of cytokinin as a regulator of cellular differentiation [J]. Curr Opin Plant Biol, 2008, 11(1): 23–27. doi: 10.1016/j.pbi.2007.10.006.
[70] DE SMET I, JüRGENS G. Patterning the axis in plants–auxin in control [J]. Curr Opin Genet Dev, 2007, 17(4): 337–343. doi: 10.1016/ j.gde.2007.04.012.
[71] INUKAI Y, SAKAMOTO T, UEGUCHI-TANAKA M, et al., which is essential for crown root formation in rice, is a target of an AUXIN RESPONSE FACTOR in auxin signaling [J]. Plant Cell, 2005, 17(5): 1387–1396. doi: 10.1105/tpc.105.030981.
[72] MENG W J, CHENG Z J, SANG Y L, et al. Type-B ARABIDOPSIS RESPONSE REGULATORs specify the shoot stem cell niche by dual regulation of[J]. Plant Cell, 2017, 29(6): 1357–1372. doi: 10.1105/tpc.16.00640.
[73] LIU J, HU X M, QIN P, et al. Thepathway promotes pluripotency acquisition in callus cells during de novo shoot regene- ration in tissue culture [J]. Plant Cell Physiol, 2018, 59(4): 739–748. doi: 10.1093/pcp/pcy010.
[74] Liu L, Wang Y, Shi Z Y, et al. Advances in plant stem cell culture [J]. Chin J Biotechnol, 2018, 34(11): 1734–1741. doi: 10.13345/j.cjb.180 047.
劉連, 王義, 史植元, 等. 植物干細胞培養研究進展 [J]. 生物工程學報, 2018, 34(11): 1734–1741. doi: 10.13345/j.cjb.180047.
[75] MAYER K F X, SCHOOF H, HAECKER A, et al. Role ofin regulating stem cell fate in theshoot meristem [J]. Cell, 1998, 95(6): 805–815. doi: 10.1016/s0092-8674(00)81703-1.
[76] LEIBFRIED A, TO J P C, BUSCH W, et al. WUSCHEL controls meristem function by direct regulation of cytokinin-inducible response regulators [J]. Nature, 2005, 438(7071): 1172–1175. doi: 10.1038/ nature04270.
[77] ZHOU Y, LIU X, ENGSTROM E M, et al. Control of plant stem cell function by conserved interacting transcriptional regulators [J]. Nature, 2015, 517(7534): 377–380. doi: 10.1038/nature13853.
[78] SU Y H, ZHOU C, LI Y J, et al. Integration of pluripotency pathways regulates stem cell maintenance in theshoot meristem [J]. Proc Natl Acad Sci USA, 2020, 117(36): 22561–22571. doi: 10.1073/ pnas.2015248117.
[79] STAHL Y, WINK R H, INGRAM G C, et al. A signaling module controlling the stem cell niche inroot meristems [J]. Curr Biol, 2009, 19(11): 909–914. doi: 10.1016/j.cub.2009.03.060.
[80] CHANDLER J, NARDMANN J, WERR W. Plant development revolves around axes [J]. Trends Plant Sci, 2008, 13(2): 78–84. doi: 10.1016/j. tplants.2007.11.010.
[81] IWASAKI M, PASZKOWSKI J. Epigenetic memory in plants [J]. EMBO J, 2014, 33(18): 1987–1098. doi: 10.15252/embj.201488883.
[82] SERMAN A, VLAHOVI? M, SERMAN L, et al. DNA methylation as a regulatory mechanism for gene expression in mammals [J]. Coll Antropol, 2006, 30(3): 665–671.
[83] XING L B, LI Y M, QI S Y, et al. Comparative RNA-sequencing and DNA methylation analyses of apple (Borkh.) buds with diverse flowering capabilities reveal novel insights into the regulatory mechanisms of flower bud formation [J]. Plant Cell Physiol, 2019, 60(8): 1702–1721. doi: 10.1093/pcp/pcz080.
[84] XING L B, QI S Y, ZHOU H, et al. Epigenomic regulatory mechanism in vegetative phase transition of[J]. J Agric Food Chem, 2020, 68(17): 4812–4829. doi: 10.1021/acs.jafc.0c00478.
[85] LI W, LIU H, CHENG Z J, et al. DNA methylation and histone modifications regulateshoot regeneration inby modulatingexpression and auxin signaling [J]. PLoS Genet, 2011, 7(8): e1002243. doi: 10.1371/journal.pgen.1002243.
[86] LIAN G B, DING Z W, WANG Q, et al. Origins and evolution of WUSCHEL-related homeobox protein family in plant kingdom [J]. Sci World J, 2014, 2014: 534140. doi: 10.1155/2014/534140.
[87] BERTRAND C, BERGOUNIOUX C, DOMENICHINI S, et al.histone acetyltransferase AtGCN5 regulates the floral meristem activity through thepathway [J]. J Biol Chem, 2003, 278(30): 28246–28251. doi: 10.1074/jbc.M302787200.
[88] SALVINI M, FAMBRINI M, GIORGETTI L, et al. Molecular aspects of zygotic embryogenesis in sunflower (L.): Correlation of positive histone marks withexpression and putative link[J]. Planta, 2016, 243(1): 199–215. doi: 10.1007/s00425-015-2405-2.
[89] PI L M, AICHINGER E, VAN DER GRAAFF E, et al. Organizer- derived WOX5 signal maintains root columella stem cells through chromatin-mediated repression ofexpression [J]. Dev Cell, 2015, 33(5): 576–588. doi: 10.1016/j.devcel.2015.04.024.
[90] IKEUCHI M, IWASE A, SUGIMOTO K. Control of plant cell diffe- rentiation by histone modification and DNA methylation [J]. Curr Opin Plant Biol, 2015, 28: 60–67. doi: 10.1016/j.pbi.2015.09.004.
Roles of WOX Transcription Factors in Plant Development
ZENG Danqi1,2, HE Chunmei1, YU Zhenming1, SI Can1,2, CHEN Jing1, HUANG Lei1,2, Li Dongmei3, DUAN Jun1*
(1. Guangdong Provincial Key Laboratory of Applied Botany, Key Laboratory of South China Agricultural Plant Molecular Analysis and Gene Improvement, South China Botanical Garden, Chinese Academy of Sciences, Guangzhou 510650, China; 2. University of Chinese Academy of Sciences,Beijing 100049, China; 3. Shunde Polytechnic,Foshan 528300, Guangdong,China)
WOX (WUSCHEL-related homeobox) transcription factors (TFs) are closely associated with plant development, including embryogenesis, somatic embryogenesis, flower and root development, the initiation and maintenance of callus, and stem cell maintenance in plants. Increasing studies demonstrated that WOX TFs play a crucial role in plant development. Studied on the regulatory mechanisms of WOX in plant development provide a breakthrough in promoting plant development and constructing excellent plant phenotypes. The regulation of WOX TFs in plant development was reviewed, and the developmental processes by integrating WOX and epigenetic regulation were discussed, which aimed at providing inspiration for the mechanism of WOX TFs in regulating plant development.
WOX transcription factor; Plant development; Transcriptional regulation; Review
10.11926/jtsb.4520
2021-09-03
2022-03-07
中國科學院華南農業植物分子分析與遺傳改良重點實驗室開放課題(KF202008);國家自然科學基金項目(32071819)資助
This work was supported by the Project of Key Laboratory of South China Agricultural Plant Molecular Analysis and Genetic Improvement, South China Botanical Garden, Chinese Academy of Sciences (Grant No. KF202008), and the National Natural Science Foundation of China (Grant No. 32071819).
曾丹琦(1997生),女,博士研究生,從事鐵皮石斛生物技術育種研究。E-mail: ZengDanqi20@scbg.ac.cn
. E-mail: duanj@scib.ac.cn

