楓香C4H基因的克隆及生物信息學分析
劉雄盛 尹國平 肖玉菲 王仁杰 黃榮林 王勇
楓香基因的克隆及生物信息學分析
劉雄盛 尹國平 肖玉菲 王仁杰 黃榮林 王勇
(廣西壯族自治區林業科學研究院/廣西優良用材林資源培育重點實驗室 廣西南寧 530002)
肉桂酸-4-羥基化酶(cinnamate 4- hydroxylase,C4H)是花色素苷合成過程第二步中的重要酶。以變色期楓香葉片為試驗材料,根據前期研究獲得的楓香葉片RNA-Seq數據庫中篩選的基因序列設計一對引物,利用RT-PCR技術克隆核苷酸序列,命名為;將基因上傳至GenBank數據庫獲得楓香基因的登錄號(OL871170),并對其進行生物信息學分析。結果顯示:楓香基因長度為1 551 bp,包含一個1 518 bp的完整開放閱讀框,編碼505個氨基酸;保守結構域分析顯示,基因編碼的蛋白包含一個p450家族蛋白的結構域,分子量為58.00 kDa,理論等電點為9.13,為親水性的不穩定蛋白質,定位在內質網,存在多個磷酸化位點,無信號肽,不含有跨膜結構域,蛋白的二級結構主要為-螺旋和無規則卷曲,基因在氨基酸水平與蓮同源性最高(94.5%);進化分析結果表明,與青脆枝親緣關系相對較近。本研究成功克隆了楓香基因,并分析了的蛋白質結構功能,對下一步利用基因工程技術進行楓香葉色調控及新品種培育具有重要意義。
楓香;肉桂酸-4-羥基化酶;基因克隆;生物信息學分析
楓香(Hance)為落葉喬木,屬于金縷梅科(Hamamelidaceae)楓香樹屬()[1]。楓香木材紋理美觀,耐腐耐蟲,落葉量大,可兼用于食用菌產業,經濟價值高,應用前景廣闊,是我國優良的鄉土樹種;耐火力和適應性強,天然條件下易更新,有荒山先鋒樹種之稱;樹干高大通直,枝葉繁茂,入秋后,其葉片顏色會由綠色變成紅色、紫色等,極具觀賞價值[2],已成為優良的城市景觀彩葉樹種。目前,國內外對楓香葉色方面的研究多集中在葉色變化與葉片組織結構[3]、花色素苷含量等生理基礎[4]及季節變化之間的關系[5-6],而對于楓香葉片變色的分子機理及其關鍵基因的研究鮮有報道。
楓香葉色變化過程中起決定性作用的主要是花色素苷[7],花色素苷合成需要很多酶參與。肉桂酸-4-羥基化酶(cinnamate 4- hydroxylase,)亦稱反式肉桂酸-4-單氧化酶(trans-cinnamate 4-monooxygenase)[8],是花色素苷合成過程第二步中的重要酶。歸為亞家族,是細胞色素P450氧化酶家族成員之一[9],因在植物次生代謝中具有重要作用,基因研究已獲得國內外學者的關注[10]。迄今,有關學者已從斑地錦()[11]、膜莢黃芪()[12]、滇水金鳳(I)[13]、白梨()[14]、山葡萄()[15]、芍藥()[16]、青稞()[17]、桂花()[18]等50多種植物中克隆出基因,但有關楓香基因克隆及其相關特性的研究尚未見報道。
本研究以楓香無性系葉片為試驗材料,利用RT-PCR分離克隆其基因,并借助多種生物信息學軟件分析其序列,獲得其蛋白質理化性質和結構、氨基酸同源性、系統發育等特點,為楓香花色素合成途徑中關鍵酶基因功能的研究奠定基礎,對下一步應用基因工程技術進行楓香的葉色改良及新品種的培育具有重要意義。
1 材料與方法
1.1 材料
1.1.1 供試材料 供試材料采自廣西林科院森林經營研究所苗圃,該圃地處南寧市郊,屬濕潤的亞熱帶季風氣候。選用長勢優良、變色效果良好的楓香優良無性系葉片作為供試材料,用超純水將樣品洗凈后置于液氮中處理,然后儲存于–80℃超低溫冰箱中備用。
1.1.2 主要試劑 RNA提取試劑盒(天根生化科技有限公司);反轉錄試劑盒(TaKaRa杭州公司);質粒提取和DNA凝膠回收試劑盒(美國Axygen公司);瓊脂糖(美國海晉生物);克隆載體pEASY?-T5 Zero、大腸桿菌trans1-T1(Transgen中國公司);引物合成及測序委托上海生工生物公司完成。
1.2 方法
1.2.1基因特異引物設計 根據楓香轉錄組測序得到的序列,采用Blast分析其基因保守區,利用primer3 plus(http://www.primer3p lus.com/cgi-bin/dev/primer3plus.cgi),根據expasyTr- anslate tool(https://web.expasy.org/tran slate/)譯出的最長編碼區,在編碼區兩端設計引物:F(ATGGATCTTCTCCTCTTGGAGAAAA)/R(TCAAAAGGATCTCGGCTTTGCAACA)。
1.2.2 楓香葉片RNA的提取和反轉錄 以楓香葉片作為試驗材料,使用相應試劑盒提取總RNA,瓊脂糖凝膠電泳檢測RNA質量,用合格的RNA反轉錄合成第一鏈cDNA,獲得cDNA樣品。
1.2.3 楓香基因PCR擴增 以cDNA為模板,對cDNA片段進行擴增。PCR擴增體系(50 μL):模板2.0 μL+-F 1.0 μL+-R 1.0 μL+2×Taq PCR Master Mix 25 μL+ddH2O 21.0 μL。擴增程序為:95℃預變性5 min,95℃變性30 s,54℃退火30 s,72℃延伸120 s,35個循環;72℃再延伸120 s。采用1.0%瓊脂糖凝膠電泳檢測cDNA的PCR擴增產物并進行純化回收。
1.2.4 載體構建 在16℃條件下,將回收產物與pMD19-T載體連接構建重組質粒,然后轉化大腸桿菌trans1-T1。挑取陽性克隆,用引物進行PCR驗證并測序。
1.2.5 測序序列分析 分析預測開放閱讀框:NCBI ORFFinder;分析蛋白保守結構域:NCBI CD-Search;序列的相似性及同源性查找:NCBI Protein BLAST;預測理化特性:ExPASy ProtParam tool;預測編碼蛋白的疏水性:ProtScale;亞細胞定位:Euk-mPLoc 2.0;蛋白質的N-磷酸化預測:NetPho 3.1;預測蛋白質二級結構:PSIPRED和SOPMA;同源三級結構建模分析:SWISS- MODEL;跨膜結構域分析:TMHMM Server 2.0;預測氨基酸信號肽:SignalP 4.1;序列同源性比較:DNAMAN軟件的Multiple Alignment;通過MEGA (version 7.0.26)軟件,采用相鄰連接法(neighbor- joining NJ,執行參數為Bootstrap method 1000, Pairwise deletion)構建系統進化樹。
2 結果與分析
2.1 楓香LfC4Hb基因克隆
以楓香葉片材料中RNA反轉錄得到的cDNA為模板,根據前期研究轉錄組測序得到的(GenBank:OL871171)序列設計的特異引物,進行RT-PCR擴增后獲得了1條1 500 bp左右的條帶,經瓊脂糖凝膠電泳檢測可見明顯條帶(圖1-A),與預期大小一致。將PCR產物回收純化,連接轉化大腸桿菌(Trans1-T1),挑取單菌落進行菌落PCR驗證,瓊脂糖凝膠電泳結果如圖1-B所示。將陽性菌液進行測序,所得基因和氨基酸序列見圖2。將基因測序結果與序列進行核苷酸序列比對,發現二者之間存在7個堿基差異(圖3),擴增核苷酸序列與序列基本一致,序列相似度達到97.29%;7個堿基的變化僅引起了1個氨基酸的變化(圖4),氨基酸序列一致性達到99.8%,表明克隆得到的序列為楓香基因序列,將其命名為(GenBank: OL871170)。的cDNA序列全長為1 551 bp,其中5¢非翻譯區長15 bp,3¢非翻譯區長18 bp。
2.2 LfC4Hb保守結構域分析
對基因編碼區進行翻譯,發現其包含一個以ATG為起始密碼子、TAG為終止密碼子的完整開放閱讀框,長度為1 518 bp,共編碼505個氨基酸。對氨基酸序列進行比對分析,結果(圖5)顯示,基因編碼的蛋白為多結構域蛋白,序列中包含p450家族蛋白的結構域。
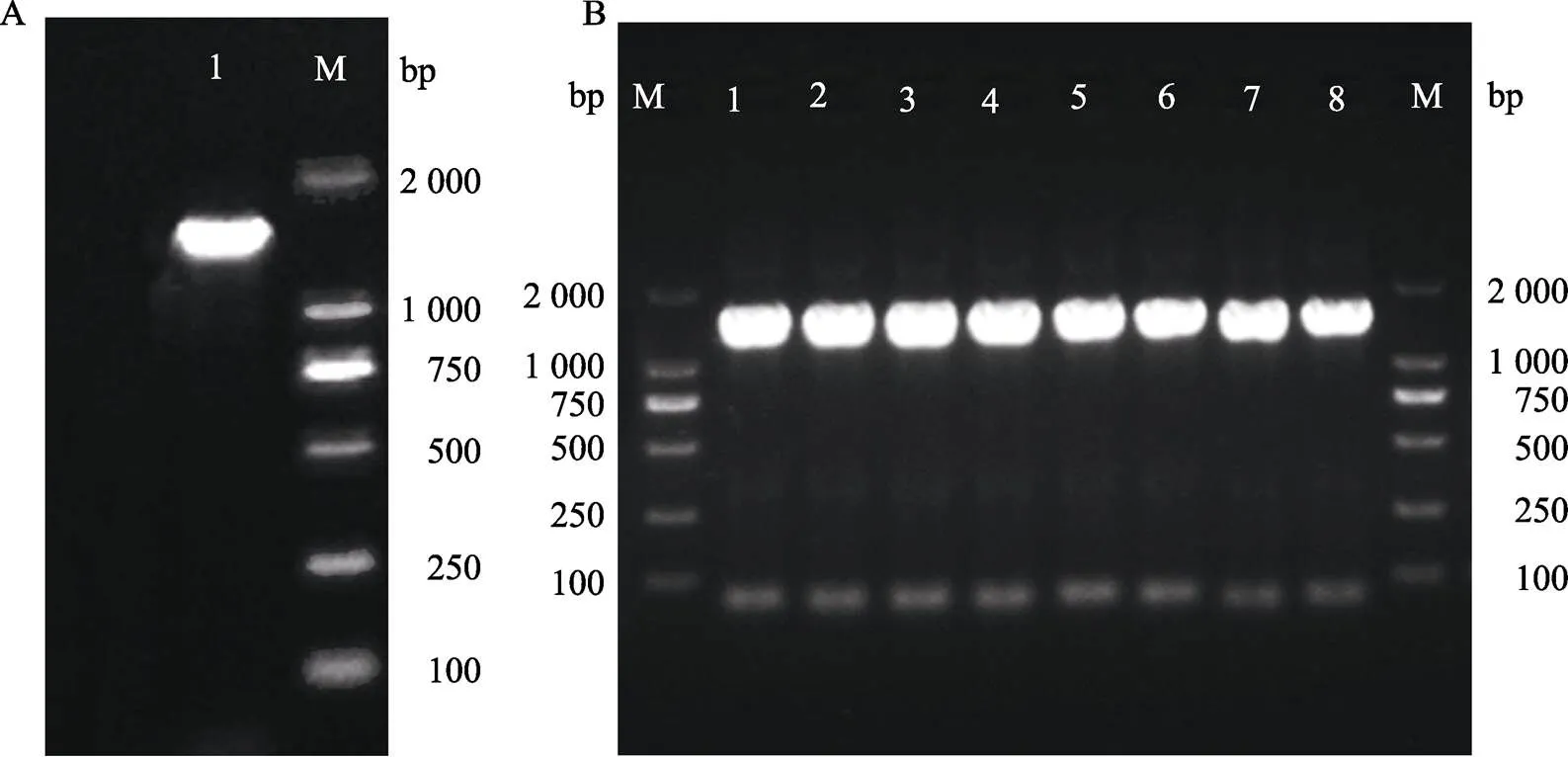
A. 目的基因克隆(1為目的基因);B. 菌落PCR驗證陽性克隆(1~8為陽性克隆)。

A. 目的基因cDNA序列;B. 目的氨基酸序列。
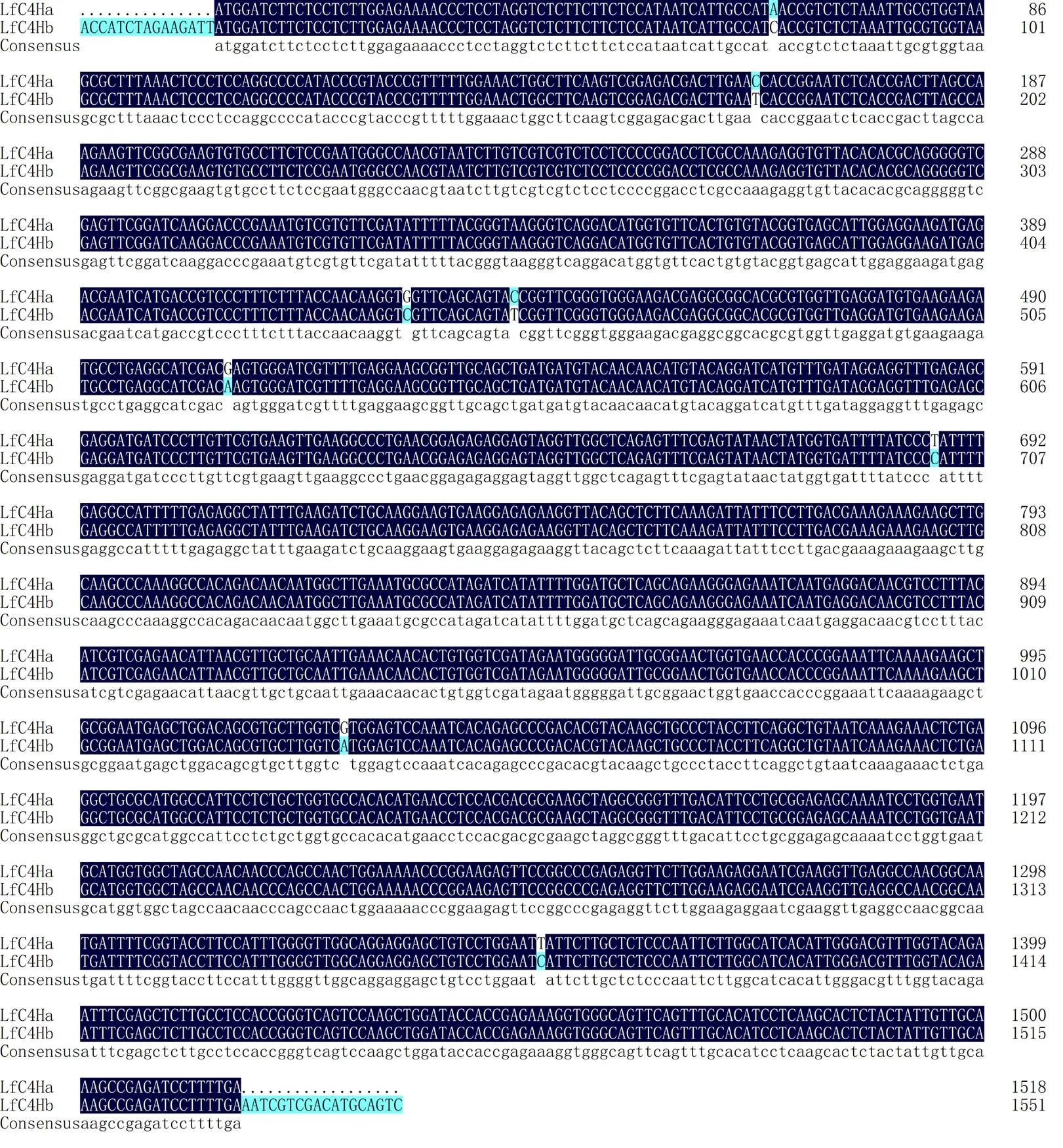
圖3 目的基因與LfC4Ha基因序列比對

圖4 目的基因與LfC4Ha氨基酸序列比對

圖5 LfC4Hb編碼蛋白的保守結構域分析
2.3 LfC4Hb蛋白理化性質分析與蛋白質結構預測
對LfC4Hb蛋白理化性質進行分析,結果發現,LfC4Hb蛋白質由505個氨基酸殘基組成,氨基酸序列的預測結果見表1。由表1可知,Leu、Lys、Val、Glu、Arg氨基酸的使用頻率相對較高,使用頻率依次為12.3%、7.1%、7.1%、7.1%、6.7%。LfC4Hb蛋白分子式為C2638H4173N715O724S16,原子總數為8 266,分子量為58.00 kDa,其中帶正電荷的氨基酸殘基(Arg+Lys)有70個,帶負電荷的氨基酸殘基(Asp+Glu)有61個,理論等電點為9.13;LfC4Hb蛋白氨基酸殘基中以A17(3.156)疏水性最強,A196(–2.722)親水性最強,疏水性氨基酸的數量小于親水性氨基酸的數量,平均親水系數為-0.231(圖6),不穩定指數為46.61,說明LfC4Hb蛋白為親水性的不穩定蛋白質。利用Euk-mPLoc 2.0進行在線亞細胞定位分析,結果表明,LfC4Hb蛋白為膜蛋白,定位于內質網。LfC4Hb蛋白序列的磷酸化位點預測結果顯示,LfC4Hb蛋白存在豐富的磷酸化位點,有潛在磷酸化位點35個(圖7)。
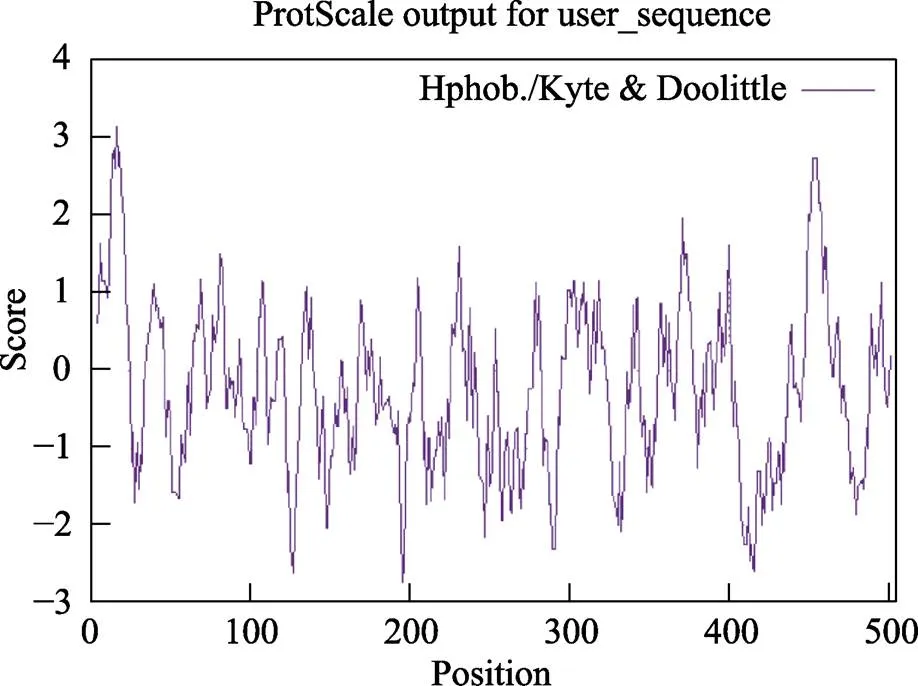
圖6 LfC4Hb蛋白氨基酸序列的親/疏水性分析
圖7 LfC4Hb蛋白氨基酸序列的磷酸化位點預測
對蛋白的二級結構進行預測分析,結果顯示,該蛋白具有20段-螺旋(Alpha helix)和9段-折疊(Beta turn)結構(圖8),-螺旋占比為46.53%,無規則卷曲(Random coil)占比為34.46%,延伸鏈(Extended strand)占比為14.26%,-折疊占比為4.75%,說明該蛋白的二級結構主要為-螺旋和無規則卷曲。
運用SWISS-MODEL對LfC4Hb進行三級結構同源建模時發現,數據庫中匹配度最高的模板是6vby.1.A,建模區域32~502位氨基酸中序列一致性為76.91%,注釋為肉桂酸-4-羥基化酶即C4H蛋白。將LfC4Hb預測的三級結構(圖9)與6vby.1.A模板相比較,可見蛋白結構組成、裝配形式基本一致,從蛋白三級結構來推測,LfC4Hb可能具有與之類似的羥化酶活性,與預期相符。
2.4 LfC4Hb信號肽、跨膜區預測
蛋白質的信號肽序列位于其N端,在蛋白質后被切除,作用是指導分泌性蛋白合成,通常包含疏水核心區、信號肽N端和C端[19]。本研究分析了楓香LfC4Hb的信號肽序列,結果發現,該蛋白序列并不存在信號肽(圖10)。同時對的跨膜區域進行預測,結果表明,LfC4Hb蛋白不含有跨膜結構域(圖11)。

圖8 LfC4Hb編碼蛋白質二級結構分析
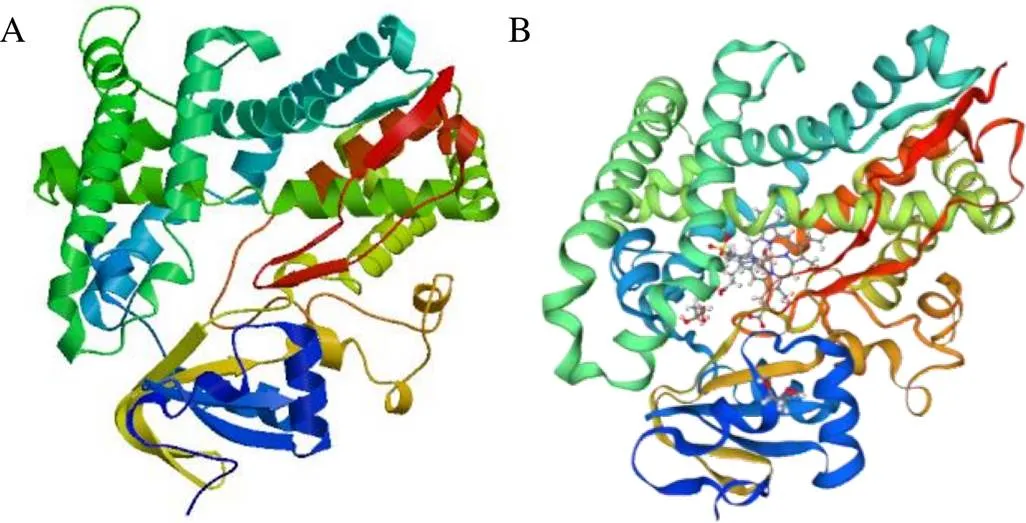
A. LfC4Hb編碼蛋白三級結構;B. 6vby.1.A模板。
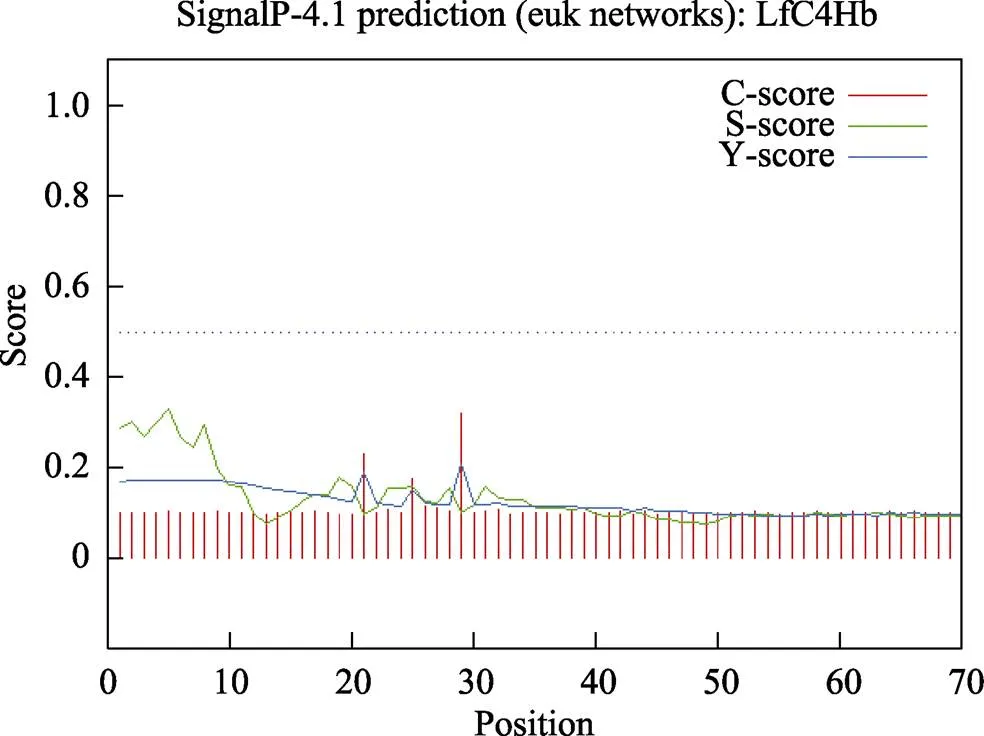
圖10 LfC4Hb信號肽預測
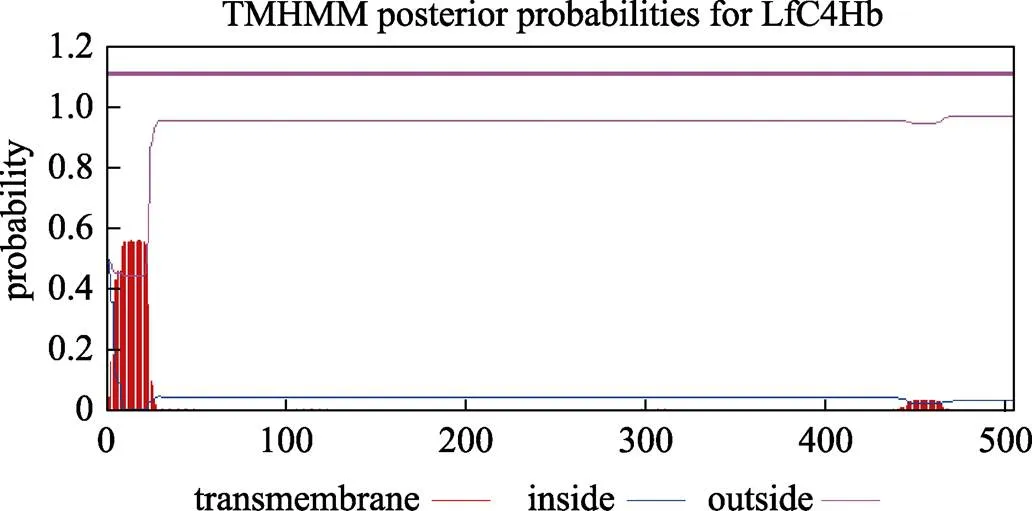
圖11 LfC4Hb跨膜區預測
2.5 LfC4Hb蛋白的同源比對分析
通過NCBI數據庫查詢到其他植物基因的氨基酸序列,利用DNAman軟件進行比對分析。結果表明,楓香LfC4Hb蛋白與10個不同的植物均有較高的同源性。楓香基因在氨基酸水平與蓮()同源性最高(94.5%),與青脆枝()同源性為94.1%,與喜樹()同源性為93.9%,與蒂羅花()同源性為92.5%,與澳洲堅果()同源性為91.7%,與紫竹()同源性為91.3%,與蕓香唐松草()同源性90.9%,與芍藥()和雷公藤()同源性均為90.7%,與大麻槿()同源性為90.5%。分析還顯示,LfC4Hb蛋白含有P450超家族的特征序列:FGVGRRSCPG(血紅素結合域)和PPGPIPVG(富含脯氨酸的保守區域)(圖12),同時還具有CYP73A1的5個特征性底物結合位點(substrate recognition sites, SRS):SRTRNVVFDTFTGKGQ MVFTVY(SRS1)、YNYGDFIPI(SRS2)、VENIN VAAIETTLWS(SRS3)、RMAIPLLVPHMNL(SRS4)以及KGGQFSLHI(SRS5)。
2.6 LfC4Hb蛋白的系統進化樹構建
從NCBI數據庫中篩選出與LfC4Hb氨基酸序列同源性較高的蛋白共9個,分別為芍藥(,AGG55322.1)、雷公藤(,XP_038693505.1)、蕓香唐松草(,KAF5177303.1)、青脆枝(,QNS29969.1)、蓮(,XP_010252045.1)、蒂羅花(,XP_043723676.1)、澳洲堅果(,XP_042501716.1)、紫竹(,XP_021297241.1)、大麻槿(,AGJ84133.1)。利用MEGA 7.0.26軟件的Neighbor-Joining法構建系統進化樹(圖13),發現楓香LfC4Hb蛋白與青脆枝親緣關系相對較近,與紫竹、大麻槿親緣關系較遠。

紅線方框. 富含脯氨酸和血紅素結合域的保守區域; 黑線方框. 底物結合位點(SRS1~SRS5)。
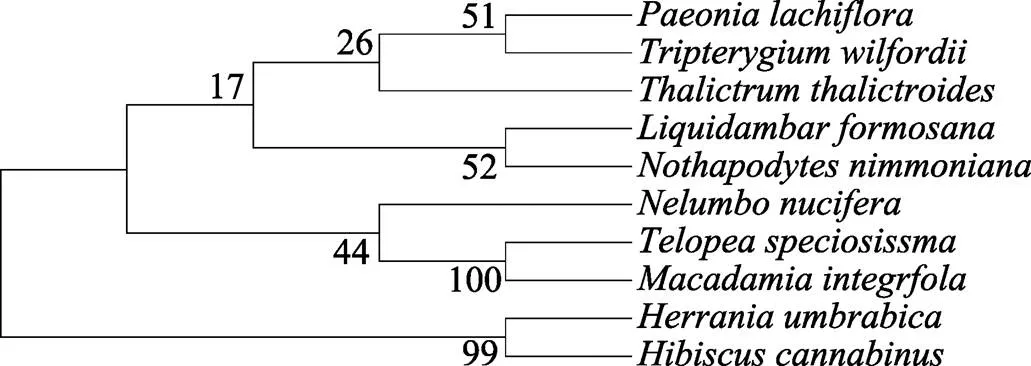
圖13 LfC4Hb系統進化樹
3 討論與結論
苯丙烷代謝途徑是植物次生代謝的重要途徑之一,對于植物的生長發育具有重要作用[20]。通常認為,此途徑參與一系列生物反應形成花色素苷,并由幾種酶共同參與完成催化[21],而C4H是其中的關鍵酶之一,對基因的克隆與功能研究已有一些報道[14,22]。
本研究克隆得到的楓香基因cDNA全長序列,命名為;該基因編碼區存在一個長度為1 518 bp的完整開放閱讀框,共編碼505個氨基酸,與斑地錦[11]、龍血樹[23]一致。基因相對而言,以小基因家族存在,是遺傳多樣性低、保守性較高的基因,杜仲(,Unigene9197)基因與川椒(ACF19421.1)、馬鈴薯(ABC69046.1)、紫草(, BAB71716.1)的C4H氨基酸序列相似性高達90%以上[24],龍血樹與擬南芥的基因同源性亦達到92%以上[23]。本研究中基因在氨基酸水平與蓮、青脆枝、喜樹等的相似性也均高達90%以上,再次印證了植物中C4H蛋白具保守性這一結論。對LfC4Hb蛋白親水性分析發現,其為親水性的不穩定蛋白,與榅桲C4H[25]蛋白一致。預測LfC4Hb蛋白的二級結構發現,其由無規卷曲、-螺旋、-折疊構成,這與爬山虎、山葡萄等C4H蛋白二級結構相同[15],再次證明了為楓香的基因,且與已報道的基因的同源性極高。
本研究分離克隆了楓香的基因,豐富了基因資源,也為探討楓香苯丙烷類次生代謝途徑(包括黃酮類化合物生物合成途徑)奠定了一定的基礎,同時為楓香葉色新品種的培育提供了可操作基因。但楓香葉色的變化也受光照、溫度等條件的影響,這些因素是否影響基因的表達尚不明確。在開展下一步研究時,可在本研究基礎上探討楓香的轉錄因子對結構基因的作用以及表達特性,從而進一步在分子層面揭示楓香葉片的呈色和變色機理,實現楓香的葉色調控。
[1] 李勇. 楓香速生優良無性系組織培養技術體系[J]. 廣西林業科學, 2019, 48(2): 252-256.
[2] 楊繼生. 楓香葉色變化過程中葉片結構和生理特征的研究[D]. 南寧: 廣西大學, 2020.
[3] 尹國平, 劉雄盛, 蔣燚, 等. 楓香變紅過程中葉片組織結構、光合特性及色素含量變化研究[J]. 廣西植物, 2021: 5-10. https: //kns. cnki. net/kcms/detail/detail. aspx?dbcode= CAPJ&dbname=CAPJLAST&filename=GXZW20210719001&uniplatform=NZKPT&v=nld-w7thijbKahM_pf_mFmvAP b0rj61gXmeGojguZnooWaZqSfDmG1PJ_oUy4r3E
[4] 唐生森, 陳虎, 覃永康, 等. 楓香秋季變色期葉色變化及其生理基礎[J]. 廣西植物, 2021, 41(12): 2 061-2 068.
[5] 王冬雪, 孫海菁, 德永軍, 等. 不同光質處理對楓香幼苗葉色的影響[J]. 林業科學研究, 2019, 32(4): 158-164.
[6] 賴玖鑫. 楓香葉色季節性變化及常彩葉機理研究[D]. 北京: 中國林業科學研究院, 2020.
[7] 李文鑫. 楓香優良無性系篩選及紫葉品系葉色變化相關基因模塊鑒定[D]. 長沙: 中南林業科技大學, 2020.
[8] 王安娜, 王嬋嬋, 吳蕾, 等. 大豆基因克隆及生物信息學分析[J]. 東北農業大學學報, 2010, 41(4): 12-16+161.
[9] 趙樂, 馬利剛, 楊澤岸, 等. 獨行菜基因克隆與表達分析[J]. 藥學學報, 2017, 52(5): 821-831.
[10] 魏建華, 宋艷茹. 木質素生物合成途徑及調控的研究進展[J]. 植物學報, 2001, 43(8): 771-779.
[11] 郭三保, 黃勝和, 鄒嘉軒, 等. 斑地錦肉桂酸4-羥基化酶基因及啟動子的克隆與分析[J]. 分子植物育種,2021:1-10. https: //kns. cnki. net/kcms/detail/detail. aspx?dbcode= CAPJ&dbname=CAPJLAST&filename=FZZW20210312004&uniplatform=NZKPT&v=_MB1PS_Ch2CPdB0xVtwonH OaDpHHW3tgUsQ87cWvL_MdHvb-jt6X9lAvz9DbX9CY.
[12] 馮藝川, 趙洋, 全雪麗, 等. 膜莢黃芪基因的克隆及表達分析[J]. 分子植物育種, 2021, 19(1): 130-136.
[13] 朱佳鵬, 高珊, 羅超, 等. 滇水金鳳C4H基因的克隆及序列分析[J]. 分子植物育種, 2020, 18(19): 6 247-6 255.
[14] LI G, LIU X, ZHANG Y, et al. Cloning and functional characterization of two cinnamate 4-hydroxylase genes from[J]. Plant Physiology and Biochemistry, 2020, 156(11): 135-145.
[15] 陳蒙, 劉海峰. 山葡萄基因的克隆表達及遺傳轉化分析[J]. 西南大學學報(自然科學版), 2019, 41(10): 11- 21.
[16] 陳德偉, 王昕, 李紅葉, 等. 芍藥基因的克隆及其在鈣處理下的表達特性分析[J]. 分子植物育種, 2018, 16(9): 2 795-2 801.
[17] 羅小嬌, 劉新春, 楊曉云, 等. 青稞基因的克隆及組織表達分析[J]. 植物遺傳資源學報, 2014, 15(3): 589-596.
[18] 曾祥玲, 鄭日如, 羅靖, 等. 桂花基因的克隆與表達特性分析[J]. 園藝學報, 2016, 43(3): 525-537.
[19] 張弢. 不同植物查爾酮合成酶基因的生物信息學分析[J]. 江西農業學報, 2012, 24(6): 5-8.
[20] 張雪, 唐銘浩, 陳蒙, 等. 山葡萄不同著色時期果皮轉錄組測序分析[J]. 果樹學報, 2017, 34(7): 781-789.
[21] 王安娜. 大豆基因克隆及生物信息學分析[D]. 哈爾濱: 東北農業大學, 2010.
[22] Millar D J, Long M, Dononvan G, et al. Introduction of sense constructs of cinnamate 4-hydroxylase (CYP73A24) in transgenic tomato plants shows opposite effects on flux into stem lignin and fruit flavonoids[J]. Phytochemistry, 2007, 68(11): 1 497-1 509.
[23] 李爽, 楊春勇, 李戈, 等. 國產龍血竭基原植物肉桂酸-4-羥基化酶基因(C4H)分離比較[J]. 分子植物育種, 2021: 1-12. https: //kns. cnki. net/kcms/detail/detail. aspx?dbcode= CAPJ&dbname=CAPJLAST&filename=FZZW20211102001&uniplatform=NZKPT&v=_MB1PS_Ch2AQtWkHZl1T3N 1TSU4AupSkByJqbaqvO8Ayhe5ccomAVF8e1bAJh0vq.
[24] 李鐵柱, 杜紅巖, 王淋. 杜仲基因cDNA全長序列特征分析[J]. 經濟林研究, 2014, 32(1): 34-39.
[25] 車玉紅, 楊波, 郭春苗, 等. 榅桲基因的克隆、序列分析及表達[J]. 經濟林研究, 2020, 38(2): 1-8.
Cloning and Bioinformatics Analysis ofGene of
LIU Xiongsheng YIN Guoping XIAO Yufei WANG Renjie HUANG Ronglin WANG Yong
(Guangxi Forestry Research Institute/Guangxi Key Laboratory of Superior Trees Resource Cultivation, Nanning, Guangxi 530002, China)
Cinnamate 4- hydroxylase() is the second important enzyme in anthocyanin synthesis. A pair of primers were designed based on thegene sequence screened from the RNA-Seq database ofleaves obtained in previous studies and then the nucleotide sequence ofwas cloned by RT-PCR and named. The gene was uploaded to GenBank database to obtain the gene login number (OL871170) of, and the bioinformatics analysis was conducted. The results showed that the length ofgene was 1 551 bp, which contained a complete open reading frame of 1 518 bp and encoded 505 amino acids. The conserved domain analysis showed that there was a p450 family protein domain. The molecular weight was 58.00 kDa and the theoretical isoelectric point was 9.13. The protein encoded bywas an unstable hydrophilic protein located in the endoplasmic reticulum. There were multiple phosphorylation sites,there was no signal peptide and no transmembrane domain. The secondary structure of the protein was mainly α -helix and random coil. The homology ofgene withwas the highest(94.5%).The phylogenetic analysis showed thatwas relatively close to.In this study,gene ofwas successfully cloned and the protein structure and function ofwere analyzed. It will be of great significance for further application of genetic engineering technology in leaf color regulation and breeding of new varieties of
; cinnamate 4- hydroxylase; gene cloning; bioinformatic analysis
S533
A
10.12008/j.issn.1009-2196.2022.06.008
2022-02-24;
2022-03-21
廣西優良用材林資源培育重點實驗室自主課題(No.2019-A-03-02);2021年自治區林業科技推廣示范項目(No.桂林科研[2021]26號)。
劉雄盛(1988—),男,碩士,副研究員,研究方向為森林培育,E-mail:517261654@qq.com。
王勇(1983—),男,碩士,副高級工程師,研究方向為森林培育,E-mail:742722538@qq.com。
(責任編輯 林海妹)

