豌豆蚜可溶型海藻糖酶基因克隆及RNA干擾效應
許靜靜,常永梅,任夢圓,董艷玲,李永強
西北農林科技大學 植物保護學院, 陜西 楊凌 712100
海藻糖是一種非還原性雙糖, 廣泛存在于細菌、 真菌、 昆蟲和植物等許多生物體內[1]. 海藻糖酶能將一分子海藻糖水解為兩分子葡萄糖, 通過糖酵解為各個組織器官提供能量, 或為幾丁質合成提供原料, 因而海藻糖酶在昆蟲體內起著非常重要的作用[2]. 根據是否含有跨膜結構域將昆蟲海藻糖酶基因分為兩類: ① 可溶型海藻糖酶(Tre1), 主要分解細胞內的海藻糖;② 膜結合型海藻糖酶(Tre2), 主要水解食物中的海藻糖[3-4]. 海藻糖酶是海藻糖分解代謝的關鍵酶, 廣泛參與并調控昆蟲的生長發育、 非生物脅迫以及激素反應等, 與昆蟲能量代謝、 幾丁質合成和誘導滯育密切相關. 目前, 已經在甜菜夜蛾Spodopteraexigua[5]、 赤擬谷盜Triboliumcastaneum[6]、 飛蝗Locustamigratoria[7]和灰飛虱Laodelphaxstriatellus[8]等昆蟲中, 通過RNAi技術證實了海藻糖酶基因在能量代謝和幾丁質合成方面發揮著重要作用. Chen等[5]通過顯微注射法對甜菜夜蛾海藻糖酶基因SeTre1和SeTre2進行RNAi研究發現, 基因轉錄水平及幾丁質含量均顯著降低, 且SeTre1和SeTre2分別在昆蟲的表皮和中腸的幾丁質合成中起重要作用. 張倩等[8]通過飼喂法干擾灰飛虱海藻糖酶基因LSTre1和LSTre2, 發現基因轉錄水平分別降低49%和41%, 并導致昆蟲體質量減輕、 死亡率顯著升高. 由于哺乳動物沒有海藻糖代謝系統, 以海藻糖酶為靶標的新農藥對人畜等非靶標生物可能不存在毒害作用[8-9], 因此海藻糖酶便成為新型高效殺蟲劑設計和開發的潛在優良靶標.
豌豆蚜Acyrthosiphonpisum是影響糧食生產的重要害蟲之一, 主要通過取食植物汁液、 誘發煤污病和傳播植物病毒的方式[10-12]為害農作物. 目前, 關于豌豆蚜海藻糖酶的RNA干擾研究尚未見相關報道. 本研究以豌豆蚜為試蟲, 通過基因克隆得到可溶型海藻糖酶基因ApTre-1的全長編碼序列, 運用生物信息學方法對ApTre-1基因和蛋白氨基酸序列進行序列特征分析, 構建系統進化樹分析豌豆蚜海藻糖酶ApTre-1的進化關系, 利用熒光定量PCR(qPCR)技術明確ApTre-1基因在豌豆蚜不同生長發育階段的表達水平;在上述基礎上, 進一步通過顯微注射法和飼喂法測定兩種dsRNA片段對海藻糖酶ApTre-1基因的干擾效率, 以及對試蟲生長發育的影響和致死效應. 本研究對基于RNAi技術的海藻糖酶基因在害蟲防治中的應用進行了初步探索, 可為今后深入研究提供有價值的前期參考.
1 材料與方法
1.1 供試昆蟲
豌豆蚜由西北農林科技大學應用昆蟲學重點實驗室提供, 飼養于人工氣候培養箱內的盆栽蠶豆(“晉農”精選蠶豆)上, 飼養條件: 溫度(21±1) ℃, 相對濕度70%~75%, 光照周期L(光照)∶D(黑暗)=16∶8.
1.2 豌豆蚜海藻糖酶基因的克隆
按照TRNzol Universal Reagent試劑盒(天根)說明書, 對豌豆蚜3齡若蟲進行總RNA提取. 取1 μg總RNA作為模板, 按照HiFiScript gDNA Removal cDNA Synthesis Kit試劑盒(康為世紀)說明書, 去除基因組DNA后, 反轉錄合成cDNA第一條鏈.
基于NCBI(National Center for Biotechnology Information)數據庫中豌豆蚜的基因組序列, 本研究利用大豆蚜Aphisglycines海藻糖酶基因(GeneBank登錄號: JQ246351.1)進行BLAST搜索, 獲得豌豆蚜全基因組數據中編號為Contig13577的序列(GeneBank登錄號: ABLF02013269.1). 通過軟件進一步分析找到豌豆蚜海藻糖酶編碼基因閱讀框后, 利用Primer Premier 5軟件設計特異性引物(表1), 以豌豆蚜cDAN第一鏈為模板, 擴增豌豆蚜的海藻糖酶編碼基因. PCR反應條件: 98 ℃預變性1 min, 98 ℃變性10 s、 55 ℃退火30 s、 72 ℃延伸3 min, 共35個循環. 擴增產物經1%瓊脂糖凝膠檢測后, 進行回收純化. 純化后的DNA通過DNA A-Tailing Kit(TaKaRa)加“A”反應后, 與pMD 19-T Vector(TaKaRa)連接, 構建重組質粒. 將重組質粒轉入大腸桿菌感受態細胞EscherichiacoliDH5α, 菌落PCR鑒定正確后, 送至生工生物工程(上海)股份有限公司(Sangon Biotech)測序.

表1 引物名稱與序列
1.3 豌豆蚜海藻糖酶蛋白ApTre-1生物信息學分析
利用在線軟件Softberry(http: //www.softberry.com)分析豌豆蚜基因組中海藻糖酶基因的內含子和外顯子組成;利用在線軟件ExPASy(https: //web.expasy.org/protparam)預測蛋白質的相對分子質量和理論等電點;通過在線軟件Signal P(https: //services.healthtech.dtu.dk/)和TMHMM(https: //services.healthtech.dtu.dk/)分別對蛋白質的信號肽和跨膜區域進行分析;通過NCBI在線工具BLAST(https: //blast.ncbi.nlm.nih.gov/Blast.cgi)進行蛋白質同源搜索, 應用MEGA 6軟件中的Clustal W進行多重序列比對, 采用鄰位相連法(Neighbor-joining)進行聚類分析, 構建系統進化樹. 重復次數為1 000次, 樹枝上的數字表示bootstrap驗證中該樹枝可信度百分比大于50%的數值(圖1).

圖1 豌豆蚜ApTre-1蛋白與其他昆蟲海藻糖酶系統進化樹
1.4 實時熒光定量PCR檢測ApTre-1基因在不同齡期的表達水平
采用qPCR技術分析ApTre-1基因在豌豆蚜不同齡期(1齡、 2齡、 3齡、 4齡若蚜和無翅成蚜)的表達水平. 根據獲得的豌豆蚜ApTre基因序列設計qRCP特異性引物(表1), 選擇EF-1a和RPS-20作為雙內參基因[13]. 以豌豆蚜1齡若蟲的相對表達量為基準, 以無核酸酶水代替cDNA為陰性對照, 以不加反轉錄酶的核酸代替cDNA排除核酸樣品中基因組DNA污染的可能性. qPCR反應體系(20 μL)為: 5倍稀釋的cDNA模板2 μL, 上、 下游引物各0.8 μL(10 mmol/L), 2×TB GreenPremixExTaqII 10 μL, 無核酸酶水6.4 μL. 反應于Roche Light Cycler 480 實時熒光定量PCR儀(德國Roche Diagnostics GmbH)上進行. qPCR反應條件為: 95 ℃ 5 min, 95 ℃ 5 s, 60 ℃ 1 min, 共40個循環. 根據熔解曲線, 確定引物及擴增特異性. 實驗設置3個生物學重復, 每個生物學重復設置3個技術重復. 采用2-ΔΔCt法計算基因的相對表達量[14].
1.5 體外合成dsRNA
以序列驗證無誤的ApTre為模板進行PCR擴增, 引物見表1. 使用Gel Extraction Kit試劑盒(天根)對PCR產物進行純化回收. 根據T7 RiboMAXTM Express RNAi System試劑盒(Promega)說明書, 進行體外dsRNA合成. 取2 μL dsRNA稀釋10倍后, 使用分光光度計測定dsRNA的濃度, 并通過1%的瓊脂糖凝膠進行檢測, 置于-80 ℃冰箱保存.
1.6 顯微注射法測定ApTre-1基因不同dsRNA片段的RNAi效率
參照葉超[15]的方法, 選擇生長健康、 狀態一致的4齡若蚜, 使用顯微注射器分別注射2種不同的dsApTre-1片段, 以dsGFP為對照. 每只蚜蟲注射dsRNA(1 μg/μL)60 nL, 每個處理10只蚜蟲, 設6個生物學重復. 注射完成后, 將豌豆蚜放入培養皿中新鮮的蠶豆葉片上飼養. 采用qPCR技術檢測注射試蟲2 d后的ApTre-1基因相對表達水平.
1.7 飼喂法測定ApTre-1基因dsRNA片段的RNAi效應檢測
根據Auclair等[16]蚜蟲人工飼料配方配置豌豆蚜的人工飼料, 采用飼蚜器進行飼喂. 飼蚜器為兩端開口的玻璃管, 將封口膜(美國Parafilm)拉至最薄, 附于玻璃管的一端, 使用移液槍將40 μL的人工飼料滴加至封口膜中央(dsRNA的濃度為1 μg/μL), 然后再拉伸一張封口膜蓋在人工飼料上, 使人工飼料均勻平鋪于兩膜之間. 將飼蚜器置于智能人工光照氣候培養箱中, 每2 d更換1次人工飼料. 每個處理10頭若蚜, 重復6次. 每天觀察并統計存活數, 每2 d稱質量記錄1次豌豆蚜體質量. 采用qPCR技術檢測飼喂2 d后ApTre-1基因的相對表達水平.
2 結果與分析
2.1 豌豆蚜海藻糖酶基因ApTre-1克隆及序列分析
利用特異性引物對豌豆蚜海藻糖酶基因進行PCR擴增, 擴增產物經1%瓊脂糖凝膠電泳檢測, 獲得1條大小約為2 300 bp的電泳條帶(圖2). 將目的條帶回收純化后, 經連接、 轉化和測序, 篩選陽性克隆, 得到海藻糖酶基因(ApTre-1)的cDNA序列. 通過Vector NT 軟件對PCR擴增序列進行分析, 確定了豌豆蚜海藻糖酶基因的開放閱讀框. 該基因的開放閱讀框(ORF)為1 770 bp, 編碼589個氨基酸. 在基因組中DNA水平上該基因由9個外顯子和8個內含子組成(圖3). 其編碼的氨基酸序列與NCBI中注釋和預測的豌豆蚜海藻糖酶氨基酸序列(GenBank登錄號: XM001950229.4)完全一致.
使用在線軟件ExPASy預測豌豆蚜海藻糖酶ApTre-1蛋白的相對分子質量為6.83×104;理論等電點(PI)為6.10. 利用Signal P在線預測該蛋白的信號肽, 結果表明ApTre-1蛋白的N段含有一段長為20 aa的信號肽序列. 利用TMHMM預測該蛋白的跨膜結構域, 結果表明ApTre-1蛋白同其他已知昆蟲的可溶型海藻糖酶一樣無跨膜區域. 通過NetNGlyc軟件預測, 發現ApTre蛋白含有4個糖基化位點, 分別位于124,234,346和473位氨基酸. 通過GeneDoc軟件進行蛋白質同源序列比對, 發現ApTre-1蛋白含有“PGGRFRELYYWDTY” “QWDFPNAWPP”2個標簽序列和一個富甘氨酸區域“GGGGEY”. 并且, 發現在蚜蟲等半翅目昆蟲中還含有“YYLNRSQPP” “PRPESYREDY” “IIPVDLN” “MFEKYD”和“GFGWXNG”共5個高度保守區域(圖4).
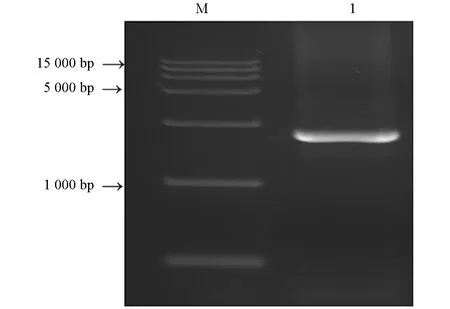
圖2 ApTre-1基因PCR擴增產物
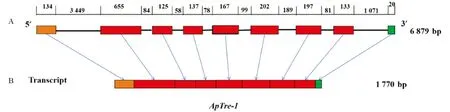
圖A中實心矩形框表示ApTre-1基因的外顯子, 矩形框之間的短線段表示ApTre-1基因的內含子;圖B表示ApTre-1基因轉錄后不含內含子的編碼序列.圖3 ApTre-1基因DNA結構圖

半翅目昆蟲可溶型海藻糖酶GeneBank登錄號: 大豆蚜AgTre-1(AFJ00065.1)、 綠盲蝽AlTre-1(AGK89798.1)、 煙粉虱BtTre-1(JX024261.1)、 灰飛虱LsTre-1(AFL03409.1)、 褐飛虱NlTre-1(ACN85420.1)、 白背飛虱SfTre-1(AQS60672.1). 標簽序列用紅色方框標注, 高度保守區域用藍色方框標注, 富甘氨酸區域用下劃線標注, 信號肽用紫色矩形框標注, 糖基化位點用黑色三角形標注.圖4 ApTre-1蛋白與其他半翅目昆蟲Tre-1氨基酸序列多重比較結果
2.2 豌豆蚜ApTre-1蛋白的分子進化分析
通過ClustalW軟件將豌豆蚜ApTre-1蛋白與其他已知的13種昆蟲海藻糖酶蛋白氨基酸序列進行同源比對, 利用MEGA 6 軟件中的Neighbor-Joining方法進行分子系統進化分析, 得到昆蟲海藻糖酶蛋白分子進化樹(圖1). 從該系統發育樹可以看出, 海藻糖酶分為可溶型海藻糖酶和膜結合型海藻糖酶兩大類, 在分類上屬于同一目的昆蟲均單獨形成一個亞分支, 這與傳統的分類結果一致. 本實驗克隆得到的豌豆蚜ApTre-1蛋白與大豆蚜、 飛虱等可溶型海藻糖酶歸為一個亞分支, 其中豌豆蚜ApTre-1蛋白與大豆蚜Aphisglycines的可溶型海藻糖酶(AgTre-1)聚在更小的一個分支內, 表明其氨基酸序列相似性較高, 與煙粉虱Bemisiatabaci、 褐飛虱Nilaparvatalugens及白背飛虱Sogatellafurcifera可溶型海藻糖酶的氨基酸序列相似性則相對較低.
2.3 豌豆蚜海藻糖酶基因ApTre-1在不同發育時期的表達分析
通過qPCR技術, 對ApTre-1基因在豌豆蚜1~4齡若蟲和成蟲中的表達水平進行檢測分析(圖5). 結果表明:ApTre-1基因在豌豆蚜整個生長發育期間均有表達, 但不同發育階段表達量具有明顯差異, 其mRNA在1齡若蚜中表達水平最高, 顯著高于其他各階段, 約為2齡若蚜的5倍、 成蟲的12倍.
2.4 豌豆蚜海藻糖酶基因ApTre-1的RNA干擾效率
對4齡若蚜進行顯微注射dsRNA后, 放入培養皿中用新鮮蠶豆葉片進行飼喂, 2 d后采用qPCR技術檢測蚜蟲體內ApTre-1基因表達量的變化(圖6), 以綠色熒光蛋白(GFP)的dsGFP為對照. 由圖6可見, 經過顯微注射兩種dsRNA后, 蚜蟲體內ApTre-1基因轉錄水平均受到不同程度的抑制. 在分別注射dsApTre-1-1和dsApTre-1-2片段2 d后,ApTre-1基因的相對表達量分別降低了48.0%和30.3%.
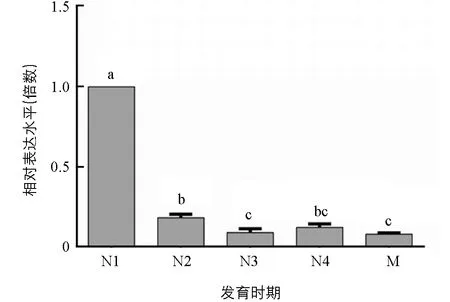
N1,N2,N3,N4和M分別代表1,2,3,4齡若蚜和成蚜;不同小寫字母表示差異具有統計學意義(p<0.05).圖5 ApTre-1基因在豌豆蚜不同發育階段的相對表達量
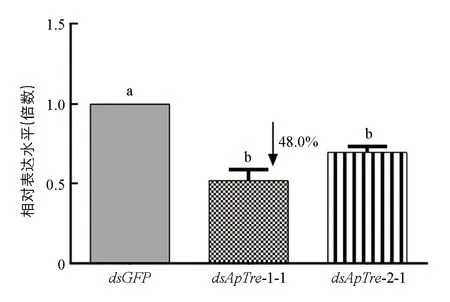
不同小寫字母表示差異具有統計學意義(p<0.05).圖6 不同dsRNA注射2 d后豌豆蚜ApTre-1基因的相對表達量
2.5 飼喂dsApTre-1-1片段對豌豆蚜RNA干擾效率及生長的影響
2.5.1 飼喂dsApTre-1-1片段對豌豆蚜ApTre-1基因的RNA干擾效率
根據以上結果, 選擇其中干擾效率較高的dsApTre-1-1片段與人工飼料混合, 進一步采用飼喂法測定其對靶基因ApTre-1的RNA干擾效率. 以取食含dsGFP飼料的蚜蟲為對照, 檢測取食含dsApTre-1-1飼料2 d后的蚜蟲體內可溶型海藻糖酶基因表達量的變化(圖7). 從圖7中可以看出, 蚜蟲取食含dsApTre-1-1的飼料后與對照組(含dsGFP飼料)試蟲相比,ApTre-1基因表達量降低了27.3%, 差異具有統計學意義(p<0.5%).
2.5.2 飼喂dsApTre-1-1片段對豌豆蚜生長的影響
以飼喂含有質量濃度為1 μg/μLdsGFP和dsApTre-1-1的飼料分別作為非靶標對照組和處理組, 統計第0,2,4,6 d存活豌豆蚜的平均體質量(圖8). 從圖8中可以看出, 連續飼喂含有dsApTre-1的飼料2 d后, 試蟲平均體質量與對照組相比開始顯著降低. 到第6 d時, 蚜蟲實驗組平均體質量為0.88 mg, 與對照組的平均體質量(1.13 mg)差異具有統計學意義(p<1%), 表明試蟲取食dsApTre-1-1片段后, 生長發育受到明顯抑制.
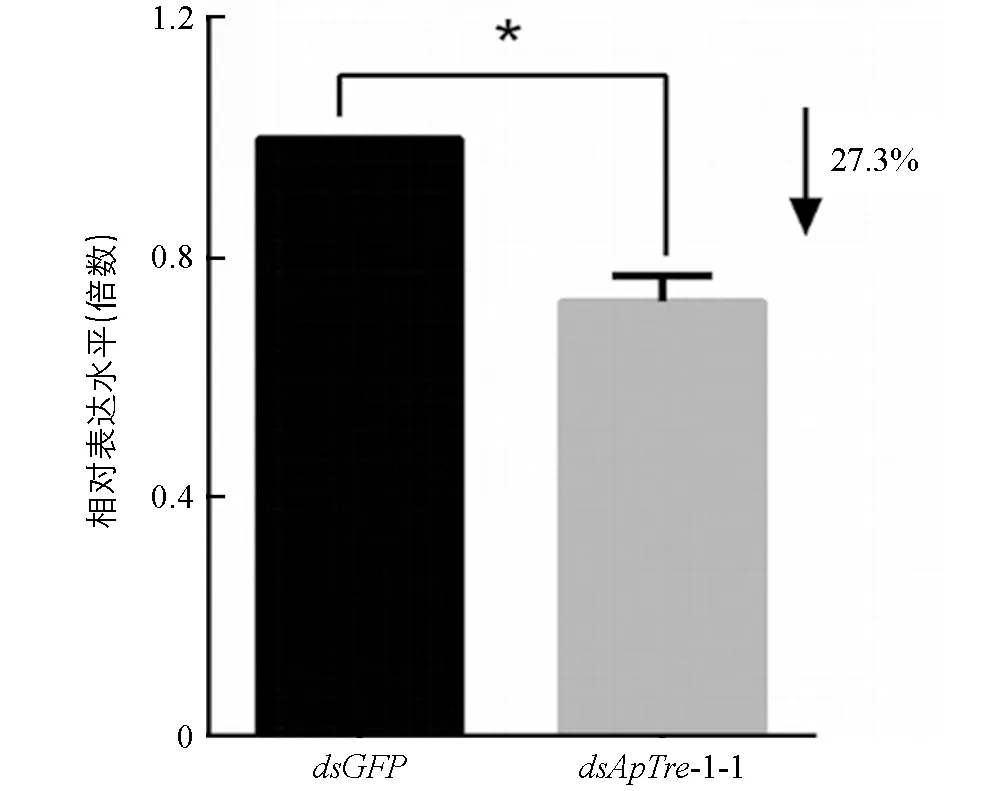
*表示差異具有統計學意義(p<0.05).圖7 dsApTre-1-1飼喂2 d后豌豆蚜ApTre-1基因的相對表達量
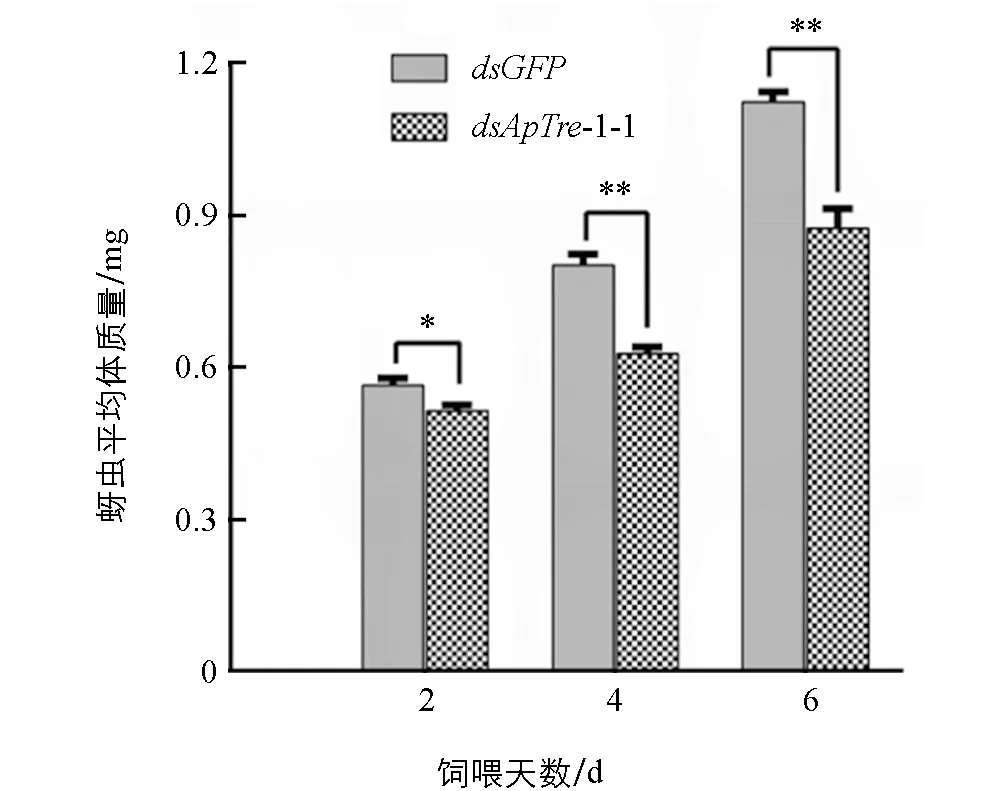
*表示差異具有統計學意義p<0.05, **表示差異具有統計學意義p<0.01.圖8 dsApTre-1-1飼喂后豌豆蚜平均體質量
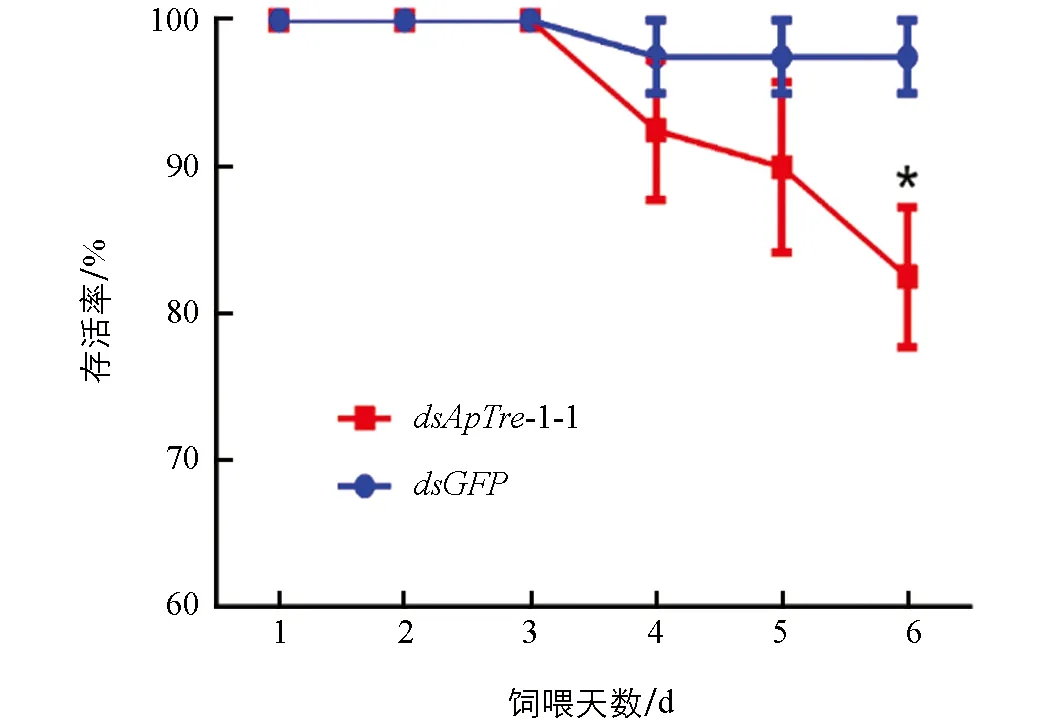
*表示差異具有統計學意義p<0.05.圖9 dsApTre-1-1飼喂后豌豆蚜ApTre-1基因的存活率
2.5.3 飼喂dsApTre-1-1片段對豌豆蚜的致死率
以飼喂含有質量濃度為1 μg/μLdsGFP和dsApTre-1-1的飼料分別作為非靶標對照組和處理組, 統計2齡若蚜取食dsRNA后1~6 d的存活率(圖9). 由圖9可以看出, 連續飼喂含有dsApTre-1-1飼料3 d后, 蚜蟲的存活率開始降低, 到第6 d時蚜蟲的存活率為82.5%, 與對照組之間差異具有統計學意義(p<0.05).
3 討論與結論
海藻糖酶是昆蟲體內重要的調控酶, 自從1992年和2005年分別鑒定出黃粉蟲Tenebriomolitor的可溶型海藻糖酶[17]和家蠶的膜結合型海藻糖酶[3]后, 學者們很快就發現昆蟲普遍具有這兩種類型的海藻糖酶. 本研究基于已公布的豌豆蚜基因組數據庫, 擴增獲得了豌豆蚜可溶型海藻糖酶基因ApTre-1, 其全長為1 770 bp, 編碼589個氨基酸, 與NCBI中預測的豌豆蚜海藻糖酶基因(GenBank登錄號: XM001950229.4)氨基酸序列完全一致, 揭示了海藻糖酶氨基酸序列在不同地域豌豆蚜中的高度保守性. 序列分析表明ApTre-1氨基酸序列具有典型的標簽序列和富甘氨酸序列, 系統進化樹揭示其與大豆蚜海藻糖酶基因序列相似性最高, 并且與其他半翅目昆蟲, 包括褐飛虱、 白背飛虱以及煙粉虱的可溶型海藻糖酶聚為一類.
海藻糖酶基因在豌豆蚜體內的表達水平處于動態變化過程. Bansal等[18]研究發現, 海藻糖酶基因在大豆蚜1,2齡若蚜期的表達水平較高, 隨后其表達水平持續降低, 在成蚜中表達水平最低. 本研究中, 可溶型海藻糖酶基因ApTre-1在豌豆蚜不同齡期表達水平差異具有統計學意義, 其在1齡若蚜期的表達量最高, 且顯著高于其他各生長發育階段. 與文獻數據相比, 本研究中ApTre-1基因在豌豆蚜不同齡期的表達模式與其在大豆蚜不同齡期的表達模式較為一致, 均在1,2齡若蚜期表達水平最高. 此外, 在飛蝗研究中發現, 海藻糖酶基因LmTre-1在卵發育前期和中期表達量極低, 在末期表達量顯著增加, 在卵期最后1天表達量最高且顯著高于其他時期[7]. 結合本研究, 推測ApTre-1基因在豌豆蚜1齡若蟲中的高表達, 可能是因為1齡若蚜剛開始取食且處于快速生長發育階段, 與需要大量的能量有關. 但是, 該推測尚需進一步開展相關研究工作去證實.
RNAi的效率與試蟲體內靶基因的選擇密切相關, 因此在利用RNAi技術防治害蟲的研究中需要從害蟲體內篩選和鑒定高效靶標基因[19-20]. 目前研究較多的靶基因主要包括致死基因、 抗性和免疫基因、 生長發育相關基因和產卵相關基因等[21]. 例如, Terenius等[22]通過對鱗翅目昆蟲RNAi研究結果進行比較發現, 免疫相關基因的干擾效率顯著高于其他類型的基因. 此外, 基于已發現的害蟲體內的高效靶基因, 學者們利用植物表達這些靶基因的dsRNA來防治害蟲已經取得了較大進展. 例如, 植物介導針對幾丁質合成酶基因(CHS)的RNA干擾技術已經在麥長管蚜Sitobionavanae[23]中進行了較為深入的研究, 發現試蟲取食表達dsRNA的第3代轉基因小麥后, 其體內的靶基因CHS1表達水平下降了45%~50%, 試蟲總蛻皮率也顯著下降. 在褐色橘蚜Toxopteracitricida[24]的研究中, 也發現取食植物傳導的dsRNA后, 試蟲體內CHS的表達水平下降了48%, 且多數試蟲不能蛻皮進入到下一個齡期. Mao等[25]研究發現, 桃蚜Myzuspersicae連續取食植物表達的間隙基因(Mphb)dsRNA后, 降低了靶基因的表達量, 抑制了桃蚜的繁殖.
針對靶基因不同區域設計的dsRNA片段也會導致干擾效率的顯著差異性. 本研究首先采用顯微注射法針對靶標基因ApTre-1不同區域進行RNAi效率分析, 發現dsApTre-1-1,dsApTre-1-2對靶基因ApTre-1沉默效率分別為47.7%和30.3%. Chen等[5]針對甜菜夜蛾2種海藻糖酶的RNAi研究發現,dsSeTre-1和dsSeTre-2片段注射試蟲24 h后, 2種海藻糖酶靶基因的沉默效率從50%逐漸上升, 至72 h時達到最高(近80%). 相較而言, 本研究中針對豌豆蚜海藻糖酶基因ApTre-1的RNAi效率相對較低, 主要原因可能是本研究中所設計的2種dsRNA片段長度或位置不是最佳所致, 其他原因尚需進一步分析研究.
目前, 實驗室研究中dsRNA導入昆蟲體內的方法主要為顯微注射法和飼喂法. 由于顯微注射法過程較為繁瑣、 處理樣本量少, 較適用于昆蟲基因功能的研究;而飼喂法過程較為簡單, 更接近昆蟲的自然取食過程, 有助于最終在實踐生產中應用. 因此, 飼喂法的應用實踐性顯著優于顯微注射法. 在采用顯微注射法分析的基礎上, 本研究選用其中RNA干擾效率較高的片段dsApTre-1-1進一步通過飼喂法測定其對豌豆蚜的RNA干擾效應, 結果發現dsApTre-1-1對靶基因ApTre-1的沉默效率較注射法有較明顯下降, 靶基因的表達水平下降了27.3%, 試蟲第6 d時死亡率為17.5%. 張倩等[8]采用飼喂法對灰飛虱的2種海藻糖酶基因(LSTre-1和LSTre-2)進行RNA干擾效應研究, 發現dsTre-1和dsTre-2對靶基因的干擾效率分別為49.1%和41.5%, 在第2 d時致死率為10%~15%, 以后逐漸上升, 到第5 d時試蟲死亡率為20%~40%. 二者相比, 采用飼喂法測定的dsRNA干擾效率及致死率略高于本實驗結果, 但總體上致死率也相對較低. 上述研究結果說明在不同種類的試蟲中, 與顯微注射法相比, 昆蟲取食dsRNA后對靶基因的干擾效率及致死率均相對較低很可能是一種普遍現象. 已有諸多研究表明, 昆蟲腸道中含有豐富的核酸酶, 能夠降解dsRNA, 從而影響RNAi效率, 并對通過飼喂方式讓dsRAN進入試蟲體內后保持穩定和傳遞極為不利[26-27], 這可能是導致上述研究中通過飼喂法測定dsRNA的RNAi效率降低和致死率較低的重要原因之一. 據文獻報道, 針對靶基因不同長度的dsRNA片段也可能導致不同的RNA干擾效應, 包括靶基因沉默效率、 試蟲生長發育、 死亡與繁殖率等. 因此, 在本實驗的后續研究中, 仍需針對靶基因設計合成更多不同類型的dsRNA片段, 以便能篩選出對靶基因干擾效率更高的dsRNA片段, 為針對海藻糖酶的RNAi技術應用于害蟲防治提供有價值的參考.
本研究獲得了豌豆蚜可溶型海藻糖酶基因(ApTre-1)序列, 明確了其基因結構特點及在豌豆蚜不同生長發育階段的表達差異, 探索了顯微注射和飼喂ApTre-1基因的dsRNA片段對豌豆蚜RNA的干擾效應, 為今后以ApTre-1基因為靶標的RNA干擾技術在害蟲綜合防治中的應用奠定了基礎.

