橄欖果實轉錄組SSR和SNP/InDel位點特征
賴瑞聯 沈朝貴 馮新 陳義挺 韋曉霞 吳如健
關鍵詞:橄欖;轉錄組;簡單重復序列;單核苷酸多態性;插入缺失標記
中圖分類號:S667.5 文獻標識碼:A
橄欖(Canarium album)是我國熱帶亞熱帶地區特色果樹,其果實富含多種營養和藥用成分,在我國福建、廣東、四川、廣西等地區廣泛栽培。福州市是我國橄欖最主要的產地之一,“福州橄欖”品牌于2011 年獲得農業部地理標志保護品牌,2017 年品牌價值評估達20.80 億元人民幣,入選全國農產品百強[1]。福州市傳統栽培的橄欖品種主要包括長營、惠圓、檀香、自來圓等。其中,長營和惠圓的果實性狀存在明顯差異,長營可食率約為78.40%,可供鮮食或加工,在長營基礎上選育出了一系列鮮食品種,例如福欖1 號(光甜)[2]、清欖1 號[3]、福欖2 號[4]等;惠圓可食率約為85.20%,因鮮食品質較差,主要以加工為主,通過惠圓選育出了惠圓1 號[5]、惠圓3 號[6]等橄欖加工品種。作為我國特色果樹產業,品質性狀提升是橄欖科研創新的重要命題。目前,在橄欖品種選育[2, 7]、性狀評價[8-9]、品質模型[10]、遺傳背景[11]等方面都開展了大量研究。值得注意的是,橄欖童期較長,開發果實性狀相關分子標記用于育種材料初步篩選可有效提高種質創新和育種效率。而現階段,橄欖果實性狀形成分子機制研究及相關標記的開發仍處于空白,限制了橄欖分子輔助育種進程。
DNA 分子標記鑒定是植物分子輔助育種重要技術手段,尤其是具有高靈敏度、高特異性的簡單重復序列標記(simple sequence repeats, SSR)和單核苷酸多態性標記(single nucleotide polymorphism,SNP)在許多植物中得到了有效應用。?AHIN 等[12] 篩選了抗霜霉病相關的向日葵(Helianthus annuus)SSR 標記并成功應用于向日葵輔助育種;GHARSALLAH 等[13]結合表型和SSR 標記挖掘了番茄(Lycopersicon esculentum)品種耐鹽特性相關位點,有效提高了番茄耐鹽脅迫育種效率;TERAKAMI 等[14]采用SSR 標記對中國梨(Pyrus ussuriensis)黑斑病易感基因進行了定位,為梨抗黑斑病輔助育種提供了重要途徑。
在SNP 開發相關研究中,TAN 等[15]開發了小麥(Triticum aestivum)抗黑森癭蚊基因的SNP 標記,可用于進一步指導小麥育種;KIM 等[16]通過轉錄組測序挖掘了蘿卜(Raphanus sativus)SNP標記用于蘿卜種子的鑒定、純度檢測和親本組合的調整;FROUIN 等[17]鑒定了水稻(Oryza sativa)預防砷吸收和積累相關的SNP 位點,為水稻相關育種提供了參考依據。目前,橄欖轉錄組測序相關研究也取得了一定進展,為分子標記開發奠定了良好的基礎[18]。在前期研究基礎上,本研究基于轉錄組數據對具有不同性狀的橄欖品種長營和惠圓的果實進行SSR 和SNP/InDel 特征分析,以期為橄欖果實性狀相關分子標記開發提供數據基礎。
1 材料與方法
1.1 材料
供試材料為長營和惠圓橄欖品種的果實,取自福建省農業科學院果樹研究所橄欖種質資源圃(26° 07′40″N,119°20′16″E)。以孔祥佳等[19]建立橄欖果實成熟度評價體系為參考標準,采集不同品種成熟、健康、無機械病蟲損傷的果實,充分洗凈后用液氮速凍,并置于超低溫冰箱中保存備用。
1.2 方法
1.2.1 RNA提取與檢測 采用植物多糖多酚試劑盒E.Z.N.A.TM Plant RNA Kit 提取果實總RNA,采用1.0%瓊脂糖凝膠電泳和Agilent 2100 bioanalyzer(Agilent Technologies, USA)檢測總RNA的完整性, 隨后利用NanoPhotometer spectrophotometer(IMPLEN, USA)檢測總RNA 的純度。
1.2.2 文庫構建與質檢 取1 μg 橄欖果實總RNA,采用NEBNext? UltraTM RNA Library Kit試劑盒(Illumina, USA)進行建庫。首先使用Oligo(dT)磁珠富集帶有polyA 尾的mRNA,使用二價陽離子在高溫下在NEBNext 第一鏈合成反應緩沖液(5×)中進行裂解,使用隨機寡核苷酸引物和M-MuLV 逆轉錄酶體系合成cDNA 第一鏈。隨后使用RNaseH 酶降解RNA 鏈,再通過DNA 聚合酶I 合成cDNA 第二鏈。純化后的cDNA 進行末端修復、加A 尾、連接測序接頭,隨后使用AMPure XPbeads 篩選長度為250~300 bp 的cDNA片段,經PCR擴增后進行PCR 產物純化,最終獲得文庫。采用Qubit 2.0 Fluorometer(Invitrogen,USA)和Agilent2100 bioanalyzer 進行文庫質量檢測,隨后采用qRT-PCR 對文庫進行定量,篩選有效濃度高于2.0 nmol/L 的文庫后用于后續測序。
1.2.3 RNA 測序與數據組裝 采用TruSeq PECluster Kit v3 cBot HS(Illumina, USA)對樣本進行聚類,隨后在Illumina Novaseq 平臺上對制備的文庫進行測序,并產生150 bp 配對末端讀數。測序片段的圖像數據經CASAVA 轉化為fastq 格式的序列數據文件,去除帶測序接頭、無法確定堿基信息以及低質量的reads,最終獲得高質量的clean reads。隨后采用TRINITY 軟件[20]將cleanreads 拼接成transcripts,再采用CORSET 軟件[21]進行層次聚類后得到最長cluster 序列作為unigene用于后續分析。每個樣本進行3 次生物學重復。
1.2.4 SSR 位點鑒定 采用MISA 1.0 軟件[22]篩選和識別unigene,并對轉錄組SSR 位點進行分析,鑒定出單堿基重復、雙堿基重復、三堿基重復、四堿基重復、五堿基重復和六堿基重復等類型的橄欖果實SSR。采用Microsoft Excel 2013 軟件進行數據分析和圖表制作。
1.2.5 SNP/InDel 分型與特征分析 采用Picardtoolsv1.41 和Samtools v0.1.18 軟件進行排序并刪除重復數據,同時合并每個樣本的校準結果,并采用GATK3 軟件v3.4 版本[23]默認參數進行SNP位點檢測和數據統計。
2 結果與分析
2.1 橄欖果實的SSR 位點檢測
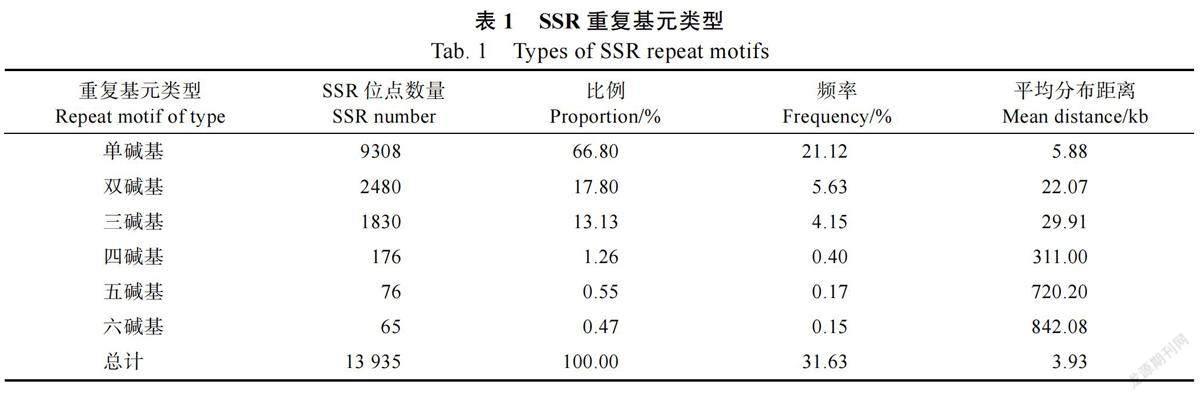
2.1.1 SSR 位點數量 通過長營和惠圓橄欖品種果實轉錄組測序, 6 個測序樣本平均獲得22 813 726 bp 的raw reads,過濾后的clean reads為22 019 057 bp,原始數據過濾后的平均堿基數為6.61 Gb,整體測序錯誤率約為0.02%,Q20 和Q30 分別為98.11%和94.19%,說明測序結果較好,可用于后續分析。拼接后,共獲得125 021條轉錄本和44 062 條unigenes,這些unigenes 包含54 735 280 bp 核苷酸。采用MISA1.0 對獲得的unigenes 進行SSR 位點鑒定,總共在10 124 條unigenes 上鑒定到13 935 個SSR 位點,發生頻率為22.98%,平均每1 kb 序列出現0.25 個SSR 位點。這些SSR 分布的unigenes 中,2657 條序列包含1 個以上的SSR 位點,1072 條序列包含復合型SSR 位點。可見,橄欖果實轉錄組中廣泛分布SSR 位點。
2.1.2 SSR 重復基元類型 橄欖果實SSR 位點的重復基元類型如表1 所示。SSR 重復基元類型較為豐富,除了含有單堿基重復到六堿基重復6 種類型,還包含977 種復雜重復類型。對單堿基重復到六堿基重復的SSR 類型進一步分析發現,單堿基重復的SSR 類型最多,達到9308 個,占全部SSR 位點的66.80%,隨著重復基元堿基數的增加,類型數量逐漸減少,六堿基重復的SSR 類型僅有65 個,占全部SSR 位點的0.47%。從SSR位點的出現頻率和分布距離來看,單堿基重復的SSR 位點類型分別為21.12%和5.88 kb,而六堿基重復的SSR 位點類型分別為0.15%和842.08 kb。結果可見,不同重復基元類型的SSR 位點數目和分布存在較大差異。
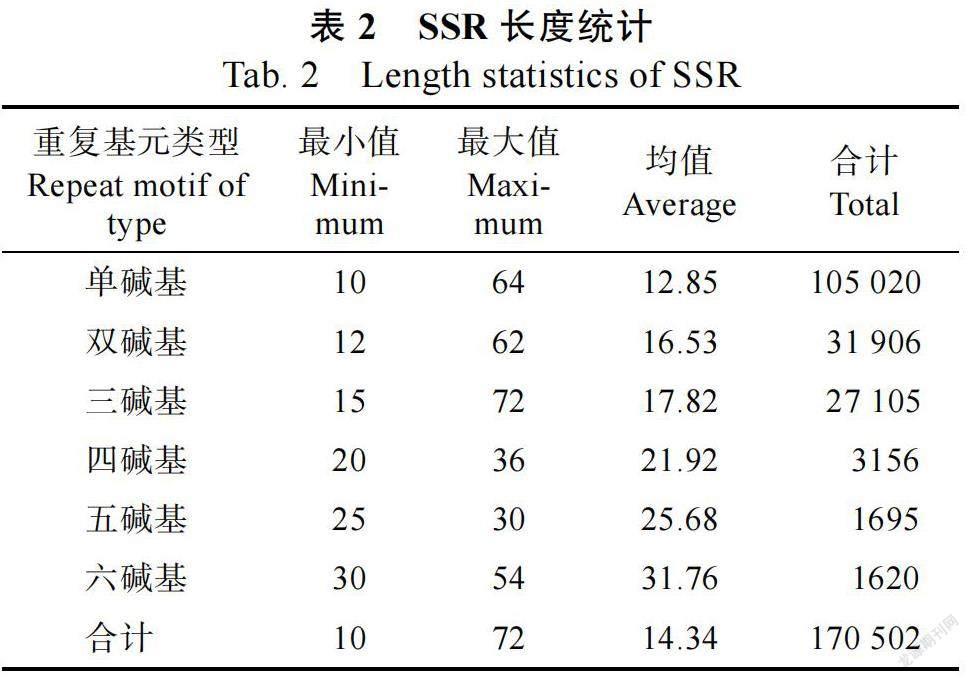
2.1.3 SSR 序列長度分布 不同重復基元的橄欖果實SSR 類型的序列長度為10~320 bp,平均長度為36.19 bp。對其中單堿基重復基元到六堿基重復基元的SSR 序列進一步分析發現(表2),整體上序列長度為10~72 bp,平均長度為14.34 bp。其中,長度差異最大的是單堿基重復的SSR 類型,介于10~64 bp 之間,平均長度為12.85 bp;差異最小的為五堿基重復的SSR類型,介于25~30 bp之間,平均長度為25.68 bp。
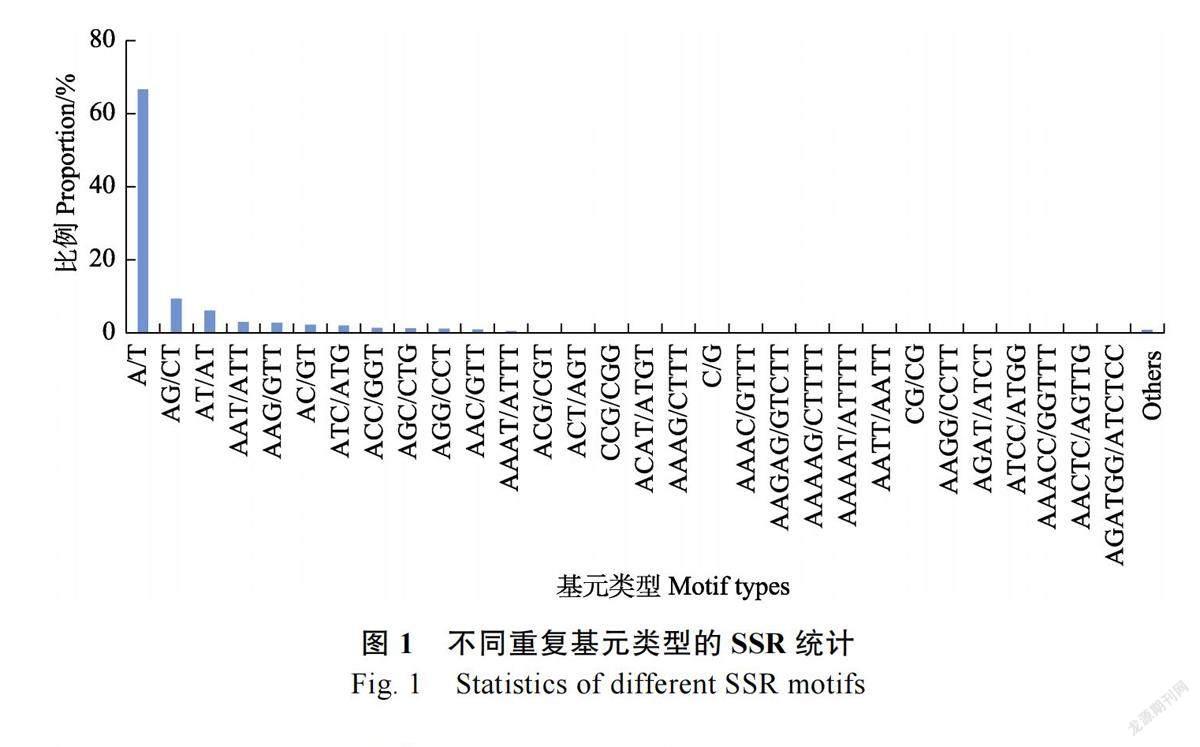
2.1.4 SSR 優勢重復基元分布特征 橄欖果實SSR 位點基元數量統計分析結果如圖1 所示。分布頻率最高的基元分別是A/T(9290個,占比66.67%)、AG/CT(1304 個,占比9.36)、AT/AT(857 個,占比6.15%)、AAT/ATT(421 個,占比3.02%)、AAG/CTT(392 個,占比2.81%)。對不同SSR 重復基元類型的出現頻數進行分析發現,所有SSR 位點共出現105 種重復基元,單堿基到六堿基重復的基元類型分別為2、4、10、21、29、39 種。單堿基重復基元到六堿基重復基元中的優勢重復基元分別為A/T、AG/CT、AAT/ATT、AAAT/ATTT ( 65 個, 占比0.47% )、AAGAG/CTCTT (10個, 占比0.07% ) 和AGATGG/ATCTCC(5 個,占比0.04%)。
2.1.5 SSR 各基元類型重復次數 橄欖果實SSR位點不同基元類型的重復次數統計結果如圖2 所示。其中,單堿基重復基元的重復次數主要分布在9~12 和13~16 之間,分別占SSR 位點總數的41.69%和17.22%,占單堿基重復基元總數的62.42%和25.77%;雙堿基到六堿基重復基元的重復次數均主要集中在5~8,分別占對應重復基元總數的68.67%、93.55%、98.30%、100%和98.46%。不同重復基元類型的SSR 位點數量隨著重復次數的增多呈遞減趨勢。
2.2 橄欖果實的SNP/InDel 的特征分析
基于轉錄組數據進行不同性狀橄欖品種果實SNP/InDel 挖掘。共獲得284 992 個SNP 位點,平均每1 kb 序列含有5.21 個。其中,轉換類型的SNP 位點共166 162 個,C/T 和A/G 發生頻率相近,每1 kb 序列分別含有1.51 個和1.52 個。顛換類型的SNP 位點共118 830 個,每1 kb 序列中A/T、A/C、T/G 和C/G 的平均個數分別為0.63、0.53、0.54 和0.47。轉換類型的SNP 位點(3.03個)的發生頻率明顯高于顛換類型(2.18 個)。其中,第1、2 和3 位上發生SNP 突變的密碼子數量分別為51 513、25 378 和50 306。在這些SNP 位點中,4479 條unigenes 包含1 個SNP 位點,3096 條unigenes 包含2 個SNP 位點,2419條unigenes 包含3 個SNP 位點,隨著SNP 位點數增多,unigenes 的數量逐漸減少(圖3A)。值得注意的是,其中的14 條unigenes 包含有100個以上的SNP 位點(表3),其中的9 條unigenes得到了功能注釋,仍有5 條unigenes 在所有數據庫中未得到有效比對,這些存在高頻SNP 變異的unigenes 可能在果實性狀差異形成過程中發揮了重要功能。
基于轉錄組測序數據,共獲得18 548 個InDel位點,平均每1 kb 序列含有2.95 個,其分布變化趨勢與SNP 相似(圖3B),unigenes 中含有1 個InDel 位點的數量最多,達到7853 條。InDel 位點最多的是Cluster-4594.16496,達到24 個InDel位點,通過比對預測,其可能是胼胝質合成酶。
3 討論
3.1 轉錄組測序可有效開發橄欖SSR 和SNP/InDel 標記
轉錄組測序是植物SSR 和SNP/InDel 標記開發有效的技術手段。VIDYA 等[24]從生姜(Zingiberofficinale)轉錄組中挖掘了16 790 個EST-SSR 位點,其中4597 個SSR 位點分布在已預測的編碼基因中;ZHOU 等[25]從楠木(Phoebe bournei)轉錄組中鑒定了40 853 個SSR 位點,并對其中23個多態性EST-SSR 標記進行應用;TULSANI 等[26]從芫荽(Coriandrum sativum)轉錄組中鑒定到了9746 個SSR 位點,為其轉錄組草圖和基因標記提供了重要信息;XU 等[27]基于轉錄組測序從草珊瑚(Sarcandra glabra)中挖掘了726 476 個SNP位點和42 939 個InDel 位點,為其資源開發奠定了基礎。本研究采用Illumina Novaseq 測序平臺,從不同性狀的橄欖果實中分別挖掘了13 935 個SSR 位點、284 992 個SNP 位點和18 548 個InDel位點,為后續橄欖分子標記開發和應用奠定了數據基礎。
3.2 橄欖果實中存在豐富多樣的SSR位點
SSR 標記具有多態性高、分辨率高、通用性好、穩定性強等優點,是物種種質資源鑒定、遺傳連鎖圖譜構建、基因定位、數量性狀基因位點分析、分子標記輔助育種等研究的重要技術手段[28]。本研究通過轉錄組高通量測序,在10 124條unigenes 上鑒定到13 935 個SSR 位點,發生頻率為22.98%。其中,單堿基重復基元類型的SSR位點最多并且單堿基重復是橄欖果實的優勢重復基元,隨著重復堿基數量的增加,SSR 位點出現頻率下降,與油梨(Persea americana)[29]、甜柿(Diospyros kaki)[30]、香椿(Toona sinensis) [31]等物種的規律一致。橄欖果實中的優勢重復基元主要包括A/T 、AG/CT 、AT/AT 、AAT/ATT、AAG/CTT 等,尤其A/T 的比例達到66.67%,與甜柿[30]、龍眼(Dimocarpus longan)[32]、油梨[29]等其他物種存在一定的相似性,但仍有差異,可見SSR 位點分布存在物種特異性。此外,橄欖果實SSR 位點中也發現了6 個CG/CG 和29 個CCG/CGG 重復基元,其在雙子葉植物中較為少見[30],可能在橄欖進化過程中發揮特殊的生物學功能。從多態性上看,通常認為長度大于20 bp的SSR 位點具有較高的多態性[33],橄欖果實中四堿基、五堿基和六堿基重復的SSR 位點長度均在20 bp 以上,具有更高的多態性,可能具有較高的應用價值。
3.3 橄欖果實中廣泛分布SNP/InDel位點
SNP/InDel 具有高效性、準確性,并且與作物性狀密切相關。本研究基于轉錄組測序,共檢測到橄欖果實284 992 個SNP 位點,平均每1 kb 序列含有5.21 個SNP 位點。其中,轉換和顛換類型的比例為1.398,與理論值0.500 存在較大偏差,說明橄欖進化過程中對不同類型的選擇并非隨機而可能存在偏向性[34]。此外,本研究共獲得橄欖果實18 548 個InDel 位點,每1 kb 序列平均包含2.95 個InDel 位點。后續還應進一步針對SNP/InDel位點進行深入研究,開發橄欖果實性狀相關的特異性分子標記,為橄欖種質資源鑒定和分子輔助育種提供技術支撐。

