基于機器學習的miRNA與疾病關系預測系統的設計與實現
夏岱婷,彭熒熒
(湖南中醫藥大學,長沙 410001)
MicroRNA(以下簡稱miRNA)是一類由22~24個核苷酸組成的非編碼RNA[1],可通過對miRNA的研究來進一步了解疾病的發病機制,區分疾病類型,還可根據其與疾病之間的相關性來預測和防治疾病。越來越多的人開始使用生物信息學的計算方法對miRNA-疾病關聯關系進行預測。基于“功能相似的miRNA往往與相似的疾病相關且反之亦然”[2]的假設,研究人員提出了許多模型。
盡管已出現了不少先進的預測方法,但對潛在的miRNA-疾病關聯關系進行預測時,部分方法利用高斯核函數計算高斯核相互作用譜相似性,未對狀態相同但意義相反的情況[(0,0)和(1,1)]進行區分,缺少預測系統進行展示。
1 數據
miRNA-疾病關聯數據。人類miRNA疾病關聯數據庫(the Human microRNA Disease Database,HMDD)[3]始建于2007年(Lu等[4]),本研究采用V 2.0版本數據。
1)miRNA功能相似性數據。Wang等在計算miRNA之間功能相似性時分別考慮了與miRNA相關的相似疾病的影響,提出了MISIM計算方法[5],將其應用于HMDD中記錄的miRNA,計算出所有miRNA之間的功能相似性,并建立了對應的網站。miRNA功能相似性數據可直接從網站上下載(http://www.cuilab.cn/files/images/cuilab/misim.zip)。
2)疾病語義相似性數據。Wang等根據美國國家醫學圖書館MeSH(https://www.ncbi.nlm.nih.gov/)中對疾病類別的描述信息,提出基于疾病關系結構的疾病語義相似性度量方法[5],用有向無環圖(DAG)描述疾病之間的關系,節點表示疾病術語,分為表示更寬泛的父節點和表示更具體的子節點,邊表示疾病之間存在關聯。
2 miRNA-疾病關聯預測模型
提出了一個半監督學習的預測模型(RGM)。對已知的關聯關系進行預處理,去除冗余數據,得到只含有miRNA信息和疾病信息的關聯矩陣。針對部分方法存在的問題,運用高斯核函數獲取高斯核相互作用譜相似性信息時,區分了狀態相同但意義相反的情況,利用譜相似性矩陣,結合miRNA功能相似性矩陣和疾病語義相似性矩陣得到新的矩陣。在新的矩陣和miRNA-疾病關系矩陣中,利用標簽傳播算法得到最終的預測結果。以下主要對獲取高斯核相互作用譜相似性進行說明。
基于“功能相似的miRNA往往與相似的疾病存在關聯關系”的假設[6],根據Liu[6]等的方法,從關聯矩陣中獲取高斯核相互作用譜相似性時,對狀態相同但意義相反的情況加以區分。以疾病信息為例,關聯矩陣A作為高斯核相互作用分布圖,矩陣中的每一列看作表示每一個疾病的特征向量D,D可以表示疾病與miRNA之間的關聯關系,D中僅包含0和1兩種數值,0表示疾病與對應的miRNA存在未知的關聯關系,1表示疾病與對應的miRNA存在已知的、經實驗驗證的關聯關系,因此可以通過式(1)和式(2)獲取疾病的高斯核相互作用譜相似性矩陣KD。
(1)
其中,γd用來控制高斯核的帶寬參數,計算方法如下:
其中
(2)
式中,δd[6]是一個新的帶寬參數,原則上可以通過交叉驗證實驗來進行設置,根據Liu等的研究,將δd簡單地設置為1。nm表示關聯矩陣中miRNA的種類數,yik表示第i列疾病與第k行miRNA對應的數值,yik表示第j列疾病與第k行miRNA對應的值。
同理,根據公式(3)、(4)可以得到miRNA的高斯核相互作用譜相似性矩陣KM。
(3)
其中
(4)
基于HMDD v2.0版本數據庫中已知的miRNA-疾病關聯關系,采用全局留一法對模型性能進行評估。為了排除實驗數據的偶然性,使實驗數據更具說服力,取5次全局留一法實驗的數據平均值作為最后的實驗結果,結果顯示,新模型的AUC值高于其他模型。
如圖1所示,在全局留一法實驗中,IMCMDA[7]、QIMCMDA[8]、MCLPMDA[9]和RGM,分別取得了0.839、0.924、0.966和0.972的AUC值。
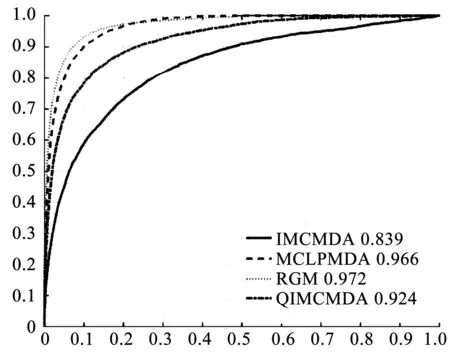
圖1 全局留一法實驗中各模型的ROC曲線及AUC值Fig.1 ROC curve and AUC value of each model in the experiment of global leave-one method
3 預測系統的設計與實現
3.1 需求分析
本系統將用戶分為普通用戶和管理員兩大類。
1)功能性需求。用戶可查看系統中收錄的疾病、miRNA、miRNA-疾病預測結果,可根據miRNA名稱或疾病名稱進行模糊查詢,也可通過疾病和miRNA的組合進行精確查詢。上傳頭像、更新昵稱、修改密碼等功能需打開“個人中心”頁面,點擊頁面中的頭像,實現上述功能。管理員可根據用戶名對用戶進行查詢、新增、修改、刪除等操作,對疾病信息和miRNA信息進行查詢、收錄、新增、修改等操作,對預測結果進行管理,預測結果以關系圖形式進行可視化展示。
2)非功能性需求。可靠性:系統前后端均采用成熟的開發框架,同時考慮了響應速度,避免出現長時間不響應導致不滿足用戶需求的情況。安全性:采用路由攔截,用戶無法在未登錄狀態下通過路由地址訪問系統。用戶密碼在前端采用SHA256進行加密,數據庫中存儲加密后的密碼。易用性:采用成熟的組件庫進行開發,風格統一,界面美觀,用戶視覺感官好。界面中包含操作說明,可操作性強。
3.2 系統總體設計
系統架構采用前后端分離技術,耦合程度低,前端基于Vue框架進行開發,后端基于SpringBoot和MyBatis實現,主要包括表示層、業務層、數據層。
系統功能模塊劃分為普通用戶模塊和管理員模塊。普通用戶可對系統收錄疾病、miRNA和miRNA-疾病關聯關系的預測結果進行查詢,還可對個人信息進行修改。管理員可對普通用戶、疾病、miRNA及miRNA-疾病關聯關系預測結果進行管理,實現對相關數據的新增、修改和刪除。
數據庫設計。采用MySQL數據庫,主要存儲用戶個人信息、疾病信息、miRNA信息、miRNA-疾病關聯關系預測結果信息等。
3.3 系統實現
系統開發環境如下:操作系統:windows10;開發語言:Java、JavaScript、Matlab;開發環境:Visual Studio Code、IntelliJ IDEA;數據庫:MySQL;數據庫可視化工具:Navicat Premium。通過Navicat Premium對存儲系統數據的MySQL數據庫進行可視化管理。Matlab主要用來進行數據處理,通過預測模型從已知的少量miRNA-疾病關聯關系、miRNA功能相似性信息及疾病語義相似性信息得到潛在的miRNA-疾病關聯關系預測結果。
目前系統已成功部署在阿里云服務器上,可通過http://47.106.112.145:8080/login進行訪問。部分關鍵界面如下:
1)系統收錄疾病頁面。主要用于展示系統中收錄的所有miRNA,包括miRNA編號及miRNA名稱。用戶可在輸入框中輸入某一特定的 miRNA名稱,點擊“搜索”按鈕。若系統中存在該種miRNA,則在表格中顯示相關信息,否則不進行顯示。也可在輸入框中輸入miRNA的部分名稱信息,點擊“搜索”按鈕,查詢系統中所有包含該部分的miRNA信息。也可不進行輸入,直接點擊“搜索”按鈕,查詢系統中所有miRNA的信息。圖2為系統收錄疾病頁面截圖。
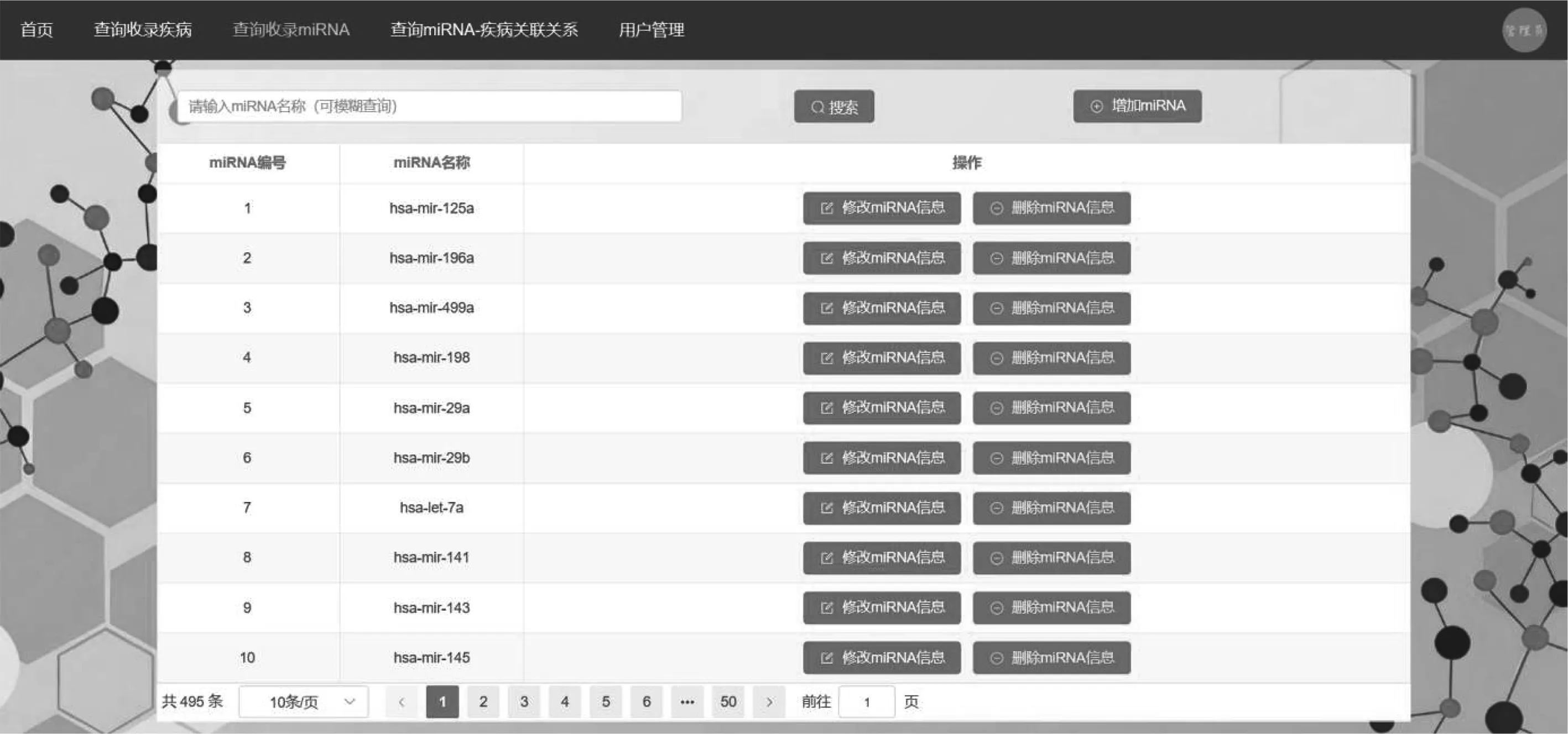
圖2 系統收錄miRNA頁面截圖Fig.2 MiRNA page snapshots included in the system
2)預測結果展示頁面。主要用于展示系統中收錄的所有預測結果,包括預測結果編號、疾病名稱及miRNA名稱。以本模型中3種疾病的部分預測結果為例進行可視化展示,使用戶更直觀地感受miRNA-疾病關聯關系。可根據疾病或miRNA名稱對預測結果進行模糊查詢,也可通過輸入疾病與miRNA的組合對預測結果進行精確查詢。圖3為預測結果可視化截圖。
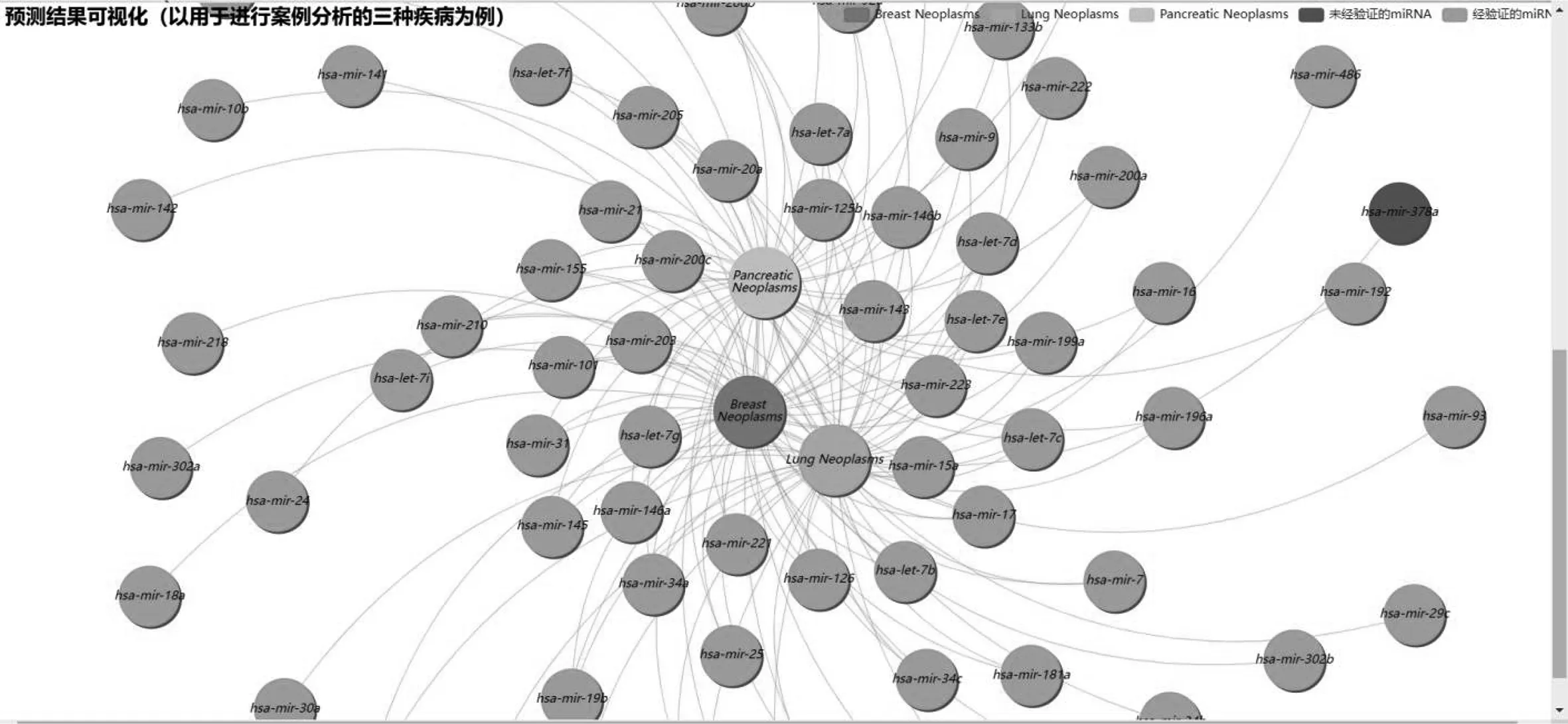
圖3 預測結果可視化截圖Fig.3 Visual screenshot of prediction results
3.4 系統測試
對本系統進行業務功能和性能測試,均達到預期。

