基于生物信息學數據庫的結腸癌SDC4基因表達調控機制及作用研究
唐梓涵 袁煒嗣 吳澤強 張粵烽 詹涵 王琛瑤 李恩民 陳滋凱 周飛
摘 要:為了揭示SDC4基因在結腸癌中的功能及其表達調控機制.通過GEPIA2、Ualcan與Oncolnc等數據庫研究分析SDC4基因在結腸癌的表達及其對患者總生存期的影響.通過Wanderer、UCSC、Cistrome DB、miRcode和miRDB等數據庫研究分析結腸癌SDC4基因啟動子區的甲基化、組蛋白修飾,調控其表達的轉錄因子和miRNAs.通過GEPIA2、Ualcan、HCMdb與DAVID等數據庫研究分析在結腸癌中與SDC4基因共表達的基因及其生物學作用.基于以上生物信息學數據庫,研究發現,在結腸癌中:SDC4基因的表達水平顯著增高,并與腫瘤組織分期、淋巴結轉移正相關,與患者預后負相關.SDC4基因啟動子區的甲基化水平顯著升高,轉錄激活性組蛋白修飾H3K4Me1與H3K27Ac顯著富集.SP1、TEAD4、JUND、FOSL1、NR1H3、PPARG、SRF、TFAP4共8個轉錄因子可能調控SDC4基因的轉錄表達.與此同時hsa-miR-194和hsa-miR-124也可能調控SDC4基因表達.hsa-miR-194和hsa-miR-124的表達下調.SERINC3、DNTTIP1、ATP9A、CHMP4B、BCL2L1、STAU1、SYS1、YWHAB、OSBPL2、SLC35C2、TM9SF4與SDC4基因共表達.SDC4基因可能參與調控結腸癌細胞凋亡、細胞通訊、物質運輸與定位等生物學過程.基于生物信息學數據庫研究發現的結果,為進一步闡明SDC4基因在結腸癌中的功能及其表達調控機制提供重要的線索.
關鍵詞:生物信息學;結腸癌;SDC4;表達調控機制
中圖分類號:R 735.3 文獻標識碼:A 文章編號:1007-6883(2023)03-0013-11
DOI:10.19986/j.cnki.1007-6883.2023.03.003
SDC4蛋白質是一種跨膜(I型)硫酸乙酰肝素蛋白聚糖,包含胞外結構域(N端)、跨膜結構域和胞質結構域(C端)[1].SDC4蛋白質常作為受體分子,通過其胞外結構域,與細胞外基質成分(如纖連蛋白)、生長因子(如FGF、PDGF、VEGF)、細胞因子(如MCP-1、CXCL12)等相互作用,傳遞細胞外信號[1-3].SDC4蛋白質的胞質結構域能結合磷脂酰肌醇4,5-二磷酸(PIP2),繼而激活 PKCα信號通路[1].SDC4蛋白質還能激活MAPK、ERK、Wnt/β-catenin信號通路[4-6].這些分子功能表明SDC4蛋白質具有重要的細胞生物學作用.據報道:SDC4蛋白質能調控細胞的生長、粘附、移動、侵襲,上皮-間質轉化(EMT)等與腫瘤進展密切相關的細胞生物學過程[1,6].而且,SDC4基因在肝癌、腎癌、雌激素受體陰性的乳腺癌、黑色素瘤、膠質瘤中高表達[7].但是,腫瘤細胞SDC4基因的表達調控機制,知之甚少[7].
基因轉錄調控是基因表達調控的重要環節.DNA甲基化、組蛋白修飾、轉錄因子在基因轉錄調控中起重要作用[8-10].DNA甲基化是一種發生在胞嘧啶-鳥嘌呤二核苷(CpGs)的化學修飾[9-10].在腫瘤中,基因啟動子的DNA甲基化水平,往往與其轉錄活性負相關[9].組蛋白修飾類型很多,不同類型的組蛋白修飾對基因表達調控的作用不同,有的抑制基因表達,也有的促進基因表達[9-10].其中H3K4me1與H3K27Ac被認為是基因轉錄激活標記,占據染色質中轉錄活躍的基因區域,通常與基因的轉錄激活正相關[9-11].轉錄因子與基因啟動子區特異DNA序列結合,調節基因對RNA聚合酶的招募作用,既可上調基因表達,也可下調基因表達[12].MicroRNAs(miRNAs)是一類小的非編碼RNA,在轉錄后水平調控基因表達,通過與靶基因mRNA完全或不完全互補配對,引起mRNA降解或翻譯阻遏[13].
結腸癌是常見的消化道惡性腫瘤,嚴重威脅人民健康.我國結腸癌發病率和死亡率逐年升高.2015年我國結腸癌新發病例38.8萬,死亡病例18.7萬[14].據報道,SDC4蛋白質在結腸癌的腫瘤細胞浸潤前緣高表達[11].而且SDC4蛋白質高表達與結腸癌進展性病理特征顯著正相關,對患者生存率不利[11].然而SDC4基因在結腸癌中的表達調控機制與作用,至今尚不清楚.為此,本研究基于多種生物信息學數據庫,研究分析SDC4基因在結腸癌中的表達調控機制及其作用,為進一步闡明SDC4基因在結直腸癌發生發展中的生物學功能奠定基礎.
1 材料與方法
1.1 結腸癌SDC4基因的表達水平
通過GEPIA2數據庫(http://gepia2.cancer-pku.cn/)和Ualcan數據庫(http://ualcan.path.uab.edu/),研究分析SDC4在人結腸癌組織及其癌旁正常組織中的表達情況.(1)GEPIA2數據庫的參數設置:“gene:SDC4”,“|Log2FC|Cutoff:0.5”,“p-value Cutoff:0.01”,“cancer name:COAD”,“Log Scale:Yes”,“Jitter Size:0.4”,“Matched Normal data:Match TCGA normal data Plot”.(2)Ualcan數據庫參數設置:“Scan by gene:SDC4”,“TCGA dataset:Colon adenocarcinoma”,“SDC4 expression based on:Sample types/Individual cancer stages/Nodal Metastasis status”.
1.2 結腸癌SDC4基因表達水平對患者生存率的影響
通過OncoLnc數據庫(http://www.oncolnc.org/)、GEPIA2數據庫,研究分析結腸癌SDC4基因表達水平對患者生存期的影響.Oncolnc數據庫的參數設置如下,“gene:SDC4”,“cancer:COAD”,“high:50,low:50”.GEPIA2數據庫的參數設置如下,“gene:SDC4”,“methods:overall survival”,“group cutoff:median”,“hazards ratio:NO”,“95%confidence interval:NO”.
1.3 結腸癌SDC4基因的甲基化修飾水平
通過Wanderer在線服務器(http://maplab.imppc.org/wanderer/),研究分析結腸癌組織及其癌旁正常組織細胞SDC4基因啟動子區(Chr20:43977064-43979064;GRCh37/hg19)的甲基化水平.參數設置如下,“Gene Symbol:SDC4”,“Project:Colon adenocarcinoma”,“Data Type:450K Methylation array”,“Specify a region:Start=43977064;End=43979064”.
1.4 結腸癌SDC4基因的組蛋白修飾水平
利用數據庫UCSC Genome Browser(http://www.genome.ucsc.edu/),研究分析SDC4基因的組蛋白修飾情況.方法如下,選擇參考基因組Human GRCh38/hg38,并用Track search工具檢索結腸癌細胞(HCT-116)SDC4基因的基因區內H3K4me1、H3K27ac修飾情況.通過Zoom in/out工具調整窗口大小.通過Vertical viewing range工具設置圖形縱坐標的最小值與最大值.
1.5 結腸癌中調控SDC4基因表達的轉錄因子
通過Cistrome DB數據庫(http://cistrome.org/db/#/)的在線工具,研究分析在結腸癌細胞中調控SDC4基因表達的轉錄因子.具體參數如下,“Species:Human hg38”,“Data type in Cistrome:Transcription factor, chromatin regulator”,“Gene or transcript:SDC4”,“The half-decay distance to transcription start site:10 kb”.同時設置,Regulatory Potential分值大于0.6.
1.6 結腸癌中調控SDC4基因的miRNA
通過miRcode在線分析工具(http://www.mircode.org/)、miRDB數據庫(http://mirdb.org/)和TargetScan數據庫(http://www.targetscan.org/vert_80/)預測調控SDC4基因表達的miRNAs.利用Jvenn數據庫(http://jvenn.toulouse.inra.fr/app/example.html),分析它們共同預測的miRNAs.利用miRCancer數據庫(http://mircancer.ecu.edu/index.jsp)檢索調控SDC4的miRNAs在結腸癌中的表達情況.
1.7 結腸癌中SDC4基因的生物學作用
通過GEPIA2數據庫、Ualcan數據庫與HCMdb數據庫(http://hcmdb.i-sanger.com/index),研究分析在結腸癌中與SDC4基因共表達的基因.研究分析時,設置這三個數據庫的皮爾森相關系數≥0.5.在利用HCMdb數據庫研究分析時,選擇其收錄的EXP00421項目數據.因為EXP00421項目檢測的結腸癌臨床樣本最多,為453例.通過DAVID數據庫對SDC4基因及與其共表達的基因進行GO注釋,從而分析SDC4基因的生物學作用.參數設置,[p<0.05],[Count<2].
2 結果與分析
2.1 結腸癌組織SDC4基因的表達水平
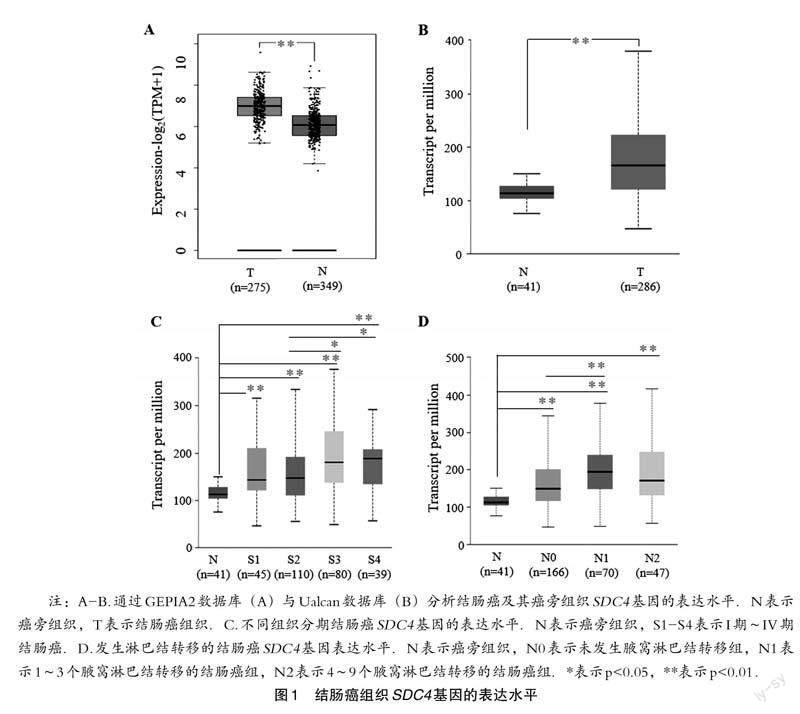
GEPIA2數據庫顯示,與相應的癌旁正常組織相比,SDC4基因在結腸癌組織中的表達水平顯著增高.而且Ualcan數據庫,也證實了此分析結果(圖1A和圖1B).本研究進一步分析發現,SDC4基因的表達水平,在癌旁正常組織、I期~Ⅳ期結腸癌組織中逐漸升高,在發生淋巴結轉移的結腸癌中顯著增高(與無淋巴結轉移的結腸癌或癌旁正常組織相比)(圖1C和圖1D).這些證據表明,SDC4基因表達可能與結腸癌的組織分期和淋巴結轉移正相關
2.2 結腸癌SDC4表達水平與患者生存率的關系
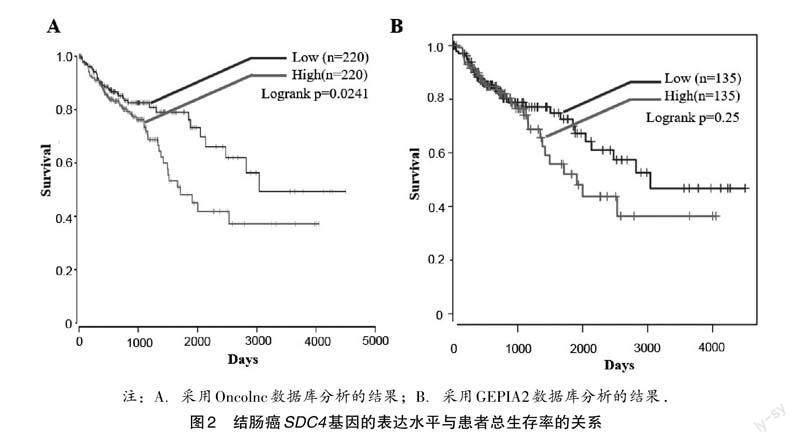
通過Oncolnc數據庫,研究分析SDC4基因表達水平對結腸癌患者總生存率的影響.結果發現,SDC4基因高表達的患者總生存率顯著較低(圖2A).為了驗證此結果,本研究又通過GEPIA2數據庫,分析SDC4基因表達水平與結腸癌患者總生存期的關系.其分析結果,也表明SDC4基因高表達對結腸癌患者預后不利(圖2B).
2.3 結腸癌SDC4基因的甲基化修飾水平
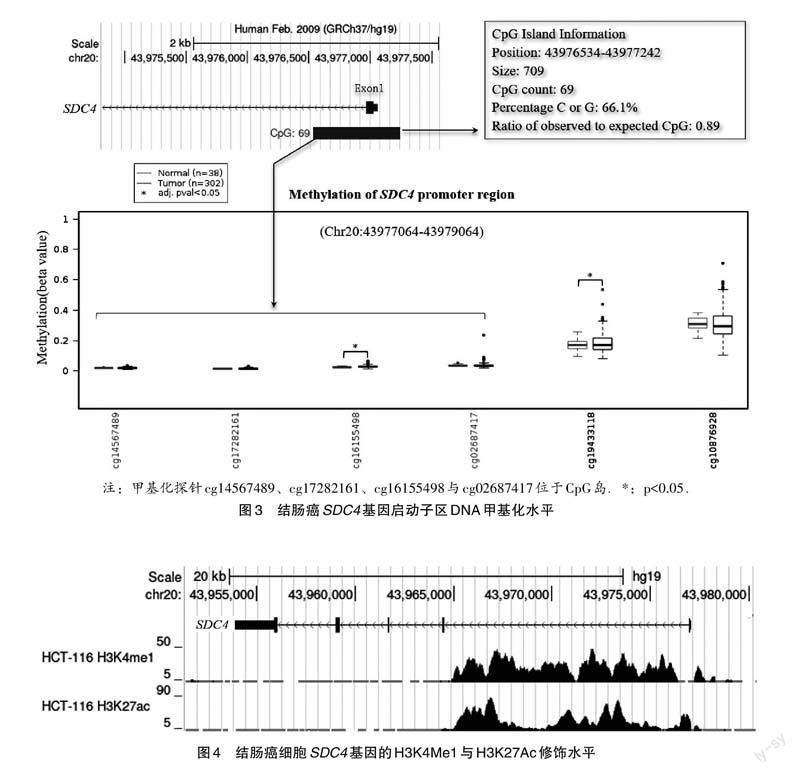
本研究認為SDC4基因轉錄起始位點上游2000 bp為其啟動子區域(Chr20:43977064-43979064,GRCh37/hg19).UCSC基因組瀏覽器顯示SDC4基因的啟動子及與其相鄰的基因組區域(chr20:43976534-43977242;GRCh37/hg19)中有一個長為709 bp的CpG島,島內包含69個CpG.Wanderer數據庫顯示SDC4基因啟動子區中存在6個甲基化位點,其中4個位于此CpG島的3'端區域,2個位于CpG島下游.與癌旁正常組織相比,其中2個位點(cg16155498與cg19433118)的甲基化水平在結腸癌中顯著升高(圖3).
2.4 結腸癌SDC4基因的組蛋白修飾情況
利用UCSC數據庫,研究分析SDC4基因在結腸癌細胞(HCT-116)內的組蛋白修飾情況.結果表明,轉錄激活相關的H3K4Me1與H3K27Ac顯著富集于SDC4基因的啟動子區域與第一內含子(圖4).此結果表明,組蛋白修飾可能對結腸癌SDC4基因的轉錄表達起重要調控作用.
2.5 調控結腸癌SDC4基因的轉錄因子
通過Cistrome DB數據庫的在線工具,分析在結腸癌中調控SDC4基因表達的轉錄因子.結果發現,ChIP-Seq或ChIRP-Seq實驗結果顯示在結腸癌細胞(HCT-116、HT29、V852或SNU175)中,SP1、TEAD4、JUND、FOSL1、NR1H3、PPARG、SRF與TFAP4共8個轉錄因子結合SDC4基因(圖5A).例如在結腸癌HCT-116細胞中,ChIP-Seq實驗檢測結果顯示SDC4基因的5'端上覆蓋多個明顯的SP1結合峰(圖5B).此結果提示,以上8個轉錄因子可能在結腸癌細胞中調控SDC4基因表達.
2.6 調控結腸癌SDC4基因的miRNA
分別通過miRcode數據庫、TargetScan數據庫和miRDB數據庫,預測能調控SDC4基因表達的miRNA.miRcode數據庫、TargetScan數據庫與miRDB數據庫分別發現能調控SDC4基因表達的miRNAs有4個、4個與82個.其中這三個數據庫均發現hsa-miR-194和hsa-miR-124調控SDC4基因的表達(圖6A).本研究進一步分析hsa-miR-194和hsa-miR-124在結腸癌中的表達情況.結果顯示,hsa-miR-194和hsa-miR-124在結腸癌中低表達(圖6B).
2.7 結腸癌中SDC4基因的作用
為了研究分析結腸癌中SDC4基因的作用,本研究首先通過GEPIA2數據庫、Ualcan數據庫與HCMdb數據庫(http://hcmdb.i-sanger.com/index),分析在結腸癌中與SDC4基因共表達的基因.為了提高分析結果的可靠性,本研究認為由這三個數據庫共同分析得到的基因,才是在結腸癌中與SDC4基因共表達的基因.GEPIA2數據庫、Ualcan數據庫與HCMdb數據庫分別發現在結腸癌中,有49個、104個與20個基因與SDC4基因共表達.其中SERINC3、DNTTIP1、ATP9A、CHMP4B、BCL2L1、STAU1、SYS1、YWHAB、OSBPL2、SLC35C2、TM9SF4這11個基因由這三個數據庫共同分析發現(圖7).本文認為這11個基因在結腸癌中與SDC4基因共表達.換句話說,本研究分析發現了一個結腸癌SDC4共表達基因集.接著,為了研究分析結腸癌SDC4基因的生物學作用.我們通過DAVID數據庫對SDC4共表達基因集進行功能富集分析.結果發現,這些基因被富集于18個GO條目,其中包含細胞凋亡相關GO條目(GO:0097190;GO:2001233;GO:2001235)、細胞通訊調控相關GO條目(GO:0023056、GO:0010647)、物質運輸、定位及其調控相關GO條目(GO:0044765、GO:0071705、GO:0016192、GO:0071702、GO:0046907、GO:0034613、GO:0060341、GO:1903827、GO:0090150)、細胞器組裝及調控相關GO條目(GO:0033043、GO:0044802、GO:0007009)、病毒參與生物學過程相關GO條目(GO:0016032)(圖8).
3 討論
SDC4蛋白質能結合生長因子、細胞外基質組分、細胞骨架蛋白,調控細胞內RAC1活性、PKCα活性、黏著斑激酶磷酸化水平、Ca2+水平,在細胞骨架重組、細胞粘附、細胞移動、細胞增殖、細胞分裂、細胞極性形成等生物學過程中起重要的調控作用[1].這些SDC4蛋白質調控的細胞生物學過程與腫瘤進展密切相關.故SDC4蛋白質可能參與腫瘤的發生發展.據報道,SDC4基因在雌激素受體陰性的乳腺癌、膠質瘤、肝癌、黑色素瘤、腎癌等惡性腫瘤中高表達[7].與永生化結腸細胞(CCD-18Co)相比,SDC4 mRNA在高轉移性的結腸癌細胞(KM1214、HCT116,KM12SM)中顯著低表達[15].最近研究表明,SDC4蛋白質在結腸癌癌巢浸潤前緣細胞高表達,特別是在腫瘤芽表達最強[16].本文發現,SDC4基因的表達水平與結腸癌臨床分期和局部淋巴結轉移正相關(圖1).SDC4基因高表達的結腸癌患者預后不良(圖2).這些證據表明,SDC4基因的表達水平與結腸癌發生發展密切相關.以往發現,缺氧條件能夠誘導結腸癌細胞上調SDC4基因表達[17].靶向抗腫瘤藥物曲妥珠單抗(Trastuzumab)與帕尼單抗(Panitumumab)能夠下調腫瘤細胞SDC4基因表達[7].除此之外,至今SDC4基因在結腸癌表達的分子調控機制并不清楚.研究闡明腫瘤相關基因表達的分子調控機制,能為揭示腫瘤發生發展病理機制提供重要線索.為此,本文通過生物信息學方法,研究分析了結腸癌中SDC4基因表達的分子調控機制.
本文發現:SDC4基因啟動子區包含6個甲基化位點.其中2個位點的甲基化水平在結腸癌組織顯著升高(圖3).H3K4Me1與H3K27Ac在SDC4基因的啟動子區、第一內含子顯著富集(圖4).SP1等共8個轉錄因子在結腸癌細胞中調控SDC4基因表達(圖5).hsa-miR-194和hsa-miR-124調控SDC4基因的表達(圖6).一般來說,基因的啟動子甲基化水平與其表達水平負相關[9].miRNA下調其靶基因的表達[13].H3K4me1、H3K27Ac與基因的轉錄激活正相關[9-11].轉錄因子既可上調基因表達,也可下調基因表達[12].本文發現,SDC4是hsa-miR-194和hsa-miR-124的靶基因.而hsa-miR-194和hsa-miR-124在結腸癌中低表達(圖6B).因此,結腸癌中hsa-miR-194和hsa-miR-124的表達下調,對維持SDC4基因的高表達有利.此外,結腸癌中SDC4基因啟動子區組蛋白修飾(H3K4Me1、H3K27Ac)顯著富集,也對維持SDC4基因高表達有利.然而,結腸癌中SDC4基因啟動子區甲基化水平上調,卻對維持SDC4基因高表達不利.本文既發現了對結腸癌SDC4基因高表達的不利因素,又發現對其有利的因素.這表明結腸癌SDC4基因表達的分子調控機制十分復雜.甲基化、轉錄因子、組蛋白修飾、miRNAs均與結腸癌SDC4基因表達調控有關.在結腸癌中,這些調控因素可能以某種方式嚴密組合、相互作用,共同決定SDC4基因的表達.
據報道,腫瘤細胞內共表達的基因之間具有緊密相關的生物學功能[18-20].為了探索SDC4基因在結腸癌發生發展中的作用.本文發現,SERINC3、DNTTIP1、ATP9A、CHMP4B、BCL2L1、STAU1、SYS1、YWHAB、OSBPL2、SLC35C2、TM9SF4共11個基因在結腸癌中與SDC4基因共表達(圖7).基因功能富集分析發現,這些基因參與18個生物學過程,其中包含3個細胞凋亡相關GO條目(圖8).失巢凋亡是由于細胞與細胞外基質失去接觸,而誘導的一種特殊的程序化細胞死亡形式[21-22].腫瘤細胞具有抗失巢凋亡的能力[23].據報道,在耐受失巢凋亡的模型細胞中,SDC4基因表達上調.敲低SDC4基因的表達,導致細胞增殖、侵襲和血管生成的能力下降[21-22].還發現,下調SDC4基因表達,可通過抑制Wnt/beta catenin信號通路的激活,促進甲狀腺癌細胞凋亡[6].本文發現YWHAB、SERINC3、BCL2L1等3個與SDC4共表達的基因,被聚類于細胞凋亡相關的GO條目.這一發現為今后研究SDC4調控細胞凋亡的分子機制提供新線索.本文還發現,SDC4基因參與調控細胞通訊等生物學過程.這與已經報道的SDC4基因的生物學功能一致.本文還發現,SDC4基因可能參與病毒的傳播、致病等病毒參與的生物學過程(GO:0016032).這是尚未報道的新發現,可能為研究拓展SDC4基因新的生物學功能提供線索.總之,本研究采用生物信息學方法揭示結腸癌SDC4基因高表達的分子調控機制與作用,為深入研究揭示結腸癌發生發展的病理機制奠定基礎.
4 結論
本研究采用多種生物信息學數據庫研究發現,在結腸癌中:SDC4基因高表達,而且其表達水平與腫瘤組織分期、淋巴結轉移正相關,與患者預后負相關.SDC4基因的表達可能被甲基化,組蛋白修飾(H3K4Me1與H3K27Ac),轉錄因子(SP1、TEAD4、JUND、FOSL1、NR1H3、PPARG、SRF、TFAP4),miRNAs(hsa-miR-194和hsa-miR-124)調控.SDC4基因通過其共表達基因參與結腸癌細胞凋亡、細胞通訊、物質運輸與定位等生物學過程.本研究為進一步闡明結腸癌SDC4基因表達調控機制與作用提供重要的新線索.
參考文獻:
[1]Keller-Pinter A,Gyulai-Nagy S,Becsky D,et al.Syndecan-4 in Tumor Cell Motility[J].Cancers(Basel),2021,13(13):1-24.
[2]Zhang Y,Li J,Partovian C,et al.Syndecan-4 modulates basic fibroblast growth factor 2 signaling in vivo[J].Am J Physiol Heart Circ Physiol,2003,284(6):2078-2082.
[3]Lambert J,Makin K,Akbareian S,et al.ADAMTS-1 and syndecan-4 intersect in the regulation of cell migration and angiogenesis[J].J Cell Sci,2020,133(7):1-22.
[4]Neel D S,Allegakoen D V,Olivas V,et al.Differential Subcellular Localization Regulates Oncogenic Signaling by ROS1 Kinase Fusion Proteins [J].Cancer Res,2019,79(3):546-556.
[5]Corti F,Finetti F,Ziche M,et al.The syndecan-4/protein kinase Calpha pathway mediates prostaglandin E2-induced extracellular regulated kinase(ERK)activation in endothelial cells and angiogenesis in vivo[J].J Biol Chem,2013,288(18):12712-12721.
[6]Chen L L,Gao G X,Shen F X,et al.SDC4 Gene Silencing Favors Human Papillary Thyroid Carcinoma Cell Apoptosis and Inhibits Epithelial Mesenchymal Transition via Wnt/beta-Catenin Pathway [J].Mol Cells,2018,41(9):853-867.
[7]Onyeisi J O S,Lopes C C,Gotte M.Syndecan-4 as a Pathogenesis Factor and Therapeutic Target in Cancer[J].Biomolecules,2021,11(4):1-16.
[8]Chuang T J,Chiang T W.Impacts of pretranscriptional DNA methylation,transcriptional transcription factor,and posttranscriptional microRNA regulations on protein evolutionary rate [J].Genome Biol Evol,2014,6(6):1530-1541.
[9]唐德平,姚慧慧,唐金舟,等.癌癥中microRNAs和表觀遺傳之間的相互調控作用[J].生物技術通報,2020,36(8):194-200.
[10]馬梅燕,冀慎英,張湘豫,等.基于生物信息學方法的人類GLTP在胰腺腺癌發生發展中的作用及其表觀遺傳調控機制分析[J].激光生物學報,2021,30(4):348-354.
[11]Beacon T H,Delcuve G P,Lopez C,et al.The dynamic broad epigenetic(H3K4me3,H3K27ac)domain as a mark of essential genes[J].Clin Epigenetics,2021,13(138):1-17.
[12]馮琳瓔,張利絨.表達相關的轉錄因子相互作用模式[J].內蒙古大學學報(自然科學版),2018,49(5):476-486.
[13]路玉盼,董憲喆,王瑾,等.Micrornas調控胃癌相關基因的研究進展[J].中國藥理學通報,2018,34(11):1481-1485.
[14]劉正,王錫山.國內外結直腸癌篩查的對比分析及啟示[J].中華結直腸疾病電子雜志,2022,11(1):18-23.
[15]Park H,Kim Y,Lim Y,et al.Syndecan-2 mediates adhesion and proliferation of colon carcinoma cells [J].J Biol Chem,2002,277(33):29730-29736.
[16]Jechorek D,Haeusler-Pliske I,Meyer F,et al.Diagnostic value of syndecan-4 protein expression in colorectal cancer [J].Pathol Res Pract,2021,222:1-9.
[17]Koike T,Kimura N,Miyazaki K,et al.Hypoxia induces adhesion molecules on cancer cells:A missing link between Warburg effect and induction of selectin-ligand carbohydrates [J].Proc Natl Acad Sci USA,2004,101(21):8132-8137.
[18]李雪霞,鄒焱堂,李炫坤,等.基于生物信息學數據庫分析IER2在腫瘤中高表達及其作用機制[J].中國生物化學與分子生物學報,2020,36(11):1333-1345.
[19]Stuart J M,Segal E,Koller D,et al.A gene-coexpression network for global discovery of conserved genetic modules [J].Science,2003,302(5643):249-255.
[20]王夢男,韓明飛,劉炳輝,等.基于集成的共表達網絡分析方法研究3種癌癥的腫瘤相關模塊[J].生物工程學報,2021,37(11):4111-4123.
[21]Onyeisi JOS,Pernambuco Filho PCA,Mesquita APS,et al.Effects of syndecan-4 gene silencing by micro RNA interference in anoikis resistant endothelial cells:Syndecan-4 silencing and anoikis resistance [J].Int J Biochem Cell Biol,2020,128:1-11.
[22]Carneiro B R,Pernambuco Filho P C,Mesquita A P,et al.Acquisition of anoikis resistance up-regulates syndecan-4 expression in endothelial cells [J].PLoS One,2014,9(12):1-23.
[23]Onyeisi JOS,Castanho de Almeida Pernambuco Filho P,de Araujo Lopes S,et al.Heparan sulfate proteoglycans as trastuzumab targets in anoikis-resistant endothelial cells [J].J Cell Biochem,2019,120(8):13826-13840.
The Study on the Expression Regulation Mechanism
and the Role of SDC4 Gene in Colon Cancer
Based on Bioinformatics Databases
TANG Zi-han1, YUAN Wei-si2, WU Ze-qiang1, ZHANG Yue-feng1, ZHAN Han1, WANG Chen-yao3, LI En-ming3, CHEN Zi-kai1, ZHOU Fei1*
(1. College of Life Sciences and Food Engineering, Hanshan Normal University, Chaozhou, Guangdong, 521041; 2. Department of Colorectal and Anorectal Surgery, Chaozhou Central Hospital, Chaozhou, Guangdong, 521041; 3. Shantou University Medical College, Shantou, Guangdong, 515041)
Abstract:To investigate the expression regulation mechanism and the role of SDC4 gene in colon cancer, GEPIA2 database, Ualcan database and Oncolnc database were used to study the expression of SDC4 gene in colon cancer and its effect on overall survival of the patients. Wanderer database, UCSC database, Cistrome DB database, miRcode database and miRDB database were used to study methylation and histone modification of the promoter region of SDC4 gene, and transcription factors and miRNAs for regulating the gene expression. GEPIA2 database, Ualcan database, HCMdb database and DAVID database were used to analyze genes co-expressed with SDC4 gene and the biological processes where these gene were involved in the colon cancer. The following results were found in colon cancer through the usage of the above bioinformatics database: The expression of SDC4 gene was significantly higher than that in adjacent normal tissues, and was positively correlated with tissue stage and lymph node metastasis, and negatively correlated with prognosis of patients in colon cancer. The methylation level of SDC4 gene promoter region was elevated in colon cancer. Histone modifications in transcriptional activation of H3K4Me1 and H3K27Ac were significantly enriched in the promoter of the SDC4 gene. A total of 8 transcription factors including SP1, TEAD4, JUND, FOSL1, NR1H3, PPARG, SRF, and TFAP4 might transcriptionally regulate the expression of SDC4 gene in colon cancer. Meanwhile, hsa-miR-194 and hsa-miR-124 might also regulate SDC4 gene expression. The expression of hsa-miR-194 and the expression of hsa-miR-124 were down-regulated in colon cancer. A total of 11 genes, namely SERINC3, DNTTIP1, ATP9A, CHMP4B, BCL2L1, STAU1, SYS1, YWHAB, OSBPL2, SLC35C2, and TM9SF4 were co-expressed with SDC4 gene in colon cancer. SDC4 gene was possibly involved in the regulation of biological processes such as apoptosis, cell communication, material transport and localization in colon cancer cells. The findings based on the bioinformatics database can provide important clues for further elucidating the regulatory mechanism and role of SDC4 gene expression in colon cancer.
Key words:bioinformatics; colon cancer; SDC4; expression regulation mechanism
責任編輯 周春娟
收稿日期:2022-09-28
基金項目:廣東省科技創新戰略專項資金(大學生科技創新培育)項目(項目編號:PDJH2023B0342);韓山師范學院教育教學改革項目(項目編號:粵韓師教[2021]148號);韓山師范學院科研項目(項目編號:XPY202106);廣東省教育廳科研項目(項目編號:2021ZDZX2066).
作者簡介:唐梓涵(1999-),男,廣東汕頭人,韓山師范學院生命科學與食品工程學院2020級學生.周飛為通訊作者.

