基于SSR熒光標記的新疆甜瓜種質資源遺傳多樣性與群體結構分析



摘"要:【目的】""分析新疆甜瓜種質資源的遺傳多樣性與群體結構,為甜瓜種質資源收集、遺傳改良和高效利用提供理論基礎。
【方法】""選取甜瓜種質182份,利用152對SSR引物對其進行檢測。篩選出19對多態性較高的引物,利用TP-M13-SSR分子標記分析182份新疆甜瓜種質材料的遺傳多樣性,并根據Nei's遺傳距離(D)進行聚類分析,以Structure軟件的混合模型聚類法分析其群體遺傳結構。
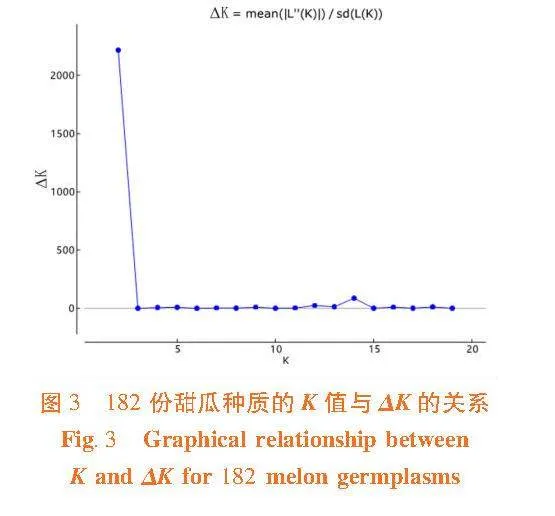
【結果】""SSR標記共檢測到160個等位基因,等位基因數(Na)的變化幅度為3~16個,平均每對引物8.421 1個,有效等位基因數(Ne)的變化幅度為1.092 0~6.087 9,平均為3.077 0;觀測雜合度(Ho)為0.011 0~1.000 0,平均0.309 7;期望雜合度(He)為0.084 5~0.838 0,平均為0.599 1;香農信息指數(I)范圍為0.190 6~2.061 3,平均為1.239 9;各位點的多態性信息含量(PIC)為0.081 1~0.815 8,平均為0.555 8,大多數位點表現為高度多態性。182份甜瓜種質材料被分為兩大亞群,亞群Ⅰ為88份甜瓜種質材料,亞群Ⅱ為94份甜瓜材料。當K=2時,ΔK出現明顯的峰值,182份甜瓜種質材料分為2個亞群較為合適,且存在過度的純合性。
【結論】""新疆甜瓜種質資源遺傳多樣性較為豐富,但與傳統4個變種的品種群分類結果不同。新疆甜瓜種質資源可分為兩大亞群,群體中純合個體較多,但其他雜合個體間存在一定的基因交流。
關鍵詞:""甜瓜種質;遺傳多樣性;SSR;熒光標記;群體結構
中圖分類號:"S652;S603.2""""文獻標志碼:"A""""文章編號:"1001-4330(2024)11-2614-12
0"引 言
【研究意義】甜瓜(Cucumis melo L., 2n=2x=24)屬葫蘆科黃瓜屬1年生草本植物,具有高度多樣化和多態化的特點[1-2]。甜瓜可分為長毛亞種C.melon L.subsp.melo和短毛亞種C.melo subsp.agrestis (Naudin) Pangalo)及其若干變種[3-6] 。分析新疆甜瓜種質資源的多樣性,對于甜瓜新品種選育、物種進化及分類具有重要意義[7-8]。【前人研究進展】形態學方法可用于甜瓜種質資源類群劃分和馴化聚類結構分析[7,9,10],中國新疆甜瓜地方品種與塔吉克斯坦甜瓜遺傳關系較近,可分為3個類群[11-13]。但分子標記技術穩定,較傳統形態學方法更能正確反映材料的遺傳背景和親緣關系[14]。SSR(Single nucleotide polymorphism,單核苷酸多態性)因其良好的再現性、多等位性和共顯性等優點[15]被應用于不同作物的遺傳圖譜[16,17]、指紋圖譜構建[18]、遺傳多樣性和群體結構分析等研究[19,20]。在甜瓜種質資源研究方面,SSR分子標記更多地被應用于指紋圖譜的構建[21]和遺傳多樣性分析[2,5,6,22-24]。TP-M13-SSR分子標記(Tailed primer M13 microsatellite markers)技術結合了SSR分子標記和熒光測序技術的優點,具有分辨率高、重復性強、結果準確、數據記錄工作量小等優點,被廣泛應用于不同作物種質資源的遺傳多樣性[24-27]、指紋圖譜構建[28,29]和品種鑒定等[30]。【本研究切入點】新疆是厚皮甜瓜的主產區,種質資源多樣復雜。目前利用TP-M13-SSR技術對新疆甜瓜種質資源遺傳多樣性及群體結構的研究分析鮮見報道。需基于SSR熒光標記分析新疆甜瓜種質資源遺傳多樣性與群體結構。【擬解決的關鍵問題】以收集保存的182份新疆甜瓜種質資源為研究對象,應用TP-M13-SSR分子標記分析其遺傳多樣性和群體結構,研究厚皮甜瓜種質之間的遺傳關系,為甜瓜種質資源的收集、引進以及品種改良提供科學依據。"
1"材料與方法
1.1"材 料
以收集的182份新疆地方厚皮甜瓜種質(其中南疆97份,東疆58份,北疆24份,不確定的3份)為材料,目前均保存于種質庫中。所有試驗材料均種植于新疆維吾爾自治區葡萄瓜果研究所西甜瓜基地溫室中。表1
1.2"方 法
1.2.1"基因組DNA的提取與檢測
采集供試材料幼嫩葉片,采用改良CTAB法[31]提取其基因組DNA。用美國Thermo Scientific NanoDrop One 超微量分光光度計和1%瓊脂糖凝膠電泳檢測DNA濃度、純度和質量,并用ddH2O將母液質量濃度稀釋至50 ng/μL,-20℃保存,備用。
1.2.2"引物合成與篩選
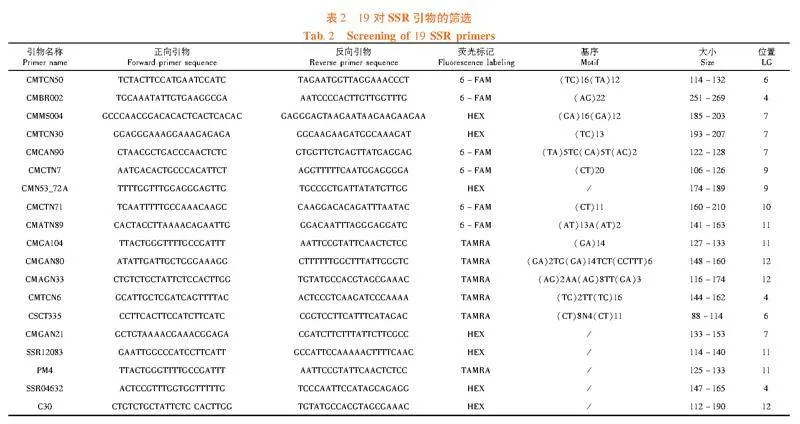
普通引物由北京華大基因科技有限公司合成。從報道的152對SSR引物中,通過8%非變性聚丙烯酰胺凝膠電泳、銀染檢測,初步篩選出條帶清晰、多態性高、穩定性強的引物。
TP-M13-SSR引物由5′端帶M13接頭序列(5′-TGTAAAACGACGGCCAGT-3′)的上下游引物和具有熒光標記(HEX-green、TAMRA-yellow、6-FAM-blue)的M13引物組成。所用引物由上海派森諾生物科技有限公司合成。
1.2.3"PCR擴增及其產物檢測
20 μL的SSR反應體系包括DNA模板1 μL,正向熒光引物(10 μmol/L)1 μL,反向普通引物(10 μmol/L)1 μL,10×buffer 2 μL,dNTP 0.5 μL,Taq酶0.5 μL,ddH2O 14 μL。PCR反應在PCR儀ABI-2720上進行,PCR反應條件為:95℃預變性5 min;95℃變性30 s,52~62℃退火30 s,72℃延伸30 s,10個循環;95℃變性30 s,52℃退火30 s,72℃延伸30 s,25個循環;72℃延伸7 min。
取1 μL 的PCR產物加入LIZ500分子量內標0.1 μL和去離子甲酰胺8.9 μL,混合加入PCR儀,95℃變性5 min,迅速冷卻到 4℃后離心,在ABI3730XL DNA遺傳分析儀上進行自動熒光檢測。
1.3"數據處理
用GeneMapper4.0軟件對ABI3730XL Date collection軟件收集的182份甜瓜種質資源的原始數據進行初步處理,分析擴增片段峰值,讀取擴增片段大小;運用PoPGen32軟件統計觀測等位基因數(Na)、有效等位基因數(Ne)、Shannon's 信息指數(I)、觀測雜合度(Ho)、期望雜合度(He)、Nei's遺傳距離值(D)、多態性信息含量指數(PIC)(由PIC_CALC計算)等遺傳學參數。在 MEGA5.1中構建遺傳聚類圖,用Origin軟件進行PCoA分析,用 STRUCTURE軟件(由structure2.3.4、CLUMPP_Windows.1.1.2、distrut1.1計算)進行群體結構分析,burnin period和MCMC iterations參數分別為10 000 和100 000,亞群數目(K)設置為1-20個,每個K值獨立運算10次,根據ΔK法確定最佳亞群數。根據最大遺傳比例進行亞群歸類。
2"結果與分析
2.1"SSR引物的篩選
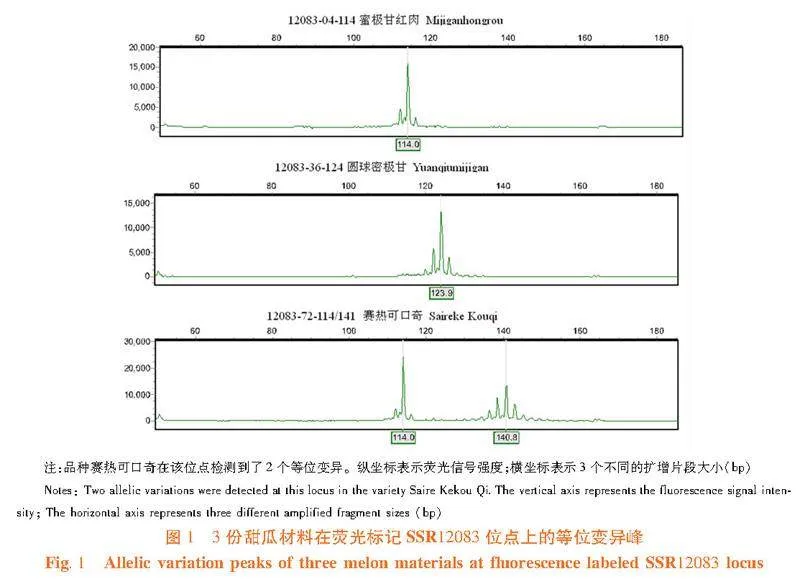
研究表明,利用156對SSR引物對21份果實差異性大的甜瓜品種進行PCR擴增,并對PCR產物進行8%非變性聚丙烯酰胺凝膠電泳檢測,篩選出19對條帶清晰、多態性高、擴增穩定的SSR引物,并用于182份甜瓜種質材料的熒光毛細管電泳試驗。其中19對SSR引物的峰圖簡單、等位變異合理,可用于試驗材料的遺傳多樣性分析。以引物SSR12083為例,毛細管電泳檢測結果能準確地讀取具體的片段大小。供試材料多為純合。表2,圖1
2.2"SSR分子標記多態性
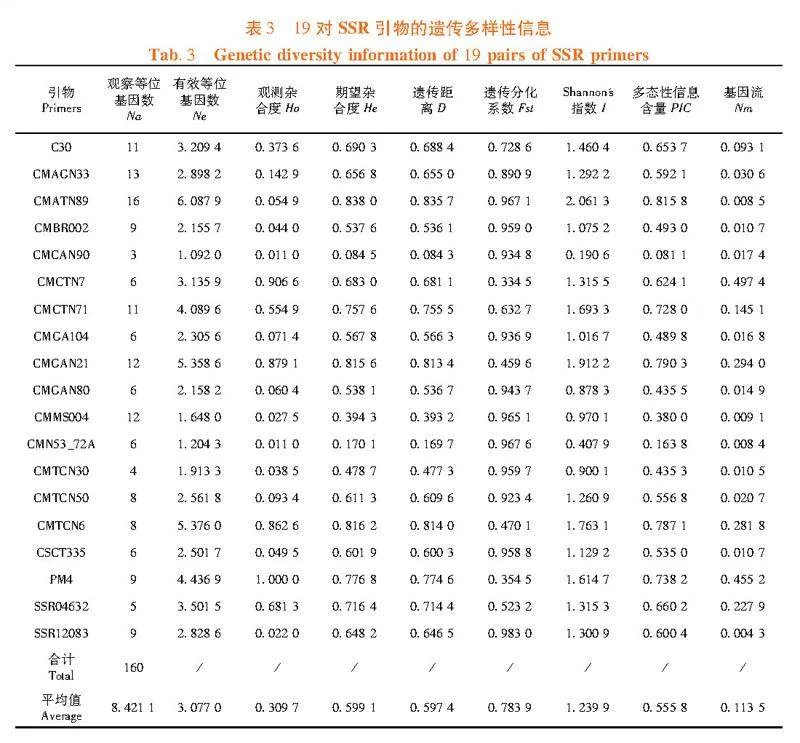
研究表明,19個SSR位點在182份新疆甜瓜種質資源中檢測到160個等位基因,Na的變化幅度為3~16個,平均為8.42個,這些引物可適于182份新疆甜瓜種質資源的遺傳多樣檢測。Ne是反映群體遺傳變異大小的指標,Ne和Na差異越大,等位基因在群體中分布越不均勻。Ne的變化幅度為1.092 0~6.087 9,平均為3.077 0,其中最高的是CMATN89,為6.087 9,其次為PM4和CMCTN71,該3對引物有較高的檢測效率。多態性信息含量(PIC)值大于 0.50 的位點為高度多態位點,處于0.25~0.50 的為中度多態位點,小于0.25 時為低度多態位。182份甜瓜種質資源材料的PIC值的變化范圍為 0.081 1~0.815 8,平均為0.555 8,其中12對引物的PIC值大于0.50,大部分屬于高度多態性SSR標記。表1
2.3"群體遺傳多樣性
研究表明,觀測雜合度(Ho)是指隨機抽取的2個樣本等位基因不同的概率;期望雜合度(He)是指理論計算得出的雜合度。He可以衡量群體的遺傳多樣性高低,He值越高,反映群體的遺傳一致性就越低,其遺傳多樣性就越豐富。Ho值范圍在0.011 0~1.000 0,平均為0.309 7;He值范圍在0.084 5~0.838 0,平均為0.599 1,且19對引物中,有13對的He均大于平均值,182份資源的遺傳多樣性豐富;Ho平均值明顯小于He平均值,且有15對引物的Ho低于He,絕大多數位點均出現了純合子偏多的現象。 Shannon's 指數(I)較He相比,更能反映遺傳多樣性程度的,I越大,群體離散度越高,遺傳多樣性越豐富。19對SSR引物的I值變化幅度為0.190 6~2.061 3,平均為1.239 9,研究群體遺傳多樣性比較豐富。SSR引物CMATN89、CMGAN21、CMTCN6對應的Nei's遺傳距離值(D)較高,分別為0.835 7、0.813 4、0.814,群體間這些基因位點的遺傳多樣性高。19對SSR引物鑒定計算的遺傳分化系數(Fst)值范圍在0.334 5~0.983 0,平均為0.783 9,大于0.25,種群間的遺傳分化水平處于較高水平,有78.39%的變異存在于甜瓜種質群體間,21.61%的變異存在于甜瓜種質群體內的個體間,182份新疆甜瓜種質資源的遺傳變異主要來源于種質群體間。基因流(Nm)值為0.004 3~0.497 4,平均為0.113 5且均小于1,種群間某些基因交流受到影響。表3
2.4"SSR分子標記的甜瓜種質聚類
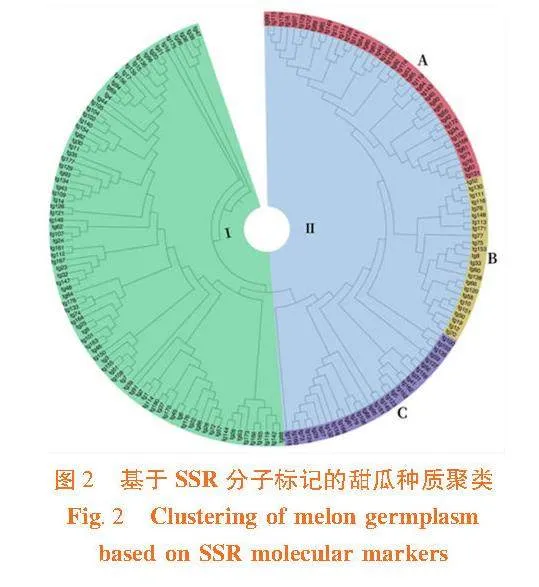
研究表明,南疆(97份)、東疆(吐魯番市、哈密市58份)、北疆(24份)及其他(3份)的182份甜瓜種質資源進行聚類可分為2個亞群。Ⅰ 類分別包含東疆甜瓜種質41份、南疆甜瓜種質29份、北疆甜瓜種質15份及其他甜瓜種質3份,共計88份,占總供試材料的48.35%,包括瓜旦甜瓜變種3份、夏甜瓜變種37份、冬甜瓜變種22份及未明確分類種質26份。其中來自皮山縣的努拉伊與其他種質遺傳距離較遠,單獨成一支,屬于夏甜瓜變種納西甘品種群;白巴登也單獨成一支;圓球密極甘(編號36)、阿比那瓦提(編號38)、一窩旦(編號47)和賽熱克吐木休克(編號96)聚為一支,其中編號38屬于夏甜瓜變種可口奇夏甜瓜品種群,編號96屬于冬甜瓜變種可口奇冬瓜品種群;Ⅱ 類分別包含南疆甜瓜種質69份、東疆甜瓜種質17份以及北疆甜瓜種質8份,共94份,占總供試材料的51.65%。Ⅱ類又可分為3個小群A、B和C。小群A包括41份甜瓜種質,其中南疆35份,東疆6份,含有夏甜瓜變種23份、冬甜瓜變種5份、未確定分類種質12份和瓜旦甜瓜變種1份;小群B包括24份甜瓜種質,其中南疆18份,東疆4份,北疆2份,包含夏甜瓜變種15份、冬甜瓜變種4份、未確定分類種質5份;小群C包括29份甜瓜種質,其中南疆16份,東疆7份,北疆6份,含有夏甜瓜變種8份、冬甜瓜變種5份、瓜旦甜瓜變種2份、卡沙巴甜瓜變種1份和未確定分類種質13份。供試材料中不同地方收集到的同名種質并未聚在1類,存在同名異物現象,如編碼1、21、72、85、92、145和159同為賽熱克可口奇,編碼67、77和176同為伯謝克辛,卻未能聚為一類。也存在疑似同名同物的,如編號93和129、編號15和136,聚類位置相同。圖2"
2.5"群體結構與主坐標
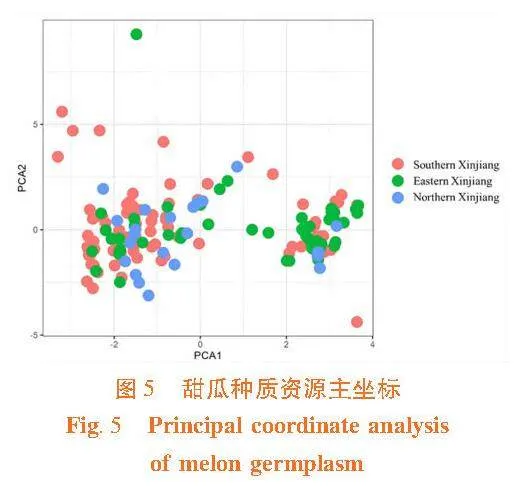
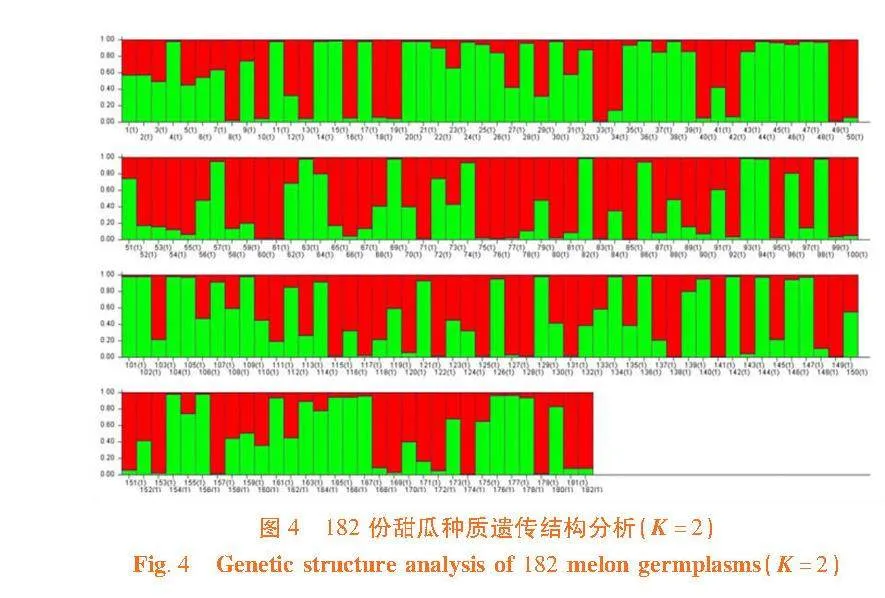
研究表明,當K=2時,ΔK出現峰值,將全部調查資源分為2個亞群。圖中每一種顏色代表一個亞群,每個小柱狀堆疊圖代表一個個體,亞群I(綠色所示)由份(48.35%)種質組成,亞群II(紅色所示)由份(51.65%)種質組成,與聚類結果一致。如果個體至少有20%的血統來自其他不同群體,則被認為是混合個體,亞群I中純合個體和混合個體的數量分別為66個(75.00%)和22個(25.00%)。亞群體II中則分別為66(70.21%)和28(29.79%)。對182份新疆甜瓜種質的SSR標記數據進行主坐標分析(PCoA),南疆(紅色)、東疆(綠色)和北疆(藍色)聚為兩大亞群,與群體結構分析相互驗證,結果一致。圖3,圖4,圖5
3"討 論
3.1
進行品種遺傳改良必須了解種質資源的遺傳信息與特點,遺傳多樣性作為生物所攜帶遺傳信息的總和,對分析重要性狀和輔助育種具有無可替代的作用[32]。
研究利用19個多態性SSR標記對中國新疆"的182份甜瓜種質進行檢測,平均等位基因數"(Na)為8.421 1,平均多態信息含量(PIC)為0.555 8,均大于Ansari等[33]研究的印度甜瓜、Carvalho等[34]研究的巴西商品甜瓜、Chikh-Rouhou等[35]研究的突尼斯甜瓜以及Kaar等[36]研究的土耳其甜瓜相應參數,說明新疆的甜瓜種質資源遺傳多樣性較為豐富,支持新疆為厚皮甜瓜的次級起源中心的假說[37]。期望雜合度值越高,反映群體的遺傳一致性就越低,其遺傳多樣性就越豐富。觀測雜合度(Ho)小于期望雜合度(He),純合子偏多。研究中觀測雜合度(Ho)值平均為0.309 7,期望雜合度(He)值平均為0.599 1,除SSR標記CMCTN7、CMGAN21、CMTCN6、PM4外其余的期望雜合度均高于觀測雜合度,大部分表現出純合子偏多的現象,可能由于早期交通閉塞形成小群體的分布模式,容易引起近交,從而導致群體內純合個體增加。基因流(Nm)表示基因在種群之間交換的多少。基因流數值大說明群體之間遺傳關系較近,反之亦然[38]。研究遺傳分化研究結果表明,基因流(Nm)為0.113 5,相對較低,說明群體間基因交流受到一定限制,可能是由于新疆地理條件影響,空間相對隔離,形成“島式”分布,導致群體間遺傳分化增大。遺傳分化系數(Fst)為0.783 9,大于0.25,新疆甜瓜群體間有很大遺傳分化。此結果與zhang等[23]研究結果一致。
3.2
試驗研究收集的各地域甜瓜種質數量分別為97份、24份、58份及3份未確定種質。利用19對SSR標記可將供試種質材料聚為2大亞群,Ⅰ類主要為新疆東疆和北疆收集的88份甜瓜種質資源,Ⅱ 類主要為南疆的94份甜瓜種質資源,Ⅱ類又可分為3個小群A、B和C,并未嚴格按地理位置區分,主坐標分析(PCoA)結果也說明這一點。張永兵等[13]利用表型數據對新疆121份甜瓜種質進行聚類分析表明,除哈密野瓜和炮彈瓜外,其余材料被聚為兩類,與研究結果一致。同時也顯示研究收集的甜瓜種質可能存在同名異物的現象,如卡拉尕西(編號49和153)、卡拉可口奇(編號49、170和178)、卡拉麥蓋(編號88、89和122)、波斯皮牙子(編號86、169、180和181)等。
群體結構分析可以揭示出種質材料的內在遺傳結構,有助于闡明其遺傳組成[39]。研究利用Structure軟件對182份新疆甜瓜種質進行分析,以確定研究群體的最佳亞群數目。參照Evanno等[40]的方法將甜瓜種質分為2個亞群,并且與張永兵等[13]利用表型分析對121份新疆甜瓜進行分類的結果相一致。類似地,Saha等[2]將印度46份甜瓜材料分為2個亞群,Andrade等[41]將巴西46份甜瓜分為2個亞群,說明中國新疆甜瓜和印度、巴西的甜瓜具有相似的遺傳馴化進程或遺傳背景或有某種聯系。陳蕓等[42]利用SRAP對61份甜瓜種質進行遺傳多樣性分析,結果顯示中國新疆甜瓜與土耳其、伊朗、阿富汗的厚皮甜瓜有共同的起源。楊永等[14]研究結果表明中國新疆甜瓜與印度、西班牙的甜瓜親緣關系較近。研究群體結構分析結果表明,供試的一些甜瓜種質存在較高的純合性,說明新疆地方甜瓜品種之間缺乏雜交或與其他群體異交[23],從而形成了獨特的基因庫。"
4"結 論
供試甜瓜種質的SSR分子標記分析揭示了新疆甜瓜種質資源存在較為豐富的遺傳多樣性。甜瓜種質聚類和PCoA分析未按照地域區分,且與傳統4個變種的品種群分類結果不同。新疆甜瓜種質資源可分為2大亞群,群體中純合個體較多,但其他混合個體間存在一定的基因交流。
參考文獻"(References)
[1]"王建玉, 鄧志斌, 張愛萍, 等.甜瓜種質資源遺傳多樣性的SSR分析[J].中國蔬菜, 2013, 1(18): 42-47.
WANG Jianyu, DENG Zhibin, ZHANG Aiping, et al.Analysis of melon germplasms genetic diversity by SSR markers [J].China Vegetables, 2013, 1(18): 42-47.
[2] Saha K, Choudhary H, Munshi A D, et al.Genetic characterization and population structure analysis among different horticultural groups of muskmelon (Cucumis melo L.) using microsatellite markers [J].Plant Genetic Resources: Characterization and Utilization, 2022, 20(2): 116-123.
[3] Liu S, Gao P, Zhu Q L, et al.Resequencing of 297 melon accessions reveals the genomic history of improvement and loci related to fruit traits in melon [J].Plant Biotechnology Journal, 2020, 18(12): 2545-2558.
[4] 崔浩楠.甜瓜白粉病抗性基因Cmpmr 2F的精細定位及候選基因分析[D].哈爾濱:東北農業大學, 2021.
CUI Haonan.Fine mapping and candidate gene analysis of powdery mildew resistance gene Cmpmr 2F in melon [D].Haerbin: Northeast Agricultural
University, 2021.
[5] 張凱歌, 胡倩梅, 靳志恒, 等.219份甜瓜種質資源的遺傳多樣性分析[J].河南農業大學學報, 2020, 54 (2): 216-230.
ZHANG Kaige, HU Qianmei, JIN Zhiheng, et al.Genetic diversity analysis of 219 melon germplasms [J].Journal of Henan Agricultural University, 2020,
54(2): 216-230.
[6] 徐小軍, 劉海英, 梁長志, 等.不同類型甜瓜種質SSR遺傳多樣性及耐冷性評價[J].植物遺傳資源學報, 2020, 21(3): 568-578.
XU Xiaojun, LU Haiying, LIANG Changzhi, et al.Genetic diversity analysis of melon germplasms using SSR markers and tests for chilling tolerance [J]. Journal of Plant Genetic Resources, 2020, 21(3): 568-578.
[7] 卿東山, 江鴻, 張露瑤, 等.甜瓜種質資源形態學性狀遺傳多樣性分析[J].中國蔬菜, 2023,(4): 39-49.
QING Dongshan, JIANG Hong, ZHANG Luyao, et al.Analysis of genetic diversity of melon(Cucumis melo L.)germplasm resources based on mor phological characters [J].China Vegetables, 2023,(4): 39-49.
[8] 王志強, 杜慧瑩, 李程, 等.甜瓜親本資源果實性狀的遺傳多樣性分析[J].內蒙古農業大學學報(自然科學版), 2022, 43(6): 7-13.
WANG Zhiqiang, DU Huiying, LI Cheng, et al.Genetic diversity analysis of fruit characters of melon parents [J]. Journal of Inner Mongolia Agricultural University(Natural Science Edition), 2022, 43(6): 7-13.
[9] 王吉明, 尚建立, 李娜, 等.引進國外的野生甜瓜種質資源果實形態多樣性分析[J].果樹學報, 2017, 34(3): 295-302.
WANG Jiming, SHANG Jianli, LI Na, et al.Genetic diversity analysis in imported wild melon (Cucumis melo L.) germplasm resources based on morphological characters [J].Journal of Fruit Science, 2017, 34(3): 295-302.
[10] 胡建斌, 馬雙武, 簡在海, 等.中國甜瓜種質資源形態性狀遺傳多樣性分析[J].植物遺傳資源學報, 2013, 14(4): 612-619.
HU Jianbin, MA Shuangwu, JIAN Zaihai, et al.Analysis of genetic diversity of Chinese melon(Cucumis melo L.)germplasm resources based on morphological characters [J].Journal of Plant Genetic Resources, 2013, 14(4): 612-619.
[11] 張永兵, 伊鴻平, 馬新力, 等.新疆甜瓜地方品種資源核心種質構建[J].植物遺傳資源學報, 2013, 14(1): 52-57.
ZHANG Yongbing, YI Hongping, MA Xinli, et al.Construction of core collection of melon landraces in Xinjiang [J].Journal of Plant Genetic Resources, 2013, 14(1): 52-57.
[12] 郭麗霞, 李寐華, 王豪杰, 等.塔吉克斯坦甜瓜地方品種資源表型遺傳多樣性[J].新疆農業科學, 2015, 52 (5): 837-842.
GUO Lixia, LI Meihua, WANG Haojie, et al.Genetic diversity of melon landraces (Cucumis melo L.) in Tajikistan based on phenotypic characters [J].Xinjiang Agricultural Science, 2015, 52(5): 837-842.
[13] 張永兵, 李寐華, 吳海波, 等.新疆甜瓜地方品種資源的表型遺傳多樣性[J].園藝學報, 2012, 39(2): 305-314.
ZHANG Yongbing, LI Meihua, WU Haibo, et al.Genetic diversity of melon landraces(Cucumis melo L.)in Xinjiang based on phenotypic characters [J]. Acta Horticulturae Sinica, 2012, 39(2): 305-314.
[14] 楊永, 王豪杰, 張學軍, 等.新疆甜瓜地方種質資源遺傳多樣性的SRAP分析[J].植物遺傳資源學報, 2017, 18(3): 436-448.
YANG Yong, WANG Haojie, ZHANG Xuejun, et al.Genetic diversity analysis of melon landraces (Cucumis melo) in Xinjiang based on SRAP markers [J].Journal of Plant Genetic Resources, 2017, 18(3): 436-448.
[15] Zhu H Y, Guo L Q, Song P Y, et al.Development of genome-wide SSR markers in melon with their cross-species transferability analysis and utilization in genetic diversity study [J].Molecular Breeding2016, 36(11): 153-167.
[16] 張金鵬.遺傳作圖群體中SSR基因型分型軟件開發及楊樹SSR遺傳圖譜構建[D].南京: 南京林業大學, 2022.
ZHANG Jinpeng.Software development of SSR genotyping across a genetic mapping population and construction of SSR genetic maps in Populus [D].Nanjing: Nanjing Forestry University, 2022.
[17] Wang X L, Yang X Q, Yao J T, et al.Genetic linkage map construction and QTL mapping of blade length and width in Saccharina japonica using SSR and SNP markers [J].Frontiers in Marine Science, 2023, 10: 1116412.
[18] 冉昆, 隋靜, 王宏偉, 等.利用SSR熒光標記構建山東地方梨種質資源分子身份證.果樹學報[J], 2018, 35(增刊): 71-78.
RAN Kun, SUI Jing, WANG Hongwei, et al.Using the fluorescent labeled SSR markers to establish the molecular ID of pear germplasm resources in Shandong [JJournal of Fruit Science.2018, 35(S): 71-78.
[19] Alhariri A, Behera T K, Jat G S, et al.Analysis of ge- netic diversity and population structure in bitter gourd (Momordica charantia L.) using morphological and SSR markers [J].Plants, 2021(9): 1860.
[20] Zhang Q H, Zang X X, Yang Y C, et al.Genetic diversity of Juglans mandshurica populations in northeast china based on SSR markers [J].Frontiers in Plant Science, 2022, 13: 931578.
[21] 李超, 楊英, 陳偉, 等.西州密系列甜瓜 SSR 指紋圖譜構建及聚類分析[J].園藝學報, 2022, 49(3): 622-632.
LI Chao, YANG Ying, CHEN Wei, et al.Construction of DNA fingerprinting and clustering analysis with SSR markers for the muskmelon of Xizhoumi Series [J].Acta Horticulturae Sinica, 2022, 49(3): 622-632.
[22] 翟文強, 王豪杰, 李俊華, 等.喀什地區厚皮甜瓜品種資源遺傳多樣性分析[J].新疆農業科學, 2011, 48(1): 64-69.
ZHAI Weiqiang, WANG Haojie, LI Junhua, et al.Analysis of genetic diversity in melon variety resources in Kashgar region [J].Xinjiang Agricultural Sciences, 2011, 48(1): 64-69.
[23] Zhang Y B, Fan X B, Aierken Y, et al.Genetic diversity of melon landraces (Cucumis melo L.) in the Xinjiang "Autonomous Region on the basis of simple sequence repeat markers [J].Genetic Resources and CropEvolution, 2017, 64: 1023-1035.
[24] 王璐偉, 沈志軍, 李賀歡, 等.基于SSR 熒光標記的79份桃種質遺傳多樣性分析[J].中國農業科學, 2022, 55(15): 3002-3017.
Wang Luwei, SHEN Zhijun, LI Hehuan, et al.Analysis of genetic diversity of 79 cultivars based on SSR fluorescence markers for peach [J].Scientia Agricultura Sinica, 2022, 55(15): 3002-3017.
[25] 劉松, 聶興華, 李伊然, 等.基于SSR熒光標記構建板栗品種(系)核心種質群[J].果樹學報, 2023, 40(2): 230-241.
LIU Song, NIE Xinghua, LI Yiran, et al.Construction of core germplasm collection of Chinese chestnut cultivars (lines) based on SSR fluorescence markrs [J].Journal of Fruit Science, 2023, 40(2): 230-241.
[26] 侯麗媛, 董艷輝, 聶園軍, 等.蘋果屬種質 TP-M13-SSR 親緣關系及遺傳多樣性分析[J].山西農業科學, 2020, 48(9): 1371- 1378, 1522.
HOU Liyuan, DONG Yanhui, NIE Yuanjun, et al.Analysis of phylogenetic relationship and genetic diversity of TP-M13-SSR technique for some malus germplasm[J].Journal of Shanxi Agricultural Sciences, 2020, 48(9): 1371-1378, 1522.
[27] 解華云, 高崇敏, 葉云峰, 等.基于TP-M13-SSR分子標記技術的133份薄皮甜瓜種質資源遺傳多樣性分析[J].南方農業學報.2022, 53(9): 2547-2556.
XIE Huayun, GAO Chongmin, YE Yunfeng, et al.Genetic diversity analysis of 133 pellicle melon germplasm resources based on TP-M13-SSR molecular marker technology[J].Journal of Southern Agriculture, 2022, 53(9): 2547-2556.
[28] 吳仕蔓, 婁兵海, 陳傳武, 等.應用SSR熒光標記法構建22個柚類品種的分子身份證[J].果樹學報, 2023, 40(4): 605-614.
WU Shiman, LOU Binghai, CHEN Chuanwu, et al.Establishment of molecular identity of 22 pomelo varieties using fluorescent labeled SSR markers [J].
Journal of Fruit Science, 2023, 40(4): 605-614.
[29] 位明明, 黃肖, 李維國, 等.利用 SSR 熒光標記構建 37 個中國橡膠樹栽培品種指紋圖譜[J].熱帶作物學報, 2022, 43(8): 1565-1576.
WEI Mingming, HUANG Xiao, LI Weiguo, et al.Using the SSR fluorescent labeling to establish SSR fingerprints for 37 cultivars in rubber tree (Hevea brasiliensis) [J].Chinese Journal of Tropical Crops, 2022, 43(8): 1565-1576.
[30] 張志軍, 黃克興, 岳杰, 等.TP-M13-SSR技術在麥芽品種鑒定中的應用[J].食品科學.2018, 39(24): 183-188.
ZHANG Zhijun, HUANG Kexing, YUE Jie, et al.DNA fingerprinting of malt varieties using tailed primer M13 mi crosatellite (TP-M13-SSR) markers [J].Food Science, 2018, 39(24): 183-188.
[31] 李超, 孫玉萍, 楊英, 等.應用 SSR 標記鑒定甜瓜新品種純度與真實性研究[J].分子植物育種, 2017, 15(8): 3088- 3096.
LI Chao, SUN Yuping, YANG Ying, et al.Application of SSR markers in purity and authenticity identification for melon new hybrid [J]. Molecular Plant Breeding, 2017, 15(8): 3088-3096.
[32] 穆小婷, 廖偵成, 凌彩金, 等.基于SSR標記的55份清遠野生茶樹種質資源遺傳多樣性和親緣關系分析[J].中國茶葉, 2023, 45(2): 36-43.
MU Xiaoting, LIAO Zhicheng, LING Caijin, et al.Genetic Diversity and Relationship Analysis of 55 Wild Tea Germplasm from Qingyuan based on SSR Markers [J]. China Tea, 2023, 45(2): 36-43.
[33] Ansari W A, Atri N, Yang L, et al.Genetic diversity in muskmelon based on SSR markers and morphological traits under well-watered and water-deficit [J]. Biocatalysis and Agricultural Biotechnology, 2020, 26(4): 101630.
[34] Carvalho N, Canela F M, Leite P H S, et al.Research Article Analysis of genetic variability of commercial melon cultivars using SSR molecular markers [J].Genetics and Molecular Research, 2017.16(3): 16039739.
[35] Chikh-rouhou H, Mezghani N, Mnasri S, et al.Assessing the Genetic Diversity and Population Structure of a Tunisian Melon (Cucumis melo L.) Collection Using Phenotypic Traits and SSR Molecular Markers [J].Agronomy, 2021, 11(6): 11061121.
[36] Kacar Y A, Simsek O, Solmaz I, et al.Genetic diversity among melon accessions (Cucumis melo) from Turkey based on SSR markers [J].Genetics and Molecular Research.2012, 11(4): 4622.
[37] Luan F S, Delannay I, Staub J E.Chinese melon(Cucumis melo L.)diversity analyses provide strategies for germplasm curation, genetic improvement, and evidentiary support of domestication patterns [J].Euphytica2008, 164(2): 445-461.
[38] 吳嫻, 徐海峰, 張志斌, 等.基于KASP標記的貴州禾群體遺傳多樣性與結構分析[J].分子植物育種, 2021, 19(4): 1345-1353.
WU Xian, XU Haifeng, ZHANG Zhibin, et al.Genetic Diversity and Population Structure Study of Guizhou He Based on KASP Marker [J].Molecular Plant Breeding, 2021, 19(4): 1345-1353.
[39] 王美榮, 許勇, 詹永樂, 等.甜瓜育種親本材料遺傳多樣性及群體結構的SSR標記分析[J], 華北農學報, 2010, 25(增刊): 41-46.
WANG Meirong, XU Yong, ZHAN Yongle, et al.Analysis of Genentic Diversity and Population Structure in Melon (Cucumis melo L.) Breeding Materials by SSR Markers [J], Acta Agriculture Boreali-Sinica, 2010, 25(S): 41-46.
[40] Evanno G, Regnaut S, Goudet J.Detecting the number of clusters of individuals using the software structure: a simulation study [J].Molecular Ecology, 2005, 14(8): 2611-2620.
[41] Andrade I S, de Melo C A F, de Sousa Nunes G H, et al.Phenotypic variability, diversity and genet ic population structure in melon (Cucumis melo L.) Associated with total soluble solids [J].Scientia Horticulturae, 2021, 278 (1): 109844.
[42] 陳蕓, 李冠, 王賢磊.甜瓜種質資源遺傳多樣性的SRAP分析[J].遺傳, 2010, 32(7): 744-751.
CHEN Yun, LI Guan, WANG Xianlei.Genetic diversity of a germplasm collection of Cucumis melo L.using SRAP markers [J].Hereditas (Beijing), 2010, 32(7): 744-751.
Genetic diversity and population structure analysis of melon"""germplasm resources in Xinjiang based on SSR fluorescence markers
LI Chao, YANG Ying, ZHENG Heyun, YANG Jianli, CHEN Wei, YANG Mi, SUN Yuping
(Research Institute of Grape and Melon of Xinjiang Uighur Autonomous Region, Shanshan Xinjiang 838200, China)
Abstract:【Objective】 ""To analyze the genetic diversity and population structure of Xinjiang melon germplasm resources in the hope of providing a theoretical basis for future collection, genetic improvement, and efficient utilization of melon germplasm resources.
【Methods】 """182 melon germplasms were selected, and 19 pairs of primers with high polymorphism were selected from 152 pairs of SSR primers.The genetic diversity of 182 Xinjiang melon germplasms was analyzed using TP-M13-SSR molecular markers, and the cluster analysis was conducted according to Nei's genetic distance (D), and the population genetic structure was analyzed using the hybrid model cluster method of Structure software.
【Results】 """The results showed that a total of 160 alleles were detected using SSR markers, with a variation range of 3-16 allele numbers (Na), an average of 8.421,1 per primer pair, and a variation range of 1.092,0-6.087,9 effective allele numbers (Ne), an average of 3.077,0; The observed heterozygosity (Ho) ranges from 0.011,0 to 1.000,0, with an average of 0.309,7; The expected heterozygosity (He) is 0.084,5-0.838,0, with an average of 0.599,1; The Shannon’ S Diversity Index (I) from 0.190,6 to 2.061,3, with an average of 1.239,9; The polymorphism information content (PIC) of each site from 0.081,1 to 0.815,8, with an average of 0.555,8.Most sites exhibit high polymorphism.The cluster analysis results showed that 182 melon germplasm materials were divided into two major subgroups, with subgroup I consisting of 88 melon germplasm materials and subgroup II consisting of 94 melon materials.The analysis of population genetic structure showed that when K=2, ΔK showed a significant peak, indicating that 182 melon germplasm materials were more suitable to be divided into two subgroups and there was excessive homozygosity.
【Conclusion】 """The genetic diversity of Xinjiang melon germplasm resources is relatively rich, but the classification results are different from the four traditional varieties.It can be seen that Xinjiang melon germplasm resources can be divided into two major subgroups from the analysis of population structure.There are more homozygous individuals in the population, but there is some gene exchange between other heterozygous individuals, which may be due to variety improvement or germplasm innovation by breeders.
Key words:""melon germplasm; genetic diversity; SSR; fluorescence markers; population structure analysis
Fund projects:""Tianshan Youth Program Project (2020Q140);Key Ramp;D Projects in Turpan City (2023003) ; Tianshan Innovation Team Plan (2021D14009) ; Basic Scientific R amp;D Program of Public Welfare Research Institutions of Xinjiang Uygur Autonomous Region (KY2022109); National Consortium Research Project on Muskmelons Breeding of Characteristic Species; Tianshan Talent Plan Phase III of Xinjiang Uygur Autonomous Region
Correspondence author:"""SUN Yuping (1979-) , female, from Gansu,research associate,research direction:breeding of watermelon and melon, (E-mail) 1501218842@qq.com

