2019-2022年中國H6N1亞型禽流感病毒的生物學特性分析
陳源,崔鵬飛,施建忠,張元成,于晴晴,顏成,張亞萍,王叢叢,張潔,王燕,鄧國華,陳化蘭
2019-2022年中國H6N1亞型禽流感病毒的生物學特性分析
陳源,崔鵬飛,施建忠,張元成,于晴晴,顏成,張亞萍,王叢叢,張潔,王燕,鄧國華,陳化蘭
中國農業科學院哈爾濱獸醫研究所/動物疫病防控全國重點實驗室/國家禽類實驗動物資源庫,哈爾濱 150069
【背景】H6亞型禽流感病毒(avian influenza virus,AIV)廣泛流行于我國南方地區,是我國家禽中最常見AIV之一。H6N1亞型AIV頻繁地與其他野鳥源毒株重配,并且可以作為供體為高致病性AIV提供內部基因片段,產生新型重組病毒,進而威脅人類健康。【目的】通過對我國H6N1亞型AIV的演化動態及其相關生物學特性分析研究,為我國禽流感的綜合防控提供數據支持。【方法】采集2019—2022年我國25個省(直轄市、自治區)的活禽交易市場及養殖場家禽喉和泄殖腔拭子,通過接種雞胚分離到7株H6N1亞型AIVs并對其進行全基因組測序,分析其遺傳演化特征、受體結合特性及其對SPF雞和BALB/c小鼠的感染性。【結果】遺傳演化分析表明,7株病毒的基因與分離于北美及東南亞地區的野鳥源病毒基因同源性較高,基因來源復雜,具有明顯的遺傳多樣性。貝葉斯演化分析表明,H6亞型AIV HA基因曾發生過多次的跨洲際傳播,歐亞譜系病毒在北美地區也有著較長時間流行。1株病毒HA基因與北美地區毒株基因高度同源,根據貝葉斯演化分析結果,推測該病毒在野鳥體內經歷了復雜的基因重配后形成,后經野鳥傳入我國。特殊氨基酸位點分析結果顯示,病毒HA蛋白裂解位點序列均為PQIETR↓GLF,符合低致病性AIV特征;此外,另有1株病毒的NP蛋白發生Y52H突變,據報道,該突變對AIV獲得抵抗人干擾素刺激基因BTN3A3的能力起到關鍵作用。受體結合特性分析表明,部分毒株具有雙受體結合特性,但結合人源受體能力弱于結合禽源受體能力。病毒對SPF雞的感染性試驗表明,雞感染A/chicken/Jiangxi/S40445/2019(H6N1)后能通過呼吸道及消化道排毒,并且病毒可在雞群內通過接觸傳播。雞感染A/duck/Jiangxi/S10941/2019(H6N1)后僅有少數雞通過呼吸道排毒,病毒無法在雞群間通過接觸傳播。BALB/c小鼠的感染性試驗表明,H6N1亞型AIV無需提前適應便可在小鼠呼吸道內有效復制,但對小鼠仍呈低致病力。【結論】2019—2022年分離于我國的H6N1亞型AIV基因大部分來源于野鳥源病毒,候鳥可經東亞-澳大利亞遷徙路線將病毒傳入我國;部分病毒能夠結合人源唾液酸受體并在小鼠呼吸道內有效復制,表明該亞型病毒對公共衛生安全構成潛在威脅。
H6N1;禽流感病毒;跨洲際傳播;重配;感染性
0 引言
【研究意義】禽流感病毒(avian influenza virus,AIV)是單股負鏈分節段的RNA病毒,病毒基因組包含8個基因片段。不同亞型AIV之間常發生基因的交換,這個過程稱為基因重配。一些通過基因重配產生的新型病毒不僅會對家禽養殖業造成嚴重經濟損失,甚至還能突破種間屏障感染人。因此,持續地對AIV進行監測并分析其生物學特性,具有重要的意義。【前人研究進展】H6亞型AIV于1965年被首次報道分離自火雞,隨后逐漸在多種野鳥和家禽中分離到不同NA亞型的H6亞型AIV[1-4]。禽流感監測數據表明,H6亞型AIV在我國南方地區廣泛存在,是水禽中最常見的AIV之一[5]。2000—2005年,中國香港學者對我國南方地區分離到的H6亞型AIV進行研究發現,H6N2亞型為病毒的優勢亞型,依據其遺傳演化關系,鴨源H6N2亞型AIV的HA進化樹可分為3個分支,分別為ST339-like分支、ST2853-like分支和HN573- like分支[6]。伴隨著時間的推移,H6亞型AIV不斷演化,H6N6亞型AIV取代H6N2亞型AIV,成為我國水禽中最常見的H6亞型AIV[7-8]。H6亞型AIV不僅在水禽中廣泛流行,甚至能跨物種感染哺乳動物和人類。2011年,ZHANG等[9]在廣東省豬群中分離到1株H6N6亞型AIV;2013年,臺灣報道了全球首例人感染了H6N1亞型AIV病例[10-11]。H6N1病毒在我國H6亞型AIV中分離率較低,主要分布于我國臺灣、香港地區以及我國大陸東南地區[12-13]。我國鴨群中的H6N1亞型AIV頻繁地與野鳥源病毒重配,內部基因來源復雜[8]。【本研究切入點】目前對近年來我國流行的H6N1亞型AIV的研究較少,其相關生物學特性尚未被系統分析。本研究對2019—2022年期間分離自我國的H6N1亞型AIV進行了系統的生物學特性研究,包括基因溯源分析、分離株的受體結合特性分析以及分離株對SPF雞、BALB/c小鼠的感染性分析。【擬解決的關鍵問題】了解我國最新分離到的H6N1亞型AIV的演化動態,評價其對雞的感染性和跨物種感染人的風險。
1 材料與方法
1.1 試驗時間與地點
本研究于2019—2023年在中國農業科學院哈爾濱獸醫研究所完成。
1.2 毒株
本研究中的7株H6N1亞型AIVs由國家禽流感參考實驗室分離、鑒定并保存。
1.3 實驗動物、雞胚
9—11日齡的SPF雞胚購自國家禽類實驗動物資源庫。BALB/c小鼠購自北京維通利華實驗動物技術有限公司。
1.4 主要實驗試劑
病毒RNA提取試劑盒購自天根生化科技(北京)有限公司;M-MLV逆轉錄酶購自美國Promega公司;2× Taq Master Mix購自北京康為世紀生物科技有限公司;測序反應試劑盒Big Dye Terminator 3.1購自美國ABI公司。
1.5 病毒分離及亞型鑒定
將家禽咽喉及泄殖腔拭子旋渦震蕩、離心后取上清液,以0.2 mL/枚接種于9—11日齡雞胚,37℃孵化48 h后收集雞胚尿囊液。參照血凝試驗國家參考標準測定尿囊液血凝性,并對血凝陽性尿囊液通過H1—H16亞型AIV單因子血清及新城疫血清確定其HA亞型。
1.6 病毒純化與EID50測定
將病毒懸液與磷酸緩沖鹽溶液(phosphate buffer solution,PBS)以1﹕9的比例混合,置于渦旋振蕩器上使其充分混勻,隨后用PBS 10倍連續稀釋,以0.1 mL/枚接種于雞胚。將雞胚置于37℃恒溫孵化箱中,48 h后測定雞胚尿囊液的血凝性,將最高稀釋度、最高血凝價的雞胚尿囊液分裝凍存于-70℃冰箱中。取純化后病毒,將病毒用PBS 10倍連續稀釋,每個稀釋度病毒懸液以0.1 mL重復接種5個雞胚。接種完畢后將雞胚置于37℃恒溫孵化箱,48 h后將雞胚取出,收集雞胚尿囊液,測定尿囊液血凝性。利用Reed- Muench方法計算各病毒的雞胚半數感染量(fifty percent embryo infectious dose, EID50)。
1.7 病毒全基因組測序及遺傳演化分析
參照核酸提取試劑盒說明書,取140 μL純化后的病毒,提取病毒RNA,隨后以引物(5′-AGCRAAA GCAGG-3′)為反轉錄引物,參照M-MLV逆轉錄酶使用說明書,將病毒RNA反轉錄得到cDNA。利用Hoffmann等[14]設計的引物,以病毒cDNA為模板,擴增病毒全基因組。將擴增產物利用磁珠純化后,參照Big Dye Terminator 3.1測序反應說明書,進行測序反應,反應完成后利用磁珠將測序反應產物純化,純化后的產物在測序儀上進行基因序列的測定。利用DNA-Star軟件拼接序列后,在Phylosuite[15]中進行序列的比對以及ML樹的重建。采用貝葉斯節點標記法,在BEAST V1.10.4[16]重建系統發生關系,并獲得進化樹上各個節點的絕對分化時間及其可信區間。在iTOL(https://itol.embl.de/)對進化樹美化。
1.8 病毒對BALB/c小鼠的感染性試驗
將病毒以106EID50/50 μL的劑量經鼻腔感染6周齡BALB/c小鼠,每組感染8只,在感染后的第3天隨機安樂死3只小鼠,采集小鼠的鼻甲、腦、肺臟、脾臟、腎臟,定容于1 mL PBS中,臟器經研磨后,用PBS對臟器研磨液10倍倍比稀釋,在每個稀釋度的研磨液中取0.1 mL接種于9—11日齡雞胚,48 h后計算每個稀釋度研磨液的血凝陽性雞胚數量,利用Reed-Muench方法計算小鼠臟器內的病毒滴度。其余組內的5只小鼠繼續稱重至第14天,計算其體重變化以及觀察其健康情況。
1.9 病毒對SPF雞的感染性以及傳播性試驗
每個隔離器內設置10只感染組雞、3只復制組雞,另設置3只接觸傳播組雞。將感染組雞和復制組雞以106EID50/100 μL的劑量經鼻腔滴注病毒,感染24 h后,將接觸傳播組雞放入隔離器中。感染后第3、5、7、9、11和13天采集感染組雞和接觸傳播組雞的咽喉和泄殖腔拭子;感染后第3天安樂死復制組雞,取其腦、氣管、胸腺、肺臟、腎臟、脾臟、胰腺、心臟、肝臟、法氏囊和盲腸,用于病毒滴定。在感染后第21天,測定感染組雞及接觸傳播組雞血清轉陽情況。
1.10 病毒的受體結合特性分析
參照文獻[17]的方法,將初始濃度為1 ng·μL-1糖鏈進行2倍倍比稀釋,以100 μL/孔的標準加入96孔ELISA板中,4℃條件下包被過夜。棄去96孔板中液體,用預冷的PBS洗滌4次。將病毒配置成64單位抗原后,以100 μL/孔的標準加入ELISA板中,4℃條件下孵育6 h。用含Tween-20的磷酸鹽緩沖液(PBS with tween-20, PBST)洗滌ELISA板4次,用預冷的PBS洗滌1次。加入1﹕300比例稀釋的雞多抗血清100 μL/孔,4℃孵育2 h。PBST洗滌4次后,加入辣根過氧化物酶標記的羊抗雞二抗,37℃孵育1 h后洗滌。加入顯色液,避光顯色5 min。ELISA板中每孔加入0.5 mol·L-1硫酸50 μL,終止顯色反應。在490 nm讀取OD值,利用GraphPad分析結果并作圖。
2 結果
2.1 病毒分布
2019—2022年于我國25個省(直轄市、自治區)的活禽交易市場及養殖場內對家禽進行禽流感主動監測,共分離到7株H6N1亞型AIVs。7株病毒均分離自我國南方地區,其中湖南省分離到2株,江西省分離到2株,另外在我國貴州省、湖北省、福建省各分離到1株病毒。7株病毒中有5株分離于養殖場,2株分離于市場。僅有1株病毒分離自雞,其余病毒均分離自鴨(表1)。

表1 H6N1亞型AIVs信息
2.2 病毒基因組序列的同源性分析
在GISAID數據庫中通過BLAST得到與7株H6N1亞型AIVs各節段基因核苷酸相似性最高的病毒(表2)。結果顯示,7株H6N1亞型 AIVs 的全基因組與數據庫中分離地為東亞及北美的野鳥源病毒的基因同源性較高,達到98%以上。推測H6N1病毒在野鳥群體中經歷了復雜的重配后再傳入我國家禽,其內部基因來源十分復雜,具有明顯的遺傳多樣性。
2.3 病毒HA基因演化分析
對7株病毒HA基因同源性進行分析,結果顯示核苷酸相似性在91.3%—99.2%之間。通過BEAST V1.10.4對H6亞型AIVs HA基因重建最大分支可信度(MCC)樹,以推斷HA基因的演化關系。結果顯示,H6亞型AIV分為歐亞譜系及北美譜系,其最近共同祖先可追溯至1916年(圖1)。歐亞譜系的病毒在20世紀80年代就已在北美的野鳥群體中監測到,直至2022年仍可在北美野鳥群體中監測到歐亞譜系的病毒。在歐亞譜系中,出現了多起洲際基因交換事件,結合進化樹中毒株的宿主信息,推斷病毒隨候鳥遷徙跨洲際傳播。與 DK/JX/941/19 HA基因親緣關系較近的基因來自韓國以及阿拉斯加的野鳥源病毒,與分離于北美地區和韓國的病毒聚在一簇;其余分離株HA基因與東亞野鳥源病毒基因具有較高的同源性。以上結果表明,H6N1亞型AIVs HA基因與來自野鳥的H6亞型AIVs高度同源,推斷DK/JX/941/19 HA基因節段是由候鳥遷徙傳入我國。
2.4 病毒NA基因遺傳演化分析
NA基因核苷酸相似性在95.9%—99.6%之間,以相似性高于95%為依據進行分組,7株病毒的NA基因均位于同一組內,與東亞地區野鳥源 H1N1、H5N1、H6N1 AIVs的N1基因親緣關系較近(圖2)。
2.5 病毒內部基因遺傳演化分析
分離株內部基因之間的核苷酸相似性分別為PB2:93.7%—99.7%,PB1:92.7%—98.9%,PA:93%— 96.5%,NP:93.5%—99.6%,M:96%—99.4%,NS:70.6%—99.8%。以相似性高于95%為依據分組,M 基因分為1組,其余內部基因分組情況為PB2:2組,PB1:2組,PA:3組,NP:3組,NS:2組。NS基因可分為2個等位基因群,其余內部基因均位于歐亞分支且均與野鳥源毒株基因親緣關系較近(圖3)。
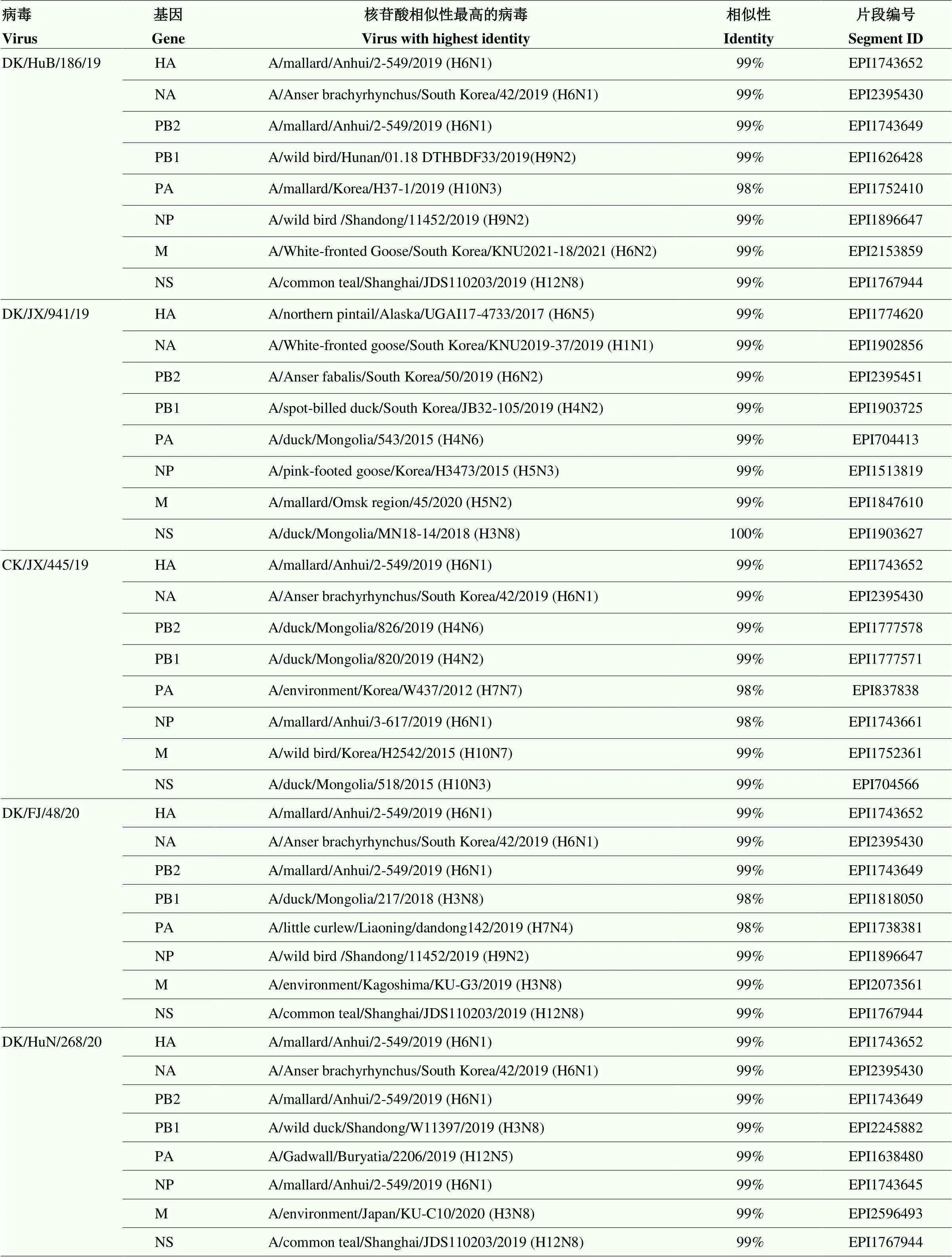
表2 7株H6N1亞型AIVs各基因節段同源性的分析
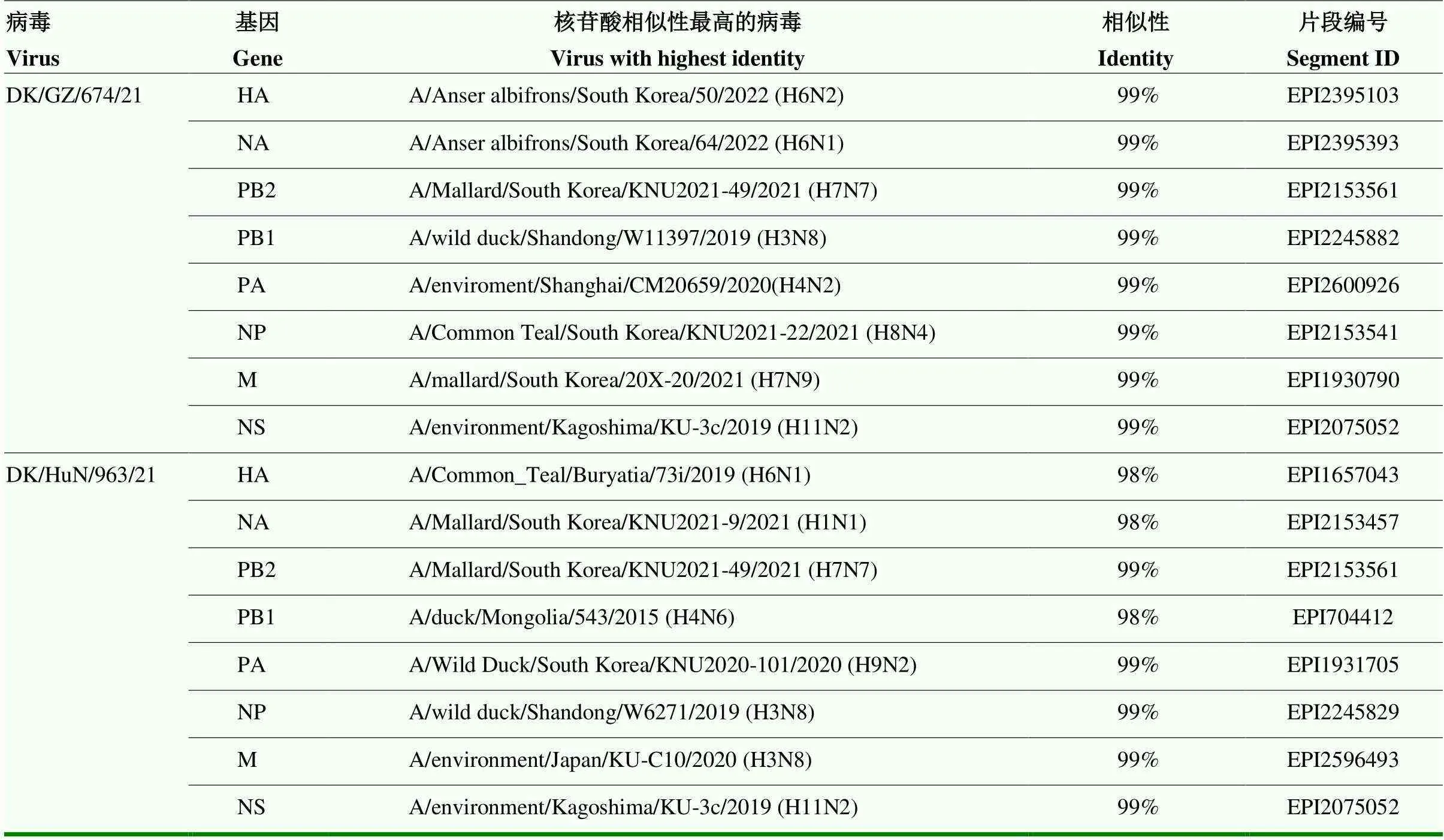
續表2 Continued table 2
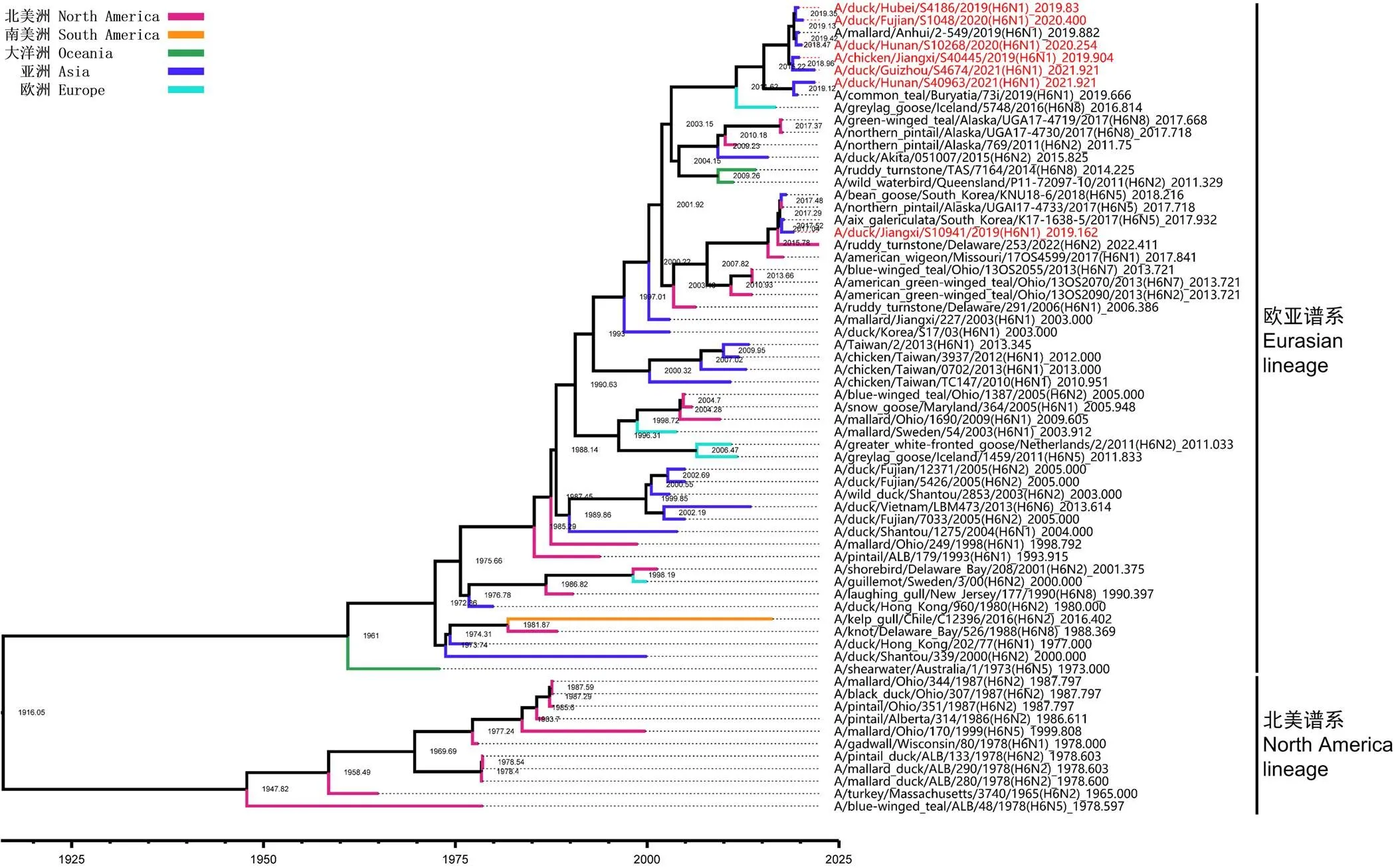
圖1 HA基因貝葉斯分析最大分支可信度(MCC)樹

圖2 NA基因遺傳演化分析
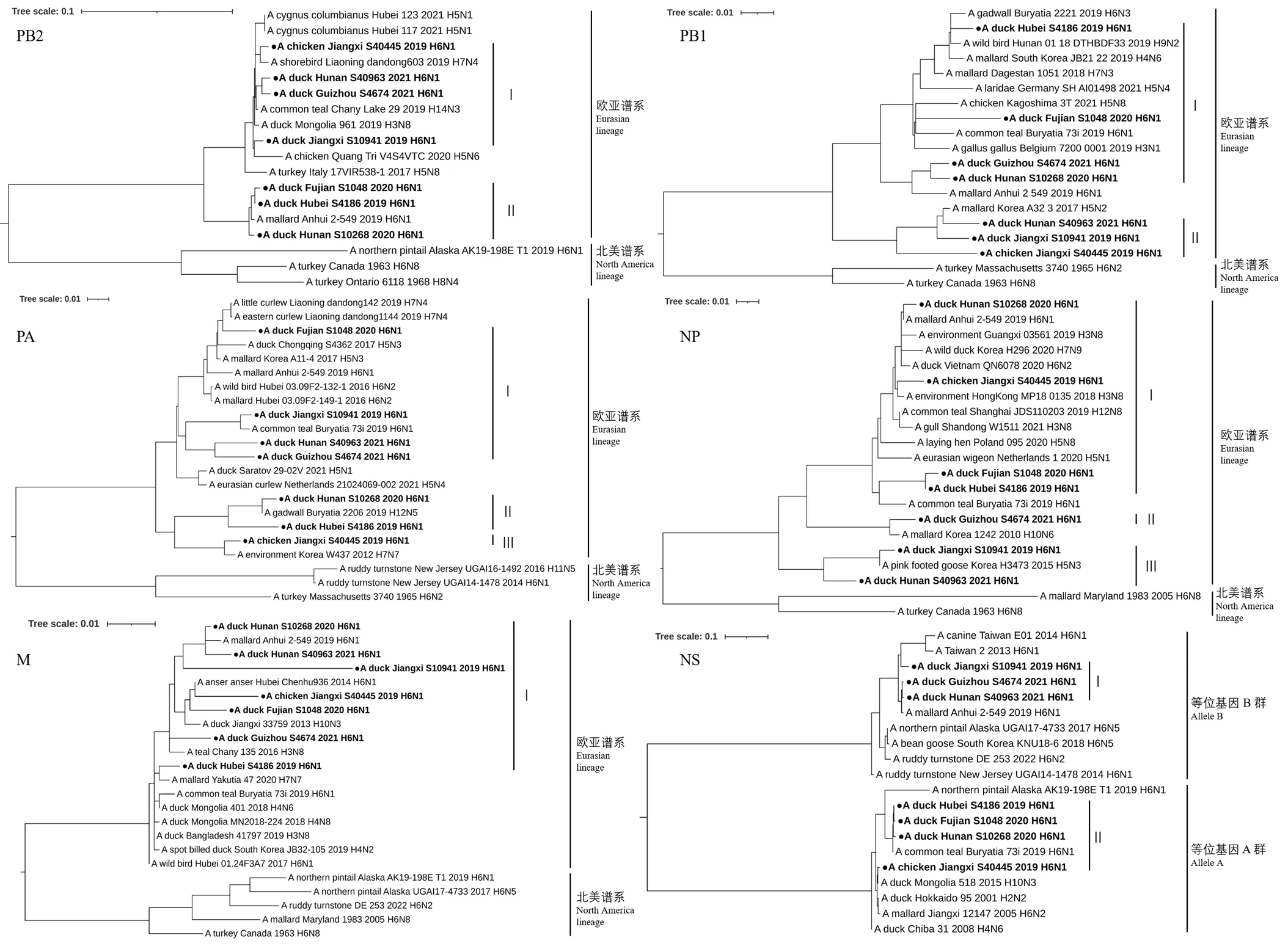
圖3 PB2、PB1、PA、NP、M、NS 基因遺傳演化分析
2.6 DK/JX/941/19的演化關系推斷
由于DK/JX/941/19 HA基因與分離于北美地區的病毒聚在一簇,而其余基因片段均與東亞野鳥源、鴨源病毒有著較高的同源性,表明東亞與北美的病毒基因庫發生基因漂移,因此,有必要對此病毒進行演化分析,以分析其完成重配的時間。
通過BEAST V1.10.4對DK/JX/941/19的8個基因片段進行定年分析,結果顯示8個內部基因片段的最近共同祖先最早可追溯至2016年5月,最晚為2019年1月,推斷病毒在2019年初完成重配(表3)。
DK/JX/941/19 HA基因與來自阿拉斯加、韓國的野鳥源病毒 HA 基因高度同源,而其余的基因也多與韓國的野鳥源病毒基因相似。基于阿拉斯加、韓國與我國均位于東亞-澳大利亞候鳥遷徙路線,推測起始于阿拉斯加地區的候鳥經該路線遷徙至韓國,其攜帶的北美地區HA基因與韓國野鳥群體中的東亞分支基因重配形成新型病毒,最后通過候鳥遷徙傳入我國鴨場。

表3 DK/JX/941/19演化關系推斷
2.7 病毒基因組特殊氨基酸位點分析
7株H6N1亞型AIVs HA裂解位點基序均為PQIETR↓GLF,符合LPAIVs特征。
分析影響病毒受體結合特性的相關位點,顯示有5株病毒獲得了T160A的突變,據報道,獲得該突變的H5N1病毒增強了對人源受體的結合[18]。
內部基因分子特征分析顯示病毒PB2蛋白的627、701位點未發生突變,但DK/GZ/674/21在NP蛋白上發生Y52H的突變,據報道,該突變對AIV獲得抵抗人干擾素刺激基因BTN3A3的能力起到關鍵作用[19]。5株病毒發生NP M105V的突變,有研究表明,獲得該突變的H5N1病毒增強了對雞的致病性[20]。
2.8 病毒對小鼠的感染性
將7株H6N1亞型AIVs分別以106EID50的劑量經鼻腔感染小鼠。在感染后第3天安樂死小鼠,取其腦、鼻甲、脾、腎、肺進行病毒滴定。結果顯示,所有小鼠均未死亡或出現明顯臨床癥狀,部分小鼠體重有輕微下降(圖4)。病毒在小鼠肺臟及鼻甲均能有效復制,其中在肺臟復制的平均滴度范圍為5.4— 6.4 log10EID50/mL,在鼻甲復制的平均滴度范圍為0.98—3.58 log10EID50/mL。以上結果表明,7株H6N1亞型AIVs能在小鼠呼吸器官中有效復制,但對小鼠呈低致病力。
2.9 病毒對雞的感染性
為探究來自不同宿主的H6N1亞型AIV對雞的感染能力,選取雞源病毒(CK/JX/445/19)及鴨源病毒(DK/JX/941/19),以106EID50/100 μL的劑量經鼻腔感染SPF雞。結果顯示,感染組的雞感染 CK/JX/445/19后,10只雞的血清均轉陽,僅有4只雞排毒(圖5),持續到感染后第7天;此外,3只接觸組的雞中有2只可通過泄殖腔向外排毒,其血清也發生轉陽,表明該病毒可在雞群中通過接觸傳播。雞感染 DK/JX/ 941/19后,感染組的雞的血清均轉陽,但僅在2只感染組的雞的咽喉中檢測到病毒,排毒持續到第3天;未在接觸組的雞中檢測到病毒,其血清也并未轉陽,表明該病毒無法在雞群中通過接觸傳播。在感染后第3天對復制組的雞的臟器進行病毒滴定,均未在臟器中檢測到病毒。雞感染CK/JX/445/19后,以泄殖腔排毒為主;雞感染 DK/JX/941/19后,排毒能力較弱,僅能通過咽喉排毒。
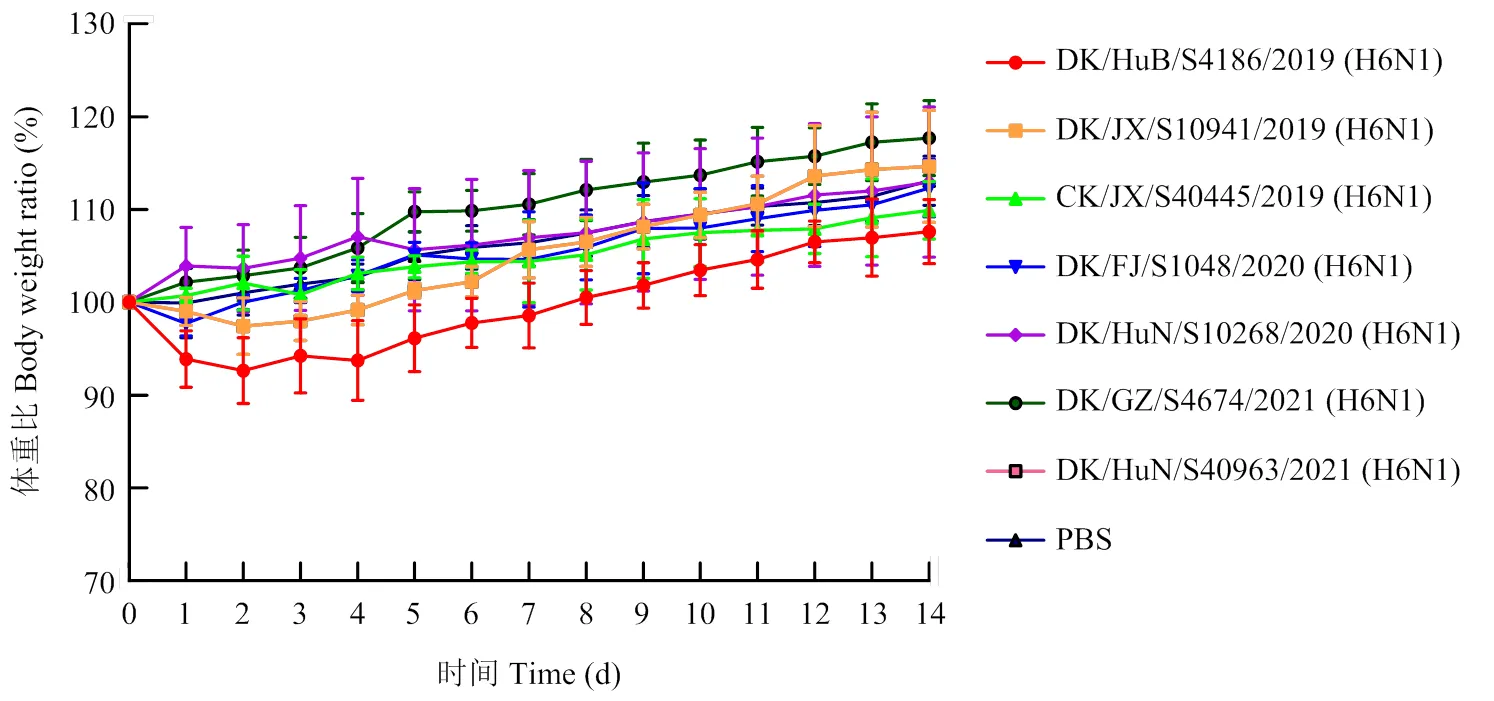
圖4 小鼠體重變化
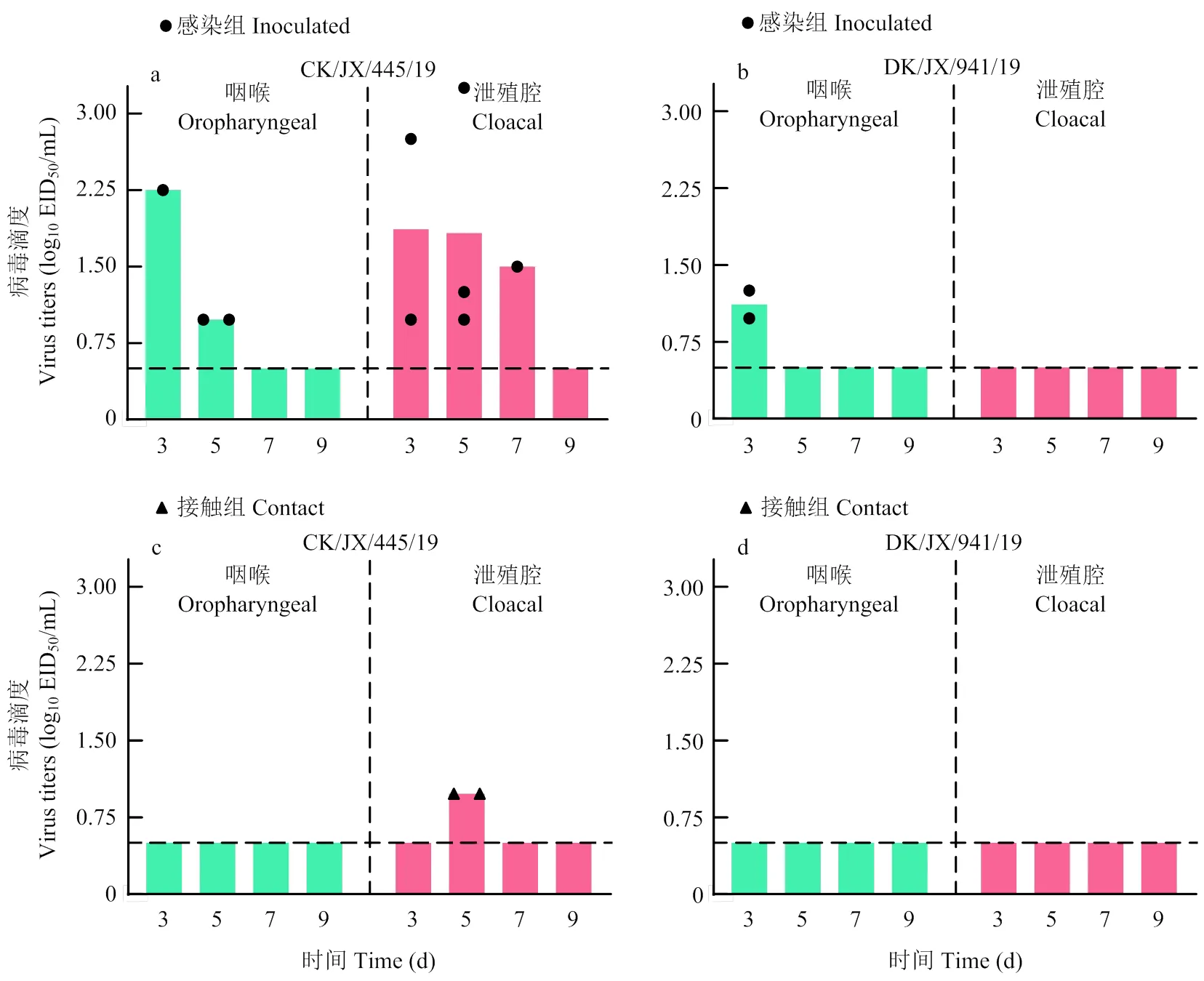
a:雞接種 CK/JX/445/19 排毒情況;b:SPF 雞接種 DK/JX/941/19 排毒情況;c:接觸組雞(CK/JX/445/19)排毒情況;d:接觸組雞(DK/JX/941/19)排毒情況
2.10 病毒的受體結合特性分析
對7株H6N1亞型AIVs的受體結合特性進行測定,結果顯示,所有毒株均對禽源唾液酸(sialic acid, SA)α2,3-Gal受體有著較強的結合能力,此外,DK/GZ/ 674/21與DK/HuB/186/19對人源唾液酸SAα2,6-Gal受體還有著較低的結合水平(圖6)。
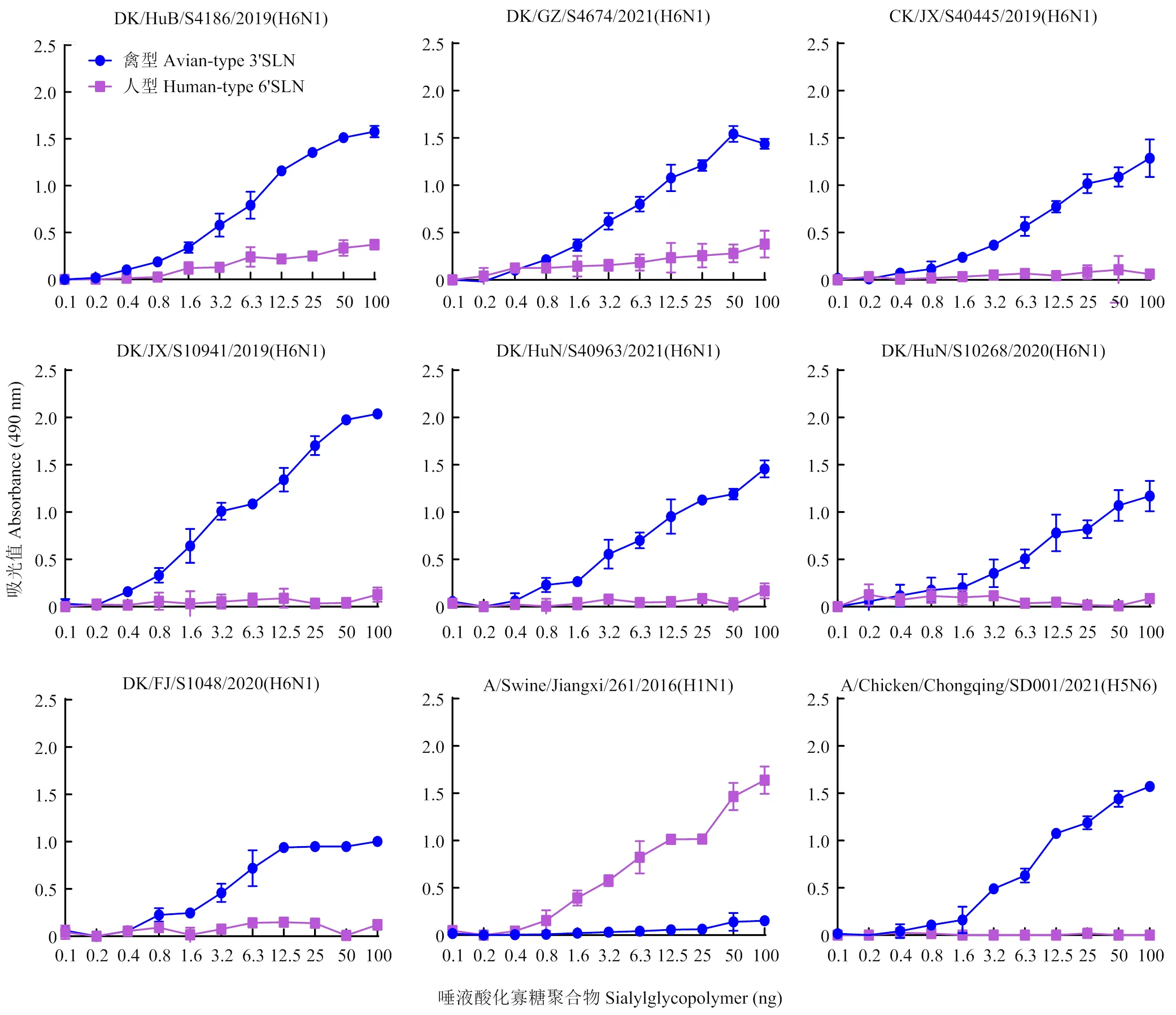
圖6 H6N1亞型AIVs受體結合特性
3 討論
3.1 H6N1亞型AIVs的跨洲際傳播與候鳥遷徙密切相關
本文中的H6N1亞型AIV基因與野鳥源AIV基因高度同源,表明野鳥是H6N1亞型AIV天然的基因儲存庫。相關研究表明,H6亞型AIV與其他亞型AIV相比,在野鳥體內的適應性更好,是在野鳥中檢出率最高的 AIV之一[21-23]。鑒于H6亞型AIV在野鳥中具有較好的適應性及高檢出率,推測該亞型病毒應更容易發生跨洲際傳播。BAHL等[24]研究發現,歐亞譜系的H6亞型AIV可隨著候鳥的遷徙,跨洲際傳播到北美,其通過對易感宿主的競爭導致部分北美譜系病毒被替代。本研究通過Beast構建最大分支可信度(MCC)樹,證實了H6基因發生過多次跨洲際的傳播,歐亞譜系中的部分病毒在北美地區有著較長時間的流行。在本研究中,還發現DK/JX/941/19 HA基因與北美阿拉斯加地區、韓國 H6N5 亞型 AIV HA 基因聚在一簇,基于地理位置及進化關系,推斷該病毒可能在2019年初完成重配,并通過候鳥經東亞-澳大利亞遷徙路線傳入我國。值得注意的是,本文中有5株H6N1亞型AIVs分離自我國養鴨場,其地理位置均處于東亞-澳大利亞遷徙路線所在區域。我國開放的水禽養殖環境使得候鳥有機會進入養殖場并為候鳥將病毒傳播給水禽提供了有利條件。
大量的H6亞型AIV分離自水禽,鴨源或鵝源H6亞型AIV一般需要適應后才可在陸生家禽體內有效復制。本研究中的鴨源病毒DK/JX/941/19感染雞后無法在雞體內有效復制,而雞源病毒CK/JX/445/19感染雞后則表現出較好的適應性。CK/JX/445/19的基因與分離自韓國和蒙古的野鴨源病毒基因高度同源,這表明來源于野鴨的H6N1亞型AIV傳入我國后,部分病毒經過適應后能在雞體內有效復制并在雞群間傳播。
綜上所述,候鳥遷徙是H6N1亞型AIV跨洲際傳播的重要因素。應加強水禽養殖場的飼養管理,避免水禽與野鳥和陸生家禽的接觸,阻斷病毒的傳播鏈。
3.2 H6N1亞型AIVs對公共衛生安全構成潛在威脅
AIV不僅對家禽養殖業影響巨大,部分亞型的病毒還對人類健康構成了嚴重威脅,如H3N8、H5N1、H5N6、H7N9、H9N2、H10N3亞型AIVs[25-31]。甲型流感病毒的宿主特異性決定了其一般難以跨物種感染人,有多種因素阻止了流感病毒的外溢,如病毒對宿主細胞的受體結合特異性[32]、病毒聚合酶的活性[33]以及相關宿主因子的限制[34]。本研究中的DK/GZ/674/21在NP蛋白上發生了Y52H的突變,提示該病毒可能不受人體內干擾素刺激基因BTN3A3的限制[19];對病毒受體結合特性的測定顯示了該病毒已具備了低水平結合人源受體的能力。已有研究表明,H6亞型AIV HA蛋白上Q226L突變能使其結合人源受體能力強于結合禽源受體能力,并使其能在豚鼠間通過飛沫傳播[35];在感染人的H6N1亞型AIV發現其HA蛋白發生了E190V和G228S突變,使得病毒獲得了結合人源受體的能力。本文中所研究的病毒雖然并未獲得以上突變,但病毒已能不用預先適應便能在哺乳動物體內有效復制并有部分病毒具備結合人源唾液酸受體的能力,表明H6N1亞型AIV對公共衛生安全構成了潛在威脅。
4 結論
本研究系統性地分析了2019—2022年我國H6N1亞型禽流感病毒(AIV)生物學特性,結果表明,H6N1亞型AIV多分離自水禽,其與北美和東亞的野鳥源病毒親緣關系密切,來源于水禽的 H6N1 亞型 AIV 傳入我國雞群后,部分病毒經過適應后可在雞體內有效復制并能在雞群間傳播。H6N1病毒可在小鼠呼吸道內有效復制并且部分病毒獲得結合人源受體能力,具有跨種感染人的潛在風險,對人類健康構成了一定的威脅。因此,應持續開展對H6N1亞型AIV的監測,了解其進化動態及跨種感染人的潛在風險。
[1] LANG G, FERGUSON A E, CONNELL M C, WILLS C G. Isolation of an unidentified hemagglutinating virus from the respiratory tract of turkeys. Avian Diseases, 1965, 9(4): 495-504.
[2] OLESIUK O M, SNOEYENBOS G H, ROBERTS D H. An influenza A virus isolated from turkeys. Avian Diseases, 1967, 11(2): 203.
[3] DOWNIE J C, LAVER W G. Isolation of a type A influenza virus from an Australian pelagic bird. Virology, 1973, 51(2): 259-269.
[4] ZHAO G, LU X L, GU X B, ZHAO K K, SONG Q Q, PAN J J, XU Q G, DUAN Z Q, PENG D X, HU S L, WANG X Q, LIU X F. Molecular evolution of the H6 subtype influenza A viruses from poultry in Eastern China from 2002 to 2010. Virology Journal, 2011, 8: 470.
[5] BI Y H, CHEN Q J, WANG Q L, CHEN J J, JIN T, WONG G, QUAN C S, LIU J, WU J, YIN R F, et al. Genesis, evolution and prevalence of H5N6 avian influenza viruses in China. Cell Host & Microbe, 2016, 20(6): 810-821.
[6] HUANG K, BAHL J, FAN X H, VIJAYKRISHNA D, CHEUNG C L, WEBBY R J, WEBSTER R G, CHEN H, SMITH G J D, PEIRIS J S M, GUAN Y. Establishment of an H6N2 influenza virus lineage in domestic ducks in Southern China. Journal of Virology, 2010, 84(14): 6978-6986.
[7] HUANG K, ZHU H C, FAN X H, WANG J, CHEUNG C L, DUAN L, HONG W S, LIU Y M, LI L F, SMITH D K, CHEN H L, WEBSTER R G, WEBBY R J, PEIRIS M, GUAN Y. Establishment and lineage replacement of H6 influenza viruses in domestic ducks in Southern China. Journal of Virology, 2012, 86(11): 6075-6083.
[8] CUI J Q, CUI P F, SHI J Z, FAN W F, XING X, GU W L, ZHANG Y C, ZHANG Y P, ZENG X Y, JIANG Y P, et al. Continued evolution of H6 avian influenza viruses isolated from farms in China between 2014 and 2018. Transboundary and Emerging Diseases, 2022, 69(4): 2156-2172.
[9] ZHANG G H, KONG W L, QI W B, LONG L P, CAO Z X, HUANG L Z, QI H T, CAO N, WANG W H, ZHAO F R, et al. Identification of an H6N6 swine influenza virus in Southern China. Infection, Genetics and Evolution: Journal of Molecular Epidemiology and Evolutionary Genetics in Infectious Diseases, 2011, 11(5): 1174-1177.
[10] WEI S H, YANG J R, WU H S, CHANG M C, LIN J S, LIN C Y, LIU Y L, LO Y C, YANG C H, CHUANG J H, et al. Human infection with avian influenza A H6N1 virus: An epidemiological analysis. The Lancet Respiratory Medicine, 2013, 1(10): 771-778.
[11] YUAN J, ZHANG L, KAN X Z, JIANG L, YANG J K, GUO Z C, REN Q Q. Origin and molecular characteristics of a novel 2013 avian influenza a(H6N1) virus causing human infection in Taiwan. Clinical Infectious Diseases, 2013, 57(9): 1367-1368.
[12] CHEUNG C L, VIJAYKRISHNA D, SMITH G J D, FAN X H, ZHANG J X, BAHL J, DUAN L, HUANG K, TAI H, WANG J, et al. Establishment of influenza A virus (H6N1) in minor poultry species in Southern China. Journal of Virology, 2007, 81(19): 10402-10412.
[13] LEE M S, CHANG P C, SHIEN J H, CHENG M C, CHEN C L, SHIEH H K. Genetic and pathogenic characterization of H6N1 avian influenza viruses isolated in Taiwan between 1972 and 2005. Avian Diseases, 2006, 50(4): 561-571.
[14] HOFFMANN E, STECH J, GUAN Y, WEBSTER R G, PEREZ D R. Universal primer set for the full-length amplification of all influenza A viruses. Archives of Virology, 2001, 146(12): 2275-2289.
[15] ZHANG D, GAO F L, JAKOVLI? I, ZOU H, ZHANG J, LI W X, WANG G T. PhyloSuite: An integrated and scalable desktop platform for streamlined molecular sequence data management and evolutionary phylogenetics studies. Molecular Ecology Resources, 2020, 20(1): 348-355.
[16] SUCHARD M A, LEMEY P, BAELE G, AYRES D L, DRUMMOND A J, RAMBAUT A. Bayesian phylogenetic and phylodynamic data integration using BEAST 1.10. Virus Evolution, 2018, 4(1): vey016.
[17] 崔鵬飛, 盧昆鵬, 陳思, 肖麗, 關立崢, 鄧國華, 陳化蘭. 兩株H3N8亞型禽流感病毒(AIV)的生物學特性. 農業生物技術學報, 2016, 24(7): 980-986.
CUI P F, LU K P, CHEN S, XIAO L, GUAN L Z, DENG G H, CHEN H L. Biological Characteristic of Two H3N8 Subtype Avian influenza viruses(AIV). Journal of Agricultural Biotechnology, 2016, 24(7): 980-986. (in Chinese)
[18] GAO Y W, ZHANG Y, SHINYA K, DENG G H, JIANG Y P, LI Z J, GUAN Y T, TIAN G B, LI Y B, SHI J Z, et al. Identification of amino acids in HA and PB2 critical for the transmission of H5N1 avian influenza viruses in a mammalian host. PLoS Pathogens, 2009, 5(12): e1000709.
[19] PINTO R M, BAKSHI S, LYTRAS S, ZAKARIA M K, SWINGLER S, WORRELL J C, HERDER V, HARGRAVE K E, VARJAK M, CAMERON-RUIZ N, et al. BTN3A3 evasion promotes the zoonotic potential of influenza A viruses. Nature, 2023, 619: 338-347.
[20] TADA T, SUZUKI K, SAKURAI Y, KUBO M, OKADA H, ITOH T, TSUKAMOTO K. NP body domain and PB2 contribute to increased virulence of H5N1 highly pathogenic avian influenza viruses in chickens. Journal of Virology, 2011, 85(4): 1834-1846.
[21] MUNSTER V J, BAAS C, LEXMOND P, WALDENSTR?M J, WALLENSTEN A, FRANSSON T, RIMMELZWAAN G F, BEYER W E P, SCHUTTEN M, OLSEN B, et al. Spatial, temporal, and species variation in prevalence of influenza A viruses in wild migratory birds. PLoS Pathogens, 2007, 3(5): e61.
[22] YAO Z Z, ZHENG H B, XIONG J S, MA L P, GUI R, ZHU G L, LI Y, YANG G X, CHEN G, ZHANG J, CHEN Q J. Genetic and pathogenic characterization of avian influenza virus in migratory birds between 2015 and 2019 in central China. Microbiology Spectrum, 2022, 10(4): e0165222.
[23] KANG H M, JEONG O M, KIM M C, KWON J S, PAEK M R, CHOI J G, LEE E K, KIM Y J, KWON J H, LEE Y J. Surveillance of avian influenza virus in wild bird fecal samples from South Korea, 2003-2008. Journal of Wildlife Diseases, 2010, 46(3): 878-888.
[24] BAHL J, VIJAYKRISHNA D, HOLMES E C, SMITH G J D, GUAN Y. Gene flow and competitive exclusion of avian influenza A virus in natural reservoir hosts. Virology, 2009, 390(2): 289-297.
[25] GU W L, SHI J Z, CUI P F, YAN C, ZHANG Y P, WANG C C, ZHANG Y C, XING X, ZENG X Y, LIU L L, et al. Novel H5N6 reassortants bearing the clade 2.3.4.4b HA gene of H5N8 virus have been detected in poultry and caused multiple human infections in China. Emerging Microbes & Infections, 2022, 11(1): 1174-1185.
[26] YIN X, DENG G H, ZENG X Y, CUI P F, HOU Y J, LIU Y J, FANG J Z, PAN S X, WANG D X, CHEN X H, et al. Genetic and biological properties of H7N9 avian influenza viruses detected after application of the H7N9 poultry vaccine in China. PLoS Pathogens, 2021, 17(4): e1009561.
[27] ZHANG Y C, SHI J Z, CUI P F, ZHANG Y P, CHEN Y, HOU Y J, LIU L L, JIANG Y P, GUAN Y T, CHEN H L, et al. Genetic analysis and biological characterization of H10N3 influenza A viruses isolated in China from 2014 to 2021. Journal of Medical Virology, 2023, 95(2): e28476.
[28] YANG R G, SUN H L, GAO F, LUO K W, HUANG Z, TONG Q, SONG H, HAN Q Q, LIU J Y, LAN Y, et al. Human infection of avian influenza A H3N8 virus and the viral origins: a descriptive study. The Lancet Microbe, 2022, 3(11): e824-e834.
[29] ZENG X Y, HE X W, MENG F, MA Q, WANG Y, BAO H M, LIU Y J, DENG G H, SHI J Z, LI Y B, et al. Protective efficacy of an H5/H7 trivalent inactivated vaccine (H5-Re13, H5-Re14, and H7-Re4 strains) in chickens, ducks, and geese against newly detected H5N1, H5N6, H5N8, and H7N9 viruses. Journal of Integrative Agriculture, 2022, 21(7): 2086-2094.
[30] KANDEIL A, PATTON C, JONES J C, JEEVAN T, HARRINGTON W N, TRIFKOVIC S, SEILER J P, FABRIZIO T, WOODARD K, TURNER J C, et al. Rapid evolution of a(H5N1) influenza viruses after intercontinental spread to North America. Nature Communications, 2023, 14: 3082.
[31] JIN Y F, CUI H, JIANG L N, ZHANG C, LI J J, CHENG H L, CHEN Z H, ZHENG J, ZHANG Y D, FU Y Y, et al. Evidence for human infection with avian influenza a(H9N2) virus via environmental transmission inside live poultry market in Xiamen, China. Journal of Medical Virology, 2023, 95(1): e28242.
[32] ROGERS G N, PAULSON J C. Receptor determinants of human and animal influenza virus isolates: Differences in receptor specificity of the H3 hemagglutinin based on species of origin. Virology, 1983, 127(2): 361-373.
[33] LONG J S, GIOTIS E S, MONCORGé O, FRISE R, MISTRY B, JAMES J, MORISSON M, IQBAL M, VIGNAL A, SKINNER M A, BARCLAY W S. Species difference in ANP32A underlies influenza A virus polymerase host restriction. Nature, 2016, 529: 101-104.
[34] M?NZ B, DORNFELD D, G?TZ V, ZELL R, ZIMMERMANN P, HALLER O, KOCHS G, SCHWEMMLE M. Pandemic influenza A viruses escape from restriction by human MxA through adaptive mutations in the nucleoprotein. PLoS Pathogens, 2013, 9(3): e1003279.
[35] QU Z Y, MA S J, KONG H H, DENG G H, SHI J Z, LIU L L, SUZUKI Y, CHEN H L. Identification of a key amino acid in hemagglutinin that increases human-type receptor binding and transmission of an H6N2 avian influenza virus. Microbes and Infection, 2017, 19(12): 655-660.
Biological characteristics of H6N1 subtype avian influenza virus from 2019 to 2022 in China
Chen Yuan,Cui PengFei, SHI Jianzhong, Zhang YuanCheng, Yu QingQing, Yan Cheng, Zhang YaPing, Wang CongCong,Zhang Jie, Wang Yan, Deng GuoHua, Chen HuaLan
Harbin Veterinary Research Institute, Chinese Academy of Agricultural Sciences/State Key Lab for Animal Disease Control and Prevention/National Poultry Laboratory Animal Resource Center, Harbin 150069
【Background】H6 avian influenza virus (AIV) is widely prevalent in southern China, which is one of the most common subtypes of AIVs circulating in poultry in China. H6N1 AIVs frequently undergo gene reassortment events with other wild bird-origin virus, which can be a donor to provide internal gene segments to highly pathogenic AIVs, which may lead to the emergence of novel virus and threaten human health.【Objective】 The aim of this study was to investigate the evolution and biological characteristics of H6N1 AIVs in China, so as to provide valuable data for the prevention and control of avian influenza in China.【Method】From 2019 to 2022, cloacal and throat swabs were collected from live poultry markets and breeding farms across 25 provinces or autonomous regions in China. 7 H6N1 viruses were isolated by inoculating into chicken embryos. The complete genome sequences were determined, followed by analyzing their phylogenetic relationships, receptor binding properties, as well as replication in SPF chickens and BALB/c mice.【Result】The phylogenetic analysis revealed that the genes of the 7 H6N1 AIVs were highly homologous to those of wild bird-origin viruses from North America and Southeast Asia, indicating a complex genetic origin with significant genetic diversity. According to the Bayesian evolution analysis, the HA gene of H6 subtype AIVs had many times of intercontinental transmission in the history, and the Eurasian lineage strains also had a long time of circulation in North America.The HA gene of one strain of the virus was highly homologous to that of the North American strain. Based on the results of Bayesian evolutionary analysis, it was hypothesized that the virus was introduced to China via wild birds after undergoing complex genetic reassortment in wild birds. Analysis of specific amino acid sites revealed that the cleavage site of HA protein was PQIETR↓GLF, which was the signature of low pathogenic AIVs. In addition, one other virus had a Y52H mutation in the NP protein, which was critical in BTN3A3 evasion. Receptor binding analysis demonstrated that some of the H6N1 AIVs bound to both avian-type receptor and human-type receptor, however, their affinity towards human receptors was weaker compared with avian receptors. Infectivity experiments on SPF chickens indicated that flocks was still able to shed virus up through oropharyngeal and cloacal routes after infection with A/chicken/Jiangxi/S40445/2019(H6N1) and that the virus could be transmitted via contact within flocks. Only a few chickens infected with A/duck/Jiangxi/S10941/2019(H6N1) shed virus through their oropharyngeal tract, and the virus could not be transmitted via contact among chickens. The infectivity experiments conducted on mice showed that H6N1 subtype AIVs could replicate in the respiratory organs of infected mice without prior adaptation, but display low pathogenicity levels in mice.【Conclusion】Most of the genes of H6N1 subtype AIV isolated in China from 2019 to 2022 were derived from wild bird-origin viruses, and those migratory birds could introduce the viruses into China via the East Asia-Australasian migratory flyway. Some of the H6 AIVs bound to human-type receptors and replicate in the respiratory organs of mice suggested that H6N1 viruses posed a potential threat to human health.
H6N1; avian influenza virus; intercontinental transmission; reassortment; infectivity

10.3864/j.issn.0578-1752.2024.09.015
2023-10-27;
2023-12-24
國家重點研發計劃(2021YFD1800201)
陳源,E-mail:charlesyg1@163.com。通信作者鄧國華,Tel:0451-51051806;E-mail:dengguohua01@caas.cn
(責任編輯 林鑒非)

