基于H&E圖像和基因表達數據的多模態深度學習模型預測胃癌生存風險


關鍵詞:胃癌;H&E染色圖像;基因表達;深度學習;多模態
中圖分類號:TP391.41 文獻標志碼:A
0 引言(Introduction)
癌癥是導致患者死亡的主要原因,其中胃癌是常見的消化系統惡性腫瘤之一,其發病率和死亡率近年來居高不下,嚴重威脅人類健康[1]。準確預測胃癌患者的死亡風險對于臨床決策、治療計劃和患者心理調整至關重要。病理學家一般通過蘇木精和伊紅(Hematoxylin-Eosinstaining,H&E)染色的圖像獲得腫瘤形態和組織學信息。數字成像技術的進步使得全幻燈片掃描儀在病理學圖像數字化中得到廣泛應用。深度卷積神經網絡(CNN)已成為一種重要的圖像分析工具,數字成像技術和CNN結合應用,極大地提高了病理學領域的專家、學者及研究人員的工作效率,為腫瘤診斷和治療提供了更準確的依據[2-4]。
轉錄組學分析關注mRNA作為DNA和蛋白質之間的中間分子,其表達水平和表達模式與腫瘤發展、細胞功能及患者預后密切相關[5]。綜合考慮組織病理學和基因組學的互補信息可以提高生存風險預測的潛力。然而,以往的研究多專注于各自領域的個體模式,缺乏對信息的整合,限制了對癌癥生物學的全面理解[6]。
因此,本研究通過整合H&E染色圖像的特征和基因組數據,構建了一個多模態生存預測模型,以提高癌癥患者生存預測的準確性。具體而言,首先,利用ResNet18從H&E染色區域提取深層特征;其次,采用多模態緊湊雙線性池化(MCB)方法融合病理學和基因組學的特征;最后,將融合后的多模態特征輸入Cox回歸層,得到癌癥患者的風險分數,為個體化的治療和關懷提供有力支持。
1 相關理論(Related theory)
1.1 H&E染色圖像
H&E染色圖像是常見的組織病理學圖像,通過蘇木精和伊紅染色,使得細胞核和細胞質分別呈現藍色和粉紅色,從而提供了關于組織結構和細胞形態的重要信息。制備過程包括組織固定、脫水、清理、染色、包埋、切片、脫蠟、染色和封片等步驟。H&E染色圖像的高分辨率導致每張圖像的像素值達到10億左右,對圖像處理帶來了巨大的挑戰。像素數量龐大可能會引起計算的問題,因此通常需要將圖像切割成小塊以提高處理效率。此外,圖像在不同設備和條件下可能存在顏色偏差,會影響可比性和分析的準確性。因此,采用顏色歸一化處理操作是必要的,它可以確保圖像的一致性,并提升建模和分析的可靠性。
1.2 生存風險預測問題
生存風險預測問題通常被視為一個重要的臨床挑戰,其中的關鍵在于預測患者在特定時間點(例如3年)的生存狀態,并將其二分化為生存或死亡[7]。然而,使用傳統的分類方法建立模型存在一些局限性,尤其在隨訪數據不完整的情況下更為明顯。在這種情況下,時間-事件模型,如Cox回歸[8]和隨機生存森林[9]展現出了更為靈活的能力。
與傳統分類方法不同,時間-事件模型可以利用所有受試者的信息進行訓練,并在各種時間點預測患者的生存概率。這一方法不僅更全面地考量了患者的生存狀況,還能有效應對隨訪數據缺失的問題,建模過程更穩健和可靠。醫生和研究人員借助時間-事件模型,可以更精準地評估患者的生存風險,并制定個性化的治療方案和臨床管理策略。
2 數據與方法(Data and method)
2.1 數據預處理
2.1.1 數據集下載及預處理
本文使用的H&E染色圖像和轉錄組數據均來自https:∥gdc.cancer.gov/的癌癥基因組圖譜(TCGA)。通過患者識別號(TCGA ID)進行數據整合和匹配,最終得到320例胃癌(STAD)患者的H&E圖像數據和基因表達數據。
2.1.2 H&E圖像的預處理
為了從H&E圖像中提取特征,本研究采取了一系列預處理操作,H&E圖像預處理步驟如圖1所示。首先,由一位經驗豐富的病理學家對腫瘤區域進行了視覺注釋,確保分析區域的正確性。其次,針對像素極大的H&E染色圖像,為了得到適配模型的大小,對H&E圖像進行切割,形成512×512的小圖像塊,針對切割后的圖像,計算出腫瘤區域與總區域的比率,并舍棄了比率小于0.3的存在噪聲的小圖塊(如空白或模糊的區域)。最后,使用文獻[10]中提到的顏色歸一化算法對帶有有用信息的小切片進行顏色歸一化,消除不同H&E圖像的染色差異[10]。
2.1.3 基因表達數據的預處理
為了減少基因表達數據的冗余信息,首先,刪除在少于50個樣本中表達的基因,此操作將胃癌的基因表達數據減少至19 531維。其次,利用單因素Cox回歸對與生存相關的特征進行篩選,并設定P 值小于0.05為閾值。在此過程中,共篩選出1 385個與生存相關的基因。
2.2 基于H&E染色圖像的深度學習模型建立
為了解決深度神經網絡訓練過程中的一系列問題,HE等[11]提出了殘差網絡(ResNet),這一結構由一系列殘差塊組成。每個殘差塊經過多次卷積操作后,將其輸出與輸入相加。通過引入殘差連接,ResNet允許某一層的輸出直接跳過一個或多個層,連接到后續層的輸入。這種結構使得網絡能夠更有效地學習復雜的特征,同時緩解了深度網絡訓練過程中的退化、梯度消失和梯度爆炸等問題。
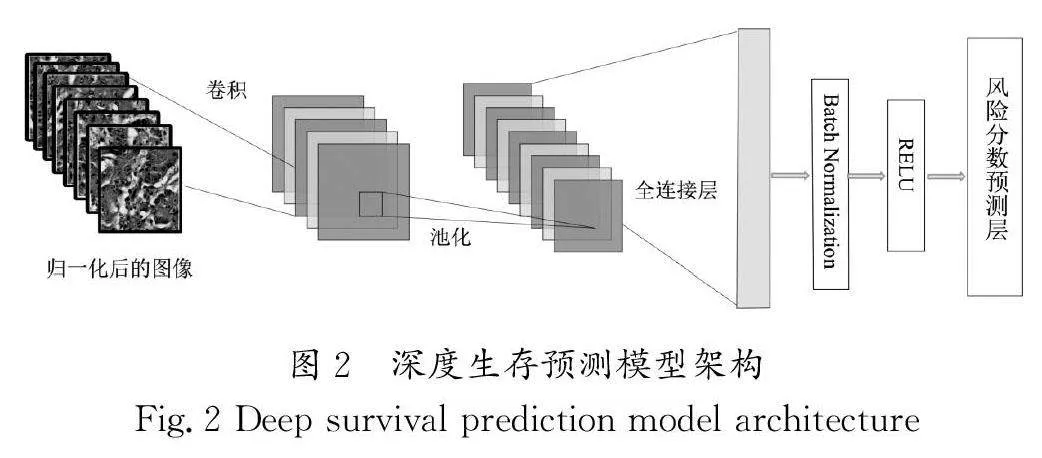
ResNet18作為一種經典的深度卷積神經網絡,其構架由17個卷積層和1個全連接層組成。該網絡的結構可以劃分為4個主要模塊,從第一層開始,每個模塊包含兩個基本塊,基本塊由一對卷積層組成,每個卷積層后面跟隨著ReLU激活函數和批量歸一化層(Batch Normalization),以增強模型的非線性建模能力和穩定性。在整個ResNet18中,這些基本塊的堆疊形成一個強大的特征提取和學習結構,使得網絡能夠有效地捕獲和表示輸入數據的復雜特征。此外,每個基本塊中的卷積層都設計有殘差連接,以促進梯度的順暢傳播和模型的訓練收斂。最終的全連接層將卷積層提取的高層次特征映射為一維向量。深度生存預測模型架構如圖2所示。
2.3 基于多模態深度學習的生存模型的建立
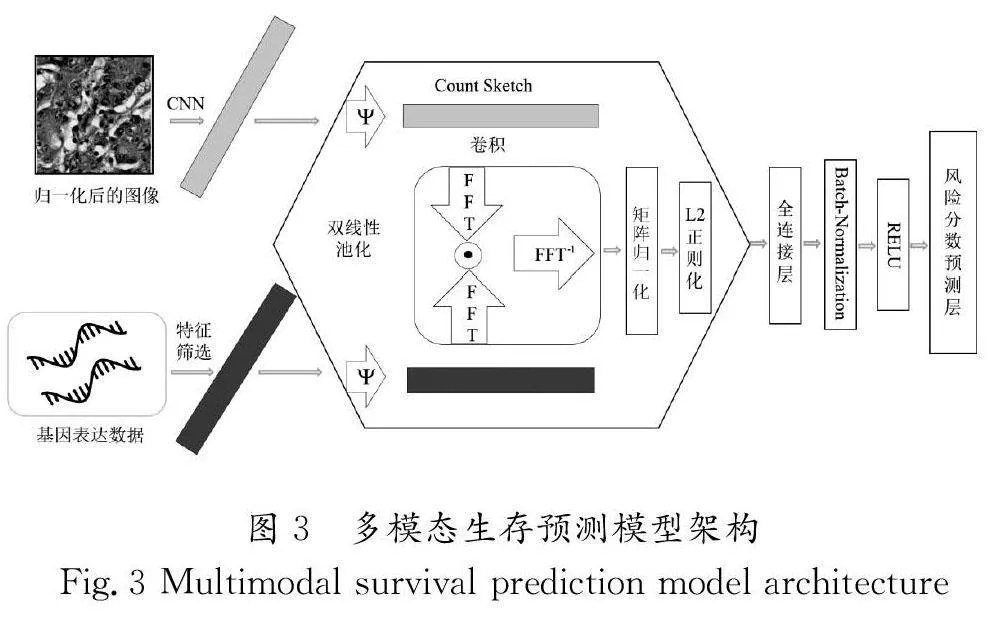
本研究采用基于多模態緊湊型雙線性池化(MCB)的多模態融合算法[12],旨在將H&E圖像的特征與基因表達特征融合為一個新的特征向量。相對于傳統的向量融合方法(如拼接、逐位相乘、逐位相加等),MCB采用了一種端到端學習的反向傳播方法,在處理雙線性特征時,MCB通過多項式核的內積近似降低特征維度,確保模型性能損失最小。
雙線性池化是通過計算特征向量的外積實現融合,充分利用了向量元素之間的交互作用。該過程包括對每個位置的特征向量進行向量外積計算,將所有位置的計算結果進行求和池化,最終得到特征向量。為了應對高維特征的挑戰,采用了近似多項式核的算法(Tensor Sketching)進行降維,即緊湊型雙線性池化(CBP)。
在多模態情境下,MCB對CBP中的近似多項式核的算法進行改進。具體工作流程包括提取各模態對應的特征,通過預訓練的CNN提取圖像高層特征,采用多層感知機(MLP)對其他模態進行特征提取;利用Count Sketch方法對兩個模態的特征進行逼近,得到降維后的特征;在頻域內進行向量內積計算,通過FFT-1 逆變換至時域空間,最后對得到的時域特征進行矩陣歸一化和L2正則化。
多模態生存預測模型架構如圖3所示,首先將歸一化后的病理圖像塊輸入ResNet18網絡,在全連接層得到512維的特征向量。同時,采用MLP處理經預處理后的基因表達數據,得到相應的特征向量。其次利用MCB方法將H&E圖像特征和基因表達特征的特征進行融合,并將融合后的特征輸入生存風險預測層,進行端到端訓練。這一整合方法旨在綜合利用不同模態的信息,提高生存風險預測模型的預測準確性。
3 實驗結果與分析(Experimental results andanalysis)
3.1 數據集劃分與模型訓練
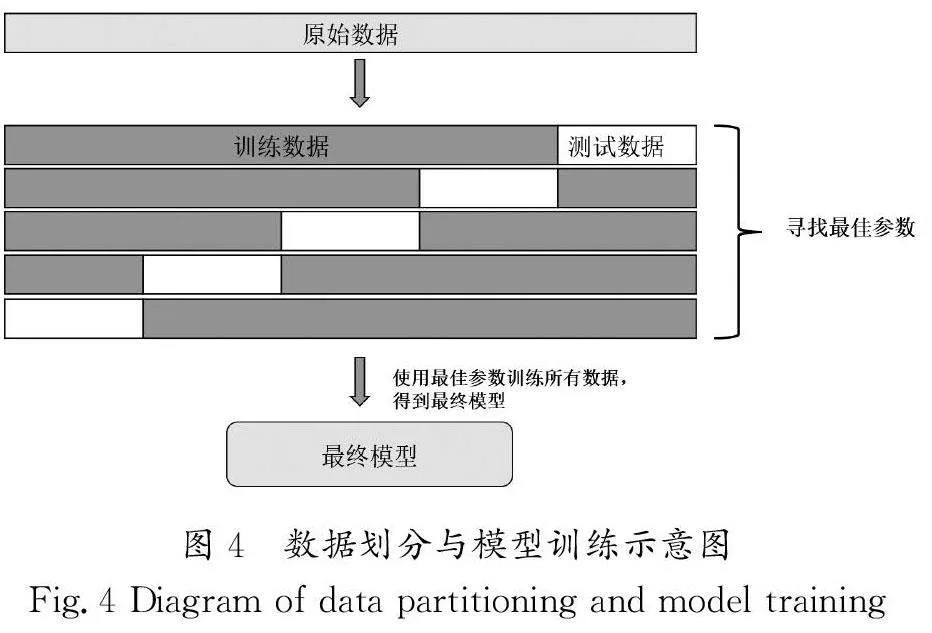
本研究按照五折交叉驗證的方式將樣本劃分為訓練集和測試集,確保每折在訓練集和測試集中存活樣本和死亡樣本的比例相同,其數據劃分與模型訓練示意圖如圖4所示。
在訓練數據方面,采用數據擴充和標準化策略。具體而言,通過隨機水平翻轉和面片的隨機仿射變換(保持中心不變)進行數據增強。在RGB通道上進行z-score歸一化處理后,將增強的小圖像塊進行中心裁剪(像素為224×224),確保訓練數據的高質量和一致性。對于測試數據,僅進行標準化處理,在確保數據高質量和一致性的同時避免引發不必要的變化。
在模型選擇方面,使用隨機梯度下降(SGD)算法更新模型的參數,將學習率設置為2×10-3。所有模型至少訓練20個epochs,最多可達300個epochs。在訓練過程中監控模型的性能,若在第50個epochs之后,訓練損失連續10個epochs未減少,則提前停止。最低損失的最佳模型將用于測試模型性能。
在風險分數的預測階段,針對每位患者,取其所有小圖像塊預測出的風險分數的平均值作為該患者的風險分數。
3.2 評價指標
本研究采用c-index,即一致性指數評價模型的預測能力。c-index的計算方式如下。
(1)把所有研究對象隨機地兩兩組成對子。
(2)對于每一對患者,若生存時間較長的樣本,其預測的生存時間也較長,預測生存概率較高的樣本,其實際生存概率也較高,則表示預測結果與實際結果一致。
(3)計算c-index=K/M,其中:K是一致的對子數,M 是有用的對子數。
c-index的值為0.5~1,0.5表示預測結果與實際結果完全不一致,說明該模型沒有預測作用;1表示預測結果與實際結果完全一致,說明該模型具有預測作用。
3.3 模型的性能比較
3.3.1 不同模態的性能比較
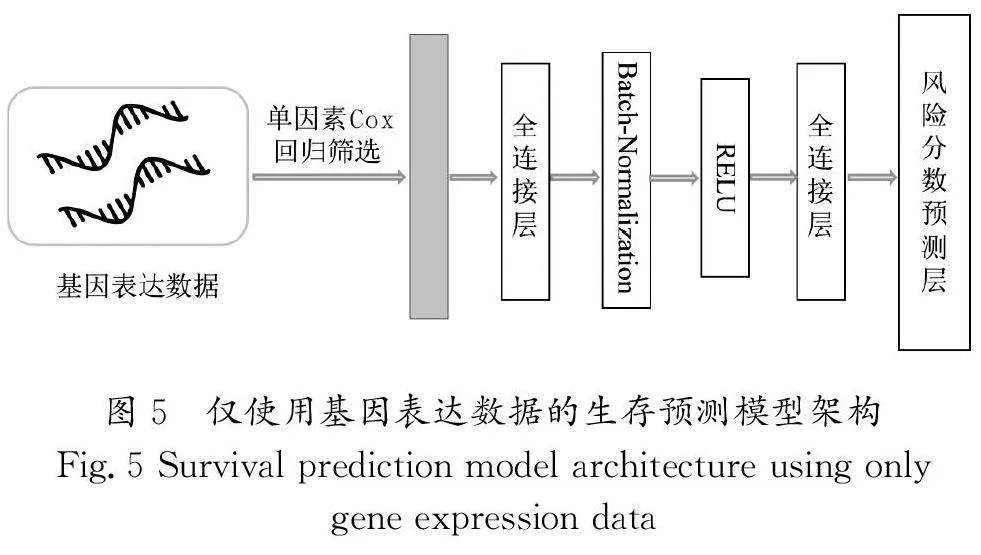
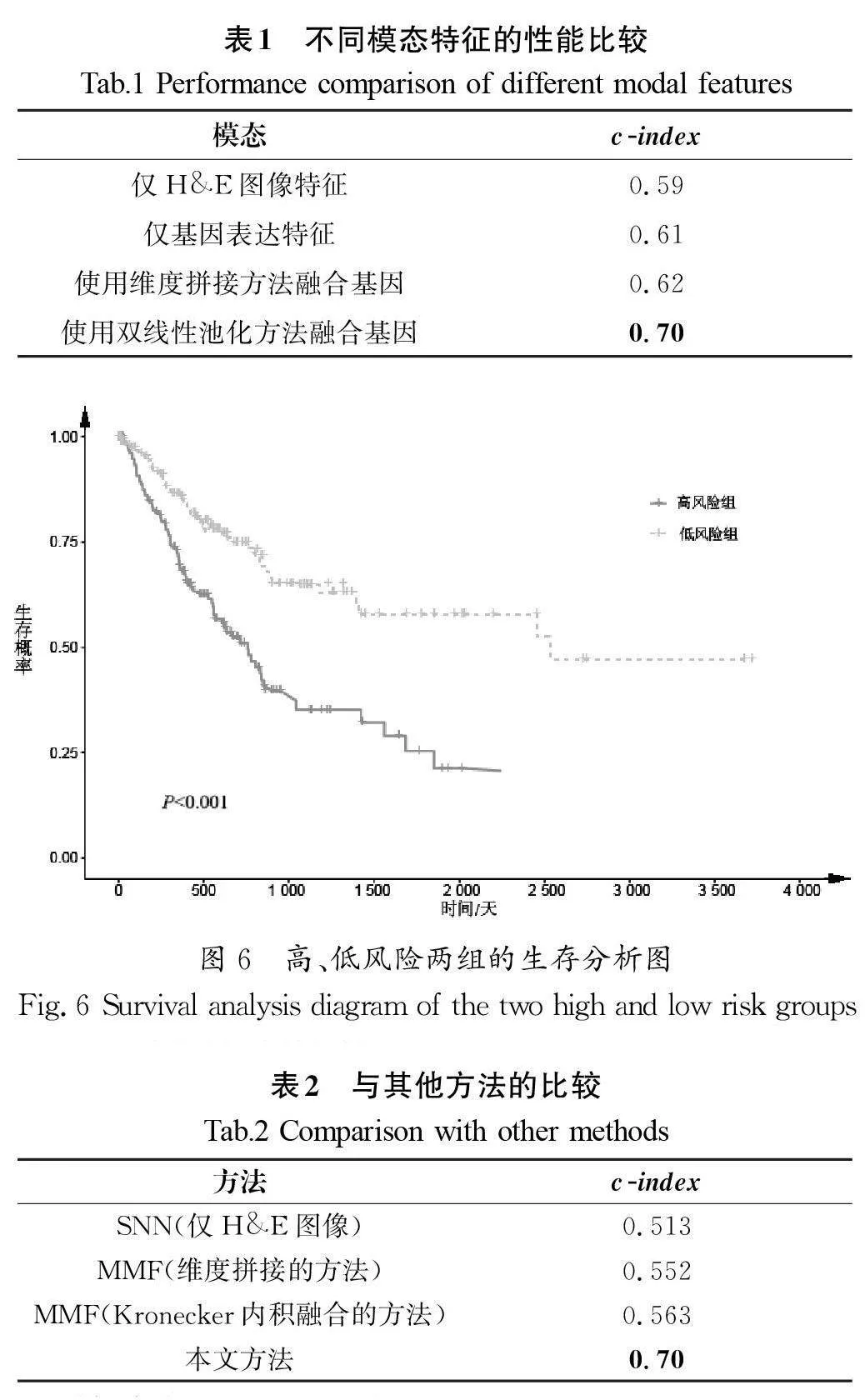
為了評估多模態方法的有效性,本研究使用MCB方法的模型與僅使用H&E圖像數據的模型以及僅使用基因表達數據的模型進行了比較。針對僅使用基因表達數據的建模流程,使用單因素Cox回歸篩選出的1385維基因表達特征作為輸入,保持全連接層結構不變,用于預測胃癌患者的風險分數,僅使用基因表達數據的生存預測模型架構如圖5所示。研究人員對各模態特征的預測能力進行了對比,結果如表1所示。
通過對比表1中的結果發現,使用MCB融合方法的模型性能明顯優于僅使用單一模態特征和使用維度拼接方法的模型性能,表明使用雙線性池化方法融合基因的方法在胃癌生存風險預測中具有顯著的優勢。
在測試集上,將模型預測出的風險分數以中位數為閾值,將樣本分為兩組,即高風險組和低風險組,并對這兩組樣本進行生存分析。為了檢查兩組樣本之間的差異,使用了log-rank 進行檢驗,生存分析的結果如圖6所示,兩組樣本之間的P 值遠小于0.05。以上結果表明,該模型能夠有效地將胃癌患者分成兩組,并且這兩組之間存在顯著差異。
3.3.2 與不同方法的比較
為了評估本研究提出的模型的性能,將其與文獻[4]中提到的所有方法進行比較,具體結果如表2所示。表2中結果表明,本研究提出的方法明顯優于其他方法。
4 結論(Conclusion)
本研究采用了基于MCB的多模態融合算法,成功地將H&E圖像的特征與基因表達特征融合為一個新的特征向量,用于預測胃癌患者的生存風險。與僅使用H&E染色圖像或采用維度拼接的方法相比,MCB算法顯著提高了模型性能。這一多模態融合方法為胃癌患者生存風險預測提供了更全面的信息。傳統的單一模態方法可能無法充分捕捉到患者病理特征與基因表達之間的復雜關系,采用MCB算法能夠將不同模態的信息有機地結合起來,從而獲得更準確、更全面的特征向量,進而提高了預測模型的性能。
此外,通過對高、低風險兩組患者的生存情況進行分析發現,該多模態算法模型能夠有效地將胃癌患者分層。這一發現不僅對個體患者的治療和管理提供了重要的指導,也為臨床實踐提供了有力支持。
作者簡介:
馬艷雨(1998-),女,碩士生。研究領域:人工智能,生物信息學。
賀平安(1969-),男,博士,教授。研究領域:計算生物學,生物信息學。

