牡荊屬葉綠體基因組比較分析及系統發育分析






摘 要: 牡荊屬(Vitex L.)廣布于熱帶和亞熱帶地區,多為喬木或灌木,包含眾多具有藥用價值、觀賞價值和生態價值的植物。為了解牡荊屬的葉綠體基因組特征、系統位置及種間關系,該研究從頭組裝、注釋獲得穗花牡荊(V. agnus-castus)的葉綠體基因組,與牡荊屬公布的11條葉綠體基因組序列進行基因組結構、密碼子偏好性、高變區和重復序列分析,并進行系統發育分析。結果表明:(1)穗花牡荊葉綠體基因組呈典型的四段式結構,全長154 444 bp,其中大單拷貝(LSC)區、小單拷貝(SSC)區、反向重復(IR)區長度分別是85 229、17 915、51 400 bp,編碼132個基因,包含87個蛋白編碼基因、37個tRNA基因和8個rRNA基因。(2)牡荊屬12個葉綠體基因組在長度、反向重復區邊界位置、編碼的基因及數目、GC含量方面均具有高保守性。(3)共檢測到31個高頻密碼子和6個共有最優密碼子,ENC-plot、PR2-plot和中性繪圖分析表明密碼子偏好性主要受自然選擇的影響。(4)共檢測到14個葉綠體基因組高變區和519個簡單重復序列。(5)分子系統發育結果支持牡荊屬隸屬于唇形科牡荊亞科,而非馬鞭草科。另外,系統發育推斷也為存在爭議的蔓荊(V. trifolia)、V. bicolor和單葉蔓荊(V. rotundifolia)之間關系的理解提供一定參考,建議將單葉蔓荊作為獨立的種。該研究結果不僅豐富了穗花牡荊的遺傳資源信息,增加了對牡荊屬葉綠體基因組的理解,提供了牡荊屬群體遺傳學研究的候選分子標記,還證明了葉綠體基因組序列在牡荊屬系統發育重建中的有效性。
關鍵詞: 牡荊屬, 葉綠體基因組, 密碼子偏好性, 系統發育, 唇形科, 馬鞭草科
中圖分類號: Q943; Q949
文獻標識碼: A
文章編號: 1000-3142(2024)09-1755-17
Comparative analysis of chloroplast genomes
and phylogenetic analysis of Vitex
Abstract: Vitex L. is widely distributed in tropical and subtropical areas, mostly as woody trees or shrubs, and contains many plants with medicinal, ornamental and ecological values. To understand the characteristics of Vitex chloroplast genomes, the phylogenetic position and interspecific relationships of the genus, the chloroplast genome sequence of V. agnus-castus was firstly obtained by de novo assembly and annotation, the genome structure, codon preference, high variation region and repeat sequence were then analyzed with 11 published chloroplast genome sequences of Vitex, and then phylogenetic analysis was conducted. The results were as follows: (1) The chloroplast genome of V. agnus-castus was a typically quadripartite structure with a total length of 154 444 bp, in which the length of the large single copy (LSC) region, the small single copy (SSC) region and the inverted repeat (IR) region were 85 229, 17 915 and 51 400 bp, respectively. The chloroplast genome encoded 132 genes, including 87 protein-coding genes, 37 tRNA genes and 8 rRNA genes. (2) The 12 chloroplast genomes of Vitex were highly conserved in terms of genome length, boundary position of IR region, number of encoded genes and GC content. (3) A total of 31 high frequency codons and six common optimal codons were detected in 12 chloroplast genomes of Vitex. Further, the results of ENC-plot, PR2-plot and neutral plot analysis indicated that codon preference was mainly influenced by natural selection. (4) In addition, a total of 14 highly variable regions and 519 simple sequence repeats (SSRs) were detected in the 12 chloroplast genomes of Vitex. (5) The molecular phylogenetic inference in this study supported that Vitex belonged to the subfamily Viticoideae (Lamiaceae), not Verbenaceae. Moreover, phylogenetic inference also provided some understanding of the controversial relationships among V. trifolia, V. bicolor and V. rotundifolia, suggesting that V. rotundifolia was considered as a separate species. This study not only enriches the information of genetic resources of V. agnus-castus, increases the understanding of the chloroplast genomes of Vitex, provides candidate molecular markers for population genetics studies of Vitex, but also proves the effectiveness of chloroplast genome sequence in the phylogenetic reconstruction of Vitex.
Key words: Vitex, chloroplast genome, codon preference, phylogeny, Lamiaceae, Verbenaceae
牡荊屬(Vitex L.)隸屬于唇形科(Lamiaceae)牡荊亞科(Viticoideae),包含的植物約250種,主要分布在熱帶區域,少數分布在北半球溫帶地區(Bramley et al., 2009;Li et al., 2016)。在許多國家,該屬的穗花牡荊(V. agnus-castus)、黃荊(V. negundo)、蔓荊(V. trifolia)等入藥歷史悠久,廣泛用于治療婦產科疾病、炎癥性疾病、風濕性疼痛、癌癥等(Vishwanathan & Basavaraju, 2010; Jangwan et al., 2013;Niroumand et al., 2018;Kamal et al., 2022)。除藥用價值外,該屬植物還具有觀賞價值、經濟價值和生態價值,如穗花牡荊花色美麗且花期較長,觀賞效果佳,是良好的園林植物(Karaguzel & Girmen, 2009),蔓荊、荊條(V. negundo var. heterophylla)和單葉蔓荊(V. rotundifolia)是水土保持、防風固沙的良好樹種(辛海量,2005;Sun et al., 2019;孫小涵,2020),穗花牡荊和荊條是良好的蜜源植物(孫小涵,2020;易仁知,2022)。
唇形科一直被認為是由馬鞭草科類的祖先進化而來,長久以來,唇形科和馬鞭草科(Verbenaceae)的界定發生了顯著變化,一些學者將馬鞭草科中的蕕亞科(Caryopteridoide331d1881ffc19b980bc5ab02d8d37f1aae)、Chloanthoideae亞科、牡荊亞科和Monochileae族,約50個屬劃分到唇形科(Cantino, 1992; Li et al., 2016)。近年的分子系統發育研究顯示唇形科包含12個亞科,230余屬,7 000種植物,是被子植物的第六大科(Li et al., 2016;Zhao et al., 2021);馬鞭草科則僅保留馬鞭草亞科的大多數物種,包含8族,約35屬,1 000種(Marx et al., 2010)。
牡荊屬是上述系統位置發生了變化的植物類群之一。早期一些學者可能因牡荊屬植物的木本或灌木、肉質果實等特征而將其劃分在馬鞭草科(Munir,1987),《中國植物志》同樣采用該分類系統(中國科學院中國植物志編輯委員會,1982);近來許多分子研究表明與狹義馬鞭草科相比,牡荊屬與傳統唇形科有更近的親緣關系,并提議將其從馬鞭草科劃分到唇形科(Schferhoff et al., 2010; Refulio-Rodriguez & Olmstead, 2014; Li et al., 2016)。另外,牡荊屬的界定存在一定爭議,如Li等(2016)認為應將Harley等(2004)分類系統中的屬Viticipremna、Tsoongia、Paravitex和Petitia歸并到牡荊屬,Bramley等(2009)建議將Harley等(2004)分類系統中的屬Viticipremna、Tsoongia和Paravitex歸并到牡荊屬。
已有的研究雖加深了對牡荊亞科及牡荊屬系統關系的理解,但受到傳統分子標記提供的信息位點有限的影響,屬間及屬內的一些系統關系仍存在爭議。高通量測序技術可為探究系統關系提供更多的分子數據,這使得葉綠體基因組數據廣泛用于探究不同分類單元的系統發育關系并獲得高分辨率(Cauz-Santos et al., 2020;Yang et al., 2021;Zhao et al., 2021)。因此,本研究從頭組裝注釋獲得穗花牡荊的完整葉綠體基因組,與牡荊屬所有公布的11條完整準確的葉綠體基因組進行密碼子偏好性分析和比較分析,如反向重復(inverted repeat, IR)區收縮擴張分析、mVISTA分析、重排分析,并且選擇35條代表唇形科所有12個亞科和馬鞭草科3個族的葉綠體基因組進行系統發育分析,擬探究以下問題:(1)牡荊屬葉綠體基因組的結構特征;(2)牡荊屬葉綠體基因組的密碼子偏好性;(3)牡荊屬的系統位置及其種間關系推斷。以期增加對牡荊屬葉綠體基因組的理解,為重建牡荊屬全面系統發育關系提供一定思路。
1 材料與方法
1.1 植物材料
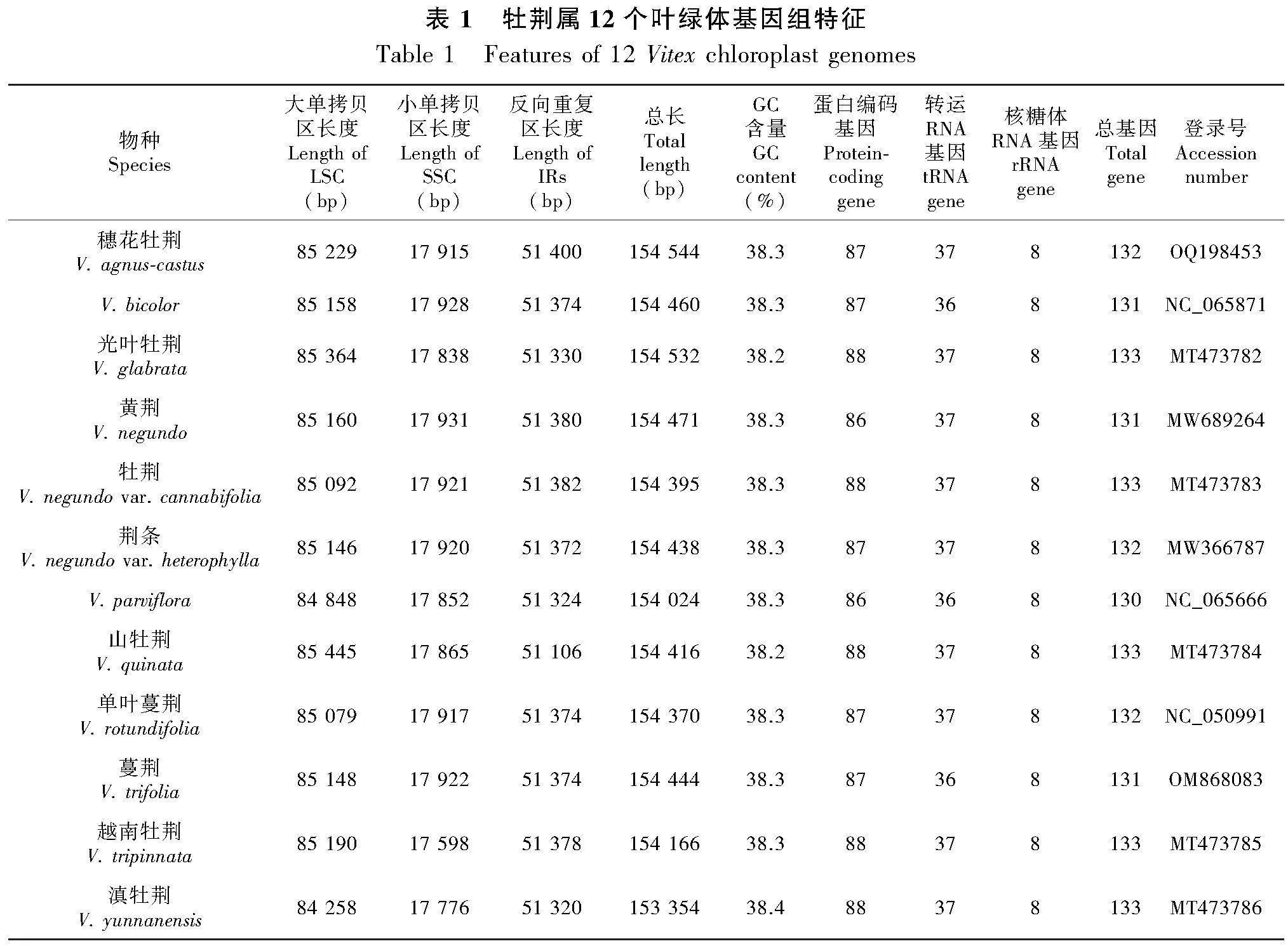
穗花牡荊樣品采集自西安植物園(陜西,西安),采集的植物葉片硅膠干燥保存。目前,NCBI(https://www.ncbi.nlm.nih.gov)共公布了牡荊屬9種2變種的16條葉綠體基因組序列,我們選取并下載其中11條完整序列以代表這9種2變種,這12種/變種分別是穗花牡荊、V. bicolor、光葉牡荊(V. glabrata)、黃荊、牡荊(V. negundo var. cannabifolia)、荊條、V. parviflora、山牡荊(V. quinata)、單葉蔓荊、蔓荊、越南牡荊(V. tripinnata)和滇牡荊(V. yunnanensis)。
1.2 DNA提取、測序和組裝注釋
采用改良的CTAB法提取穗花牡荊葉片的總DNA(Doyle, 1987)。使用Illumina Hiseq平臺完成高通量測序。總DNA提取和高通量測序工作均委托百邁克生物科技公司完成。使用MIRA v.4.0.2(Chevreux et al., 2004)和MITObim v.1.7(Hahn et al., 2013) 組裝葉綠體基因組序列;在組裝過程中,近緣種越南牡荊的葉綠體基因組作為參考基因組。葉綠體基因組注釋則使用GENEIOUS v.12(Biomatters Ltd.,Auckland,New Zealand)。使用在線程序OGDRAW(http://ogdraw.mpimp-golm.mpg.de/index.shtml)繪制穗花牡荊的葉綠體基因組圖譜。
1.3 牡荊屬葉綠體基因組比較分析
1.3.1 IR區收縮擴張分析、mVISTA分析和重排分析 為檢測可能的IR區收縮擴張事件,我們分析了12個葉綠體基因組的IR/SC邊界位置。牡荊屬12個葉綠體基因組的可視化比對結果由mVISTA程序(Frazer et al., 2004)獲取,該分析以黃荊葉綠體基因組為參考。此外,使用Progressive Mauve v.2.4.0(Darling et al., 2004)檢測12個葉綠體基因組中可能存在的重排事件,并使用擬南芥(Arabidopsis thaliana, GenBank accession number: NC_000932)和煙草(Nicotiana tabacum, GenBank accession number: Z00044)的葉綠體基因組作為參考。
1.3.2 序列分化和高變區分析 為評估牡荊屬12個葉綠體基因組序列的分化水平,首先使用MAFFT v.7.520(Katoh & Standley, 2013)進行葉綠體基因組序列比對,之后使用DnaSP v.5.0(Librado & Rozas, 2009)分別計算完整葉綠體基因組、蛋白編碼區和非編碼區的變異位點數目以及核苷酸多態性(Pi)。為尋找牡荊屬葉綠體基因組的高變區,使用DnaSP v.5.0進行滑動窗口分析(sliding window analysis)以計算Pi,其中窗口長度和步幅長度分別設置為600 bp和200 bp。
1.3.3 密碼子偏好分析
1.3.3.1 同義密碼子分析 相對同義密碼子使用度(relative synonymous codon usage, RSCU)是密碼子使用偏好的良好指標,RSCU值大于1是指該密碼子使用較高,等于1是指該密碼子沒有使用偏好性,小于1是指該密碼子使用較低(Sharp & Li, 1986)。使用MEGA v.5.0(Tamura et al., 2011)計算牡荊屬12個葉綠體基因組的RSCU值,之后使用HemI(Deng et al.,2014)制作密碼子偏好熱圖。
1.3.3.2 中性繪圖分析 對牡荊屬12個葉綠體基因組進行中性繪圖分析。中性圖是以GC3值作為橫坐標,GC12作為縱坐標進行散點圖的繪制,插入y=x的函數圖像。
1.3.3.3 ENC-Plot分析 以GC3值為橫坐標,ENC值為縱坐標繪制散點圖,并根據下方公式繪制標準曲線,標準曲線表示基因突變是密碼子偏好性的決定因素,進一步分析突變壓力對密碼子偏好性的影響。
計算公式:ENC=2+GC3+29/ [GC32+(1-GC3)2]。
1.3.3.4 PR2-Plot分析 以G3/(G3+C3)為橫坐標,A3/(A3+T3)為縱坐標繪制散點圖,通過中心點(A=T,C=G)發出的矢量,分析各核苷酸偏移的程度和方向。
1.3.3.5 最優密碼子分析 選取RSCU>1的密碼子為高頻密碼子,將提取的CDS基因的ENC值按照從低到高排列,從兩端各自選取基因總數的10%(5條)建立高低表達庫并計算RSCU值,2個庫的△RSCU≥0.08(△RSCU=RSCU高表達-RSCU低表達)的密碼子作為高表達密碼子。將既為高頻又為高表達的密碼子定義為最優密碼子。
1.3.4 重復序列分析 分別檢測分布于12個葉綠體基因組序列中的散在重復(dispersed repeat)、串聯重復(tandem repeat)和簡單重復序列(simple sequence repeats, SSRs)。首先,使用REPuter(Kurtz et al., 2001)識別各種散在重復,包括正向(forward)、反向(reverse)、互補(complement)和回文(palindromic)重復;散在重復的重復單元長度需不少于30 bp且高于90%的相似性。其次,使用在線程序Tandem Repeats Finder(TRF)(Benson, 1999)(默認參數)檢測重復單元長度大于10 bp的串聯重復。所有檢測到的散在重復和串聯重復都需手工校對,并去除冗余結果。最后,使用Msatcommander(Faircloth, 2008)檢測SSRs,單核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重復的次數分別設置為不低于10、5、4、3、3和3。
1.4 牡荊屬系統關系分析
為探究牡荊屬的系統位置,根據近來的分子研究結果,系統發育分析包含了目前所有公布了可靠葉綠體基因組數據的牡荊屬和馬鞭草科物種,除牡荊亞科之外,在唇形科的11個亞科各隨機選擇1個物種,鑒于《中國植物志》中劃分在馬鞭草科的蕕亞科、六苞藤亞科(Symphorematoideae)和牡荊亞科在近來的分子研究中均屬于唇形科,我們對《中國植物志》中描述的這3個亞科14屬進行葉綠體基因組數據查詢,發現其中11個屬公布了葉綠體基因組數據。最終,共有35條序列用于構建系統發育樹,包含牡荊屬10種、2變種,共12條葉綠體基因組序列,唇形科除牡荊亞科之外所有亞科的17個物種,即短柄野芝麻(Lamium album)、歧傘花(Cymaria dichotoma)、藏黃芩(Scutellaria kingiana)、冬紅(Holmskioldia sanguinea)、膜萼藤(Hymenopyramis cana)、辣蕕(Garrettia siamensis)、赪桐(Clerodendrum japonicum)、九味一枝蒿(Ajuga bracteosa)、蒙古蕕(Caryopteris mongholica)、豆腐柴(Premna microphylla)、苦梓(Gmelina hainanensis)、柚木(Tectona grandis)、絨苞藤(Congea tomentosa)、毛楔翅藤(Sphenodesme mollis)、藏荊芥(Nepeta hemsleyana)、枇杷葉紫珠(Callicarpa kochiana)和Dasymalla teckiana的葉綠體基因組序列以及馬鞭草科3個族的6個物種,即馬櫻丹(Lantana camara)、Lippia origanoides、Aloysia citrodora、細葉美女櫻(Glandularia tenera)、馬鞭草(Verbena officinalis)和假連翹(Duranta erecta)的葉綠體基因組序列。此外,選擇列當科(Orobanchaceae)的歐亞馬先蒿(Pedicularis oederi)和返顧馬先蒿(P. resupinata)為外類群。使用上述37條完整葉綠體基因組的比對序列分別構建最大似然(maximum likelihood, ML)樹和貝葉斯(Bayesian inference, BI)樹,ML樹由RAxML v.7.2.8(Stamatakis,2006)構建,BI樹由MrBayes v.3.1.2(Ronquist & Huelsenbeck,2003)構建。
2 結果與分析
2.1 葉綠體基因組特征
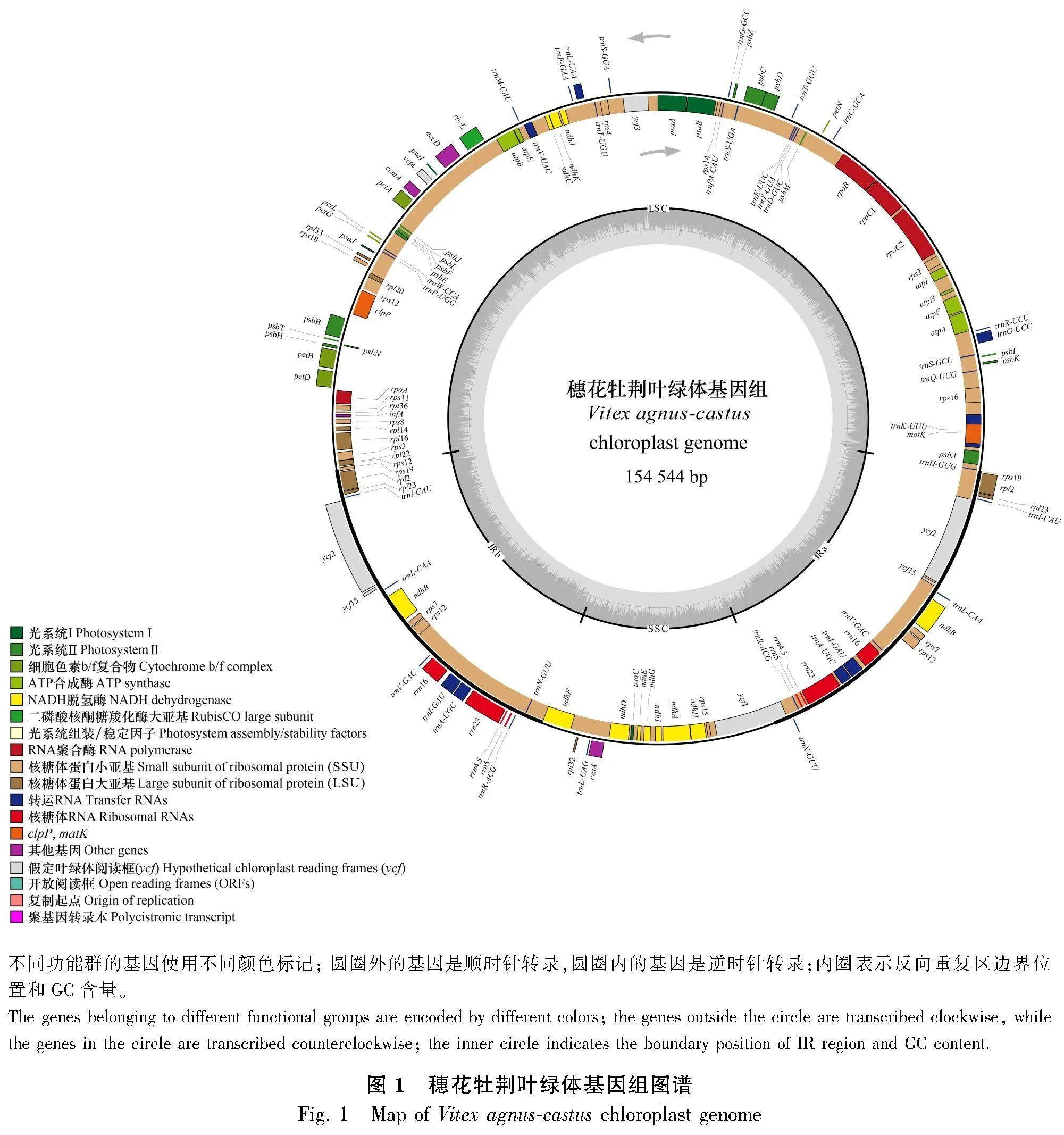
本研究獲得的穗花牡荊葉綠體基因組呈典型的四段式結構,全長154 544 bp,其中大單拷貝[JP2](large single copy, LSC)區、小單拷貝(small single copy, SSC)區、2個IR區長度分別是85 229、17 915、51 400 bp。共編碼132個基因,包含87個蛋白編碼基因、37個tRNA基因和8個rRNA基因(圖1)。
牡荊屬12個葉綠體基因組均呈四段式結構。由表1可知,這些葉綠體基因組總長度在153 354~154 444 bp之間;LSC區長度在84 258~85 445 bp之間;SSC區長度在17 598~17 931 bp之間;IR區長度在51 106 bp~51 400 bp之間。總基因數在130~133個之間;GC含量在38.2%~38.4%之間。
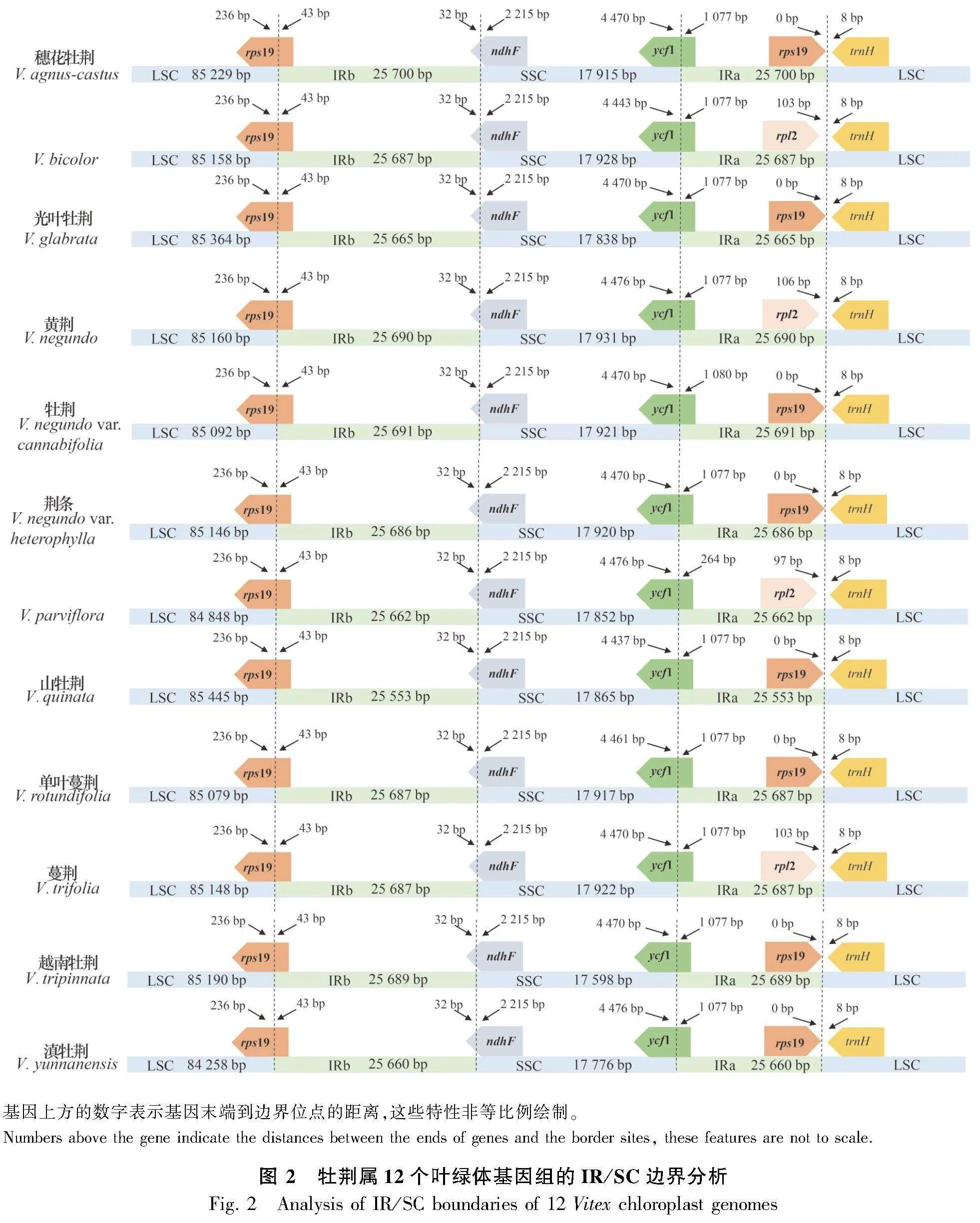
2.2 葉綠體基因組IR區收縮擴張和重排分析
IR區收縮擴張分析結果顯示,牡荊屬12個葉綠體基因組的LSC/IRa、IRa/SSC和邊界位置較為穩定,而SSC/IRb和IRb/LSC邊界位置則呈現出一定變化,如ycf1 3′端到SSC/IRb邊界位置在V. parviflora中為264 bp,在牡荊中為1 080 bp,
在剩余物種中為1 077 bp(圖2)。牡荊屬植物12條葉綠體基因組序列顯示出保守的線性基因順序,并未檢測到重排事件(附圖1)。
2.3 葉綠體基因組的mVISTA分析和序列分化
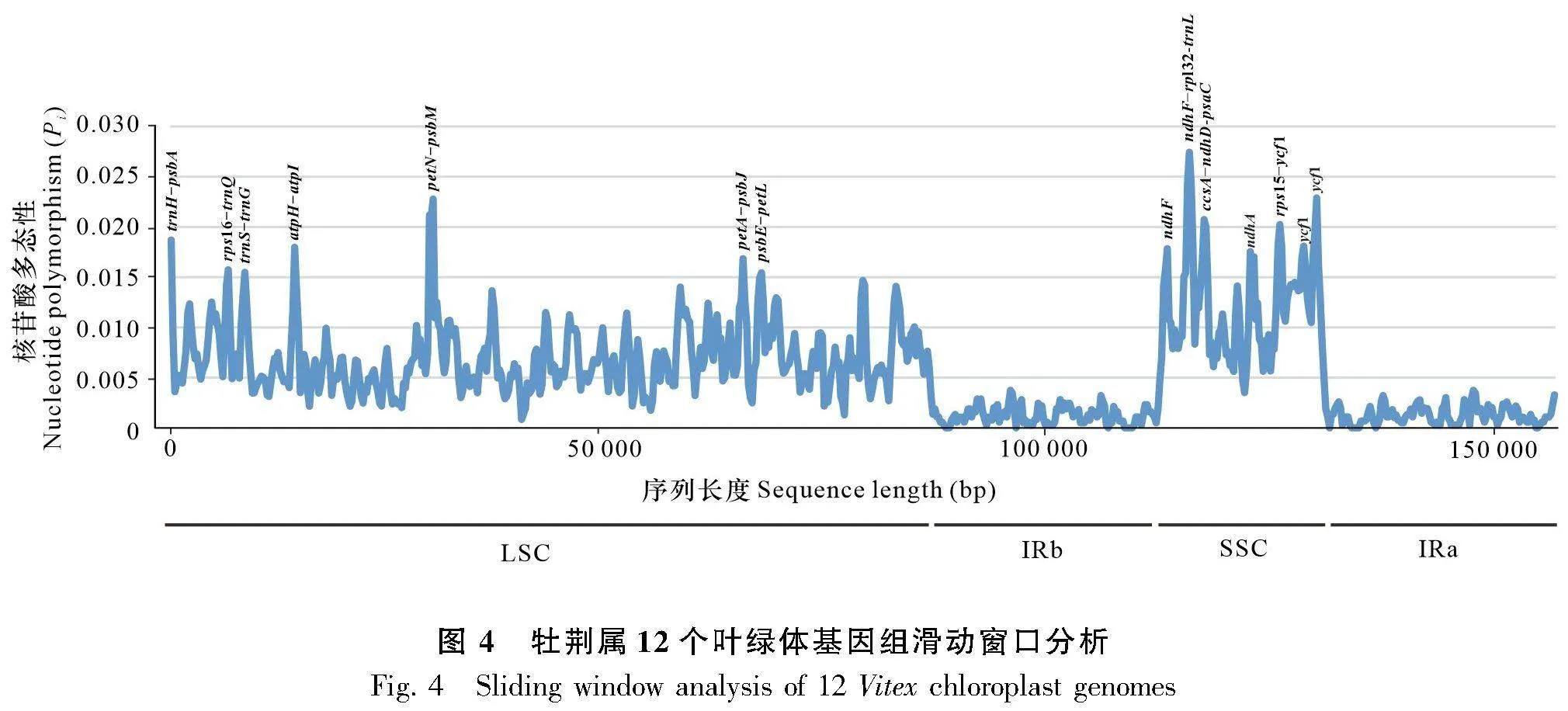
mVISTA分析顯示,牡荊屬12個物種的葉綠體基因組具有高相似性,差異主要存在于非編碼區,如rps16-trnQ(UUG)、ycf3-trnS(GGA)、ndhF-rpl32-trnL(UAG)(圖3)。牡荊屬12個葉綠體基因組全長比對長度為156 965 bp,包含3 087個變異位點、1 423個信息位點和598個插入缺失,核苷酸多態性(Pi)為0.005 69。另外,蛋白編碼區顯示出高保守性,具有最低的變異位點數目和Pi;反之,非編碼區具有最高的變異位點數目和Pi(表2)。滑動窗口分析結果顯示Pi在0~0.027 45之間,并檢測到14個Pi大于0.015的高變區(trnH-psbA、rps16-trnQ、trnS-trnG、atpH-atpI、petN-psbM、petA-psbJ、psbE-petL、ndhF-rpl32-trnL、ccsA-ndhD-psaC、rps15-ycf1、ndhF、ndhA和ycf1)。14個高變區中的7個位于LSC區、7個位于SSC區。這些高變區中10個位于基因間隔區,剩余4個位于ndhF、ndhA和ycf1基因區(圖4)。
2.4 葉綠體基因組密碼子偏好分析
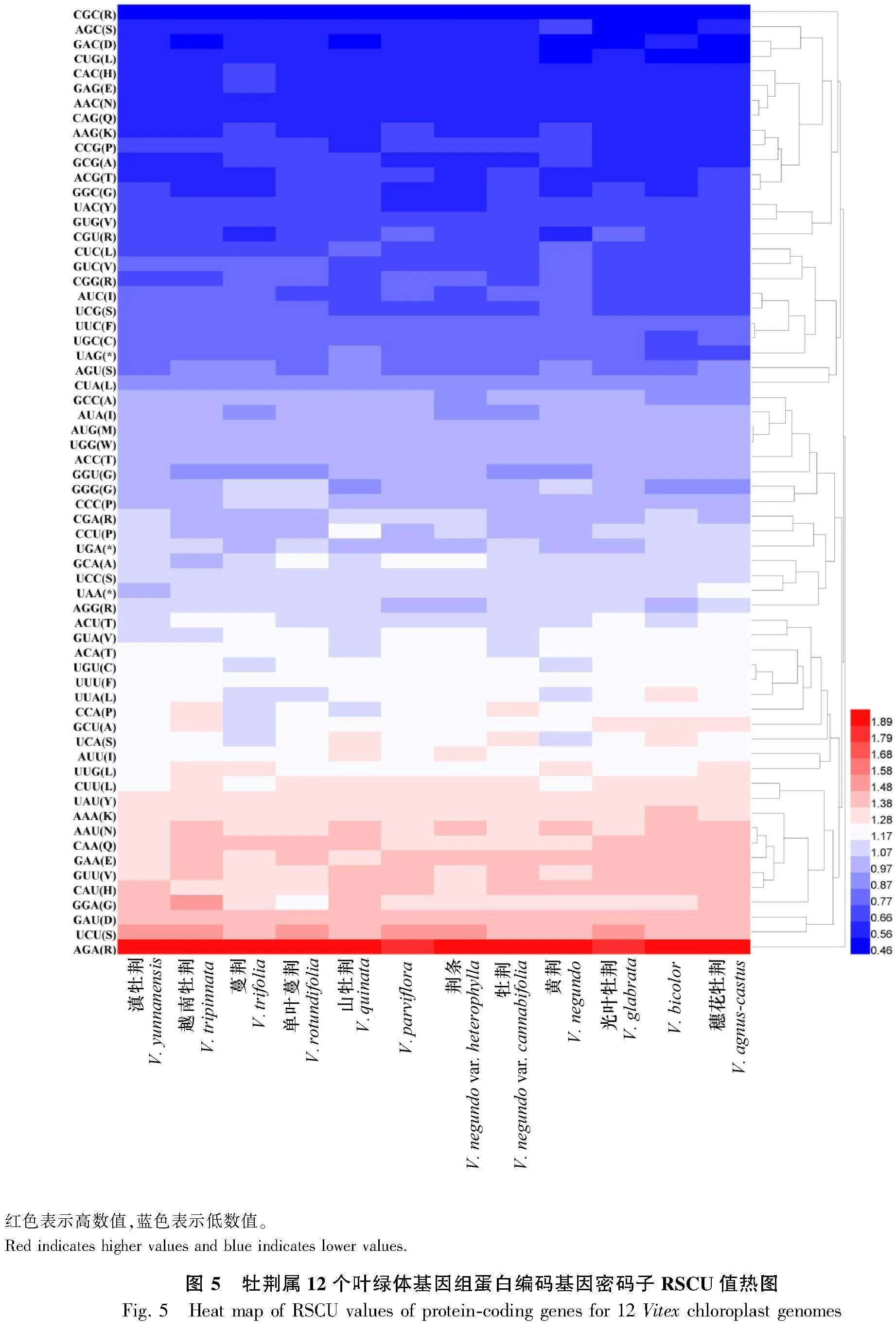
2.4.1 同義密碼子分析 牡荊屬12個葉綠體基因組的密碼子數目在26 037個(V. parviflora)~26 796個(蔓荊)之間。64種密碼子中,甲硫氨酸(M)和色氨酸(W)分別僅由密碼子AUG和UGG編碼,因此這2種密碼子未顯示出使用偏好性(RSCU=1);有31種密碼子顯示出偏好性(RSCU>1)。值得注意的是,除密碼子UCC和UUG外,其他具有使用偏好性的密碼子第3位均為嘌呤(A/U)(圖5)。
2.4.2 中性繪圖分析 若基因均勻地分布在坐標軸對角線兩側且回歸系數接近1,則表明GC12與GC3變異更多地受到突變的影響,自然選擇對其影響較小;若基因呈現不對稱分布且回歸系數離1較遠,則表明GC12與GC3的變異主要受自然選擇的影響,突變對其的影響較小。由附圖2可知,GC12和GC3的分布范圍較窄,表明突變對密碼子使用偏好性影響較小,中性圖斜率為0.080 4~0.179 9,表明突變對密碼子使用偏好性影響僅占8.04%~17.99%,而選擇壓力占82.01%~91.96%。結果表明,牡荊屬12個葉綠體基因組密碼子使用偏好性主要受選擇壓力的影響。
2.4.3 ENC-Plot分析 以12個牡荊屬植物葉綠體基因組的GC3s為橫坐標,ENC值為縱坐標繪制ENC圖。附圖3中牡荊屬12個葉綠體基因組的ENC-Plot圖相似,大多數基因位于標準曲線的下方,離標準曲線距離較遠,說明大多數基因密碼子偏好性主要受自然選擇的影響,有少部分基因緊靠標準曲線上方,表明這部分基因密碼子偏好性受突變的影響。
2.4.4 PR2-Plot分析 附圖4結果顯示,坐標點不相等地分布在4個區域內,坐標點主要分布在右下方區域,這表明密碼子第3位核苷酸使用頻率為T>A、G>C,即密碼子的第3位核苷酸在選擇上具有偏好性。在其他區域也有部分基因散落,說明密碼子使用偏好性不僅受突變壓力的影響,還受選擇壓力等因素的影響。
2.4.5 最優密碼子分析 由表3可知,最優密碼子數量介于10~19個之間,其中V. parviflora和滇牡荊的最優密碼子數量最多,穗花牡荊、牡荊、荊條、單葉蔓荊和越南牡荊的最優密碼子數量最少。分析它們的最優密碼子數據可知,牡荊屬12個葉綠體基因組的最優密碼子大多以A或U作為第3位核苷酸,說明其最優密碼子偏向于使用A和U作為結尾。對其共有最優密碼子進行分析,發現其共有最優密碼子6個,分別為UGU、CAC、AUU、UUG、ACU和GUU。
2.5 葉綠體基因組重復序列分析
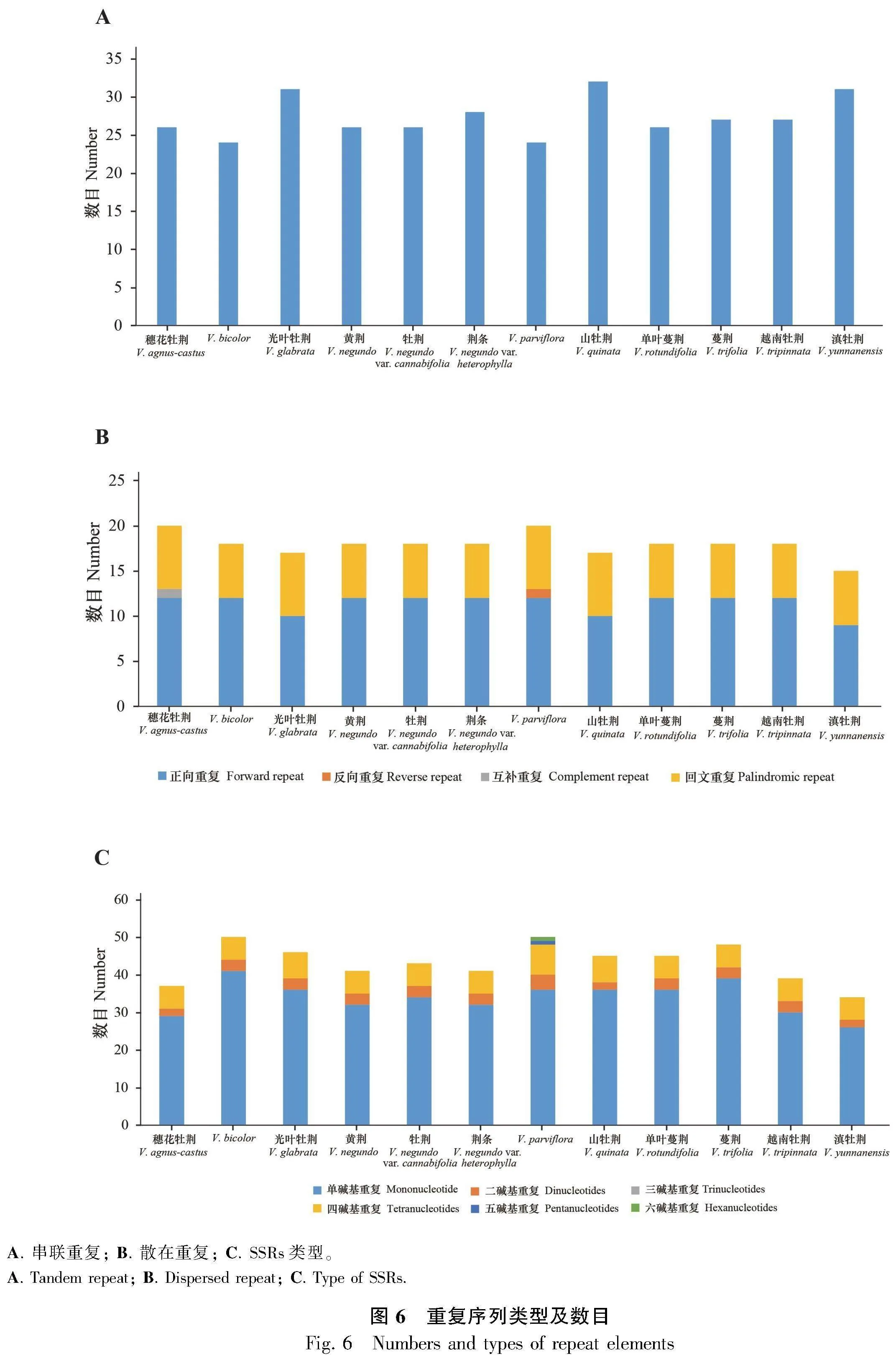
牡荊屬12個葉綠體基因組中共檢測到328個串聯重復,其中山牡荊具有最多的串聯重復(32個),V. bicolor和V. parviflora具有最少的串聯重復,均為24個(圖6:A)。串聯重復的長度范圍為22~90 bp;串聯重復單元長度范圍為11~42 bp;串聯重復次數為2~5次。另外,在12條葉綠體基因組序列中共檢測到215個散在重復,其中數目最多的是正向重復(137個,63.7%);回文重復數目次之(76個,35.3%);反向重復和互補重復僅各檢測到1個,分別存在于V. parviflora和穗花牡荊(圖6:B)。12條葉綠體基因組序列中共檢測到519個SSRs,其中占比最大的是單核苷酸重復(407個,78.4%),其次是四核苷酸重復(76個,14.6%)和二核苷酸重復(34個,6.6%),五核苷酸重復和六核苷酸重復各檢測到1個,未檢測到三核苷酸重復(圖6:C)。
2.6 系統發育關系分析
共37條完整葉綠體基因組用于分析系統發育關系,具體的物種信息見表4。基于完整葉綠體基因組序列構建的ML樹和BI樹得到了一致的拓撲結構并獲得了較高支持率(圖7)。內類群分為兩大支,分別對應唇形科和馬鞭草科。唇形科中,12個亞科均為單系支且獲得了高度支持。牡荊屬所在的牡荊亞科和六苞藤亞科擁有最近的親緣關系。牡荊屬分為兩支,其中一支包含V. parviflora、滇牡荊、山牡荊和光葉牡荊;第二支包含剩余的物種和變種。值得注意的是,黃荊與其變種牡荊聚為一支,但其另一變種荊條卻與其他物種聚為另一小支,支持率較低(MLBS=48%,BIPP=0.95)。馬鞭草科中,6個物種劃分在3個單系族,其中馬纓丹族(Lantaneae)和馬鞭草族(Verbeneae)具有更近的親緣關系。
3 討論與結論
3.1 牡荊屬葉綠體基因組特征
與大多數陸地植物葉綠體基因組一樣,牡荊屬的12個葉綠體基因組均呈現典型的環狀四分體結構,并且長度、GC含量和基因組成均展示出高度保守性。大多數陸地植物葉綠體基因組的IR邊界會發生一定變化(可達數百個核苷酸),但是顯著的IR區邊界變化(高達數千核苷酸)相對較少(Zhu et al., 2016)。與一些唇形科植物,如鼠尾草屬(Salvia)(Zhao et al., 2020a)、青蘭屬(Dracocephalum)及近緣屬(Fu et al., 2022)、刺蕊草屬(Pogostemon)(Zhang et al., 2020)、黃芩亞科(Zhao et al., 2020b)的葉綠體基因組類似,牡荊屬12個葉綠體基因組的IR區發生了低程度的收縮擴張且僅發生在SSC/IRb和IRb/LSC邊界。與其他11個葉綠體基因組相比,V. parviflora 的SSC/IRb邊界發生了較明顯的不同,這可能是由ycf1基因注釋誤差引起的;IRb/LSC邊界發生的收縮擴張現象同樣與基因注釋差異相關。mVISTA的結果顯示,12個葉綠體基因組序列相似性較高且與預期一致,物種間的序列差異主要發生在非編碼區。滑動窗口分析共檢測到14個高變區,可用作推斷牡荊屬系統發育關系的候選分子標記。高變區中的trnH-psbA是植物DNA條形碼推薦的片段之一(Hollingsworth et al., 2009, 2011);ycf1編碼葉綠體內膜蛋白易位子組分蛋白,是葉綠體基因組中最大的基因之一(Kikuchi et al., 2013),ycf1基因在不同分類群(甚至是低等分類單元)間高度變異,常用作系統發育研究的分子標記(Dong et al., 2012)。
3.2 密碼子使用偏好和重復序列
牡荊屬12個葉綠體基因組具有一致的密碼子使用偏好,31種密碼子均顯示出使用偏好性(RSCU>1),除密碼子UCC和UUG外,剩余29個密碼子的第3位均為嘌呤(A/U),這可能是葉綠體基因組進化歷程中受自然選擇和突變偏好性共同作用的結果(Xu et al., 2011; Li et al., 2021)。密碼子偏好性分析結果顯示,影響牡荊屬12個物種密碼子使用偏好性的因素有選擇壓力、突變壓力等,但受選擇壓力的影響較大,這與櫟屬青岡亞屬(Quercus subg. Cyclobalanopsis)(黃婷等,2023)、紫花苜蓿(Medicago sativa)(喻鳳和韓明,2021)等葉綠體基因組密碼子偏好性的研究結果相似。
作為一類重要的分子標記,SSRs廣泛用于群體遺傳學研究(Powell et al., 1996)。與多數被子植物的研究結果一樣,如櫟屬(Quercus)(Yang et al., 2016)、駝蹄瓣屬(Zygophyllum)(Yang et al., 2022)和蒺藜科(Zygophyllaceae)(Zhang et al., 2021),牡荊屬葉綠體基因組中占比最高的SSRs是單核苷酸重復;不同于上述列舉的被子植物,牡荊屬占比第二的SSR類型是四核苷酸重復,而非二核苷酸重復。相比之下,牡荊屬各類型SSRs占比情況與唇形科鼠尾草屬植物的研究結果高度相似(Zhao et al., 2020a)。另外,本研究檢測到的SSRs可作為該屬群體遺傳學研究的候選分子標記。
一些研究認為長而復雜的重復序列在葉綠體基因組重排和序列分化中發揮著重要作用(Timme et al., 2007; Weng et al., 2014)。一些葉綠體基因組發生高度重排的植物類群中檢測到了大量長而復雜的重復序列,如Geranium palmatum的葉綠體基因組中檢測到505個重復序列(Weng et al., 2014),Passiflora costaricensis的葉綠體基因組中最長的重復序列為1 070 bp(Cauz-Santos et al., 2020)。本研究在12個葉綠體基因組中共檢測到328個串聯重復和215個散在重復,并且這些重復序列的長度較短(均未超過100 bp),與牡荊屬葉綠體基因組序列的高度保守、未檢測到重排的結果一致。
3.3 牡荊屬系統位置推斷及種間關系分析
近年來,在唇形科7個亞科,即筋骨草亞科(Ajugoideae)、野芝麻亞科(Lamioideae)、荊芥亞科(Nepetoideae)、Prostantheroideae、黃芩亞科(Scutellarioideae)、六苞藤亞科(Symphorematoideae)和牡荊亞科的分類基礎上(Harley et al., 2004),Li等(2016)的分子研究結果首次描述發表了歧傘花亞科(Cymarioideae)、佩龍木亞科(Peronematoideae)和豆腐柴亞科(Premnoideae)這3個新的亞科,之后分別對紫珠屬和柚木屬構成的進化支命名為亞科,即紫珠亞[JP2]科(Callicarpoideae)和柚木亞科(Tectonoideae)(Li & Olmstead, 2017);Zhao等(2021)的分子研究結果同樣高度支持唇形科的12個單系亞科。本研究的進化樹同樣支持上述分子研究結果,即牡荊屬應劃分到唇形科的牡荊亞科且牡荊亞科與六苞藤亞科擁有最近的親緣關系。
目前牡荊屬的物種的界定仍存在一定爭議(Harley et al., 2004; Bramley et al., 2009; Li et al., 2016),已有的研究仍未能全面地厘清該屬的系統發育關系。例如,形態學、解剖學和孢粉學結果揭示非洲南部的牡荊屬植物(9種、3亞種和2變種)分為Vitex subg. Vitex和Vitex subg. Holmskioldiopsis2個亞屬(Bredenkamp & Botha, 1993);《中國植物志》將我國分布的14種牡荊屬植物分為腋序組(Vitex sect. Axillares)和頂序組(Vitex sect. Vitex);Bramley等(2009)和Li等(2016)基于分子片段探究了一定數量牡荊類植物的關系,前者顯示牡荊類植物分為多支,后者顯示牡荊類植物分為2支(支持率較低)。本研究對牡荊屬10種和2變種的系統學分析顯示,大多數分支具有較高的支持率,這表明葉綠體全基因組序列在牡荊屬系統發育重建中具有一定有效性,可為后續進一步全面探究該屬系統發育關系提供研究思路。另外,本研究為一些具有爭議的種間關系的確定提供了一定參考。蔓荊、V. bicolor和單葉蔓荊三者間的關系目前存在不同看法:單葉蔓荊或被看作是蔓荊的亞種,命名為V. trifolia subsp. littoralis(de Kok, 2008; Gentallan et al., 2022),或是蔓荊的變種(中國科學院中國植物志編輯委員會,1982),或是獨立的種(Flora of China Editorial Committee, 1994; Sun et al., 2019),或將三者看作蔓荊復合體(V. trifolia complex)(Gentallan et al., 2023);V. bicolor或被看作是蔓荊的異名(Flora of China Editorial Committee, 1994),或被看作是蔓荊的變種,即V. trifolia var. bicolor(Munir, 1987; Gentallan et al., 2023)。在本研究中,單葉蔓荊與V. bicolor和蔓荊的姐妹支聚為一支,結合Sun等(2019)研究得到的蔓荊和單葉蔓荊具有顯著遺傳分化的結果,我們更支持Flora of China的分類結果,即將單葉蔓荊看作是獨立的種;而V. bicolor是蔓荊的異名或是變種,仍需要更多的數據去探究。本研究另一值得注意的種間關系:黃荊與其兩變種并未展示出姐妹關系,而是黃荊與牡荊聚為一支,但荊條與其他物種聚在另一支(支持率較低),這表明黃荊可能存在較大的種內變異(至少在葉綠體基因組水平)。黃荊種下分類群聚類的研究結果顯示,黃荊與其兩變種雖均為花序狹窄型,但荊條和牡荊的關系更近(路端正等,2015),與我們的研究結果不同。因此,黃荊種下不同類群是否因適應不同生態環境而發生了遺傳分化,是否處于物種形成階段,仍需進一步探究。
參考文獻:
BENSON G, 1999. Tandem repeats finder: a program to analyze DNA sequences [J]. Nucl Acid Res, 27(2): 573-580.
BRAMLEY GL, FOREST F, DE KOK RP, 2009. Troublesome tropical mints: re-examining generic limits of Vitex and relations (Lamiaceae) in South East Asia [J]. Taxon, 58(2): 500-510.
BREDENKAMP CL, BOTHA DJ, 1993. A synopsis of the genus Vitex L. (Verbenaceae) in southern Africa [J]. S Afr J Bot, 59(6): 611-622.
CANTINO PD, 1992. Evidence for a polyphyletic origin of the Labiatae [J]. Ann Mo Bot Gard, 79(2): 361-379.
CAUZ-SANTOS LA, DA COSTA ZP, CALLOT C, et al., 2020. Repertory of rearrangements and the loss of an inverted repeat region in Passiflora chloroplast genomes [J]. Genome Biol Evol, 12(10): 1841-1857.
CHEVREUX B, PFISTERER T, DRESCHER B, et al., 2004. Using the miraEST assembler for reliable and automated mRNA transcript assembly and SNP detection in sequenced ESTs [J]. Genome Res, 14(6): 1147-1159.
DARLING ACE, MAU B, BLATTNER FR, et al., 2004. Mauve: multiple alignment of conserved genomic sequence with rearrangements [J]. Genome Res, 14(7): 1394-1403.
DE KOK R, 2008. The genus Vitex (Labiatae) in the flora Malesiana region, excluding New Guinea [J]. Kew Bull, 63: 17-40.
DENG W, WANG Y, LIU Z, et al., 2014. HemI: a toolkit for illustrating heatmaps [J]. PLoS ONE, 9(11): e111988.
DONG W, LIU J, YU J, et al., 2012. Highly variable chloroplast markers for evaluating plant phylogeny at low taxonomic levels and for DNA barcoding [J]. PLoS ONE, 7(4): e35071.
DOYLE JJ, 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue [J]. Biol Pharm Bull, 19(1): 11-15.
Editorial Committee of China Flora of Chinese Academy of Sciences, 1982. Flora Reipublicae Popularis Sinicae (Verbenaceae): Vol. 65 [M]. Beijing: Science Press: 131-150. [中國科學院中國植物志編輯委員會, 1982. 中國植物志 (馬鞭草科): 第65卷 [M]. 北京: 科學出版社: 131-150.]
FAIRCLOTH BC, 2008. MSATCOMMANDER: detection of microsatellite repeat arrays and automated, locus-specific primer design [J]. Mol Ecol Resour, 8(1): 92-94.
Flora of China Editorial Committee, 1994. Flora of China: Verbenaceae (Vol. 17) [M]. Beijing: Science Press; St. Louis: Missouri Botanical Garden Press: 28-32.
FRAZER KA, PACHTER L, POLIAKOV K, et al., 2004. VISTA: Computational tools for comparative genomics [J]. Nucl Acid Res, 32(Suppl. 2): W273-W279.
FU G, LIU Y, CARABALLO-ORTIZ MA, et al., 2022. Characterization of the complete chloroplast genome of the dragonhead herb, Dracocephalum heterophyllum (Lamiaceae), and comparative analyses with related species [J]. Diversity, 14(2): 110.
GENTALLAN RP, QUINONES KJO, BARTOLOME MCB, et al., 2022. The complete chloroplast genome of Vitex trifolia L. (Lamiaceae) [J]. Mitochondrial DNA B, 7(7): 1316-1318.
GENTALLAN RP, QUINONES KJO, BARTOLOME MCB, et al., 2023. Characterization of the complete chloroplast genome of Vitex bicolor Willd. and its comparative analyses with other species belonging to the Vitex trifolia complex [J]. Plant Genet Resour-C, 21(1): 19-27.
HAHN C, BACHMANN L, CHEVREUX B, 2013. Reconstructing mitochondrial genomes directly from genomic next-generation sequencing reads — a baiting and iterative mapping approach [J]. Nucl Acid Res, 41(13): e129.
HARLEY RM, ATKINS S, BUDANTSEV AL, et al., 2004. The families and genera of vascular plants: Labiatae [M]. Berlin: Springer Verlag: 167-275.
HOLLINGSWORTH PM, FORREST LL, SPOUGE JL, et al., 2009. A DNA barcode for land plants [J]. Proc Nat Acad Sci USA, 106(31): 12794-12797.
HOLLINGSWORTH PM, GRAHAM SW, LITTLE DP, 2011. Choosing and using a plant DNA barcode [J]. PLoS ONE, 6: e19254.
HUANG T, TANG M, CHEN XL, et al., 2023. Comparison of chloroplast genomes and phylogenic analysis of four species of Quercus subg. Cyclobalanopsis [J]. Guihaia, 43 (4): 741-754. [黃婷, 唐夢, 陳曉麗, 等, 2023. 四種櫟屬青岡亞屬植物葉綠體基因組特征及系統發育研究 [J]. 廣西植物, 43(4): 741-754.]
JANGWAN JS, AQUINO RP, MENCHERINI T, et al., 2013. Chemical constituents of ethanol extract of leaves and molluscicidal activity of crude extracts from Vitex trifolia Linn [J]. Herba Polonic, 59(4): 19-32.
KAMAL N, MIO ASNI NS, ROZLAN INA, et al., 2022. Traditional medicinal uses, phytochemistry, biological properties, and health applications of Vitex sp. [J]. Plants, 11(15): 1SdJgOGrSox95s8+BBeGl6gnOYdmhpmyNAYQBDVXu5A=1944.
KARAGUZEL O, GIRMEN B, 2009. Morphological variations of chaste tree (Vitex agnus-castus) genotypes from southern Anatolia, Turkey [J]. New Zeal J Crop Hortic, 37(3): 253-261.
KATOH K, STANDLEY DM, 2013. MAFFT multiple sequence alignment software version 7: improvements in performance and usability [J]. Mol Biol Evol, 30(4): 772-780.
KIKUCHI S, BEDARD J, HIRANO M, et al., 2013. Uncovering the protein translocon at the chloroplast inner envelope membrane [J]. Science, 339: 571-574.
KURTZ S, CHOUDHURI JV, OHLEBUSCH E, et al., 2001. REPuter: the manifold applications of repeat analysis on a genomic scale [J]. Nucl Acid Res, 29(22): 4633-4642.
LI B, CANTINO PD, OLMSTEAD RGghHW2kVOcwGrxmZufeJlqXYJwUiwNZvrMippQkuUqQM=, et al., 2016. A large-scale chloroplast phylogeny of the Lamiaceae sheds new light on its subfamilial classification [J]. Sci Rep, 6(1): 34343.
LI B, OLMSTEAD RG, 2017. Two new subfamilies in Lamiaceae [J]. Phytotaxa, 313(2): 222-226.
LI G, ZHANG L, XUE P, 2021. Codon usage pattern and genetic diversity in chloroplast genomes of Panicum species [J]. Gene, 802: 145866.
LIBRADO P, ROZAS J, 2009.DnaSP v5: a software for comprehensive analysis of DNA polymorphism data [J]. Bioinformatics, 25(11): 1451-1452.
LU DZ, WANG JL, XIE L, 2015. Analysis on clustering and evolutionary trends of infraspecific taxa of Vitex negundo L. [J]. J Beijing For Univ, 37 (2): 55-58. [路端正, 王九麗, 謝磊, 2015. 黃荊種以下分類群的聚類與演化趨勢分析 [J]. 北京林業大學學報, 37(2): 55-58.]
MARX HE, O′LEARY N, YUAN YW, et al., 2010. A molecular phylogeny and classification of Verbenaceae [J]. Am J Bot, 97(10): 1647-1663.
MUNIR AA, 1987. A taxonomic revision of the genus Vitex L. (Verbenaceae) in Australia [J]. J Adelaide Bot Gard, 31-79.
NIROUMAND MC, HEYDARPOUR F, FARZAEI MH, 2018. Pharmacological and therapeutic effects of Vitex agnus-castus L.: A review [J]. Pharmacol Rev, 12(23): 103.
POWELL W, MORGANTE M, ANDRE C, et al., 1996. The comparison of RFLP, RAPD, AFLP and SSR (microsatellite) markers for germplasm analysis [J]. Mol Breed, 2(3): 225-238.
REFULIO-RODRIGUEZ NF, OLMSTEAD RG, 2014. Phylogeny of Lamiidae [J]. Am J Bot, 101(2): 287-299.
RONQUIST F, HUELSENBECK JP, 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models [J]. Bioinformatics, 19(12): 1572-1574.
SCHFERHOFF B, FLEISCHMANN A, FISCHER E, et al., 2010. Towards resolving Lamiales relationships: insights from rapidly evolving chloroplast sequences [J]. BMC Evol Biol, 10: 352.
SHARP PM, LI WH, 1986. An evolutionary perspective on synonymous codon usage in unicellular organisms [J]. J Mol Evol, 24(1): 28-38.
STAMATAKIS A, 2006. RAxML-VI-HPC: maximum likelihood-based phylogenetic analysis with thousands of taxa and mixed models [J]. Bioinformatics, 22(21): 2688-2690.
SUN XH, 2020. Floral biology characteristics and mating system of Vitex negundo L. var. heterophylla (Franch.) Rehder. [D]. Jinan: Shandong University. [孫小涵, 2020. 荊條開花生物學特性和交配系統研究 [D]. 濟南: 山東大學.]
SUN Y, YANG H, ZHANG Q, et al., 2019. Genetic diversity and its conservation implications of Vitex rotundifolia (Lamiaceae) populations in East Asia [J]. PeerJ, 7: e6194.
TAMURA K, PETERSON D, PETERSON N, et al., 2011. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Mol Biol Evol, 28(10): 2731-2739.
TIMME RE, KUEHL JV, BOORE JL, et al., 2007. A comparative analysis of the Lactuca and Helianthus (Asteraceae) plastid genomes: identification of divergent regions and categorization of shared repeats [J]. Am J Bot, 94(3): 302-312.
VISHWANATHAN AS, BASAVARAJU RA, 2010. A review on Vitex negundo L.: A medicinally important plant [J]. Eur J Soil Biol, 3(1): 30-42.
WENG ML, BLAZIER JC, GOVINDU M, et al., 2014. Reconstruction of the ancestral plastid genome in Geraniaceae reveals a correlation between genome rearrangements, repeats and nucleotide substitution rates [J]. Mol Biol Evol, 31(3): 645-659.
XIN HM, 2005. The chemical constituents of anti-PMS activity, intraspecific variation and quality evaluation of Fructus Viticis [D]. Shanghai: Second Military Medical University. [辛海量, 2005. 蔓荊子抗PMS物質基礎、種內變異和品質評價研究 [D]. 上海: 第二軍醫大學.]
XU C, CAI XN, CHEN QZ, et al., 2011. Factors affecting synonymous codon usage bias in chloroplast genome of Oncidium Gower Ramsey [J]. Evol Bioinform, 7: 271-278.
YANG YC, JIA Y, ZHAO YL, et al., 2022. Comparative chloroplast genomics provides insights into the genealogical relationships of endangered Tetraena mongolica and the chloroplast genome evolution of related Zygophyllaceae species [J]. Front Genet: 13: 1026919.
YANG YC, ZHOU T, DUAN D, et al., 2016. Comparative analysis of the complete chloroplast genomes of five Quercus species [J]. Front Plant Sci, 7: 959.
YANG YC, ZHOU T, QIAN Z, et al., 2021. Phylogenetic relationships in Chinese oaks (Fagaceae, Quercus): Evidence from plastid genome using low-coverage whole genome sequencing [J]. Genomics, 113(3): 1438-1447.
YI RZ, 2022. Efh4YqEMsc25TNqTIKWZulHIouWUcGsZIPb/f3ZvlI8Mw=fects of pruning and paclobutrazol on ornamental qualities of Vitex agnus-castus [D]. Shanghai: Shanghai University of Applied Technology. [易仁知, 2022. 修剪和多效唑對穗花牡荊觀賞品質的影響 [D]. 上海: 上海應用技術大學.]
YU F, HAN M, 2021.Analysis of codon usage bias in the chloroplast genome of alfalfa (Medicago sativa) [J]. Guihaia, 41 (12): 2069-2076. [喻鳳, 韓明, 2021. 紫花苜蓿葉綠體基因組密碼子偏好性分析 [J]. 廣西植物, 41(12): 2069-2076.]
ZHANG CY, LIU TJ, MO XL, et al., 2020. Comparative analyses of the chloroplast genomes of patchouli plants and their relatives in Pogostemon (Lamiaceae) [J]. Plants, 9(11): 1497.
ZHANG L, WANG S, SU C, et al., 2021. Comparative chloroplast genomics and phylogenetic analysis of Zygophyllum (Zygophyllaceae) of China [J]. Front Plant Sci, 12: 723622.
ZHAO F, CHEN YP, SALMAKI Y, et al., 2021. An updated tribal classification of Lamiaceae based on plastome phylogenomics [J]. BMC Biol, 19(1): 1-27.
ZHAO F, DREW BT, CHEN YP, et al., 2020a. The chloroplast genome of Salvia: Genomic characterization and phylogenetic analysis [J]. Int J Plant Sci, 181(8): 812-830.
ZHAO F, LI B, DREW BT, et al., 2020b. Leveraging plastomes for comparative analysis and phylogenomic inference within Scutellarioideae (Lamiaceae) [J]. PLoS ONE, 15(5): e0232602.
ZHU A, GUO WH, GUPTA S, et al., 2016. Evolutionary dynamics of the plastid inverted repeat: the effects of expansion, contraction, and loss on substitution rates [J]. New Phytol, 209(4): 1747-1756.

