嗜熱毀絲霉木糖代謝調控機制研究



摘要:為提高發酵菌株的木糖利用速率,以嗜熱毀絲霉為體系,利用遺傳學手段及轉錄組學技術,探究絲狀真菌嗜熱毀絲霉木糖代謝的調控機制。結果表明,在嗜熱毀絲霉中,轉錄因子XlnR能夠正向調控木糖代謝和轉運以及半纖維酶的表達,是木糖條件下孢子萌發的必需因子。在木糖條件下,突變體ΔxlnR孢子喪失了萌發的能力,并且菌絲木糖利用速率顯著降低,而過表達xlnR 使得木糖利用速率提升14.2%。轉錄組數據表明,敲除xlnR導致木聚糖水解酶基因、木糖轉運蛋白及代謝相關基因的轉錄水平顯著降低。全局調控因子Cre-1是木糖及葡萄糖代謝的抑制因子,敲除cre-1 后顯著提升嗜熱毀絲霉木糖和葡萄糖利用速率;同時,Cre-1可直接抑制xlnR 及自身的表達。通過過表達木糖代謝激活因子XlnR編碼基因xlnR,同時敲除抑制因子Cre-1編碼基因cre-1,嗜熱毀絲霉木糖利用速率提高100%。以上研究結果為生物質高效利用底盤細胞的創建提供了良好的出發菌株。
關鍵詞:嗜熱毀絲霉;木糖代謝;轉錄調控;Cre-1;XlnRdoi:10.13304/j.nykjdb.2023.0151
中圖分類號:S182;Q933 文獻標志碼:A 文章編號:10080864(2024)12007711
生物質資源是地球上最豐富的可再生有機原料,實現生物質轉化生產生物燃料以及生物化學品,是解決目前發酵原料短缺、實現雙碳目標的關鍵。木質纖維素由纖維素、半纖維素以及木質素構成,三者之間通過共價鍵和氫鍵緊密纏繞連接[1],形成堅硬的、難降解的剛性結構。在生物質中纖維素含量為38%~50%,半纖維素含量為23%~32%[2],二者通過多種碳水化合物活性酶協同作用進行降解[3],降解后產生的纖維寡糖由β-葡萄糖苷酶 (β-glucosidase, BGLs)降解成葡萄糖,木寡糖由β-木糖苷酶 (β-xylosidase, BXLs) 降解成木糖。傳統生物質利用技術主要包括預處理、纖維素酶生產和木質纖維素酶解糖化以及產品發酵等工藝[4]。目前,國內外纖維素酶工業生產主要體系均為絲狀真菌,包括里氏木霉(Trichoderma reesei)[5]、嗜熱毀絲霉(Myceliopthorathermophila ) 以及青霉菌 (Penicillium)[67]。經過遺傳改造以及人工誘變后,工業菌株嗜熱毀絲霉和里氏木霉的纖維素酶產量均達到100 g·L-1[8-10],顯著降低了生物質發酵成本。生物質水解液的主要成分為葡萄糖和木糖,實現生物質全糖的高效率利用是生物質高效轉化的關鍵。盡管研究者已經鑒定了木糖主要代謝途徑[11],但對于其調控網絡尚未明晰。
絲狀真菌是纖維素酶的主要生產菌,同時也被開發為重要的有機酸等工業發酵菌種[7, 1213]。絲狀真菌在利用生物質水解糖的過程中,由于碳分解代謝物阻遏效應(carbon cataboliterepression,CCR)會優先利用葡萄糖,而木糖利用則嚴重滯后[14]。研究發現,碳阻遏調控因子Cre-1(CreA)是Cys2His2型轉錄因子[1516],已被證實存在于多種絲狀真菌中,如里氏木霉(T. reesei)[17]。Cre-1通過調控糖轉運蛋白基因、關鍵轉錄因子、糖代謝途徑基因和纖維素酶基因的表達來實現對生物質中纖維素和半纖維素降解的調控[13,17],同時,Cre-1可直接與木聚糖酶、半乳糖苷酶和阿拉伯聚糖酶基因及其代謝途徑關鍵酶基因的啟動子結合[15,17]。但是,Cre-1對絲狀真菌木糖代謝的調控機制尚未明晰,有待進一步研究。在絲狀真菌中,XlnR (Xyr1/Xlr1)是木聚糖酶和纖維素酶的主要調控因子[1819]。在粗糙脈孢菌和嗜熱毀絲霉中,XlnR 主要參與木聚糖降解和木糖代謝[2021]。在黑曲霉中,XlnR 的同源蛋白Xyr1[22]參與調控20多個編碼半纖維素酶基因和纖維素酶基因的表達[23],被認為是纖維素酶和半纖維素酶基因表達的主要激活因子[15],該基因缺失后,導致所有誘導物喪失對纖維素酶的誘導作用,同時削弱誘導物對木聚糖水解酶基因和阿拉伯聚糖水解酶基因的誘導作用[2425]。
嗜熱毀絲霉為天然高效降解纖維素菌種,具有發酵溫度高、纖維素降解能力強、蛋白分泌水平高、能夠天然利用五碳糖和六碳糖等優勢,是生產工業酶和生物基化學品的優秀底盤微生物[26]。目前,研究者已開發了基于CRISPR/Cas9 和CRISPR/Cpf1的基因組編輯技術[27-29],為嗜熱毀絲霉的遺傳改造奠定了技術基礎。嗜熱毀絲霉已被開發為有機酸及有機醇細胞工廠,實現了生物質直接發酵合成目標產品[30],即真菌整合生物煉制(consolidated bioprocessing,CBP)技術[31-33]。在真菌CBP 技術中,產酶、生物質酶解和目標產品合成均由單一菌種來完成,極大降低了生物質煉制成本。因此,本研究以嗜熱毀絲霉為體系,研究其木糖代謝調控機制,通過人工干預使嗜熱毀絲霉的木糖利用效率顯著提升,為實現生物質全糖高效率利用奠定了理論及技術基礎。
1 材料與方法
1.1 材料
1.1.1 菌種和試劑 嗜熱毀絲霉ATCC 42464購于美國模式培養物集存庫(American Type CultureCollection,ATCC);質粒ppK2-Bar-GFP、pAN52-PtrpC-TN、pGEX-4T-1和AT-U6-gRNA均保藏于本實驗室;大腸桿菌感受態Mach1-T1和BL21 (DE3)購于北京全式金生物技術股份有限公司;PCR試劑盒、PCR 產物回收試劑盒、質粒小提試劑盒和GST磁珠純化試劑盒均購于康為世紀生物科技股份有限公司;限制性內切酶和T4 DNA 連接酶均購于南京諾唯贊生物科技股份有限公司;GibsonAssembly?克隆試劑盒和RNA純化回收試劑盒購于紐英倫生物技術(北京)有限公司;非變性聚丙烯酰胺凝膠購于賽默飛世爾科技公司;新霉素(neomycin)和草銨膦 (glufosinate ammonium) 購于北京吉普賽生物技術有限公司,氨芐青霉素購于Amresco;異丙基-β-D-硫代半乳糖苷 (isopropylthio-β-galactoside, IPTG) 購于上海阿拉丁生化科技股份有限公司;葡萄糖、木糖和阿拉伯糖購于Sigma公司;木二糖和木三糖購于Megazyme公司。
1.1.2 培養基 LB 培養基:酵母提取物5 g,胰蛋白胨10 g,氯化鈉10 g;定容至1 L,121 ℃滅菌20 min。
50×vogel’salt:檸檬酸鈉130 g,硝酸鉀126 g,磷酸二氫銨144 g,磷酸二氫鉀80 g,七水硫酸鎂10 g,二水氯化鈣5 g,微量元素5 mL。100 mL微量元素溶液含5 g C6H8O·7H2O,5 g ZnSO4·7H2O,1 g Fe(NH4)2(SO4)·6H2O,0.25 g CuSO4·5H2O,0.05 gMnSO4·H2O,0.05 g H3BO3,0.05 g NaMnO4·2H2O,0.1 mg·mL-1的生物素溶液2.5 mL;定容至1 L,加入1 mL氯仿作為保護劑,室溫保存。
2%葡萄糖/木糖/阿拉伯糖培養基:1 × vogel’salt 1 L, 碳源(葡萄糖、木糖或阿拉伯糖)20 g,115 ℃滅菌25 min。
1.2 試驗方法
1.2.1 質粒構建 靶基因敲除質粒構建:在聯合基因組研究所(Joint Genome Institute,JGI)網站查找Mycth_2310145 (xlnR) 和Mycth_2310085 (cre-1)序列信息,按照文獻[31]方法確定PAM序列,以嗜熱毀絲霉基因組DNA為模板,利用表1中引物擴增xlnR 和cre-1 的5’和3’片段。以p0380-bar 質粒為模板,設計引物(bar-F/R)并擴增PtrpC-bar 片段;以p0380-neo 質粒為模板,設計引物(neo-F/R)并擴增PtrpC-neo 片段。使用Gibson Assembly?克隆試劑盒分別將xlnR 的5’片段、3’片段和PtrpC-neo片段連接到用 BamHⅠ和EcoRⅠ 切開的ppK2-Bar-GFP 載體中,獲得質粒 donor-xlnR-neo。類似地,利用cre-1 基因的5’和3’片段以及PtrpC-bar 片段,構建質粒donor-cre-1-bar。靶向xlnR 和cre-1的sgRNA由嗜熱毀絲霉的U6啟動子控制,以ATU6-gRNA 質粒為模板,以引物 (U6p-F/U6p-cre-1-R、cre1-gRNA-F/gRNA-R)擴增U6p-cre-1 和cre-1-gRNA 片段。二者融合獲得sgRNA 片段后,將其克隆到pJET1.2/blunt 載體上,得到U6p-cre-1-sgRNA重組質粒。類似的,設計引物 U6p-F/U6pxlnR-R 和xlnR-gRNA-F/gRNA-R 分別獲得U6pxlnR和 xlnR-gRNA片段,二者融合獲得sgRNA片段后,將其插入pJET1.2/blunt載體,得到重組質粒U6p-xlnR-sgRNA。以Cas9 為模板,引物為cas-F/R,PCR擴增出Cas9表達盒。
靶基因過表達質粒構建:設計引物OE xlnRF/R,以嗜熱毀絲霉cDNA為模板,擴增xlnR 片段;將Ptef-1 啟動子、翻譯延長因子Tef-1(translationelongation factor Tef-1)啟動子和xlnR 片段插入pAN52-PtrpC-TN 載體(Spe Ⅰ 和 BamH Ⅰ 雙酶切),獲得過表達質粒pAN52-ptef-1-xlnR-PtrpCneo-TN。
1.2.2 嗜熱毀絲霉原生質體轉化及驗證 嗜熱毀絲霉原生質體轉化參照文獻[3132]。利用CRISPR/Cas9系統進行基因敲除,將sgRNA片段、donor片段和Cas9片段以1∶1∶1混合,濃縮后轉化到野生型或突變體原生質體中。在靶基因過表達過程中,將10 μg線性化質粒轉化到嗜熱毀絲霉原生質體中,并用相應的抗生素進行篩選,在35 ℃培養5 d 后,挑取少量菌絲于裂解緩沖液中,沸水浴20 min,高速離心(10 000 r·min-1,10 min)后加入150 μL滅菌水,作為模板使用。后續利用PCR驗證獲得陽性轉化子,驗證引物詳見表2。
1.2.3 突變體的形態學表征 將3 μL孢子懸液(1×104 cell·μL-1)接種到含有2%碳源(葡萄糖、木糖或阿拉伯糖)的固體培養基中,置于45 ℃條件下培養,觀察菌絲長勢,實時拍照記錄。
1.2.4 嗜熱毀絲霉糖利用分析 將成熟孢子接種到2% 葡萄糖或木糖液體培養基,接種量為106cell·mL-1,45 ℃下培養18 h,而后收集菌絲并用滅菌水洗滌3次。將菌絲接種于24孔板中,每孔接種3 mL 菌絲,分別加入木糖 (xylose)、木二糖(xylobiose) 和木三糖 (xylotriose),終含量為5 g·L-1,45 ℃、 700 r·min-1培養24 h。
1.2.5 凝膠遷移試驗 (electrophoretic mobilityshift assay, EMSA)
①獲取融合蛋白:以嗜熱毀絲霉野生型DNA為模板,用表1中的引物( cre-1-emsa-F/R 和xlnR-emsa-F/R) 擴增xlnR 和cre-1 片段,而后插入經BamHⅠ/XhoⅠ雙酶切后的載體pGEX-4T-1 中,分別獲得重組質粒pGEX-4T-1-xlnR 和pGEX-4T-1-cre-1。將重組質粒及對照質粒pGEX-4T-1 轉入到大腸桿菌BL21(DE3) 中,37 ℃ 200 r·min-1 培養至OD 值為0.38~0.42;而后加入20 μL 1 mmol·L-1 IPTG,15 ℃ 200 r·min-1 低溫誘導20 h;收集菌體后低溫破碎,并使用GST磁珠純化試劑盒純化出融合GST標簽的目的蛋白。
②獲取探針:以嗜熱毀絲霉野生型基因組作為模板,利用PCR 擴增相應基因的啟動子區片段,引物詳見表3。
③ 反應體系:50 mmol·L-1 MgCl2 2 μL、10 mg·mL-1 BSA 1 μL、50%甘油2 μL、Protein (0~200 nmol·L-1) 1 μL、探針(10 ng·μL-1) 1 μL,最后用ddH2O補到20 μL。將反應體系在25 ℃條件下孵育30 min。
④反應程序:預電泳150 V 40 min,沖洗膠孔;正式電泳150 V 50~60 min;電泳結束后,小心取出變性膠,使用SYBR Green 染色10 min,結束后,清水沖洗1次,而后在凝膠成像儀下成像。
1.2.6 糖耗分析 按時間點取發酵液1 mL,經高速離心(10 000 r·min-1,10 min)后,取出上清液過0.22 μm 濾膜,利用高效液相色譜(highperformance liquid chromatography,HPLC)測定發酵液中糖含量。檢測器為 Waters 2414示差檢測器;分離柱為Aminex HPX-87H 柱 (Bio-Rad,Hercules,CA,USA);檢測溫度45 ℃ ;流速0.5 mL·min-1;流動相為5 mmol·L-1 H2SO4。
1.2.7 RNA提取及轉錄組數據分析
①RNA提取:將嗜熱毀絲霉成熟孢子接種于2%葡萄糖培養基中,孢子終水平為106個·mL-1,于45 ℃ 150 r·min-1培養18 h,而后收集菌絲并用滅菌水洗滌3 次(40 mL·次-1);將菌絲轉移至2%木糖培養基中誘導 4 h,而后利用布氏漏斗收集菌絲,置于-80 ℃冰箱保存。利用液氮預冷的研缽,將樣品磨至粉末,而后按照RNA純化回收試劑盒和文獻[27]方法提取純化RNA。
②轉錄組數據分析:將檢測合格的RNA[OD260/OD280 gt; 1.8,RIN (RNA integrity number)gt;7.0]送至由諾禾致源公司(天津),利用IlluminaHiSeqTM 2000平臺進行測序,每個樣本2個平行,根據文獻[34]方法處理和分析轉錄組數據。
2 結果與分析
2.1 轉錄因子XlnR 在嗜熱毀絲霉木糖利用中的功能分析
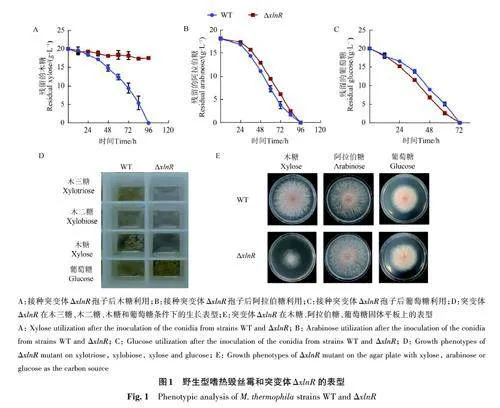
轉錄激活因子XlnR及同源蛋白具有調控半纖維素酶基因表達及木糖利用的功能[20]。為了研究XlnR (Mycth_2310145) 在嗜熱毀絲霉木糖利用中的調控作用,利用CRISPR/Cas9 基因組編輯技術[31]構建了嗜熱毀絲霉突變體ΔxlnR。對突變體ΔxlnR 在木糖、阿拉伯糖及葡萄糖液體培養基中的糖利用速率進行檢測,結果(圖1)顯示,敲除xlnR 后,嗜熱毀絲霉喪失了木糖利用能力(圖1A)。阿拉伯糖與木糖同為五碳糖,二者均是半纖維素的主要構成單元,且具有相似的代謝途徑。糖消耗試驗顯示,突變體ΔxlnR 的阿拉伯糖利用能力與野生型(WT)菌種相比顯著下降(圖1B);而葡萄糖的消耗速率上升(圖1C)。突變體ΔxlnR 對木二糖和木三糖的利用能力與木糖消耗相似,xlnR 失活后,嗜熱毀絲霉喪失了在木二糖和木三糖上生長的能力(圖1D);同時,突變體ΔxlnR 在木糖固體平板上生長狀況顯著弱于野生型(圖1E)。以上結果表明,轉錄因子XlnR在嗜熱毀絲霉木糖代謝中起激活作用。
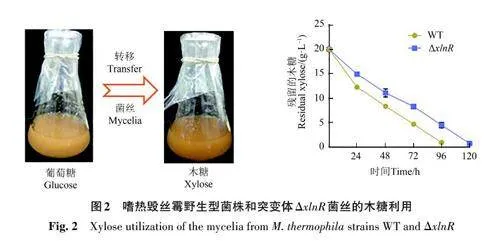
將絲狀真菌孢子接種至液體培養基中后,首先是孢子的萌發,而后菌絲體生長。xlnR 的敲除可能影響了嗜熱毀絲霉孢子在木糖條件下的萌發,進而導致突變體喪失木糖利用能力。為了驗證這一推斷,將野生型嗜熱毀絲霉和突變體ΔxlnR 在葡萄糖液體培養基中培養18 h 后轉接到木糖液體培養基中,測定木糖利用速率。結果(圖2)顯示,突變體ΔxlnR 菌絲仍然具備木糖利用的能力,但其糖耗速率顯著低于野生型菌株,說明XlnR不僅參與調控木糖代謝,而且對嗜熱毀絲霉孢子在木糖條件下萌發具有重要作用。
2.2 轉錄因子Cre-1 在嗜熱毀絲霉木糖代謝中的功能分析
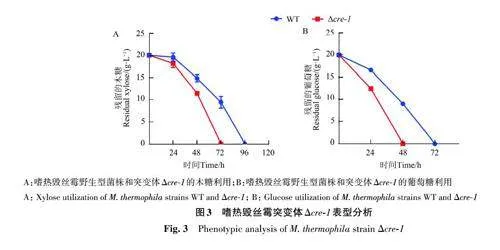
在絲狀真菌中,Cre-1是介導碳源降解物代謝阻遏的主要調控因子,參與調控轉運蛋白、轉錄因子、糖分解代謝相關因子、纖維素酶以及半纖維素酶基因的表達[17]。在嗜熱毀絲霉中,利用RNAi技術抑制cre-1 的表達后,纖維素酶及半纖維素酶基因的表達水平顯著提高[31]。為了研究Cre-1在嗜熱毀絲霉木糖代謝中的調控功能,利用基因組編輯技術構建了突變體Δcre-1,并檢測突變體木糖利用速率。結果(圖3)顯示,敲除cre-1 后,嗜熱毀絲霉的木糖利用速率顯著提升,與野生型菌株相比,突變體Δcre-1 的木糖利用速率提升25%。相似的是,在葡萄糖培養基中,突變體Δcre-1 的糖耗速率較野生型提升33%。由此表明,轉錄因子Cre-1參與調控嗜熱毀絲霉中葡萄糖和木糖的利用,是葡萄糖和木糖代謝途徑的抑制因子。
2.3 在木糖條件下突變體ΔxlnR 和Δcre-1 的轉錄組分析
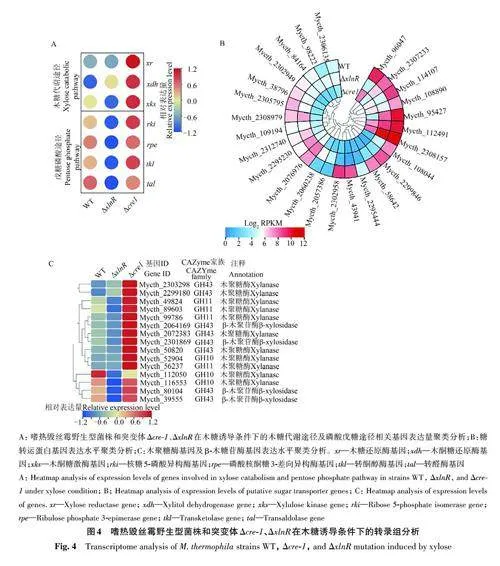
在絲狀真菌中,木糖在木糖還原酶 (xylosereductase,XR) 和木糖醇脫氫酶(xylitoldehydrogenase,XDH) 催化作用下形成木酮糖;在木酮糖激酶 (xylulokinase,XKS) 的作用下,形成5-磷酸木酮糖,然后進入戊糖磷酸途徑 (pentosephosphate pathway,PPP) 非氧化階段。為探究轉錄因子XlnR 和Cre-1 調控木糖代謝的分子機制,系統分析了嗜熱毀絲霉在木糖條件下轉錄組數據,結果(圖4A)表明,突變體ΔxlnR 中xr 和xdh的表達水平與野生型菌株相似,但木酮糖激酶基因xks 和磷酸戊糖途徑非氧化階段相關基因(rki、rpe、tkl 和tal)在突變體中的表達水平顯著低于野生型菌株,暗示XlnR可能通過調控木酮糖激酶基因表達以及PPP途徑來調節木糖代謝。在突變體Δcre-1 中,木糖代謝途徑及PPP途徑相關基因的表達水平顯著高于野生型,這與突變體Δcre-1 顯著提升的木糖利用速率一致。
糖轉運是胞內糖代謝的前提,在木糖誘導條件下,嗜熱毀絲霉中有3 個木糖轉運蛋白基因(Mycth_2308157、Mycth_96047 和Mycth_112491)被高效誘導表達,其中,Mycth_96047在粗糙脈孢菌中的同源蛋白NCU06138 為高效木糖轉運蛋白[35]。敲除xlnR 后,Mycth_96047在嗜熱毀絲霉中的表達水平下降約10倍,與突變體ΔxlnR 變弱的木糖代謝一致。另外,轉錄組數據(圖4B)表明,在突變體Δcre-1 中,11個糖轉運蛋白基因的表達水平相比野生型菌株WT顯著提高,其中11個基因的表達水平上調超過10倍。前期研究表明,粗糙脈孢菌中的Cre-1可以通過抑制糖轉運蛋白的表達,進而調控胞內糖代謝[36]。
木糖作為半纖維素構成單元,具有誘導木聚糖水解酶基因表達的作用。在木糖條件下,有5個木聚糖水解酶基因(Mycth_112050、Mycth_89603、Mycth_116553、Mycth_80104 和Mycth_39555)被高效誘導表達 (RPKMgt;100)。分析比較不同突變體木聚糖水解酶基因表達,結果(圖4C)表明,在木糖條件下,突變體ΔxlnR 中木聚糖水解酶基因的表達水平較野生型菌株顯著下調,說明XlnR在嗜熱毀絲霉中具有調控半纖維素酶基因表達的功能,這與其同源蛋白在粗糙脈孢菌[21]及里氏木霉[37]中調控作用相似;與之相反的是,敲除cre-1 后,13個木聚糖水解酶基因的表達水平顯著上調,表明Cre-1具有抑制嗜熱毀絲霉半纖維素酶基因表達的功能。
2.4 木糖利用阻遏因子Cre-1 直接抑制轉錄激活因子XlnR 的表達
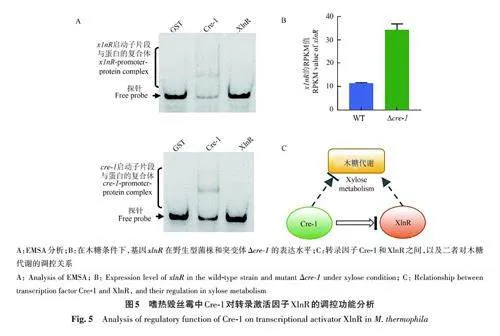
為了研究木糖利用調控因子Cre-1 與XlnR的之間的調控關系,將融合純化標簽GST 的蛋白Cre-1-GST 和XlnR-GST 在E. coli BL21 (DE3) 中表達,結果(圖5A)顯示,Cre-1可直接與xlnR 啟動子區域結合,而XlnR則不能結合于cre-1 啟動子區域。另外,比較木糖條件下嗜熱毀絲霉的轉錄組數據(圖5B)發現,與野生型菌株相比,突變體Δcre-1 中xlnR 基因 的轉錄水平上調3倍。在嗜熱毀絲霉中Cre-1能夠直接抑制調控激活因子基因xlnR 的表達(圖5C)。
2.5 嗜熱毀絲霉木糖代謝調控網絡的重構
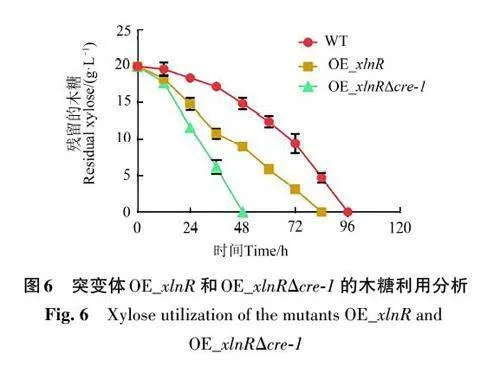
為了提升嗜熱毀絲霉木糖代謝速率,將轉錄激活因子基因xlnR 在野生型嗜熱毀絲霉中過表達,過表達突變體菌株OE_xlnR 的木糖利用速率較野生型菌株提高14.2%,完全消耗20 g·L-1木糖所需時間由96 h 降至 84 h(圖6),進一步說明轉錄因子XlnR對木糖代謝具有激活作用。
為了進一步強化木糖代謝速率,敲除突變體OE_xlnR 中基因cre-1,以實現木糖代謝的解阻遏,獲得突變體OE_xlnRΔcre-1。在木糖液體培養基中,突變體OE_xlnRΔcre-1 木糖消耗速率較對照菌株OE_xlnR 提升75%,較野生型菌種提升100%,消耗20 g·L-1 木糖所需時間縮短至48 h(圖6)。
3 討論
絲狀真菌是目前國內外纖維素酶工業生產的主要體系,同時也是眾多關鍵工業化學品的重要生產者[7]。實現絲狀真菌高效利用生物質糖是降低生物質煉制經濟成本的關鍵之一。木糖是生物質中半纖維素的重要組成部分,但由于碳代謝物阻遏效應的存在,木糖在絲狀真菌中的代謝往往受到嚴重的抑制。嗜熱毀絲霉為高效的纖維素降解菌種,同時能夠高效利用六碳糖和五碳糖,目前已被開發為工業纖維素酶和化學品細胞工廠[38]。本研究以嗜熱毀絲霉為體系,利用遺傳學手段及轉錄組學技術研究了其木糖代謝調控機制,并結合基因組編輯技術對其進行了改造,顯著提升了嗜熱毀絲霉的木糖代謝速率。
在木霉和曲霉中,轉錄因子XlnR及其同源蛋白是纖維素酶和半纖維素酶基因的主要激活因子, XlnR的失活將導致纖維素不能誘導纖維素酶和半纖維素酶基因的表達[18- 19,25]。而在粗糙脈孢菌中,XlnR 僅調控半纖維素酶基因的表達,是半纖維素利用相關基因表達所必需的[35]。此外,在曲霉和木霉中,XlnR具有調控木糖代謝的功能,參與調控木糖還原酶基因的表達,敲除xlnR 后將大幅降低突變體利用木糖和木聚糖的能力[15]。本研究利用基因編輯技術構建絲狀真菌嗜熱毀絲霉突變體ΔxlnR,表型分析發現,突變體ΔxlnR 的阿拉伯糖利用能力顯著下降,但葡萄糖利用能力顯著提升;而在木糖條件下,突變體ΔxlnR 的孢子喪失了萌發能力,且菌絲木糖的利用速率顯著降低,過表達xlnR 使得木糖利用速率提升14.2%;分析突變體ΔxlnR 在木糖條件下的轉錄組發現,敲除xlnR 導致木聚糖水解酶基因、木糖轉運蛋白基因以及磷酸戊糖途徑相關基因的轉錄水平顯著降低,說明在嗜熱毀絲霉中XlnR參與調控菌絲木糖代謝和半纖維素酶誘導,同時,對木糖條件下孢子萌發具有重要作用。
在絲狀真菌中,全局調控因子Cre-1 屬于Cys2His2 型鋅指家族,是釀酒酵母中Mig1的同源蛋白[14,39]。Cre-1 作為重要碳源代謝阻遏調控因子,在絲狀真菌生長、碳代謝及纖維素水解酶的表達中起重要調控作用[17]。在粗糙脈孢菌及里氏木霉中,敲除cre-1 能顯著提升纖維素酶和半纖維素酶的表達[16]。本研究的轉錄組數據分析結果表明,Cre-1的失活導致突變體Δcre-1 中木聚糖水解酶基因的表達水平顯著上調,暗示Cre-1對嗜熱毀絲霉半纖維素酶表達有抑制作用;同時,突變體Δcre-1 木糖消耗速率顯著提高以及木糖代謝和轉運蛋白基因高效表達,暗示Cre-1抑制嗜熱毀絲霉木糖代謝。EMSA分析表明,Cre-1能夠直接結合xlnR 啟動子區域,結合xlnR 在突變體Δcre-1 中表達水平下調,證明Cre-1能夠直接抑制xlnR 的表達;另外Cre-1能夠結合至其自身編碼基因啟動子區域,暗示Cre-1具有自我調控的功能。研究表明,在葡萄糖條件下,里氏木霉Cre-1的表達能被自身負調控[16]。這也解釋了嗜熱毀絲霉突變體Δcre-1 具有較高葡萄糖利用能力的原因。
通過代謝網絡重構可以有效地改善發酵菌株的碳源利用速率。本研究利用基因組編輯技術過表達木糖代謝激活因子XlnR編碼基因,同時敲除抑制因子Cre-1編碼基因,顯著提升了嗜熱毀絲霉木糖代謝速率,為改善發酵菌株木糖利用速率、提升生物質全糖利用提供了新思路。
參考文獻
[1] TAKKELLAPATI S, LI T, GONZALEZ M A. An overview of biorefinery derived platform chemicals from a cellulose and hemicellulose biorefinery [J]. Clean Technol. Environ. Policy,2018, 20(7):1615-1630.
[2] ALI S S, NUGENT B, MULLINS E, et al .. Fungal-mediated consolidated bioprocessing: the potential of Fusarium oxysporum for the lignocellulosic ethanol industry [J/OL]. AMB Express, 2016, 6(1):13 [2023-02-10]. https://doi.org/10.1186/s13568-016-0185-0.
[3] ZNAMEROSKI E A, CORADETTI S T, ROCHE C M, et al .. Induction of lignocellulose-degrading enzymes in Neurospora crassa by cellodextrins [J]. Proc. Natl. Acad. Sci. USA, 2012,109(16):6012-6017.
[4] BALAT M. Production of bioethanol from lignocellulosic materials via the biochemical pathway: a review [J]. Energy Convers. Manage., 2011, 52(2):858-875.
[5] VITIKAINEN M, ARVAS M, PAKULA T, et al .. Array comparative genomic hybridization analysis of Trichoderma reesei strains with enhanced cellulase production properties [J/OL].BMC Genomics, 2010, 11(1):441 [2023-02-10]. https://doi.org/10.1186/1471-2164-11-441.
[6] ALI S S, NUGENT B, MULLINS E, et al .. Insights from the fungus Fusarium oxysporum point to high affinity glucose transporters as targets for enhancing ethanol production from lignocellulose [J/OL]. PLoS One, 2013, 8(1):e54701 [2023-02-10]. https://doi.org/ 10.1371/journal.pone.0054701.
[7] LIU G, QU Y. Engineering of filamentous fungi for efficient conversion of lignocellulose: tools, recent advances and prospects [J]. Biotechnol. Adv., 2019, 37(4):519-529.
[8] VISSER H, JOOSTEN V, PUNTPETER J, et al .. Development of a mature fungal technology and production platform for industrial enzymes based on a Myceliophthora thermophila isolate, previously known as Chrysosporium lucknowense C1 [J].Ind. Biotechnol., 2011, 7:214-223.
[9] DRUZHININA I S, KUBICEK C P. Genetic engineering of Trichoderma reesei cellulases and their production [J]. Microb.Biotechnol., 2017, 10(6):1485-1499.
[10] LI N, LIU Y, LIU D, et al .. MtTRC-1, a novel transcription factor, regulates cellulase production via directly modulating the genes expression of the Mthac-1 and Mtcbh-1 in Myceliophthora thermophila [J/OL]. Appl. Environ. Microbiol.,2022, 88(19):e0126322 [2023-02-10]. https://doi.org/10.1128/aem.01263-22.
[11] HECTOR R E, QURESHI N, RHUGHES S, et al .. Expression of a heterologous xylose transporter in a Saccharomyces cerevisiae strain engineered to utilize xylose improves aerobic xylose consumption [J]. Appl. Microbiol. Biotechnol., 2008, 80(4):675-684.
[12] WU V W, THIEME N, HUBERMAN L B, et al .. The regulatory and transcriptional landscape associated with carbon utilization in a filamentous fungus [J]. Proc. Natl. Acad. Sci. USA, 2020, 117(11):6003-6013.
[13] DENG H, BAI Y, FAN T P, et al .. Advanced strategy for metabolite exploration in filamentous fungi [J]. Crit. Rev.Biotechnol., 2020, 40(2):180-198.
[14] ADNAN M, ZHENG W, ISLAM W, et al .. Carbon catabolite repression in filamentous fungi [J/OL]. Int. J. Mol. Sci., 2017,19(1):48 [2023-02-10]. https://doi.org/ 10.3390/ijms19010048.
[15] TAMAYO E N, VILLANUEVA A, HASPER A A, et al .. CreA mediates repression of the regulatory gene xlnR which controls the production of xylanolytic enzymes in Aspergillus nidulans[J]. Fungal Genet. Biol., 2008, 45(6):984-993.
[16] ANTONIETO A C, DOS SANTOS CASTRO L, SILVAROCHAR, et al .. Defining the genome-wide role of CRE1 during carbon catabolite repression in Trichoderma reesei using RNA-Seq analysis [J]. Fungal Genet. Biol., 2014, 73:93-103.
[17] PORTNOY T, MARGEOT A, LINKE R, et al .. The CRE1 carbon catabolite repressor of the fungus Trichoderma reesei: a master regulator of carbon assimilation [J/OL]. BMC Genomics,2011, 12:269 [2023-02-10]. https://doi.org/10.1186/1471-2164-12-269.
[18] FURUKAWA T, SHIDA Y, KITAGAMI N, et al .. Identification of specific binding sites for XYR1, a transcriptional activator of cellulolytic and xylanolytic genes in Trichoderma reesei [J].Fungal Genet. Biol., 2009, 46(8):564-574.
[19] PORTNOY T, MARGEOT A, SEIDL-SEIBOTH V, et al .. Differential regulation of the cellulase transcription factors XYR1, ACE2, and ACE1 in Trichoderma reesei strains producing high and low levels of cellulase [J]. Eukaryot. Cell,2011, 10(2):262-271.
[20] DOS SANTOS GOMES A C, FALKOSKI D, BATTAGLIA E, et al .. Myceliophthora thermophila Xyr1 is predominantly involved in xylan degradation and xylose catabolism [J/OL].Biotechnol. Biofuels, 2019, 12: 220 [2023-02-10]. https://doi.org/10.1186/s13068-019-1556-y.
[21] CRAIG J P, CORADETTI S T, STARR T L, et al .. Direct target network of the Neurospora crassa plant cell wall deconstruction regulators CLR-1, CLR-2, and XLR-1 [J/OL]. Mbio, 2015, 6(5):e01452-15 [2023-02-10]. https://doi.org/10.1128/mBio.01452-15.
[22] HASPER A A, TRINDADE L M, VAN DER VEEN D, et al .. Functional analysis of the transcriptional activator XlnR from Aspergillus niger [J]. Microbiology, 2004, 150(Pt5):1367-1375.
[23] HASPER A A, VISSER J, DE GRAAFF L H. The Aspergillus niger transcriptional activator XlnR, which is involved in the degradation of the polysaccharides xylan and cellulose, also regulates d-xylose reductase gene expression [J]. Mol. Microbiol.,2000, 36(1):193-200.
[24] MACH-AIGNER R, PUCHER E, STEIGER G, et al.. Transcriptional regulation of xyr1, encoding the main regulator of the xylanolytic and cellulolytic enzyme system in Hypocrea jecorina [J]. Appl. Environ. Microbiol., 2008,74(21):6554-6562.
[25] MACH-AIGNER A R, OMONY J, JOVANOVIC B, et al ..D-Xylose concentration-dependent hydrolase expression profiles and the function of CreA and XlnR in Aspergillus niger [J]. Appl. Environ. Microbiol., 2012, 78(9):3145-3155.
[26] BERKA R M, GRIGORIEV I V, OTILLAR R, et al ..Comparative genomic analysis of the thermophilic biomassdegrading fungi Myceliophthora thermophila and Thielaviaterrestris [J]. Nat. Biotechnol., 2011, 29(10):922-927.
[27] XU J, LI J, LIN L, et al .. Development of genetic tools for Myceliophthora thermophila [J/OL]. BMC Biotechnol., 2015, 15:35 [2023-02-10]. https://doi.org/10.1186/s12896-015-0165-5.
[28] SONG R, ZHAI Q, SUN L, et al .. CRISPR/Cas9 genome editing technology in filamentous fungi: progress and perspective [J].Appl. Microbiol. Biotechnol., 2019, 103(17):6919-6932.
[29] SWIAT M A, DASHKO S, DEN RIDDER M, et al .. FnCpf1:a novel and efficient genome editing tool for Saccharomyces cerevisiae [J]. Nucleic Acids Res., 2017, 45(21):12585-12598.
[30] PATEL H, RAWAT S. Thermophilic fungi: diversity, physiology, genetics, and applications [J]. New Future Dev.Microb. Biotechnol. Bioeng., 2021, 13(6):69-93.
[31] LIU Q, GAO R, LI J, et al .. Development of a genome-editing CRISPR/Cas9 system in thermophilic fungal Myceliophthora species and its application to hyper-cellulase production strain engineering [J/OL]. Biotechnol. Biofuels, 2017, 10:1 [2023-02-10]. https://doi.org/ 10.1186/s13068-016-0693-9.
[32] LI J, GU S, ZHAO Z, et al .. Dissecting cellobiose metabolic pathway and its application in biorefinery through consolidated bioprocessing in Myceliophthora thermophila [J/OL]. Fungal Biol. Biotechnol., 2019, 6: 21 [2023-02-10]. https://doi.org/10.1186/s40694-019-0083-8.
[33] XU Q, SINGH A, HIMMEL M E. Perspectives and new directions for the production of bioethanol using consolidated bioprocessing of lignocellulose [J]. Curr. Opin. Biotechnol.,2009, 20(3):364-371.
[34] WANG B, LI J, GAO J, et al .. Identification and characterization of the glucose dual-affinity transport system in Neurospora crassa: pleiotropic roles in nutrient transport, signaling, and carbon catabolite repression [J/OL]. Biotechnol.Biofuels, 2017, 10: 17 [2023-02-10]. https://doi. org/10.1186/s13068-017-0705-4.
[35] SUN J, TIAN C, DIAMOND S, et al .. Deciphering transcriptional regulatory mechanisms associated with hemicellulose degradation in Neurospora crassa [J]. Eukaryot.Cell, 2012, 11(4):482-493.
[36] SUN J, GLASS N L. Identification of the CRE-1 cellulolytic regulon in Neurospora crassa [J/OL]. PLoS One, 2011, 6(9):e25654 [2023-02-10]. https://doi.org/10.1371/journal.pone.0025654.
[37] FURUKAWA T, SHIDA Y, KITAGAMI N, et al .. Identification of specific binding sites for XYR1, a transcriptional activator of cellulolytic and xylanolytic genes in Trichoderma reesei [J].Fungal Genet. Biol., 2009, 46(8):564-574.
[38] LI J, LIN L, SUN T, et al .. Direct production of commodity chemicals from lignocellulose using Myceliophthora thermophila [J].Metab. Eng., 2020, 61:416-426.
[39] WEINHANDL K, WINKLER M, GLIEDER A, et al .. Carbon source dependent promoters in yeasts [J/OL]. Microb. Cell Fact., 2014, 13: 5[2023-02-10]. https://doi.org/10.1186/1475-2859-13-5.

