海洋鏈霉菌Streptomycessp.SCSIO 1672及其代謝產物水楊酸的分離鑒定
羅明和,汪中文,黃洪波,朱清華,田新朋,白志川,張 偲,張長生,鞠建華*
(1.西南大學園藝園林學院,重慶 400700;2.中國科學院海洋生物資源可持續利用重點實驗室,廣東廣州 510301; 3.廣東省海洋藥物重點實驗室,廣東廣州 510301;4.中國科學院南海海洋研究所,廣東廣州 510301)
海洋鏈霉菌Streptomycessp.SCSIO 1672及其代謝產物水楊酸的分離鑒定
羅明和1,汪中文2,3,4,黃洪波2,3,4,朱清華2,3,4,田新朋2,3,4,白志川1,張 偲2,3,4,張長生2,3,4,鞠建華2,3,4*
(1.西南大學園藝園林學院,重慶 400700;2.中國科學院海洋生物資源可持續利用重點實驗室,廣東廣州 510301; 3.廣東省海洋藥物重點實驗室,廣東廣州 510301;4.中國科學院南海海洋研究所,廣東廣州 510301)
從南海海洋沉積物中分離得到1株海洋放線菌,鑒定為鏈霉菌Streptom ycessp.SCSI O 1672。通過優化發酵條件,采用海蝦生物致死活性和高效液相色譜追蹤,利用有機溶劑萃取、正相硅膠、反相硅膠等各種色譜層析方法分離出活性化合物,通過波譜數據解析出海洋放線菌SCSI O 1672次級代謝產物中的該活性化合物為水楊酸。
海洋放線菌;鏈霉菌;水楊酸
微生物中的放線菌是藥物的重要來源之一。目前在已發現的23000個微生物活性次級代謝產物中有超過10000個來源于放線菌,占了近45%。這些活性物質具有抗腫瘤、細胞毒活性、抗感染及免疫抑制等廣泛的生理活性[1]。經過近幾十年的研究開發,人們從陸生放線菌中發現新抗生素的機率大大降低[2],于是把探索的目光轉向海洋。海洋環境具有低溫、高壓、高鹽等特點,這為海洋放線菌發展獨特的代謝途徑和機體防御機制并產生結構新穎、活性獨特的代謝產物提供了條件。近10 a來,人們從海洋放線菌中發現了大量結構新穎的活性物質。例如,Fenical研究小組從Salinispora tropicCNB-392分離得到的salinosporamide A具有很好的選擇性細胞毒活性[3]; Fernández-Chimeno等[4]從M icrom onosporasp.L-25-ES25-008中分離出的大環內酯類化合物I B-96212對小鼠淋巴性白血病細胞P-388有極強抑制活性(IC50=0.0001 mg/L),對腫瘤細胞系A-549、HT-29及MEL-28有明顯的細胞毒活性(I C50=1 mg/L);Riedlinger等[5]從Verrucosisporasp. AB-18-032中分離得到了一類多環聚酮類抗生素abyssomicin C,可強烈抑制革蘭陽性菌的生長。本文對從中國南海海洋沉積物中分離純化的240株海洋放線菌發酵產物進行海蝦致死活性篩選,發現編號為SCSI O 1672的海洋放線菌發酵粗提物有海蝦致死活性。對該菌株的16S r DNA進行分析,鑒定為鏈霉菌屬(Streptom yces)。采用海蝦毒性為指標的活性追蹤方法,對SCSI O 1672的活性次級代謝產物進行了研究,從中分離得到1個化合物—水楊酸,并對該菌株產水楊酸的發酵培養基進行了初步篩選優化。產水楊酸的鏈霉菌尚未見文獻報道,本文報道該菌株的16S rDNA序列分析,發酵產物中水楊酸的分離鑒定及產水楊酸的培養基的初步篩選。
1 材料與方法
1.1 材料
1.1.1 菌種 海洋放線菌SCSI O 1672分離于中國南海北部沉積物,菌種保存于中國科學院南海海洋研究所。
1.1.2 培養基 實驗采用培養基JNP1(可溶性淀粉5 g/L,魚粉2 g/L,海藻糖2 g/L,幾丁質2 g/ L,粗海鹽30 g/L)、JNP2(可溶性淀粉10 g/L,酵母膏4 g/L,蛋白胨2 g/L,粗海鹽30 g/L)和經優化的培養基AM2a(可溶性淀粉20 g/L,大豆粉10 g/L,蛋白胨2 g/L,酵母膏5 g/L,粗海鹽30 g/ L)、AM2ab(可溶性淀粉20 g/L,大豆粉10 g/L,蛋白胨2 g/L,酵母膏5 g/L,CaCO32 g/L,NaCl 4 g/L,粗海鹽30 g/L)。培養基配制時調pH 7.0~7.2,121℃滅菌20 min。
1.2 方法
1.2.1 發酵 發酵培養基的優化:將放線菌SCSI O 1672接種于裝有50 mL不同發酵培養基的250 mL三角瓶中,28℃200 r/min培養8 d。放大培養發酵;種子培養:將放線菌SCSI O 1672接種于裝有50 mL AM2a培養基的250 mL三角瓶中,28℃200 r/min培養50 h;發酵:按10%的接種量將種子接入裝有200 mL AM2ab培養基的1000 mL三角瓶中,28℃200 r/min培養7 d。
1.2.2 HPLC分析 50 mL發酵產物加80 mL丁酮,攪拌萃取2 h,靜置,取上層丁酮減壓蒸餾至干,加DMSO配成20 mg/mL供生物活性測試(HPLC分析時,50 mL發酵物提取物溶解于1 mL甲醇,12000 r/min離心2次)。色譜條件:柱溫:室溫;洗脫劑:A相為乙腈∶水∶冰醋酸(15∶85∶0.1),B相為乙腈∶水∶冰醋酸(80∶20∶0.1),利用程序設定梯度,0~20 min內A相由100%變為0,20~24 min為100%B相沖洗,流速:1.0 mL/min;檢測波長:210 nm,254 nm。取甲醇溶解的發酵產物各20μL進行HPLC(Varian ProStar高效液相色譜儀,配PDA檢測器)分析,根據HPLC的吸收峰,收集不同吸收峰的化合物,旋轉蒸干后用5μL DMSO溶解。檢測不同吸收峰化合物的海蝦致死活性,以確定活性峰的tR值及活性峰的峰面積。
1.2.3 活性測試 海蝦卵的孵化:取海蝦卵100 mg置于500 mL燒杯中,加入含3%海鹽的人工海水,28℃光照、通氣培養24 h;海蝦致死活性測試:取96孔細胞培養板,每孔加200μL含15個海蝦幼蟲的人工海水液。空白對照組加5μL DMSO,樣品組加5μL樣品液。28℃光照培養24 h,在雙目解剖鏡下計數海蝦死亡個體數目,并計算死亡率。
1.2.4 化合物的分離分析 將發酵產物用3500 r/min離心10 min,取上清液用2倍體積的丁酮萃取3次,萃取液經減壓濃縮,所得浸膏拌樣上100~200目正相硅膠柱,用氯仿-甲醇梯度洗脫(10∶0~0∶10),所接流分經HPLC分析,取氯仿∶甲醇為9∶1時的流分,上反相硅膠柱,用水-甲醇梯度洗脫(10∶0~0∶10),流分經HPLC檢驗含活性峰的流分再經Sephadex-LH20凝膠柱層析,用甲醇洗脫得化合物1。化合物1經Bruker DRX2500核磁共振儀(500/125 MHz,T MS為內標)和LCQ DECA XP液質聯用儀分析,獲得化合物1的氫譜、碳譜和分子量。
1.2.5 放線菌菌株SCSI O 1672的16S rDNA系統發育分析 用細菌16S r DNA序列的通用引物,27F primer(5′-AGAGTTTGATC(AC)TGGCTCAG-3′)和1492R primer(5′-ACGG(CT)TACCTTGTTACGACTT-3′),進行PCR擴增。PCR反應體系(20μL):DNA模板0.1μL,10×PCR Buffer 2.0μL,dNTPs(各2.5 mmol/L)1.6μL,27F primer(20μmol/L)0.4μL,1492R pri mer(20 μmol/L)0.4μL,Mg2+(25 mmol/L)1.2μL,Taq酶(5 U/μL)0.2μL,DMSO 1.0μL,ddH2O 13.1 μL。PCR反應條件:94℃預變性4 min,94℃變性1 min,57℃退火1 min,72℃延伸1.5 min;30個循環;72℃延伸10 min;4℃保溫。用凝膠回收試劑盒(Axygen)純化PCR產物,再連接到pGEMT載體(Promega)中,16S r DNA序列由上海英俊生物技術有限公司測定。根據測序結果,利用
BLAST應用軟件搜索GenBank、EMBL等數據庫
中相關菌株的16S rDNA序列(http://blast.ncbi. nlm.nih.gov/)。利用CLUSTAL X[6]進行多系列比較分析,并使用MEGA4[7]軟件構建系統發育樹。
2 結 果
2.1 不同培養基發酵產物的產量及活性
20μL不同培養基的發酵產物經HPLC分離后,檢測收集的不同吸收峰的海蝦毒性,結果表明AM2a、AM2ab培養基的發酵產物tR=10 min的峰活性最好,24 h海蝦致死率80%。AM2a、AM2ab 2種培養基發酵產物tR=10 min的峰面積較大,說明AM2a、AM2ab 2種培養基的發酵產物中tR= 10 min的化合物產率比JNP1、JNP2培養基的發酵產物的產率高,見圖1。
2.2 結構鑒定
化合物1:UVλmax205,298 nm;ESI-MS:m/z 137(M-H)-,分子量為138.0,結合氫譜、碳譜可以推測其分子式為C7H6O3。1H NMR(500 MHz, DMSO-d6):δH7.77(1H,d,J=7.5 Hz),δH7.45 (1H,m),δH6.89(1H,m),δH6.86(1H,d,J= 7.5 Hz);13C NMR(125 MHz,DMSO-d6):δC171.8(C-1),δC161.3(C-3),δC134.9(C-5),δC130.7(C-7),δC118.6(C-6),δC116.7(C-4),δC114.1(C-2)。化合物的氫譜顯示芳環1,2-取代的4個芳香質子信號峰;碳譜顯示1個共軛羧基信號峰(δ171.8)、1個連氧芳環碳信號峰(δ 161.3)和另外5個芳環碳信號,結合質譜給出的分子量,分析其結構為水楊酸,見圖2。

圖1 不同培養基發酵產物的水楊酸含量的HPLC分析Fig.1 HPLC analyses of the fermentation products under four culture conditions
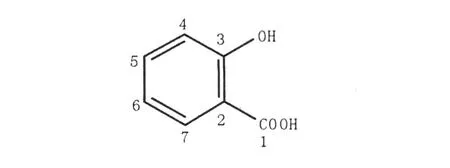
圖2 化合物1的結構Fig.2 The chemical structure of compound 1
2.3 放線菌菌株SCSI O 1672的16S rDNA系統發育分析
16S rDNA序列分析表明海洋放線菌SCSI O 1672菌株與鏈霉菌S.daliensisYI M 31724T和S. rangoonensisLMG 20295T的16S rDNA序列相似性較高,分別為98.3%和97.5%,基于16S rDNA序列的系統發育分析表明海洋放線菌SCSI O 1672菌株與鏈霉菌S.rangoonensisLMG 20295T為同一分支,支持率為77%(圖3),故鑒定為鏈霉菌Streptom ycessp.SCSI O 1672。

圖3 基于16S rDNA序列NJ系統進化樹Fig.3 Neighbour-joining tree based on nearly complete 16S rDNA sequences
3 討 論
水楊酸是生產醫藥、香料、染料等的重要化工原料。自1943年Strawinski和Stone[9]首先從微生物氧化萘的培養液中分離得到了水楊酸以來,陸續有微生物產水楊酸的報道。比如法幼華等[10]得到了2株產水楊酸較穩定的菌株,鑒定為Pseudom onasovalisAS1.593和Pseudom onas aeruginosaAS1.860;王樹青[11]用熒光極毛桿菌(Pseudom onasfluofrscens)和腎炎棒狀桿菌(Corynebacterium renals)微生物發酵制取的水楊酸濃度可達20 g/L;Ratledge和W inder[12-13]發現分枝桿菌(M ycobacterium sm egm atis)在缺鐵的培養基中能產水楊酸,并對各種離子對水楊酸產量的影響進行了研究;另外Shanmugam和Narayanasamy[14]報道在蘚樣芽胞桿菌中也產水楊酸; Indiragandhi等[15]發現Acinetobactersp.、Pseudom onassp.和Serratiasp.產水楊酸;Press等[16]報道粘質沙雷菌也產水楊酸;Ratledge和Chaudhry[17]發現Ther m oactinom yces vulgaris菌的發酵液體能分離得到水楊酸。盡管如此,目前還沒有發現從鏈霉菌中分離得到水楊酸的報道。本研究從海洋鏈霉菌Streptom ycessp.SCSI O 1672的發酵產物中分離得到了水楊酸,豐富了水楊酸的生產菌株庫,對該菌株在不同的發酵培養基中水楊酸的產量進行了初步比較研究,發現用AM2a、AM2ab 2種培養基發酵,水楊酸的產量相對較高。
[1]Berdy J.Bioactive microbial metabolites[J].J Antibiot,2005, 58:1-26.
[2]Bull AT,Ward AC,Goodfellow M.Search and discovery strategies for biotechnology:the paradigm shift[J].Microbiol Mol Biol Rev,2000,64(3):573-606.
[3]Feling RH,Buchanan GO,Mincer TJ,et al.Salinosporamide A: a highly cytotoxic proteasome inhibitor from a novel microbial source,a marine bacterium of the new genus Salinospora[J]. Angew Chem Int Ed,2003,42(3):355-357.
[4]Fernández-Chimeno R I,Canedo L,Espliego F,et al.I B-96212, a novel cytotoxic macrolide produced by a marine Micromonospora.I.taxonomy,fermentation,isolation and biological activities[J].J Antibiot,2000,53(5):474-479.
[5]Riedlinger J,Reicke A,Zāhner H,et al.Abyssomicins,inhibitors of the para-aminobenzoic acid pathwayproduced by the marine Verrucosispora Strain AB-18-032[J].J Antibiot,2004,57 (4):271-279.
[6]Thompson JD,Gibson TJ,Plewniak F,et al.The Clustal X windows interface:flexiblestrategies for multiplesequence alignment aided by quality analysis tools[J].Nucleic Acids Res,1997,25(24):4876-4882.
[7]Tamura K,Dudley J,Nei M,et al.MEGA4:Molecular evolutionary genetics analysis(MEGA)software version 4.0[J]. Mol Biol Evol,2007,24(8):1596-1599.
[8]Saitou N,Nei M.The neighbor-joining method:A new method for reconstructing phylogenetic trees[J].Mol Biol Evol,1987,4 (4):406-425.
[9]Strawinski RJ,Stone RW.Conditions governing the oxidation of naphthalene and the chemical analysis of its products[J].J Bacteriol,1943,45:16.
[10]法幼華,徐詩偉,梁家騵,等.微生物氧化萘制取水楊酸[J].微生物學報,1974,14(1):113-111.
[11]王樹青.微生物發酵法制取水楊酸[J].科技通報,1988,4 (2):34-36.
[12]Ratledge C,W inder FG.The accumulation of salicylic acid by mycobacteria suring growth on an iron-deficient medium[J]. Biochem J,1962,84:501-506.
[13]Ratledge C,Hall MJ.Influence of metal ions on the formation of mycobactin and salicylic acid in mycobacterium smegmatis grown in static culture[J].J Bacteriol,1971,108(1):314-319.
[14]Shanmugam P,Narayanasamy M.Optimization and production of salicylic acid by rhizobacterial strain Bacilluslichenifor m is MML2501[J].Int J Microbiol,2009,6(1).
[15]Indiragandhi P,Anandham R,Madhaiyan M.Characterization of plant growth-promoting traits of bacteria isolated from larval guts of diamondback moth Plutella xylostella(Lepidoptera:Plutellidae)[J].Curr Microbiol,2008,56:327-333.
[16]Press CM,Wilson M,Tuzun S,et al.Salicylic acid produced by Serratia marcescens90-166 is not the primary determinant of induced systemic resistance in cucumber or tobacco[J].Mol Plant Microbe Interact,1997,10(6):761-768.
[17]Ratledge C,Chaudhry MA.Accumulation of iron-binding phenolic acids by actinomycetales and other organisms related to the mycobacteria[J].J Gen Microbiol,1971,66:71-78.
Isolation and Identification of a Marine Stra in
Streptomyces sp.SCSIO 1672 and Its Metabolite Salicylic Acid
LUO Ming-he1,WANG Zhong-wen2,3,4,HUANG Hong-bo2,3,4,ZHU Qing-hua2,3,4, TIAN Xin-peng2,3,4,BA I Zhi-chuan1,ZHANG Si2,3,4,ZHANG Chang-sheng2,3,4,JU Jian-hua2,3,4
(1.Coll.of Horticult.&Landscape Archit.,SW Uni.,Chongqing400700; 2.Key Lab.of Marine Bio-Res.Sustainable Util.,Guangzhou510301;3.Guangdong Key Lab.of Marine Materia Medica,Guangzhou510301; 4.Ctr.for Marine Microorg.,South China Sea Inst.of Oceanology,Chinese Acad.of Sci.,Guangzhou510301)
A marine actinomycetes strain was isolated and identified as Streptomyces sp.1672 by 16S rDNA sequence analysis.Its fer mentation condition was optimized and its bioactive compound was tested by means of HPLC analysis and bioassay-guided fractionation using brine shrimp toxicity test,and was isolated by solvent extraction,normal and reverse-phase silica gel and other chromatographies.The structure of the bioactive compound was identified as salicylic acid by spectral data analyses.
marine actinomycetes;Streptomyces sp.;salicylic acid
Q939
A
1005-7021(2010)06-0022-05
中國科學院知識創新工程重要方向項目(KSCX2-Y W-G-065,KZCX2-Y W-JC202,KSCX2-Y W-G-073, KZCX2-EZCX2-EWC-G-12);中國科學院南海海洋所領域前沿項目(LYQY200805)
羅明和 男,碩士研究生。主要從事海洋微生物及其活性次級代謝產物研究。
*通訊作者。Tel:020-89023028,E-mail:jju@scsio.ac.cn
2010-08-05;
2010-11-05

