口蹄疫病毒非結構蛋白3Dpol的研究進展*
孫靜靜,邵軍軍,常惠蕓
口蹄疫(foot-and-mouth disease,FMD)是由口蹄疫病毒(Foot-and-mouth disease virus,FMDV)引起的偶蹄動物的一種急性、熱性、高度接觸性傳染病。FMD是OIE規定的A類動物疫病之一,嚴重影響畜牧業的發展,對世界公共衛生存在著巨大影響,造成巨大的經濟損失,素有“政治經濟病”之稱。FMD的致病原是小RNA病毒科,口蹄疫病毒屬成員,于1897年首次確定。FMDV是正鏈RNA分子,長約8 500bp,包裹于由60個拷貝的4種病毒結構蛋白VP1-4構成的二十面體的衣殼中。FMDV不僅宿主廣泛,傳播速度快,變異性強,而且血清型多,有7個血清型O、A、C、Asia 1、SAT 1、SAT 2、SAT 3和70多個亞型,且各血清型間無交叉免疫,同一血清型內的不同亞型或分離株發生交叉免疫的程度均不相同,這種特性給口蹄疫的診斷和疫苗免疫帶來極大的困難。
FMDV的3Dpol蛋白是RNA依賴的RNA聚合酶(RdRp),沒有型特異性,在FMDV所有非結構蛋白中,3Dpol抗原性最好,ELISA檢測方法的特異性、準確性和敏感性與檢測結構蛋白的ELISA方法相當的,是區分是否接觸過FMDV動物的重要指標。FMDV的復制也是由病毒編碼的聚合酶3Dpol催化,但3Dpol蛋白酶的保真性很差,導致病毒復制的錯誤傾向率很高,每個核苷酸復制的錯誤配對率為10-3~10-5,這種低的復制保真性導致了后代快速突變,因此口蹄疫的防控比較困難。3Dpol是非結構蛋白,只是在病毒的復制過程中出現,病毒粒子成熟后僅僅有非常少量的3Dpol。本文綜述了口蹄疫病毒基因組結構及3D基因,3Dpol蛋白的表位、結構、功能的國內外最新研究進展。
1 FMDV基因組結構
FMDV基因組為單股正鏈RNA,既是mRNA,又是負鏈RNA的模板,約有8 500個核苷酸(nts)組成,基因組的基本結構是:VPg-5′UTR(S-polyCIRES)-(L 蛋 白 )-結 構 蛋 白 P1(VP4-VP2-VP3-VP1)-非 結 構 蛋 白 (2A-2B-2C-3A-3B-3B-3B-3C-3D)-3′UTR-poly(A)。
VPg在病毒RNA開始復制時起重要作用,同時參與病毒裝配,只有病毒RNA與VPg結合后,才能夠被裝入病毒。5′-UTR約含1 300個堿基。S片段由360個堿基組成,可形成一個長莖環結構(stem-loop)。S片斷下游是約400bp的 Poly(C)序列。Poly(C)片段幾乎全部由胞嘧啶組成,約占90%,僅有少量的U與A。IRES長約1 350bp,控制不依賴于帽子結構的FMDV RNA的翻譯起始。下游3′-UTR序列可自身折疊成莖環結構,對病毒基因組的復制有重要作用,復制過程中3′-UTR可以結合病毒復制相關蛋白。3′-UTR下游是長度不一的poly(A)結構,Poly(A)尾與病毒的感染性有關,Poly(A)越長感染性越強。基因組的中部是一大的開放閱讀框(ORF),編碼一多聚蛋白。基因組的ORF分為4個區域,分別為L區、P1區、P2區、P3區。多聚蛋白的裂解過程是由三種蛋白酶:Lpro,2A和3C來完成的,多聚蛋白經3級裂解后,形成3~4種病毒結構蛋白(VP0或 VP4、VP2,VP3,VP1)和8~9種非結構蛋白(Lab、Lb、2A、2B、2C、3A、3B、3C和3D)。ORF的5′末端為 L區,長約600bp,主要編碼N末端多聚蛋白Lab和Lb,兩種蛋白作為酶能在L/P1連接處進行自我切割釋放。L區的下游為P1區,P1區全長約2 200bp,編碼FMDV的4種結構蛋白VP4、VP2、VP3和VP1,分別由85、218、220個和213個氨基酸組成。P1區的下游為P2區,約1 460bp,編碼2A、2B和2C3種非結構蛋白,分別由16、154和318個氨基酸組成。P2區的下游為P3區,長約2 720bp,編碼約908個氨基酸的聚合蛋白,該聚合蛋白含有3A、3個3B、3C和3Dpol6種多肽。
2 3Dpol抗原表位的研究
FMDV的免疫保護與中和抗體水平和親和力有關,研究發現針對B細胞表位的合成肽免疫原性低,不能產生持久的保護水平的中和抗體。Collen等[2]的研究認為機體抗口蹄疫病毒是一種依賴T細胞的體液免疫應答反應,抗體的產生需要T細胞參與。迄今為止,除了在結構蛋白區發現部分T細胞表位外,越來越多的研究顯示,T細胞表位主要分布在FMDV非結構蛋白(3ABC和3Dpol)上。國內外有關3Dpol蛋白T細胞表位的研究主要有:Collen[3]等在研究FMDV相關T細胞表位時發現,FMDV的非結構蛋白3Dpol具有刺激病毒感染牛外周血淋巴細胞增殖的能力,由此推測3Dpol上存在潛在的T淋巴細胞表位。Blanco等[4]發現,T細胞輔助表位區與B細胞表位區的串聯可以協助B細胞產生特異性抗體,提高優勢抗原表位區的免疫原性。García-Briones等[5]用 表 達 FMDV 非 結 構 蛋 白3Dpol的痘苗病毒免疫豬,即使在豬體內檢測不到中和抗體的情況下也能得到部分保護,這些豬在受到FMDV攻擊時出現典型臨床癥狀緩慢,結果與上述Collen等的推測基本一致。另外利用針對3Dpol氨基酸序列人工化學合成的重疊肽段對FMDV感染豬的外周血淋巴細胞進行刺激,結果發現很多肽段都能夠有效刺激淋巴細胞增殖,而且這些淋巴細胞能夠大量分泌IFN-γ等Th1類細胞因子。這一方面在一定程度上解釋了3Dpol蛋白能夠在無中和抗體條件下給動物提供部分保護的現象;另一方面也使人們進一步了解到3Dpol蛋白用于新型疫苗研究的潛在價值。WilhelmGerner等[6]在c/c和d/d單體型微型豬內發現了口蹄疫3Dpol非結構蛋白上被SLA識別的特異的T細胞表位,位于346-370位氨基酸,這為口蹄疫新型疫苗的設計提供了有用的信息。不同種屬宿主和不同個體的T細胞對T細胞表位的識別是受到機體MHC分子多態性的限制的,3Dpol蛋白作為不同血清型及不同毒株之間相對保守的功能蛋白,極可能存在保守的病毒T細胞表位,能夠被不同的宿主及個體T細胞所識別。
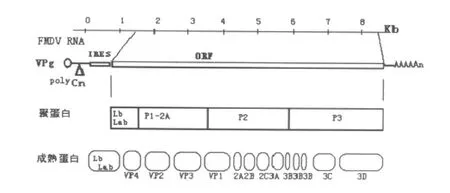
圖1 口蹄疫病毒基因組結構和成熟蛋白[1]Fig.1 FMDV genome structure and matured protein[1]
3 3Dpol的表達時相及結構
3Dpol蛋白在病毒復制的早期產生。García-Briones等[7]研究了 FMDV 在 BHK-21細胞中的分布,免疫熒光試驗發現在感染后1.5h首先檢測到非結構蛋白3Dpol,之后2~2.5h才檢測到其他的非結構蛋白2B、2C、3B、3C,他們均以間歇分散的模式出現,3A和3Dpol集中在核的一側。經共聚焦顯微鏡發現,雖然3Dpol短暫表達,但其在細胞核包括核仁中分布均勻,這與轉染3AB的細胞一致。在感染細胞中還發現3Dpol和其前體3CD,表明在FMDV感染時,這些蛋白可以同細胞核直接作用。
FMDV 3Dpol約有470個氨基酸殘基,形成典型的右手結構,與其它的聚合酶的三維結構一樣,有典型的手指、手掌和拇指亞結構。在 NH2末端(1-96位氨基酸)連接手指和拇指結構亞域的片段包圍著酶的活性位點,這個區域又稱為指套,與其他屬病毒的聚合酶相比,這是FMDV 3Dpol特有的。手指和手掌亞結構域包含6個不同的模體:A、B、C、D、E和F。手指亞結構域(97-207位和249-302位)包含8個α螺旋 (α3-α9)和7個β折疊 (β3-β7和β9-β10);手掌亞結構域 (230-248位和303-403位)包含2個螺旋(α11,α12),5個β折疊片(β8,β11,β12,β14,β15),有3個β折疊片(β8,β11,β12)位于α11 和α12之間,這段區域是所有聚合酶中最保守的區域[8],由5個保守模體A-E組成,在核苷酸結合、磷酰基轉移、手掌亞結構域的完整性和引導核苷酸結合方面發揮重要作用。拇指亞結構域(403-470位)包含4個α螺旋 (α13-α16)和2個卷曲[9]。
4 3Dpol的功能
FMDV 3Dpol首先以基因組正鏈為模板合成互補的負鏈RNA,然后再以負鏈為模板合成子代病毒的基因組的正鏈RNA。研究表明正鏈RNA病毒的復制有2種方式:引物依賴的RNA合成和引物非依賴的RNA合成即從頭合成。FMDV RNA合成是以VPg為引物的引物依賴的合成方式。之前研究已經闡明了PV、FMDV和HRVs的RdRps的三維結晶結構,要么是非結合蛋白,要么是形成引物-模板-NTP底物復合物[11-12]。正鏈和負鏈 RNA合成的第一步是形成VPg-pU(帶有一個UMP的VPg同酪氨酸Tyr3共價結合),之后發生尿苷化,緊接著進行引物延伸。CV-B3RdRp采用了一個典型的RdRp折疊方式,與其它聚合酶一樣,RdRp亞結構域中最保守的是起催化作用的拇指結構域,2個天冬氨酸和2個二價的金屬離子都是催化作用必須的[13]。FMDV 3Dpol與 VPg結合位點和 CV-B3 RdRp與VPg結合位點不同,在FMDV 3Dpol與VPg殘 基 1-GPYAGPLERQPRPLKV-15 結 合,但 是CVB3 3Dpol-VPg 復 雜 結 構 表 明,VPg 7-PNQKPRVPT-15位殘基與3Dpol拇指結構域的底部結合。其實早在研究PV RdRp時,通過一系列的遺傳學和生物學實驗已經證明了第2個結合位點的存在,并且通過結構驗證了其準確性[14-15]。3Dpol催化的FMDV RNA復制機制如下:
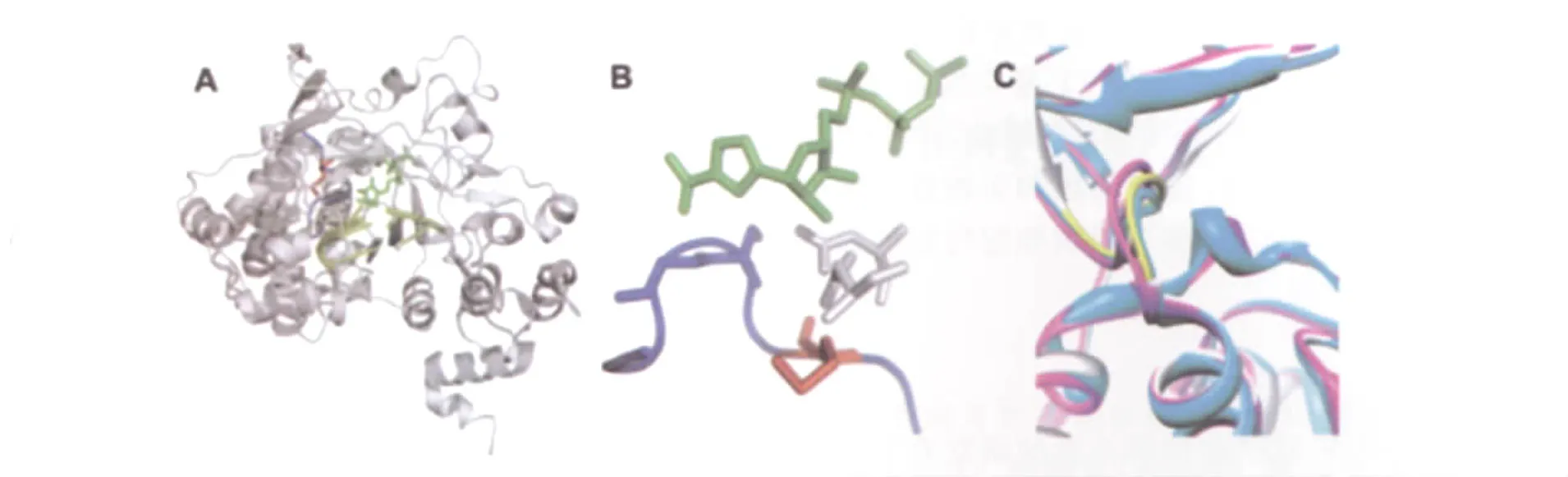
圖2 FMDV 3Dpol-RNA-RTP結晶結構[10]A:3Dpol與引物模板結合;B:3Dpol骨干;C:不同FMDV的3Dpol結構重疊Fig.2 FMDV 3Dpolcrystal structureA:3Dpol-template-pmimer molecular;B:3Dpol bone,C:superimposition of different FMDV 3Dpol
4.1 FMDV引物依賴性的復制起始 在RNA復制的起始,RdRp與病毒和宿主的許多蛋白相互作用。在小RNA病毒科中,3DPol使病毒編碼的引物蛋白VPg尿苷化,從而引發復制的起始[16]。3DPol和VPg 2個復合物的結構及其定點突變的效應分析,揭示了位于3DpolPol活性位點的殘基不僅參與與VPg結合,而且與RNA合成起始的VPg尿苷化反應密切相關[17]。
VPg采用幾乎不具二級結構的完全伸展的構相,結合在3DPol聚合酶的中心槽,VPg的N末端部分位于NTP通道附近,使突出的第3位Tyr殘基的側鏈深入活性位點區域,之后VPg滑行穿過大的RNA結合位點到達拇指結構域[18]。手指、手掌、拇指結構域的保守殘基對于VPg的結合穩定性有重要作用。在3DPol-VPg-UMP復合物中,Tyr3側鏈的-OH與UMP分子的α-磷酸共價結合,模體F帶正電的殘基參與尿苷化反應,使Tyr3和UMP穩定在一個合適的構象。模體A和C的天冬氨酸(Asp)及2個二價的陽離子也參與尿苷化反應,其機制與其它聚合酶催化的核苷酸酸轉移反應相似[19]。已有研究表明,在RNA復制起始和延伸過程中3Dpol聚合酶的結構不同。依據FMDV VPg的結構,復制起始時FMDV 3DPol模體B的保守殘基的307位天冬酰胺與尿苷化復合物的3′-OH作用,但 在 延 伸 過 程 中 是 2′-OH[20]。 氟 尿 苷 三 磷 酸(FUTP)是 VPg尿苷化的抑制劑,但不能抑制延伸。
PV蛋白酶和聚合酶前導蛋白3CD的結晶結構也完全支持FMDV的尿苷化模型,在3CD分子與VPg結合位點之間存在有大量接觸面,同樣FMDV 3Dpol-VPg復合物中3Dpol與VPg所形成的接觸界面,在病毒復制起始促進和調節VPg尿苷酰化方面發揮重要作用[21]。
4.2 FMDV 3DPol引發的引物模板識別和鏈的延伸 有序的核苷酸轉移反應是RNA延伸的基礎。核苷酸轉移反應是引物鏈的3′-OH親核進攻核苷三磷酸的α-磷酸,從而形成磷酸二酯鍵,釋放PPi。根據兩個金屬離子催化模型,金屬離子A會降低引物3′-OH的pKa,從而引發去質子化;金屬離子B確定進入的核苷酸的方向,在PPi從聚合酶上釋放之前將其質子化[22]。但最近的另一項研究表明,不同的聚合酶催化的核苷酸延伸過程中的突變分析和動力學分析,PPi的質子供體是位于酶活性位點的帶正電的氨基酸殘基[23]。
正鏈RNA病毒的聚合酶的保守氨基酸殘基位于結合槽內,是同核苷酸底物相互作用的優勢候選物,如模體A和C上的兩個天冬氨酸(第245位Asp和第338位Asp),還包模體B上的保守殘基第303位 Thr,第304位Ser和第307位 Asn[24]。在自然底物ATP和UTP存在時,或有誘變劑如FUTP和利巴韋林三磷酸(RTP)存在時,得到4種FMDV 3DPol的催化反應復合物,由此發現RNA延伸過程可以分為4步[25]:第1步,3DPol-RNA-RTP復合物表明抗病毒誘變劑占據聚合酶的活性位點,與發生磷酰基轉移反應的位點相同;第2步,3DPol-RNA-ATP復合物表明形成新的堿基對,RNA產物易位,PPi釋放;第3步,3DPol-RNA-ATP/UTP 復合物表明RNA產物易位,新的底物定位到活性位點附近;第4步,3DPol-RNA-FUTP復合物表明突變核苷酸類似物FUTP與新生RNA結合,釋放PPi。4種復合物的比較表明,位于模板和引物核苷酸的結合位置的聚合酶殘基,參與識別和定位新的參與延伸反應的核苷酸。
5 結 語
有關FMDV 3Dpol結構和功能的研究有了一定的進展,但仍有許多未知。FMDV 3Dpol是非常保守的非結構蛋白,在病毒的復制和轉錄過程中發揮重要作用,也是非常有效的抗病毒靶標。現在也發現針對結構蛋白和結構蛋白2C、3A、3C的抑制劑,相信隨著科技的發展,一定會發現更多的抗FMDV復合物,從而為有效的控制此嚴重危害畜牧業發展的傳染性疾病做出貢獻。
[1]謝慶閣.口蹄疫[M].北京:中國農業出版社,2004:21.
[2]Collen T,Pullen L,Doel TR.T cell-dependent induction of antibody against foot-and-mouth disease virus in a mouse model[J].Journal of General Virology,1989,70(2):395-403.
[3]Collen T,Baron J,Childerstone A,et al.Heterotypic recognition of recombinant FMDV proteins by bovine T-cells:the polymerase(P3Dpol)as an immunodominant T-cell immunogen[J].Virus Res,1998,56(2):125-133.
[4]Blanco E,Garcia-Briones M,Sanz-Parra A,et al.Identification of T-cellepitopes in nonstructural proteins of foot-and-mouth disease virus[J].J Virol,2001,75(7):3164-3174.
[5]Garcia-Briones MM,Blanco E,Chiva C,et al.Immunogenicity and T cell recognition in swine of foot-and-mouthdisease virus polymerase 3Dpol[J].Virolog,2004,322(2),264-275.
[6]Gerner W,Denyer MS,Takamatsu HH,et al.Identification of novel foot-and-mouth disease virus specific T-cell epitopes in c/c and d/d haplotype miniature swine[J].Virus Research,2006,121(2):223-228.
[7]Garc a-Briones M,Rosas MF,Gonz lez-Magaldi M,et al.Differential distribution of non-structural proteins of foot-and-mouth disease virus in BHK-21cells[J].Virology,2006,349(2):409-421.
[8]O'Reilly EK,Kao CC.Analysis of RNA-dependent RNA polymerase structure and function as guided by known polymerase structures and computer predictions of secondary structure[J].Virology,1998,252(2):287-303.
[9]Ferrer-Orta C,Arias A,Perez-Luque R,et al,Structure of Footand-Mouth Disease Virus RNA-dependent RNAPolymerase and Its Complex with a Template-Primer RNA[J].J Biol Chem,2004,279(45):47212-47221.
[10]Armando A,Jamie J,Macarena S,et al.Determinants of RNADependent RNA Polymerase(In)fidelity Revealed by Kinetic Analysis of the Polymerase Encoded by a Foot-and-Mouth Disease Virus Mutant with Reduced Sensitivity to Ribavirin[J].Journal Of Virology,2008,82(24):12346-12355.
[11]Ferrer-Orta C,Arias A,Agudo R,et al.The structure of a protein primer-polymerase complex in the initiation of genome replication[J].EMBO J,2006,25(4):880-888.
[12]Ferrer-Orta C,Arias A,Escarmis C,et al.A comparison of viral RNA-dependent RNA polymerases[J].Curr Opin Struct Biol,2006,16(1):27-34.
[13]Castro C,Smidansky ED,Arnold JJ,et al.Nucleic acid polymerases use a general acid for nucleotidyl transfer[J].Nat Struct Mol Biol,2009,16(2):212-218.
[14]Tellez AB,Crowder S,Spagnolo JF,et al.Nucleotide channel of RNA-dependent RNA polymerase used for intermolecular uridylylation of protein primer[J].J Mol Biol,2006,357(2):665-675.
[15]Gruez A,Selisko B,Roberts M,et al.The crystal structure of coxsackievirus B3RNA-dependent RNA polymerase in complex with its protein primer VPg confirms the existence of a second VPg binding site on Picornaviridae polymerases[J].J Virol,2008,82(19):9577-9590.
[16]Pathak HB,Oh HS,Goodfellow IG,et al.Picornavirus genome replication:roles of precursor proteins and rate-limiting steps in oriI-dependent VPg uridylylation[J].J Biol Chem,2008,283(45):30677-30688.
[17]Castro C,Smidansky E,Maksimchuk KR,et al.Two proton transfers in the transition state for nucleotidyl transfer catalyzed by RNA-and DNA-dependent RNA and DNA polymerases[J].Proc Natl Acad Sci,2007,104(11):4267-4272.
[18]Ferrer-Orta C,Arias A,Agudo R,et al.The structure of a protein primer-polymerase complex in the initiation of genome replication[J].EMBO J,2006,25(4):880-888.
[19]Steitz TA,Steitz JA.A general two-metal-ion mechanism for catalytic RNA [J].Proc Natl Acad Sci,1993,90(14):6498-6502.
[20]Korneeva VS,Cameron CE.Structure-function relationships of the viral RNA-dependent RNA polymerase:fidelity,replication speed,and initiation mechanism determined by a residue in the ribose-binding pocket[J].J Biol Chem,2007,282(22):16135-16145.
[21]Marcotte LL,Wass AB,Gohara DW,et al.Crystal structure of poliovirus 3CD protein:virally encoded protease and precursor to the RNA-dependent RNA polymerase[J].J Virol,2007,81(7):3583-3596.
[22]Castro C,Smidansky E,Maksimchuk KR,et al.Two proton transfers in the transition state for nucleotidyl transfer catalyzed by RNA-and DNA-dependent RNA and DNA polymerases[J].Proc Natl Acad Sci,2007,104(11):4267-4272.
[23]Castro C,Smidansky ED,Arnold JJ,et al.Nucleic acid polymerases use a general acid for nucleotidyl transfer[J].Nat Struct Mol Biol,2009,16(2):212-218.
[24]Hansen JL,Long AM,Schultz SC.Structure of the RNA-dependent RNA polymerase of poliovirus[J].Structure,1997,5(8):1109-1122.
[25]Ferrer-Orta C,Arias A,Perez-Luque R,et al.Sequential structures provide insights into the fidelity of RNA replication[J].Proc Natl Acad Sci,2007,104(22):9463-9468.

