Web的微生物信息資源平臺的構建
閻光甫 張利莉
微生物物種豐富、分布廣泛,是一類具有巨大開發潛力的生物資源和基因資源。因其長期以來有著重要的研究價值,世界各國對微生物資源的研究和開發競爭日益激烈。隨著對微生物資源研究不斷深入和規模的相繼擴大,出現了一系列與微生物相關的數據分析工具及網站[1],而安裝和維護這些生物信息軟件不但耗時,而且不宜普及和維護。微生物信息資源平臺的建立,旨在給微生物研究人員提供一個“一站式”的在線生物科研平臺,提高科研工作效率,加快微生物資源的相關研究。
1 材料與方法
1.1 平臺運行環境
本平臺屬于小型的科研網絡系統,初步建立在PC上,通過分配靜態IP接入校園網。采用Ubuntu Server 10.04的Linux作為服務器操作系統,Web服務由Apache 2.2.14提供,通過集成功能模塊實現具體分析功能。選擇B/S(Brower/Server,瀏覽器/服務器)結構作為其工作運行模式的結構,實現過程如圖1所示。
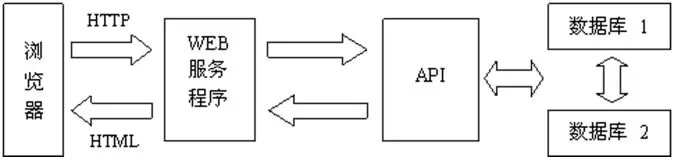
圖1 B/S結構的實現過程
本平臺所需的所有軟件均為免費開源軟件,可以從互聯網免費下載,在開源協議下可免費、自由使用。相關軟件的功能及下載地址見表1。

表1 軟件的功能描述及下載地址
1.2 系統的安裝和服務的配置[2,3]
采用光盤引導安裝。系統安裝完畢后,在 /etc/apt/sources.list中更改速度最快的更新源進行系統升級。網絡數據庫的服務包括執行Web服務的A-pache Web Server并配置CGI腳本支持。除此之外,要給執行CGI的文件夾賦予相應讀寫和運行權限。為了方便管理,還需安裝 Webmin[4],并配置 ufw實現防火墻功能,使用sudo ufw allow對內網部分ip段開啟web服務端口。
安裝apache服務:sudo apt-get install apache2安裝ufw防火墻:sudo apt-get install ufw
對CGI腳本的支持需要在httpd.conf配置文件中相應目錄處添加下列代碼:
Options ExecCGI
AddHandler cgi-script cgi pl
1.3 WWW BLAST 的構建[5,6,7,8]
1.3.1 Web 界面 Blast的配置
WWWBLAST是NCBI開發的一套使用廣泛的獨立 BLAST(Basic Local Alignment Search Tool,基本局部相似性比對搜索工具)程序,核酸和蛋白質序列的相似性分析。包含在各種平臺下使用的版本。從表1給出的地址用wget下載并解壓至 /var/www/blast。
1.3.2 數據庫的格式化
從 NCBI的 ftp 站點 ftp://ftp.ncbi.nlm.nih.gov/blast/db/中下載所需要的數據庫。對于本地自建數據庫,先將fasta格式的序列寫入一個文件里面。使用formatdb命令對數據庫進行格式化,并在目錄下的配置文件blast.rc中添加格式化好的核酸序列數據庫名稱,同時在網頁文件的源代碼中添加數據庫的鏈接和相應的數據庫名稱,為BLAST程序添加數據庫。核酸序列的格式化命令如下:
formatdb-p F -i nucleotide_db_name-o T/F
-p表示所要格式化的數據庫的類型,F表示nucleotide。
1.4 Primer3 的構建[9]
在表1的Primer3地址中下載Primer3-core、primer3-web - htdpcs-0.3.0.tar.gz和 primer3 -web- cgi- bin -0.3.0.tar.gz,并別解壓在/var/www/primer3/htdocs、/var/www/primer3/cgi - bin和/var/www/primer3/cgi-bin/primer3-web-cgi-bin-0.3.0目錄中。運行前需要從 http://cpan.org下載并安裝 perl的 cgi.pm模塊。配置 httpd.conf和增加文件夾權限使primer3中cgi-bin目錄能夠執行CGI腳本。
2 結果與分析
2.1 微生物信息資源平臺的結構
網站總體設計方案需要充分考慮到本地微生物科研的需求及可實現性。按照本地微生物科研需求,本平臺主要提供基于BLAST的在線序列比對和基于Primer3的在線引物設計,并配合Webmin實現圖形化的操作、配置和維護。整個平臺功能組成的層次結構如圖2所示。
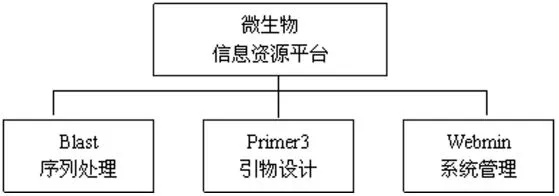
圖2 平臺的功能組成
微生物信息資源平臺的各種功能模塊通過Web服務集成在一個站點中,由發布的網站頁面鏈接集成,在瀏覽器地址欄中輸入站點ip訪問。平臺各模塊功能的實現均靠Web服務器的腳本程序,配合相互獨立的數據庫運行。
2.2 Blast在線序列比對
比較和確定某一特定的序列在某一給定的數據庫中部分或全部序列的相似性是生物信息學中最基本和最有價值的工作[10]。BLAST是一套應用廣泛的序列相似性比較的工具,基于啟發式的算法可以獲得序列匹配的近似最優解,因而BLAST的最大優勢在于運行速度快。由于本地Blast針對核酸序列,因此去掉蛋白質比對功能,模塊分為 BLASTN、TBLASTN、TBLASTX、MEGABLAST,其各部分的數據庫、檢索序列及功能注釋如表2所示。

表2 BLAST各種程序數據庫、檢測序列及其功能注釋
2.3 Primer3在線引物設計
Primer3由Whitehead Institute和Howard Hughes Medical Institute的Steve Rozen與Helen Skaletsky開發,是一款優秀的批量設計PCR引物、雜交探針、測序引物的工具,可通過設定各種標簽來指定引物設計參數,從而篩選 PCR目的引物,返回引物的相關信息。
進入Primer3引物設計的界面,在“Paste source sequence below(5'→3')”下面的文本框里面把模板序列5'→3'方向粘帖進去,不用考慮數字或者空格。通過重要參數設定,如“Product Size Ranges”、“Primer Size”和“Primer Tm”,點擊 Pick primers獲得設計的引物。
2.4 平臺的維護和安全性
Webmin能更好的實現以圖形化界面的形式對操作系統及各種服務進行配置和維護,從而簡化了通過命令符對服務器進行維護的過程。通過webmin對服務進行配置和定時備份,可提高系統的穩定性,同時簡化維護過程。
ufw是Ubuntu系統的一個簡易防火墻配置工具,底層調用 iptables處理,功能簡單實用。通過Ubuntu的ufw服務,可限制平臺對外開放的端口以及訪問該平臺的IP段,增加了系統的安全性。
3 結論與討論
使用Ubuntu Server的Linux系統建立網絡科研平臺有諸多優勢。首先,Linux系統本身具有的穩定性是諸如Windows等操作系統所無法超越的。另外,開源的軟件成本低,更新周期短。在開源環境下有豐富的開源軟件,其apt-get方式的軟件安裝和更新簡化了系統的維護和軟件的安裝過程,因而使用Ubuntu作為本科研平臺的操作系統無疑是一個很好的選擇。
在局域網構建Blast序列檢索對序列有較高的保密性,同時避免了網絡堵塞和延時,極大的縮短了序列比對操作所花費的時間。而微生物信息資源平臺中所用的Blast數據庫從NCBI中鏡像得到,這種數據庫的鏡像操作與數據庫的實時更新存在一定的延時。另外,通過構建具體的核酸序列數據庫或二次數據庫[11],消除公共數據庫中的冗余,可以進行有目的、更精準的比對,這樣會大大提高比對的目的性和結果的精確性。基于Web的Primer3在線引物設計免去了安裝應用程序的過程,直接可在線設計引物,并可結合本地的 BLAST功能對引物進行驗證。
總之,在擁有一定規模的科研單位中構建微生物信息資源平臺,對科研有著一定的支撐作用,簡化科研過程中對于數據的處理和分析。通過將該系統與本實驗室建立的微生物數據庫相關聯,可以更好的對微生物數據進行挖掘和探索。
[1] 陳潤生.生物信息學及其研究進展[J].醫學研究通訊,2002,31(12):1-5.
[2] 希爾.Ubuntu官方指南[M].北京:人民郵電出版社,2007:5-12.
[3] 李蔚澤.Ubuntu Linux入門到精通[M].北京:機械工業出版社,2007:19-52.
[4] 張旭華.用Webmin遠程管理Linux系統服務器[J].計算機與現代化,2006,(9):47-49.
[5] Altschul SF,Gish W,Miller W etc.Basic local alignment search tool[J].JMol Biol,1990,215(3):403 -410.
[6] Madden T L,Tatusov R L,Zhang J.Applications of network BLAST server[J].Methods Enzymol,1996,266:131-141.
[7] Gish W,States D J.Identification of protein coding regions by database similarity search[J].Nature Genet,1993,3(3):266-272.
[8] 張成崗,張利達,歐陽曙光等.序列同源性分析軟件Blast的WEB界面構建及其應用[J].生物化學與生物物理進展,2001,28(6):916-918.
[9] Rozen S,Skaletsky H.Primer3 on the WWW for general users and for biologist programmers[J].Methods in Molecular Biology,2000,132:365-386.
[10] 歐陽平.生物信息數據庫與序列分析[J].微生物學通報,2007,42(3):24-25.
[11] 潘雪峰.基于代理程序開發核酸序列二次數據庫[J].計算機與數字工程,2009,8(37):80-82.

