藤壺科DNA分類研究
原 帥 , 安建梅, 沙忠利
(1. 山西師范大學, 山西 臨汾 041004; 2. 中國科學院 海洋研究所, 山東 青島266071)
藤壺科DNA分類研究
原 帥1,2, 安建梅1, 沙忠利2
(1. 山西師范大學, 山西 臨汾 041004; 2. 中國科學院 海洋研究所, 山東 青島266071)
圍胸總目藤壺科的分類系統經歷了二亞科系統(小藤壺亞科 Chthamalinae,藤壺亞科 Balaninae)、三亞科系統[藤壺亞科(Balaninae), 巨藤壺亞科(Megabalaninae),凹藤壺亞科(Concavinae)], 現在采用的是四亞科系統[藤壺亞科(Balaniae)、紋藤壺亞科(Amphibalanus)、巨藤壺亞科(Megabalaninae)和凹藤壺亞科(Concavinae)], 但各亞科之間的系統演化關系尚未進行過分子系統學方面的研究。許多藤壺科物種存在趨同進化的趨勢, 致使傳統的形態分類存在困難, 不能正確地進行鑒別。本文測定了藤壺科 3個亞科里個代表種的線粒體COI,16S和12S基因的部分序列, 結合GenBank中藤壺科其他物種的12S,28S和18S等基因序列, 比較了不同基因片段作為鑒別藤壺科物種的條形碼的可行性和有效性, 并聯合16S和12S序列初步分析了藤壺科各亞科之間的一些親緣關系。研究結果表明:COI基因的種間和種內遺傳距離有明顯的間隔區,COI最小種間距離為0.122, 遠大于最大種內距離0.023, 而16S基因的種間與種內距離存在覆蓋, 最小種間距離為 0.018, 小于最大種內距離 0.023, 因此表明, 線粒體基因COI能更準確地鑒定藤壺科種間以及種內關系, 并得出閾值為種內差異小于0.023, 種間差異大于0.1。ML和BI系統發育分析結果基本一致, 支持4亞科的分類系統; 巨藤壺亞科形成明顯單系群, 支持率很高,而兩種紋藤壺和管藤壺聚成一支, 形成一個單系, 本結果支持Newman & Ross的假說, 即紋藤壺屬和管藤壺屬應合并。
藤壺科; 條形碼; DNA; 亞科; 系統發育
圍胸總目藤壺科蔓足類, 是營固著生活的海洋甲殼動物, 隸屬于甲殼動物亞門顎足綱殼甲亞綱(Thecostraca), 蔓足下綱(Cirripedia), 該類群絕大多數種類分布在熱帶、溫帶以及兩極海洋的沿岸區與亞沿岸區, 不少種類大量群棲, 分布稠密。由于藤壺幼體能隨洋流移動, 加之成體可隨附著物體如船舶、鯨、海龜等運動, 所以擴散很快, 而廣泛分布于世界海洋。此外, 因成體營固著生活, 常牢固地附著在堤壩、碼頭等水工建筑物, 水底電線等水下設施、船舶、有機體及天然石礁上, 密集群棲時對海防、海運交通、工業和漁業等均十分有害[1]。
藤壺科分類學雖經歷了漫長的歷史, 但長期以來主要是以殼板形態如幅部, 翼部的寬窄(圖1), 殼表面肋的形態、蓋板等作為分類依據。直到20世紀中期, 口器、蔓足的毛序等軟體結構才被應用到藤壺分類中(圖2)。藤壺科成體均為附著生活, 殼板經常受到海浪泥沙的沖刷且喜密集群居, 致使一些形態分類學上的特征變化較大。傳統的形態學分類法具有諸多局限性(1)受物種性別和發育階段的限制,一些物種在不同性別和不同發育階段, 形態會有差別;(2)無法鑒定許多物種中普遍存在的隱存分類單元;(3)表型可塑性和遺傳多樣性容易導致不正確的鑒定,隱存種的存在更加劇了物種鑒定的困難[2]。
DNA條形碼(DNA Barcoding)是近幾年來國際生物學研究的熱點之一。理想的DNA條形碼檢測到的同屬內種間遺傳差異應明顯大于種內遺傳差異,且在兩者之間有明顯的間隔區, 即存在Barcoding gap;它是評價DNA條形碼理想與否的一個重要指標[2]。用于DNA條形碼研究的標準基因, 一方面應該足夠保守, 能夠利用通用引物進行大范圍的擴增; 另一方面應該有足夠的變異來區分不同物種。因此, 在鑒定不同類群的物種時需要選擇有效的目的基因, 備受研究者關注的 DNA 條形碼基因為線粒體COI基因序列。近來關于藤壺科類群的DNA條形碼研究較少, 本文以藤壺科蔓足類為研究對象, 在形態學分類鑒定的基礎上, 測定多種藤壺類的線粒體基因COI,16S和12S的部分序列, 結合GenBank中的18S,28S及12S的部分序列作為參考, 重點討論線粒體基因COI和16S在藤壺類物種鑒定中的可行性和有效性,并聯合16S和12S兩個線粒體基因序列分析了藤壺科亞科間系統演化關系。
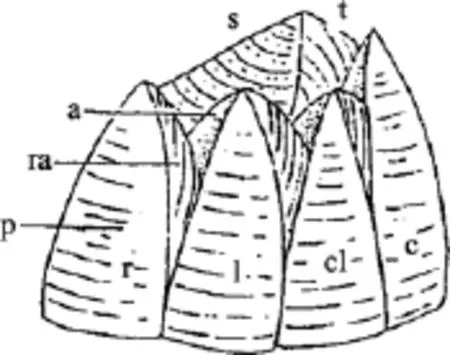
圖1 藤壺外部結構Fig. 1 Barnacle external structure
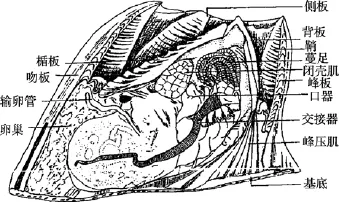
圖2 用于形態分類的藤壺內部結構Fig. 2 The barnacle internal structure for Shape classification
1 材料與方法
1.1 樣品采集及總DNA的提取
本文共收集了21個新鮮樣本, 所有樣本采集后均保存在 75%的分析純酒精溶液中, 樣本的標本號及采集地詳見表1, 所有樣品經任先秋研究員形態學鑒定后, 共包括藤壺科3屬7種。采用TIANGEN海洋動物組織提取試劑盒提取總DNA后存放于–20 ℃冰箱中備用。
1.2 目的基因的擴增及其序列的測定
1.2.1 引物的選定
本文共擴增了COI,16S和12S等3個線粒體基因片段, 引物分別如下:LCO14905'-GGTCAACAAATCATAAAGATATTGG-3', PPCO1_JR 5'-CTTTAATACCTGTAGGGACAGCA-3'[3]; 1471F 5'-CCTGTTTANCAAAAACAT-3', 1472R 5'-AGATAGAAACCAACCTGG-3'[4], 5'-GAAACCAGGATTAGATACCC-3', 5'-TTTCCCGCGAGCGACGGGCG-3'[5]。
1.2.2 基因的擴增
PCR擴增采用 25 μL反應體系, 其中包括PCRMIX12.5 μL,上、下游引物各 3 μmol/L 和 6.5 μLDNA 模板。反應設置的條件如下:94℃, 4 min;94℃, 30 min, 45℃, 1 min, 72℃, 1.5 min, 30個循環;最后72℃延伸10 min。
1.2.3 序列的測定及校對
PCR產物經凝膠檢測、純化并克隆后將菌液送至華大基因公司采用 ABIPRISMTM3730XL DNA Analyzer 測序儀雙向測序, 在BioEdit中進行正反向校正拼接。COI序列沒有插入和缺失, 可翻譯成氨基酸序列, 且在 NCBI 中 Blastn中為和圍胸類相似度最高, 排除了假基因的干擾。
1.3 數據分析
本文共獲得19條COI序列, 19條16S序列和4條12S序列, 長度分別為 887bp, 529bp, 338bp。從GenBank中下載了藤壺科其他種(表1中*標物種)共10條16S序列, 3條COI序列, 11條18S序列, 9條28S序列, 13條12S序列, 經軟件Clustalx比對后, 將同一基因序列剪切成同一長度, 最后獲得COI長度為653 bp,16S長度為529 bp,12S長度為338 bp,18S長度為1803 bp,28S長度為1785 bp。在Mega4.0軟件中計算堿基含量、遺傳距離(模型選擇為 Kimura 2-parameter)和構建 NJ樹[6]。在 Bioedit中將其相對應的種的16S和12S序列拼接在一起, 得到總長為864 bp的數據集。用Modeltest軟件選擇最優模型, 并在Paup*4.0b10軟件中構建MP和ML樹。貝葉斯分析在MrBayes 3.1.2軟件中完成[7-8]。
2 結果與討論
比較各基因最終序列片段, 變異位點和簡約位點詳見表3。COI基因序列沒有插入缺失。18S在722位點處有堿基插入, 1746位點處缺失,12S,16S和28S均有多處插入缺失。
2.1 堿基組成
用軟件 MEGA4對各基因片段進行堿基含量分
析(表2), 線粒體基因16S,COI,12S中 A+T 含量均明顯高于 G+C 含量, 均未達到飽和, 堿基組成表現出明顯的偏倚性, 核基因18S和28S則相反, A+T含量小于G+C含量。
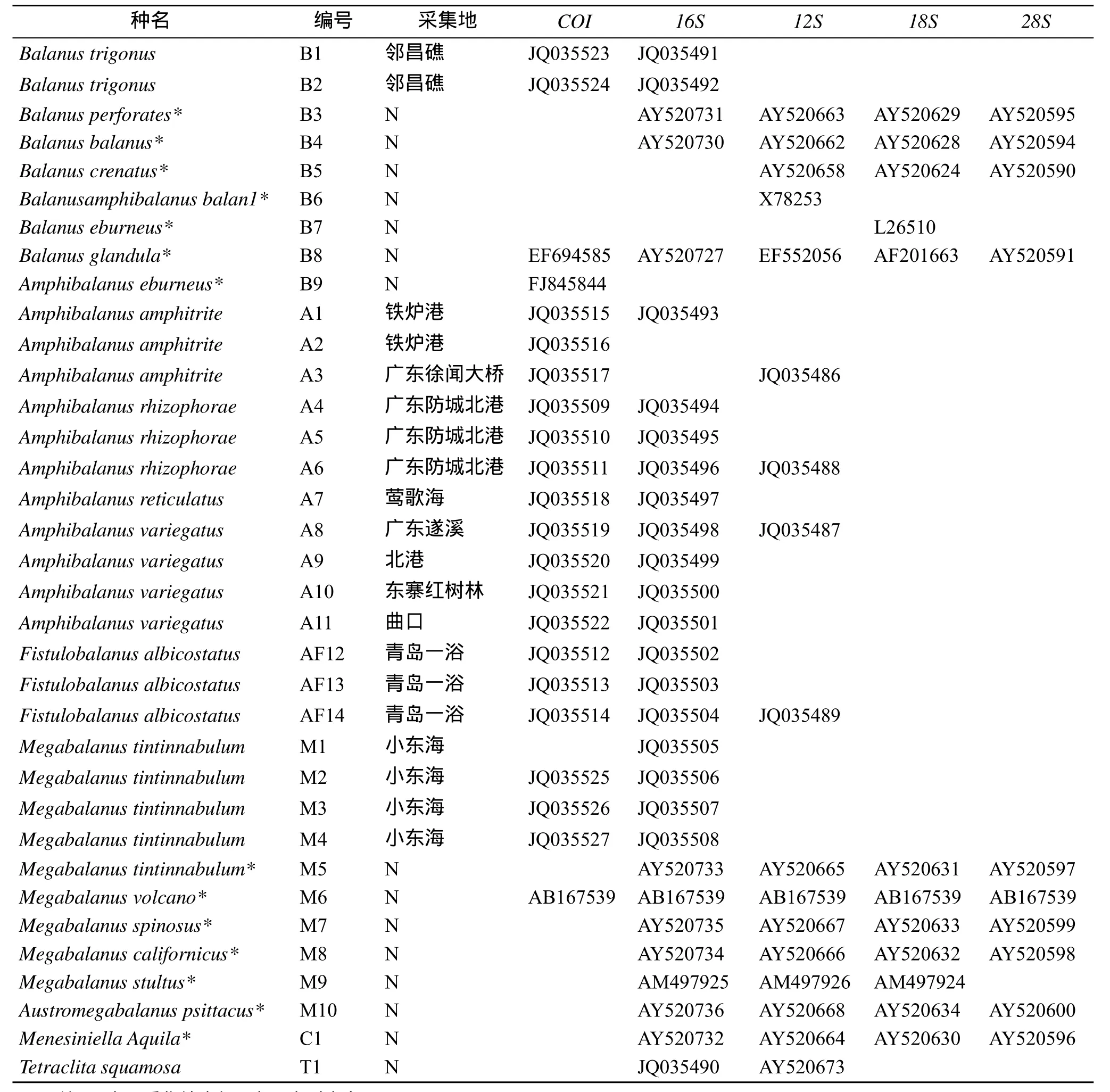
表1 樣本的序列號及采集地Tab. 1 Serial numbers and sampling locations of the samples
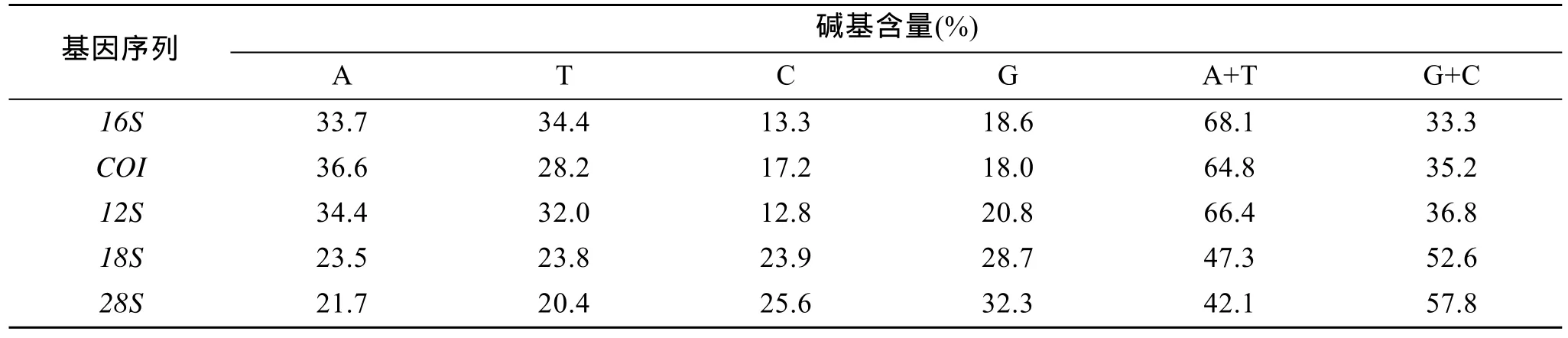
表2 5種基因片段堿基含量Tab. 2 Base contents of the fivegenefragments
2.2 藤壺科幾個物種種間及種內的遺傳距離
基于線粒體基因16S序列的平均種間遺傳距離為0.078, 其中采自廣東的紅樹紋藤壺(Amphibalanus rhizophorae)和來自美國的Austromegabalanus psittacus(AY520736)序列差異最大為 0.125, 而紅巨藤壺 (Megabalanus volcano(AB167539))和M.californicus(AY520734)序列差異最小只有 0.018,與 Hebert等[9]利用序列數據分析結果所得的種間遺傳距離大于 0.1不相符。基于COI序列的平均種間遺傳距離為 0.181, 其中采自廣東的紅樹紋藤壺(A.rhizophorae)和采自海南島的網紋紋藤壺(A.reticulatus)序列差異最小為0.140, 大于0.1, 很好地將藤壺科各個種區分開。采自青島的白脊管藤壺(Fistulobalanus albicostatus)和海南的鐘巨藤壺(Megabalanus tintinnabulum)差異最大為0.218, 且鐘巨藤壺與其他的藤壺科物種差異度大部分均為 0.2以上, 這表明巨藤壺與其他類群親緣關系較遠。基于12S序列的平均種間距離為 0.102, 其中Balanus amphibalanus balan1和M.californicus序列差異最大的為 0.155, 而采自海南小東海的鐘巨藤壺(M.tintinnabulum)和紅巨藤壺(M.volcano)差異最小只有0.000; 基于18S序列的平均種間遺傳距離為0.013,Balanus crenatus和Megabalanus spinosus之間遺傳距離最大為0.022; 基于28S序列的平均種間距離為0.038, 最大差異序列為0.061, 而最小為0.000, 結果表明12S,18S,28S種間差異均太小, 不足以區分種級階元(表3)。本研究結果表明COI最適合作為藤壺科種級階元分子鑒定的標準基因, 而16S次之,12S,28S,18S均不適合區分藤壺科種間關系。
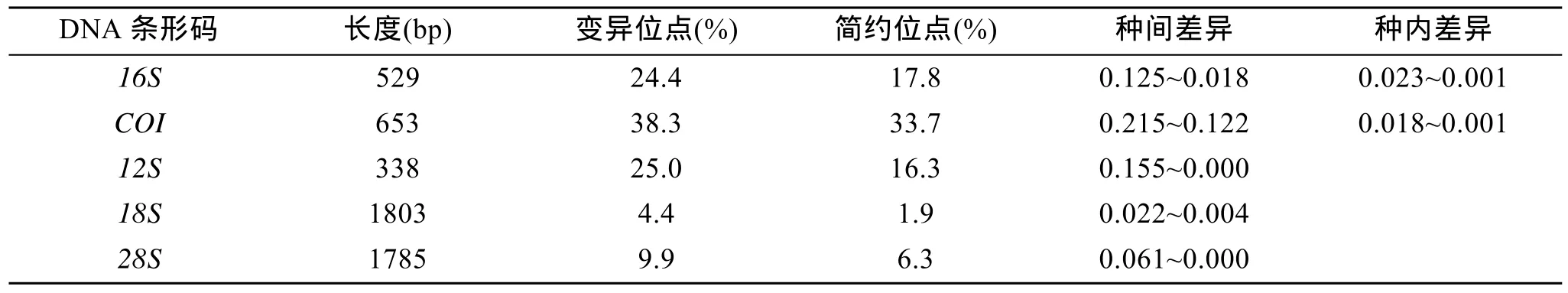
表3 基于5種DNA條形碼遺傳距離Tab. 3 Genetic distance on the basis of the 5 DNA barcodes
為了進一步探討COI和16S條形碼的適用性,將所得到的數據劃分為5個組, 分別為藤壺組BTR,紅樹紋藤壺組 ARH, 雜色紋藤壺組 AVA, 白脊管藤壺組FAL, 鐘巨藤壺組MTI。該五組的16S種內平均遺傳距離分別為0.010,0.001, 0.004, 0.001,0.023, 均居于 0~0.023區間內, 與之前16S平均種間距離0.078, 形成明顯間隔(圖3), 能較好的區分藤壺科物種的種下階元, 而基于COI序列, 該五組的種內平均距離分別為 0.005,0.001, 0.011,0.018,0.007, 其中ARH組內差異最小只有 0.001, 而 FAL組內差異最大為0.018, 均小于0.020, 而基因COI平均種間距離為0.181, 形成明顯間隔區(圖3), 結果表明COI基因和16S基因均適合作為藤壺類種下階元鑒定的分子標記。
2.3 DNA條形碼
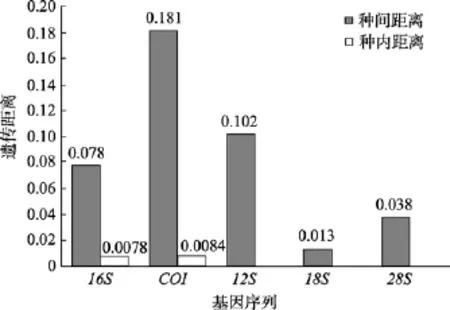
圖3 5種分子標記遺傳距離Fig. 3 Genetic distances of 5 molecular markers
DNA 條形碼(DNA Barcoding)是近幾年來國際生物學研究的熱點之一, 受零售業中作為產品通用代碼的“商品條形碼”的啟發, Herbert等[10]提出 DNA條形碼概念(DNA Barcoding), 即利用一段短的DNA序列作為物種快速鑒定的標記,并希望以此建立DNA序列和生物物種之間一一對應的關系。Hebert等[11]2003年對動物界中除刺胞動物門外的其他動物門, 共11門13320個物種的CO1基因序列進行分析,發現其序列間的差異能夠很好地區分所有研究物種,并認為在動物界中CO1基因是合適的DNA條形碼標準基因。Bucklin等[12]用線粒體COI基因序列(639 bp)分析了橈足亞綱哲水蚤目 10屬 34種系統關系,結果表明線粒體COI基因序列能很好的鑒別和區分哲水蚤的物種, 種內變異為 1%~4%, 遠小于種間變異(9%~25%)。本文嘗試利用線粒體COI序列鑒定甲殼動物, 通過研究 23目150種甲殼動物, 結果表明COI基因序列可以很好地區分目及以下階元, 在甲殼動物快速鑒定中非常有效[13]。Bucklin等[14]的研究表明DNA Barcode(COI序列, 650 bp)可以很好解決磷蝦同屬近緣種的關系和發現隱存種。Elias-Gutierrez 等[15]應用COI序列成功鑒定了來自墨西哥和危地馬拉的 61種 Cladocera 和 21種Copepoda浮游動物(代表了當地 40%的物種), 并發現了多個隱存種。本文通過對藤壺科物種COI,16S,12S,18S,28S等5種不同的DNA序列分析, 結果表明COI基因能夠準確辨別藤壺科的各物種, 其中,藤壺科各科物種之間的平均遺傳距離最小為 0.122,而各組種內平均遺傳距離最大為 0.018(表3), 與之前Hebert等[11]分析結果相似, 同屬各種COI序列的平均差異程度是 11.3%, 而種內的COI序列差異程度通常都很低, 低于2%。同時用COI作為條形碼來對藤壺科物種進行鑒定的結果與目前公認的以形態劃分的藤壺科物種分類地位相一致, 進一步證明了條形碼學說的可行性, 也驗證了COI基因適合作為藤壺科物種DNA分類的分子標記, 且閾值為種間大于10%, 種內小于2%。
2.4 白脊管藤壺和紅樹紋藤壺的關系
盡管紅樹紋藤壺和白脊管藤壺分屬為兩個屬,但在潮間帶這兩種常混棲在一起, 且條紋的顏色也彼此相似, 尤其后者的殼板經海浪泥沙沖刷后極似紅樹紋藤壺, 個體大小相似, 難于區分, 常被混淆。形態分類學上, 根據紅樹紋藤壺的殼無縱肋, 壁板管道的外壁無次級窩, 蓋板內面白色, 第3蔓足決無鋸齒剛毛等一系列特征來與白脊管藤壺進行區別,但是由于一些自然的不可抗拒的原因, 以及幼體時期無法明確判定以上的各項形態標準, 給分類鑒定工作帶來了很多困擾, 本文白脊管藤壺與紅樹紋藤壺的COI基因遺傳距離為0.189, 大于10%, 支持這兩個物種為不同物種, 且白脊管藤壺和紅樹紋藤壺的種內遺傳距離分別為0.018和0.001, 均小于2%,證明COI基因能清楚的區分白脊管藤壺和紅樹紋藤壺的種內和種間水平。本文結果表明, 白脊管藤壺和紅樹紋藤壺僅僅是可能因為混棲在相同的生境等原因造成外部形態趨同進化, 但實質上是兩種不同的物種。
2.5 藤壺科系統發育的初步分析
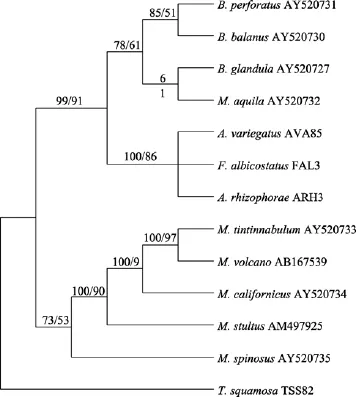
圖4 基于16S+12S基因序列數據集的進化樹Fig. 4 The phylogenetic tree of family Balanidae based on16S+12S genes
圍胸總目藤壺科的分類系統經歷了二亞科系統(小藤壺亞科 Chthamalinae,藤壺亞科 Balaninae)、三亞科系統(藤壺亞科 Balaninae, 巨藤壺亞科Megabalaninae, 凹藤壺亞科 Concavinae)及四亞科系統(藤壺亞科 Balaniae、紋藤壺亞科 Amphibalanus、巨藤壺亞科Megabalaninae和凹藤壺亞科Concavinae亞科), 目前普遍采用的 4亞科分類系統[16]。Modeltest選用的最優模型是 TVM+I+G。本文聯合16S和12S兩個線粒體基因序列分析了4亞科間的系統演化關系。MP樹支持率較低, 本文沒有采用, ML和BI兩個系統發育樹拓撲結構基本一致(圖4), 基本支持目前 4亞科的分類系統, 拓撲樹中藤壺科物種明顯分為3個分支, 且均有較高的支持率。圖中巨藤壺亞科的物種形成單系, 位于拓撲結構的根部, 起源較早, 支持率為BI:73, ML:53。巨藤壺亞科的物種壁板有管, 基底鈣質, 幅部發達, 齒隔片之間具有橫管, 藤壺科其他4個亞科的物種均不具有橫管, 且巨藤壺亞科的物種普遍比其他科物種個體大, 這些形態特征均支持巨藤壺亞科為單系群, 同時也表明這些特征在藤壺科分類中是祖征。紋藤壺亞科居于拓撲樹的中間, 成為一支, 有較高的支持率, BI:100,ML:86,但管藤壺屬與紋藤壺形成一個并群; Zullo[17]僅根據形態學上壁板有附加管將管藤壺從紋藤壺屬中分出并提升為屬, 有待商榷, 本文結果支持Newman等[18]的假說即紋藤壺屬似應與管藤壺屬合并。藤壺亞科和凹藤壺亞科聚為一支, 形成姐妹群,位于拓撲樹的內部, 但是支持率較低, 加上凹藤壺亞科僅僅包括 1種, 關于這兩個亞科間的親緣關系,有待進一步的研究探討。
[1]劉瑞玉, 任先秋. 中國動物志 甲殼動物亞門 蔓足下綱 [M]. 北京:科學出版社, 1997.
[2]Meyer C P, Paulay G. DNA Barcoding:error rates based on comprehensivesampling [J]. PLoS Biol, 2005, 3:2229-2238.
[3]VanSyoc R J, Fernandes J N,Carrison D A, et al. Molecular phylogenetics and biogeography of Pollicipes(Crustacea:Cirripedia), a Tethyan relict[J]. Elsevier,2010, 392:193-199.
[4]Crandall K A, Fitzpatrick J J R. Crayfish molecular systematics:inferences using a combination of procedures to estimate phylogeny[J]. Syst Biol, 1996, 45 (1):1-26.
[5]Mokady O Y, Loya Y, Achituv E,et al. Speciation versus phenotipic plasticity in coral inhabiting barnacles:Darwin’s observation in a phylogenetic context[J]. Mol Evol, 1999, 49:367-375.
[6]Tamura K, Dudley J, Nei M, et al. MEGA4:Molecular Evolutionary Genetics Analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24:1596-1599.
[7]Posada D, Crandall K A. Modeltest:testing the model of DNA substitution[J]. Bioinformatics, 1998, 14:817-818.
[8]Ronquist F, Huelsenbeck J P. MrBAYES 3:Bayesian phylogenetic inference under mixed models[J]. Bioinformatics, 2003, 19:1572-1574.
[9]Hebert P D N, Ratnasingham S, DeWaard J R. Barcoding animal life:Cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc R Soc Lond B Biol Sci, 2003(b), 270:596-599.
[10]Hebert P D N, Cywinska A, Ball S L ,et al. Biological identification through DNA barcodes[J]. Proc R Soc Lond B Biol Sci, 2003(a), 270:313-321.
[11]Hebert P D N, Ratnasingham S, de Waard J R. Barcoding animal Life:cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc. R. Soc.Lond. B(Suppl 1), 2003, 270:96-99.
[12]Bucklin A, Frost B W, Allen L D, et al. Molecular systematic and phylogenetic assessment of 34 calanoid copepod species of the Calanidae and Clausocalanidae[J]. Mar Biol, 2003, 142:333-343.
[13]Costa F O, deWaard J R, Boutillier J, et al. Biological identifications through DNA barcodes:the case of the Crustacea[J]. Fisheries and Aquatic Sciences, 2007, 64:272-295.
[14]Bucklin A, Wiebe P H, Smolenack S B, et al. DNA barcodes for species identification of euphausiids(Euphausiacea, Crustacea)[J]. Plankton Research, 2007,29:483-493.
[15]Elias-Gutierrez M, Jeronimo F M, Ivanova N V, et al.DNA barcodes for Cladocera and Copepoda from Mexico and Guatemala, highlights and new discoveries[J]. Zootaxa, 2008, 1839:1-42.
[16]Pitombo F B. Phylogenetic analysis of the Balanidae(Cirripedia, Balanomorpha)[J]. Zoologica Scripta, 2004,33:261-276.
[17]Zullo V A. New genera and species of balanoid barnacles from the Oligocene and Miocene of North Carolina[J]. Palaeontology, 1984, 58 (5):1312-1338.
[18]Newman W A , Ross A. Revision of the Balanomorph barnacles including a catalogue of the species[J]. San Diego Society of Natural History Memoirs, 1976,9:1-108.
DNA taxonomy of Balanidae
YUAN Shuai1,2, AN Jian-mei1, SHA Zhong-li2
(1. Shanxi Normal University, Linfen 041004, China; 2. Institute of Oceanology, Chinese Academy of Sciences,Qingdao 266071, China )
Feb., 15, 2012
Balanidae; Barcoding; DNA; Subfamily; Phylogenetic
The Balanidae (Cirripedia Thoracica) taxonomy had two subfamilies systems(Chthamalinae, Balaninae)and three subfamilies systems (Balaninae, Megabalaninae, Concavinae). The current system is the four subfamilies system. Molecular systematic research between them have never been reported. Many species of barnacles had convergent evolution so that the traditional morphological classification was not well identified. Our research determined three mitochondrial gene fragments and compared different gene fragments’ (COI, 16S, 12S, 28Sand18S) feasibility and effectiveness as barcoding to identify Barnacle species based on the12S, 16Sgene sequence.The results showed that theCOIdistance between groups and distance within groups had obvious interval.Minimum distance between species was 0.122, being much larger than the maximum distance within species (0.023).The distance between groups and distance within groups of16Shad coincidence; minimum distance between species was 0.018, less than the maximum distance within species 0.023. Therefore, the mitochondrialCOIgene could more accurately identify barnacle between species and within species, and intraspecific difference was less than 0.023, difference between species was bigger than 0.1. The analysis of ML and BI was consistent, which supported the classification of four subfamily system. Megabalaninae formed a monophyletic with a higher rate of support,which proved that transverse tubes was plesiomorphy. Amphibalanus and Fistulobalanus form a single clade. The result supported Newman & Ross’s (1976) hypothesis that the two should be merged.
Q959 文獻標識碼:A 文章編號:1000-3096(2012)09-0082-07
2012-02-15;
2012-06-11
國家科學技術基礎性工作專項(2011FY120200)
原帥(1985-), 女, 山西大同人, 碩士, 研究方向:甲殼動物 DNA 分類, E-mail:yundanfengqingde@126.com; 沙忠利, 通信作者, 副研究員, 研究方向:甲殼動物分類及分子系統學, E-mail:shazl@qdio.ac.cn
致謝:感謝中國科學院海洋研究所劉瑞玉院士和任先秋研究員在實驗設計和形態學鑒定方面給予了幫助和指導。
梁德海)

